玉米早期花药蛋白质组和磷酸化蛋白质组分析
张智敏,叶娟英,龙海飞,洪玥,陆平利
玉米早期花药蛋白质组和磷酸化蛋白质组分析
张智敏*,叶娟英*,龙海飞,洪玥,陆平利
复旦大学 生命科学学院 植物科学研究所,上海 200438
蛋白质磷酸化修饰是调控其功能的一种重要方式。植物有性生殖过程在农作物产量形成和物种繁衍过程中起着重要作用。作为植物雄性生殖器官的花药,其正常生长发育对于保证形成功能性配子 (花粉) 以及完成双受精过程至关重要。本研究以重要农作物玉米 (B73) 为材料,利用Nano UHPLC-MS/MS质谱技术对玉米早期发育的花药在蛋白质组和磷酸化蛋白质组水平进行全面分析,以探究玉米花药发育过程中的蛋白调控网络和磷酸化修饰调控网络。在蛋白质组学分析中,共鉴定到了3 016个多肽,匹配到1 032个蛋白质上。通过MapMan分析,预测到了一些和花药发育相关的蛋白质,例如受体激酶 (GRMZM2G082823_P01、GRMZM5G805485_P01等)。另外,在磷酸化蛋白质组学研究中,通过对TiO2亲和层析富集到的磷酸化多肽进行质谱分析,检测到了257个磷酸化多肽,匹配到 210个蛋白质上。我们的数据揭示了玉米花药发育过程中的 223个磷酸化位点。与已发现的玉米中的86个磷酸化蛋白质 (植物蛋白磷酸化数据库 (P3DB):http://www.p3db.org/organism.php) 相比,其中203个磷酸化蛋白和218个磷酸化位点为首次揭示。进一步生物信息学分析表明:磷酸化在14-3-3蛋白质、激酶、磷酸酶、转录因子、细胞周期和染色质结构相关的蛋白质介导的玉米早期花药发育过程中起着重要的调控作用。总之,本研究首次在蛋白质组学和磷酸化蛋白质组学水平研究了玉米早期花药发育的蛋白质调控网络,不仅丰富了玉米蛋白质和磷酸化修饰蛋白质数据库,并为利用遗传学和生物化学手段深入研究玉米花药发育的分子调控机理提供了基础。
玉米花药,蛋白质组,磷酸化,激酶,调控网络
玉米,是世界上最广泛的农作物之一,是一种重要的粮食来源。玉米的育性直接影响其产量和繁衍,研究其生殖发育有助于提高粮食作物的产量以应对日益严峻的人口增长带来的粮食短缺问题[1]。此外,玉米在植物遗传的科学研究中,也是一种重要的模式植物。随着玉米全基因组测序的完成[2],玉米相关的研究取得了进一步发展。
花药是开花植物的雄性生殖器官,大多数开花植物的花药在发育初期是由3层细胞构成的,由外而内分别是L1、L2和L3[3]。L1层细胞最终分化为表皮层;L2层细胞最终分裂分化为内皮层、中间层、绒毡层和小孢子母细胞,小孢子母细胞经过减数分裂形成小孢子,随后分裂形成花粉,绒毡层为花粉的形成和发育提供物质基础;L3层细胞最终发育成药室间隔和维管束等组织[3]。成熟的花药含有4个药室,每个药室都由5层细胞构成,外面的4层是营养细胞,最内层是生殖细胞,由外而内依次是:表皮层 (Epidermis)、内皮层 (Endothecium)、中间层 (Middle layer)、绒毡层 (Tapetum) 和小孢子母细胞 (Microsporocyte)[3]。以往的分子和遗传学研究表明,玉米中的一些基因影响花药发育过程[4-5]。随着基因芯片和新一代高通量测序技术的发展,已有一些转录水平上的玉米花药研究报道[6-8],但是蛋白质表达水平和蛋白质翻译后修饰水平上的研究还较少。
蛋白质作为细胞功能的基本单位,是生命活动的主要承担者,基因与蛋白质之间的关系极为复杂,同一个基因可能编码多个蛋白质,同一个蛋白质也可能有不同的形式和功能[9]。因此,对蛋白质的研究有助于透彻了解生物过程。中心法则以及近代蛋白质的功能研究揭示了从基因到蛋白质,中间有很多复杂的调控过程,例如,转录、翻译和翻译后修饰[10]。前体蛋白质需要经过一系列翻译后修饰 (PTM) 才能形成有功能活性的蛋白质。翻译后修饰在改变前体蛋白质的结构,酶的活性,底物的特异性,介导蛋白质与核酸、脂类、蛋白质以及其他分子之间的相互作用,调控蛋白质的亚细胞定位,蛋白复合物的形成以及蛋白质的降解等过程中具有重要作用[10]。蛋白质翻译后修饰种类较多,目前研究比较热门的修饰主要包括磷酸化、乙酰化、甲基化、糖基化、泛素化等多种修饰过程[10]。蛋白质磷酸化修饰是目前研究最多也是功能研究相对透彻的一种蛋白质翻译后修饰(http://selene.princeton.edu/PTMCuration/)。磷酸化过程是一个动态的可逆过程,由相应的激酶 和磷酸酶分别催化磷酸化过程和去磷酸化过 程[11]。可逆磷酸化修饰在花药发育过程中起着十分重要的作用[12-13]。此外,磷酸化对机体的代谢、生长、分裂和分化等过程也发挥着重要的作用[10,14]。在真核生物中,蛋白质磷酸化通常发生在丝氨酸 (Ser),苏氨酸 (Thr) 和酪氨酸 (Tyr) 残基上,也有报道,在原核生物中,磷酸化发生在组氨酸 (His)、天冬氨酸 (Asp)、谷氨酸(Glu)、赖氨酸(Lys)、精氨酸(Arg)和半胱氨酸(Cys)残基上[15]。研究表明在真核生物中,磷酸化发生在丝氨酸、苏氨酸和酪氨酸残基上的比例大致为1 800∶200∶1[16]。
在植物蛋白磷酸化数据库 (Plant protein phosphorylation data base,P3DB) 中,收录了86个玉米磷酸化蛋白质(http://www.p3db. org/ organism. php),本研究利用蛋白质组学的研究方法,鉴定到了1 242个蛋白质,其中磷酸化蛋白质有210个,并对鉴定到的玉米早期花药蛋白质和磷酸化蛋白质进行了进一步的功能分析。本文的结果不仅可以丰富植物磷酸化数据库,也能完善玉米蛋白质功能注释,促进玉米花药发育和磷酸化功能的研究。
1 材料与方法
1.1 玉米早期花药获取
将生长在大田中的B73系列玉米植株的雄穗剥出,放在冰上保鲜。取小穗放在载玻片上并滴加一定量的Mili-Q水保鲜,在立体显微镜下剥开,选取花药长度在1.5 mm左右的时期[17],迅速放入液氮中冷冻,然后储存到–80 ℃冰箱中备用。
1.2 蛋白质提取和蛋白酶消化
加入液氮,将玉米早期花药研磨成粉末状,加入10倍体积的蛋白裂解液6 mol/L 尿素, 2 mol/L 硫脲和 0.1 mol/L NH4HCO3(终浓度)悬浮,之后超声35 min (2 s超声5 s间歇),超声后14 000 r/min离心20 min,取上清。然后将蛋白质溶液在37 ℃条件下用0.005 mol/L 二硫代苏糖醇 (Dithiothreitol,DTT) 还原45 min,0.015 mol/L 碘代乙酰胺在室温条件下暗室烷基化45 min。用0.005 mol/L DTT淬灭多余的碘代乙酰胺。还原烷基化后的蛋白质用Lys-C (1 μg酶可以处理100 μg蛋白质混合物)在室温条件下消化4 h,再用6倍体积的Mili-Q水稀释之后,在37 ℃条件下用胰蛋白酶 (1 μg酶对应100 μg蛋白质混合物) 过夜消化肽段混合物。
1.3 TiO2亲和层析富集磷酸化肽段
肽段混合物酸化,离心,去除色素,上清液冻干。磷酸化肽段富集采用Thingholm等[18]的方法:用TiO2加样缓冲液 (含有80% 乙腈,1%三氟乙酸的1 mol/L 巯基乙酸) 重悬真空干燥的肽段,并使混合物缓慢的流过TiO2微柱。用20 μL上样缓冲液洗4次,20 μL清洗缓冲液 (80%乙腈,1%巯基乙酸) 洗2次,之后用15 μL洗脱缓冲液1 (2 mol/L NH3·H2O) 和2 μL洗脱缓冲液2 (含有40%乙腈的1 mol/L NH3·H2O) 各洗脱1次。
1.4 Nano UHPLC-MS/MS分析
样品采用液质联用的Easy nano-LC 1 000 LTQ-Orbitrap Elite (ThermoFisher) 进行分离和分析,液相溶剂是0.1% FA 溶解在Mili-Q水里面 (Solvent A) 和 0.1 % FA溶解在 90 % 乙腈里面 (Solvent B)。结合在分析柱上的肽段分3步分离洗脱:2%–35% solvent B 200 min,35%–90% solvent B 10 min,90% solvent B 5 min。然后90%–2% solvent B 2 min,2% solvent B 13 min平衡分析柱。进入质谱的肽段离子在Orbitrap中进行一级扫描,然后峰值最高的15个母离子 (>5 000 counts) 被选取,在线性离子阱 (LTQ) 中以MSA的碎裂模式进行二级质谱分析 (MS/MS)[19]。
1.5 数据库比对和数据解析
原始数据经过Proteome discoverer software (Version 1.4, Thermo Fisher, Germany)处理后用in-house Mascot server (Version 2.3.02,Matrix Science, London,UK)进行数据比对,搜库比对的数据库是蛋白质数据库(FASTA数据下载自ftp://ftp.plantbiology.msu.edu/pub/data/Eukaryotic_projects
/Z_mays/annotation_dbs/pseudomolecules/version 7.0/all.dir/all.pep; 2010; 66–495 sequences)。蛋白质数据库搜索参数设置为:胰蛋白酶漏切位点不超过2个;一级MS的质量误差不超过10 ppm,二级MS/MS 碎片离子的质量误差不超过0.6 Da;半胱氨酸上的烷基化作为一种固定修饰;蛋白质N末端乙酰化,甲硫氨酸的氧化,丝氨酸 (Serine)、苏氨酸 (Threonine)和酪氨酸 (Tyrosine) 的磷酸化作为可变修饰。肽段鉴定需要满足以下要求:<0.05,选取Mascot search engine (Matrix Science)排名第一位的肽段;肽段的假阳性率 (FDR)<1%;鉴定到的肽段通过Target Decoy PSM validator进一步验证,磷酸化位点通过phosphoRS3.0进一步准确定位。
1.6 生物信息学分析
利用NCBI protein Blast在线软件对鉴定到的玉米早期花药蛋白质和磷酸化蛋白质进行同源搜索,找到拟南芥中Score值最高且E-Value值最小的同源蛋白质 (http://blast.ncbi.nlm.nih. gov/Blast.cgi?PROGRAM=blastp&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome)。利用在线MapMan分析软件 (http://mapman. mpimp- golm.mpg.de/general/ora/ora.shtml)对玉米早期花药蛋白质进行功能聚类分析,<0.01的类目为显著富集的类目[20]。利用ClueGO软件对玉米磷酸化蛋白质进行生物过程、分子功能和通路途径分析[21]。利用MEGA 6.0对同源蛋白质序列进行比对。
2 结果与分析
2.1 玉米早期花药蛋白质组和磷酸化蛋白质组数据概述
经过Nano UHPLC-MS/MS分析及数据解析,本研究鉴定到了3 016个肽段,分布于1 032个蛋白质中 (附表1,具体数据作为附件,可在网络版中下载)。通过TiO2亲和层析,富集到257个磷酸化肽段,包含223个磷酸化位点,分布于210个磷酸化蛋白质中 (附表2,具体数据作为附件,可在网络版中下载)。在真核生物中,染色体的结构能够影响基因的表达、复制和修复过程,在机体的发育过程中也具有重要作 用[22]。DEK蛋白质最初在染色体易位导致的骨髓性白血病患者体内发现。体外生化研究显示,DEK蛋白质在DNA复制、DNA双链断裂修复过程、mRNA剪接和转录调控过程中都发挥重要作用,此外DEK蛋白质还具有组蛋白分子伴侣的功能,对保持异染色质的完整性尤为重 要[23]。在拟南芥中,4个基因编码DEK蛋白质:、、和4[24]。DEK3已被证明是植物染色体蛋白,调控基因表达过程,也是拟南芥中的1种抗逆调控因子[23]。拟南芥中的AT3G48710.1是1种DEK蛋白质,鉴定到的磷酸化蛋白质GRMZM2G139617_P01是其同源蛋白质。以GRMZM2G139617_P01肽段IIEDVINSMSDDEGEEGSEDEAEDDGKADNLK谱图为例,在肽段磷酸化位点S10处y23离子以及靠近肽段C末端的其他离子 (y24、y25、y26等) 处可以看到丢失 (98 Da,一个磷酸) 的峰,证明了鉴定到的磷酸化位点的可靠性 (图1)。
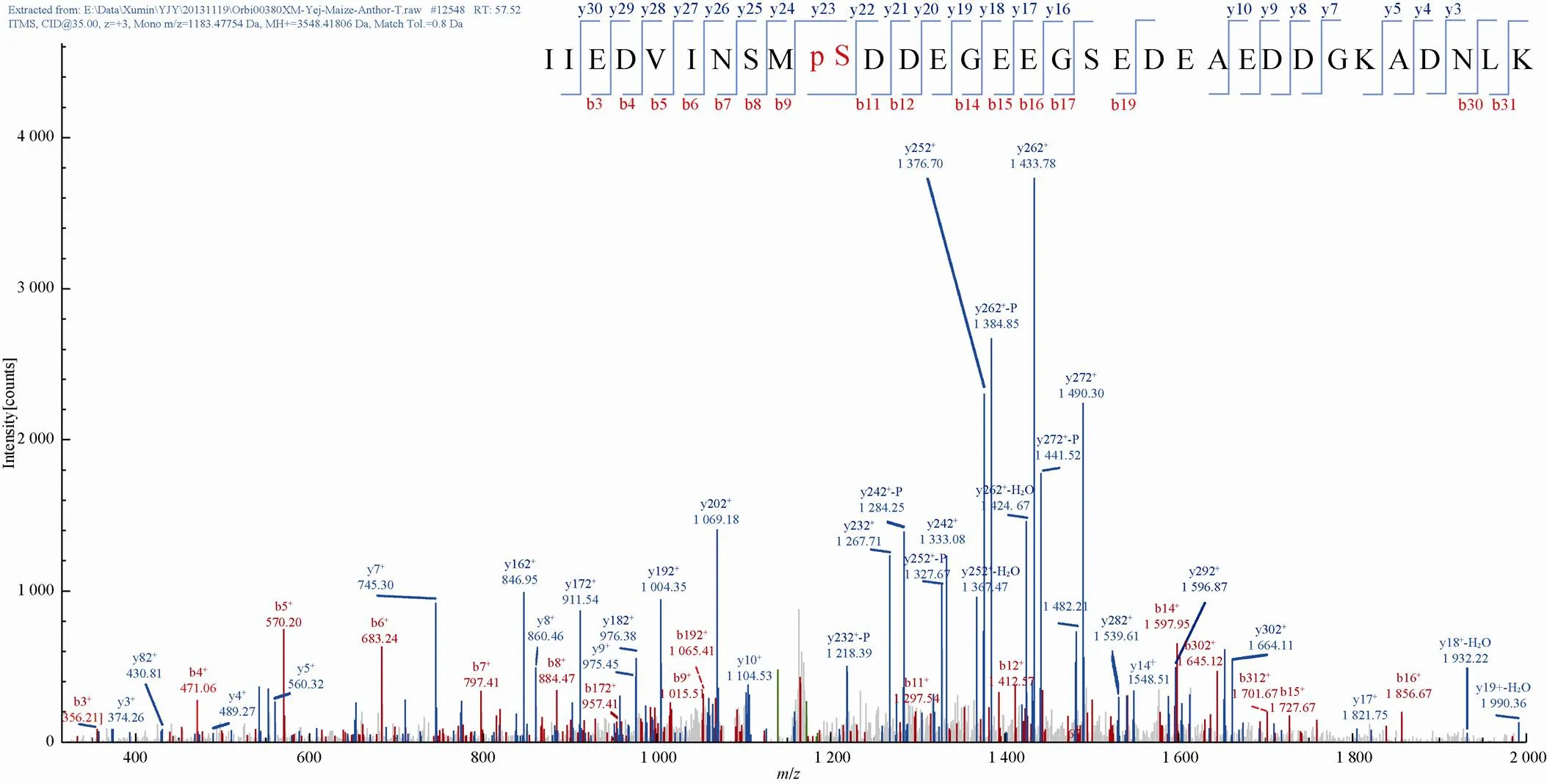
图1 玉米早期花药中鉴定到的GRMZM2G139617_P01肽段质谱图分析
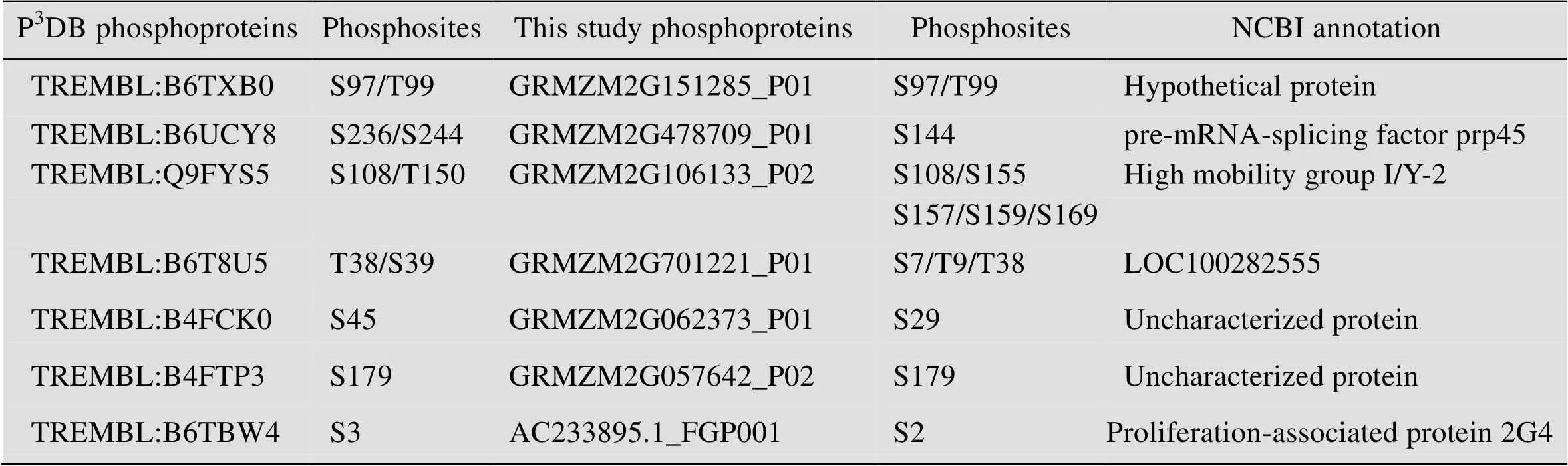
表1 与P3DB相同的磷酸化蛋白质磷酸化位点对比
Table 1 The phosphorylated sites of phosphoproteins maize phosphoproteins in P3DB and this study with the same sequence
210个磷酸化蛋白质包含的223个磷酸化位点中,80% (190个) 的位于丝氨酸 (Ser) 残基上,14% (33个) 位于苏氨酸(Thr)残基上,磷酸化同时发生在Ser残基和Thr残基上的蛋白质有13个,占磷酸化位点总数的8% (图2A)。磷酸化Thr残基的蛋白质中,82% (27个) 的蛋白质仅含有1个磷酸化位点,15% (5个) 的蛋白质含有2个,仅有1个蛋白质含有3个磷酸化位点 (图2B)。磷酸化Ser残基的蛋白质,77% (146个) 的蛋白质只含有1个磷酸化位点,17% (32个) 的蛋白质含有2个磷酸化位点,含有3个、4个、5个磷酸化位点的蛋白质分别有3个,占总数的1.5%,1.5% (3个) 的蛋白质有6–7个位点被磷酸化 (图2C)。
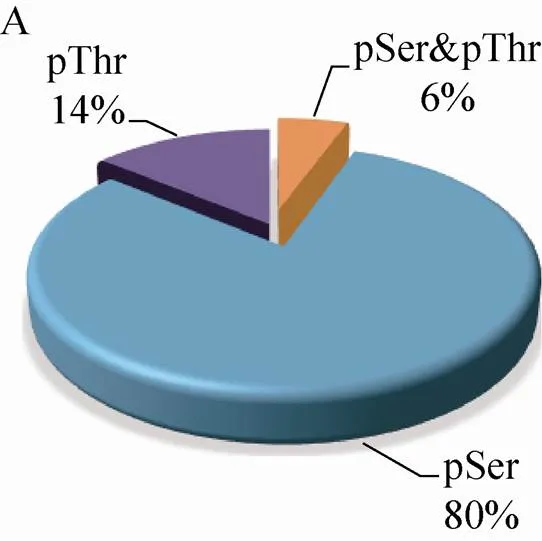
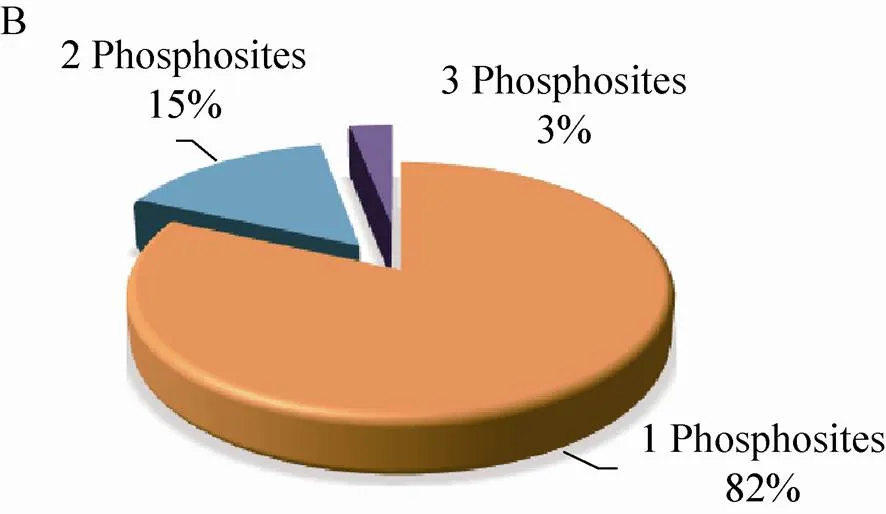
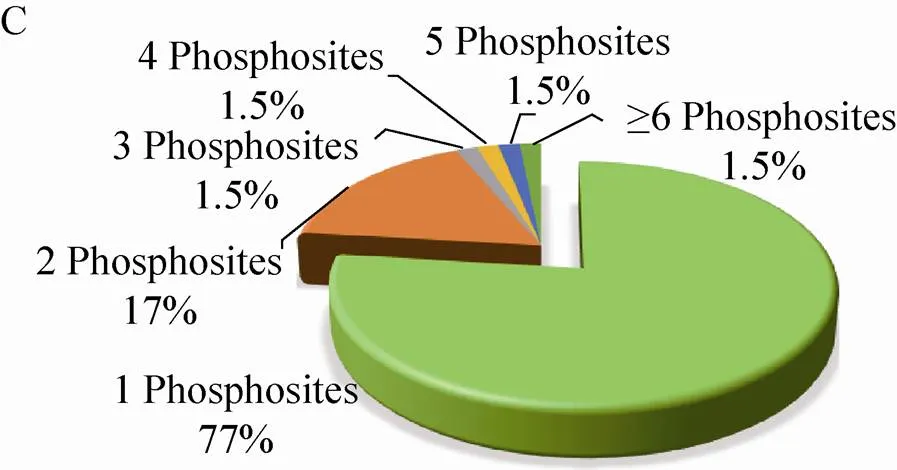
图2 玉米早期花药蛋白质组和磷酸化蛋白质组概述
在玉米早期花药中,本研究总共鉴定到 1 242个蛋白质,其中有210个磷酸化蛋白质。研究发现蛋白质组和磷酸化蛋白质组并不是简单的包含关系,鉴定到的玉米早期花药蛋白质组特有的蛋白质有997个;鉴定到的玉米早期花药磷酸化蛋白质组特有的蛋白质有172个;P3DB收录的玉米磷酸化蛋白质特有的蛋白质有77个 (图3A)。与P3DB中收录的86个磷酸化蛋白质 (http://www.p3db.org/organism.php) 相比,鉴定到的210个磷酸化蛋白质中,仅有7个蛋白质是P3DB收录的 (图3B)。此外,7个相同蛋白质中仅有2个蛋白质具有完全相同的磷酸化位点 (图3C)。这2个蛋白质是GRMZM2G151285_P01和GRMZM2G057642_P02,磷酸化位点分别是S97/T99和S179。GRMZM2G151285_P01在NCBI里面显示为hypothetical protein,GRMZM2G057642_P02是uncharacterized protein。另外5个磷酸化蛋白质鉴定到了新的磷酸化位点,其中GRMZM2G478709_P01,数据库中注释为pre-mRNA-splicing factor prp45,鉴定到的磷酸化位点是S144,P3DB报道的是S236/S244;GRMZM2G106133_P02,注释为high mobility group I/Y-2,鉴定到的磷酸化位点是S108/S155/S157/S159/S169,P3DB报道的是S108/T150;GRMZM2G701221_P01,是水稻蛋白LOC100282555的同源蛋白,鉴定到的磷酸化位点是S7/T9/T38,P3DB报道的是T38/S39;
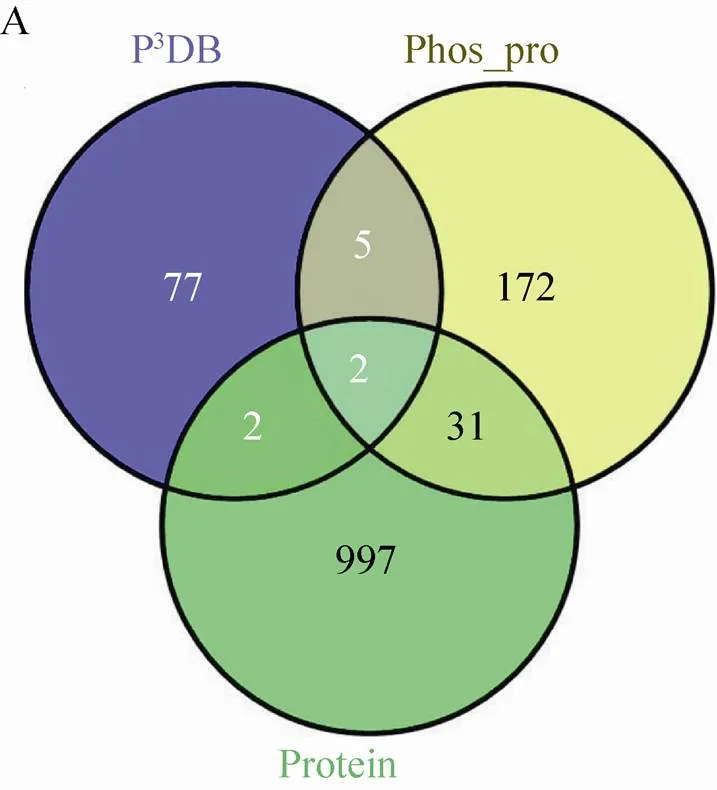

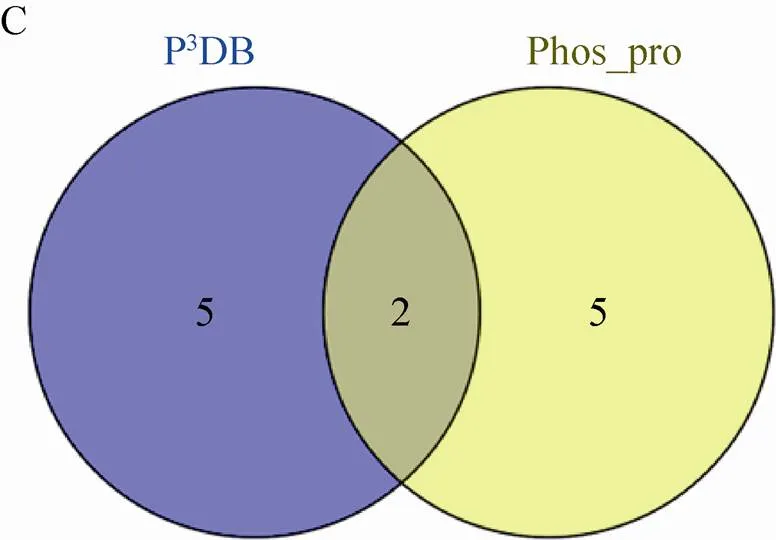
图3 玉米早期花药蛋白质组、磷酸化蛋白质组和P3DB重叠图
GRMZM2G062373_P01是未知功能的水稻蛋白LOC100192770的同源蛋白,鉴定到的磷酸化位点是S29,P3DB报道的是S45;AC233895.1_FGP001,数据库中注释为proliferation-associated protein 2G4,鉴定到的磷酸化位点是S2,P3DB报道的S3。综上所述,我们鉴定到的玉米早期花药磷酸化蛋白质和位点不仅验证了P3DB中的部分数据,更重要的是进一步丰富了P3DB中的数据。
2.2 玉米早期花药蛋白质的功能预测
为了明确鉴定到的玉米早期花药蛋白质在花药发育过程中的功能,通过NCBI Protein Blast找到拟南芥中具有最佳匹配的 (最高Score值和最小E-Value值) 同源蛋白质 (附表3,具体数据作为附件,可在网络版中下载)。利用在线MapMan分析软件对玉米早期花药蛋白质进行了分子功能预测。玉米早期花药蛋白质富集的 (<0.01) MapMan分子功能类目主要包括:信号 (209个)、RNA (166个)、转运 (163个)、逆境 (163个)、细胞壁 (133个)、细胞 (114个)、氧化还原 (98个)、氨基酸代谢 (88个)、DNA (86个)、二级代谢 (70个)、TCA (63个)、ATP合成 (57个)、光系统 (47个)、糖酵解 (43个)、主要的CHO代谢 (40个)、核苷酸代谢 (37个)、次要的CHO代谢 (32个)、N-代谢 (15个)、异生物素降解 (14个)、C1-代谢 (13个)、发酵 (8个)、OPP(8个)、乙醛酸循环 (6个) (图4)。通过对玉米早期花药蛋白质分子功能分类,富集的最多的是信号,比如鉴定到的受体激酶(GRMZM2G174708_P01,GRMZM2G176394_P03,GRMZM2G151738_P01,GRMZM2G099295_P01,GRMZM2G333811_P01,GRMZM2G149201_P01,GRMZM2G070322_P01,GRMZM2G025105_P01,GRMZM2G145440_P01,GRMZM2G038051_P01,GRMZM2G334181_P01,GRMZM2G063392_P01,GRMZM2G082823_P01,GRMZM5G805485_P01)。在拟南芥中,受体激酶 (EMS1、SERK1、SERK2、BAM1、BAM2、ER、ERL1、ERL2和RPK2) 被报道在花药发育过程中发挥着重要的作用[3,25]。因此,鉴定到的玉米早期花药蛋白质有助于推动玉米花药发育的相关研究。
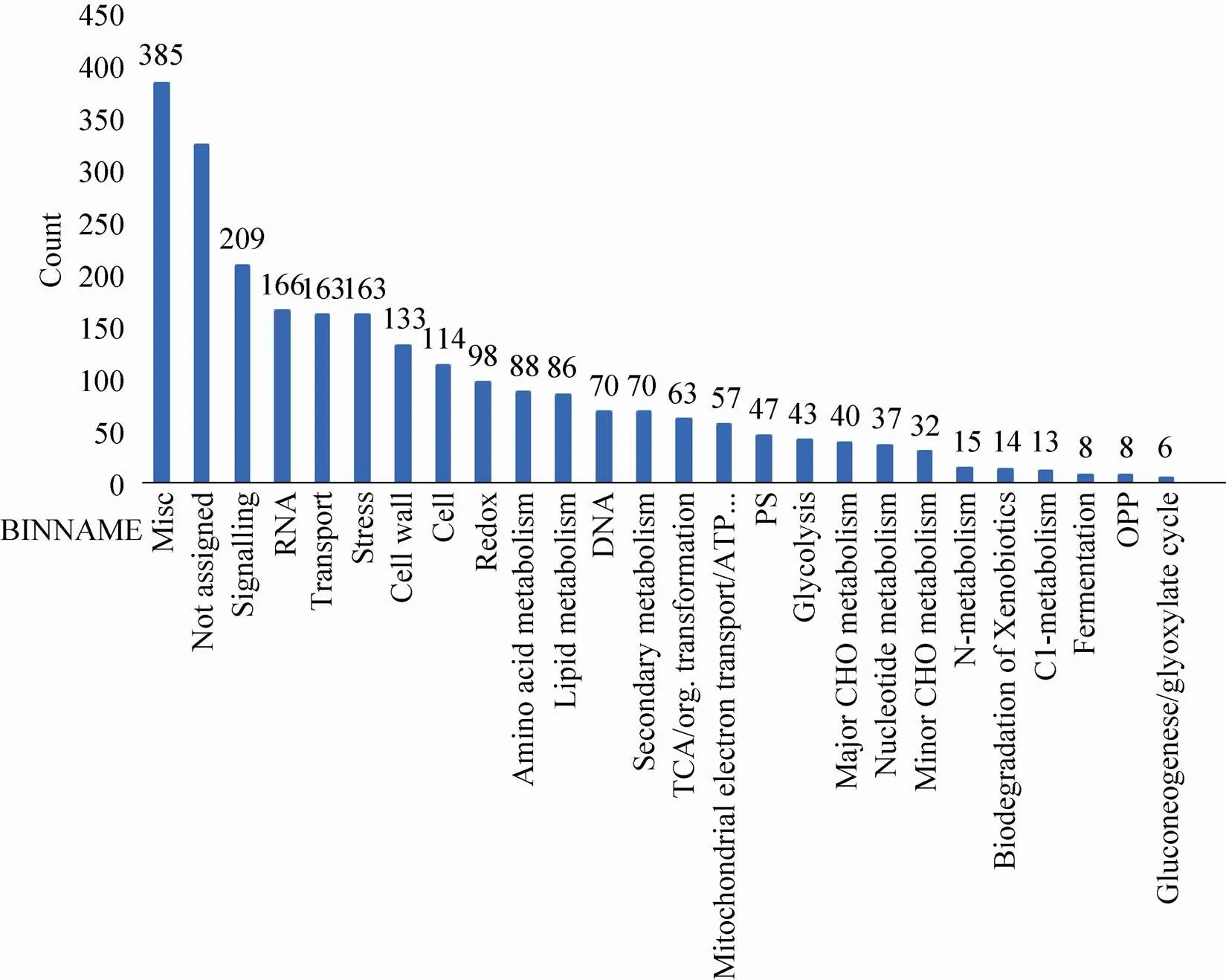
图4 玉米幼小花药蛋白质组MapMan分析
2.3 玉米早期花药磷酸化蛋白质功能概述
可逆的蛋白质磷酸化,在生物体的代谢、生长、分裂和分化等过程都发挥着重要作用[10,12-14]。通过NCBI Protein Blast找到了鉴定到的玉米早期花药磷酸化蛋白质在拟南芥中最佳匹配的 (Score值最高且E-Value值最小) 同源蛋白质 (附表4,具体数据作为附件,可在网络版中下载)。为了探究在玉米花药发育的早期,磷酸化参与的过程以及功能,我们利用ClueGO软件对玉米早期花药磷酸化蛋白质进行了生物过程、分子功能和通路分析。磷酸化蛋白质的分子功能主要分为4类:结合活性 (与DNA、钙调蛋白、某些蛋白质结构域的特异性结合);磷脂酶活性 (磷脂酶C、含磷二脂水解酶、磷脂酰肌醇磷脂酶C);磷酸转移酶活性 (分子转移酶、磷酸烯醇式丙酮酸羧激酶、磷酸烯醇式丙酮酸羧化酶);多肽受体活性 (图5A)。
磷酸化蛋白质参与的生物过程主要分为调控过程、DNA组装过程、糖代谢过程、有氧呼吸过程和蛋白质定位过程。调控过程主要包括:翻译正向调控、翻译延伸调控、细胞组分合成正向调控、细胞组分降解调控、细胞代谢调控和细胞组分合成调控、细胞蛋白代谢过程调控和氨基酸修饰等调控过程;DNA组装过程主要包括:DNA组装、DNA-蛋白质复合物亚基组装、核小体组装、核小体组装、染色质组装和蛋白质-DNA复合物组装;糖代谢过程主要包括:二糖代谢过程、二糖合成过程、蔗糖代谢过程、寡糖代谢过程、寡糖合成过程、海藻糖生物合成过程、海藻糖代谢过程和TCA循环 (图5B)。
蛋白激酶和磷酸酶介导的可逆磷酸化过程在花药发育过程中参与很多不同的信号转导途径[26],利用KEGG对玉米早期花药蛋白质磷酸化调控的通路进行分析,分析结果显示1条遗传通路、1条信号通路和4条代谢通路。遗传通路是DNA复制,信号通路是磷脂酰肌醇信号通路,4条代谢通路:碳水化合物代谢 (半乳糖代谢、氨基糖和核苷酸糖代谢、戊糖磷酸途径、TCA循环、糖异生、糖酵解);氨基酸代谢(半胱氨酸和甲硫氨酸代谢、甘氨酸代谢、精氨酸、脯氨酸、色氨酸、丝氨酸和苏氨酸代谢,赖氨酸、缬氨酸、亮氨酸、异亮氨酸降解);聚糖、脂类、维生素类的生物合成和代谢(鞘糖脂生物合成、亚麻酸代谢、不饱和脂肪酸合成、脂肪酸代谢、甘油脂代谢、花生四烯酸代谢、维生素C代谢、叶酸代谢、生物素代谢);能量代谢 (氮代谢) (图5C)。
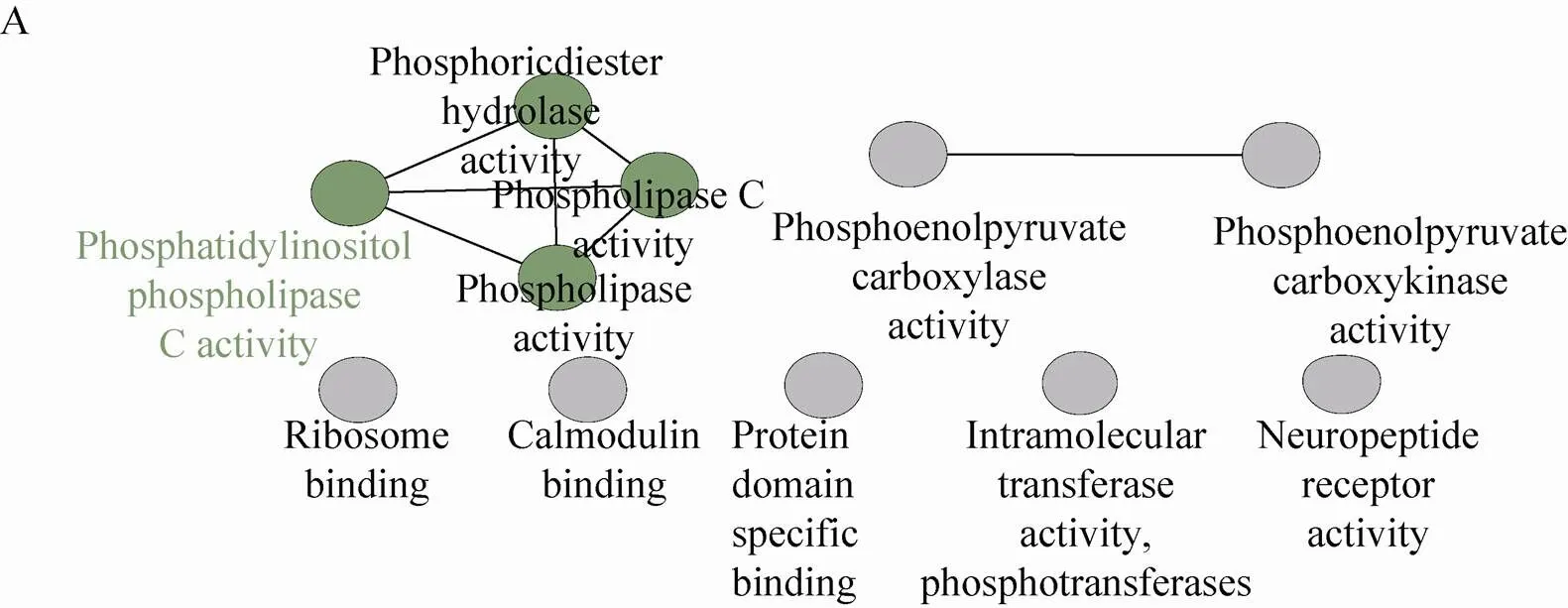
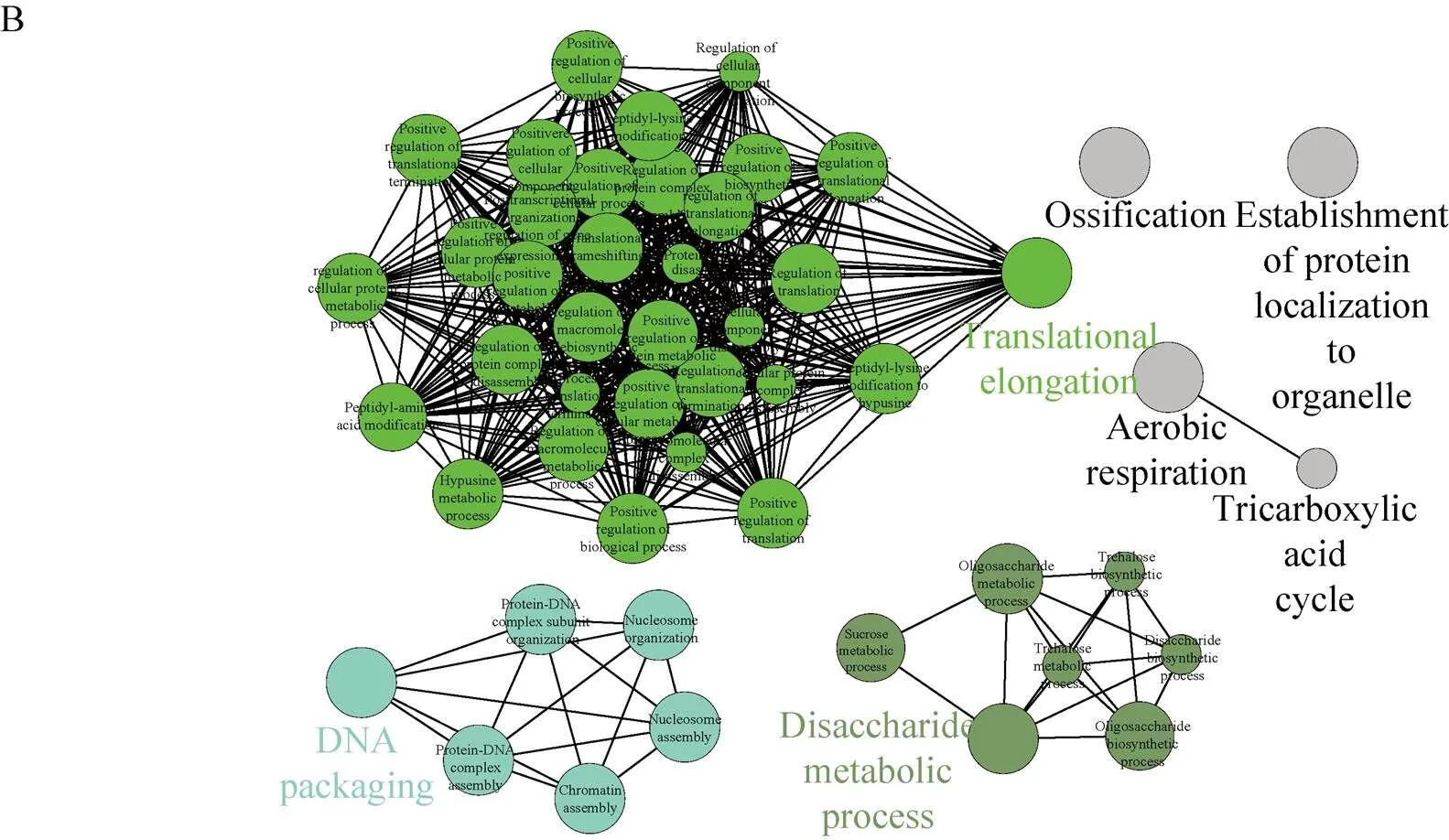
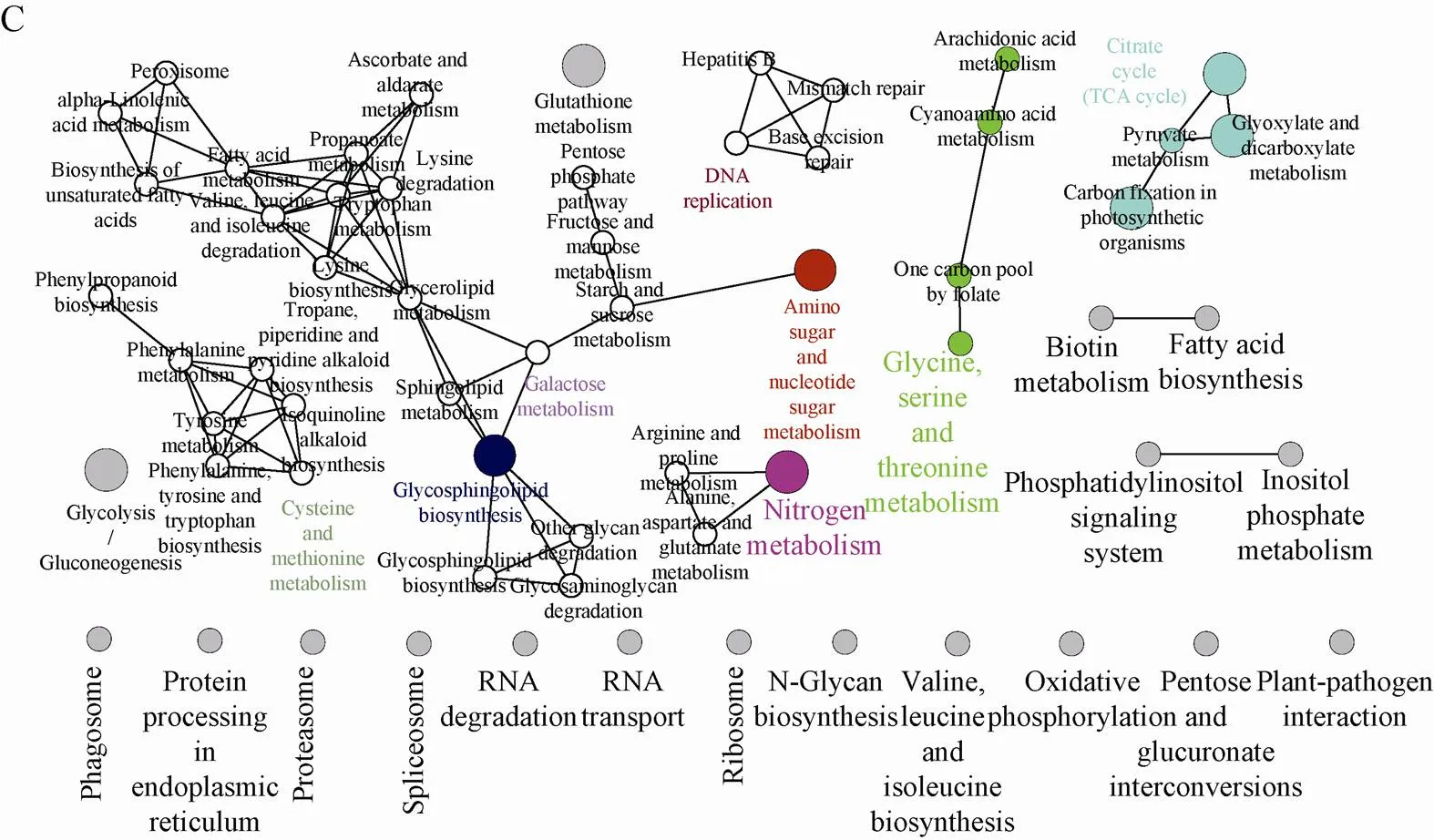
图5 玉米早期花药磷酸化蛋白质组GO分析和KEGG分析
对玉米早期花药磷酸化蛋白质的生物过程、分子功能和通路途经分析发现玉米早期花药中蛋白质磷酸化在DNA复制过程、信号通路过程、调控过程和代谢过程中发挥重要的作用。
2.4 磷酸化的14-3-3蛋白质
通过和拟南芥同源蛋白质氨基酸序列比对,推测GRMZM2G091155_P03、AC217050.4_FGP006、GRMZM2G078641_P01属于14-3-3蛋白质,GRMZM2G091155_P03拟南芥中的同源蛋白质是AT5G16050.1,AC217050.4_FGP006和GRMZM2G078641_P01在拟南芥中的同源蛋白质是AT3G02520.1;鉴定到的磷酸化位点分别是S110、T237和S257,磷酸化位点在P3DB均未曾收录 (表2)。14-3-3蛋白质最初在人脑中被发现,同时在哺乳动物、植物和其他真核生物中也有表达[27]。14-3-3蛋白质是一种酸性可溶性蛋白质,可以被上游的激酶磷酸化,其典型的功能就是可以和其他发生了磷酸化的蛋白质结合,进而调控该蛋白质的功能[28]。研究显示14-3-3蛋白质可以调控初级代谢中的酶[29],也可以与蛋白激酶发生作用,14-3-3蛋白质可以被激酶磷酸化,也可以与激酶互作进而调节其活性。研究显示14-3-3蛋白可以结合植物钙依赖性蛋白激酶 (CDPK)来激活其催化活性[30];SNF-related蛋白激酶 (SnRK) 在碳同化和胁迫适应的磷酸化级联反应中发挥核心作用,在拟南芥中,SnRK包括SnRK1、SnRK2和SnRK3,WPK4是小麦SnRK3激酶,WPK4和14-3-3蛋白具有双重连接关系,14-3-3蛋白能通过WPK4C-末端区域的磷酸化位点S388和S418结合到WPK4上,WPK4可以磷酸化14-3-3在硝酸还原酶上的结合位点S534[31]。MAP激酶参与许多细胞过程,比如细胞周期调控和非生物胁迫细胞应激过程等。番茄14-3-3蛋白7可以通过强化MAPKKKα的信号能力正向调控免疫相关的细胞程序性死 亡[32]。在百合中,14-3-3蛋白可以结合磷酸化的质子泵C-末端来调控质子泵的活性,进而影响花粉萌发和花粉管生长过程[33]。在植物生长发育中,14-3-3蛋白能够与其他蛋白质结合,从而精确的调控复杂的信号通路,影响不同的发育过程,比如植物的开花过程,细胞周期调控,脱落酸 (ABA)、赤霉素 (GA)和油菜素内酯 (BR) 的合成等过程[27–28]。GRMZM2G091155_P03、AC217050.4_ FGP006、GRMZM2G078641_P01与拟南芥 (AT5G16050.1,AT3G02520.1) 和水稻的Os08g0430500、Os04g0462500都具有高度的序列相似性,GRMZM2G091155_P03鉴定到的磷酸化位点S110和GRMZM2G078641_P01鉴定到的磷酸化位点S257,位于同一段保守的功能域内,这个磷酸化了的位点在其他物种中也是保守的,我们从而预测该磷酸化位点在多个物种间具有类似的功能 (图6)。
表2 磷酸化的玉米早期花药中的14-3-3蛋白质
Table 2 The phosphorylation of 14-3-3 proteins in maize early anther
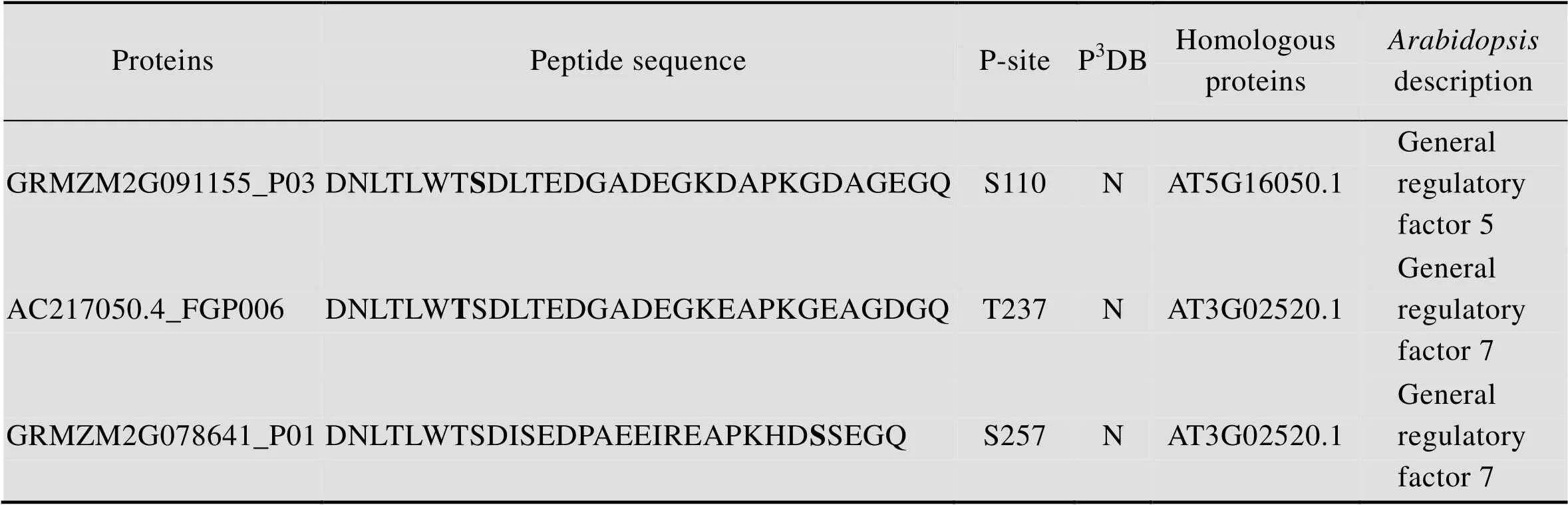
a) Bold amino acids indicated that these amino acids were phosphorylated in identified phosphoproteins. b) P-site means phosphorylation site. c) “N” means that phosphorylation site found in this study was not recorded in P3DB.

图6 玉米早期花药中鉴定到的磷酸化蛋白质与水稻和拟南芥中的同源蛋白质序列比对
2.5 磷酸化的激酶和磷酸酶
在植物中,磷酸酶、激酶构成了有机的信号转导体系,可逆的调控了蛋白质的磷酸化过程[34]。拟南芥和水稻中都报道了多个重要的磷酸酶,激酶及配基的功能及调控机制[35]。本研究中发现了一些磷酸化蛋白质GRMZM2G309025_P01/GRMZM2G180704_P01/GRMZM2G009538_P01/GRMZM2G166035_P01/GRMZM2G169182_P01/GRMZM2G127632_P01
/GRMZM2G060296_P04/GRMZM2G011070_P01预测是磷酸化的同源蛋白质是AT2G19130.1,是一种S-locus受体激酶,激酶和磷酸酶,其磷酸化位点分别是S825/T170/S469 S23/S278 S148/S153/S170/S581,这些磷酸化位点都是P3DB未报道的 (表3)。GRMZM2G 309025_ P01在拟南芥中的具有丝氨酸/苏氨酸蛋白激酶活性。S-locus受体激酶 (SRK) 基因最初在甘蓝中被发现,在十字花科植物自交不亲和反应中,SRK受体在自花授粉和异花授粉过程中发挥识别作用[36]。GRMZM2G009538_P01在拟南芥中的同源蛋白质是AT3G50690.1,是一种LRR受体激酶,LRR受体激酶在植物抗逆性反应、发育调控及激素的信号转导等生物学过程中发挥重要作用。在拟南芥中,FLS2参与植物抗逆性反应,具有防御和病原体识别的功能[37];另外拟南芥中的LRR受体激酶ER、EMS1、BAM1/BAM2,SERK1/SERK2在花药发育过程中发挥重要作 用[3,25,38-40];FLOR1介导信号转导过程,参与花发育的调控[41]。GRMZM2G166035_P01在拟南芥中的同源蛋白质是AT4G31750.1,是一种磷酸酶。拟南芥的磷酸酶PSP1在植物的雄性育性中也发挥着重要的功能[42]。GRMZM2G169182_P01在拟南芥中的同源蛋白质是AT2G01190.1,GRMZM2G127632_P01的是AT1G18160.1,GRMZM2G011070_P01的是AT2G35050.1,都属于蛋白激酶超家族。GRMZM2G060296_P04在拟南芥中的同源蛋白质是AT4G30600.1,是SRP受体α亚基家族蛋白。GRMZM2G180704_P01在拟南芥中的同源蛋白质是AT3G01090.2,是AKIN10/ SNF1-RELATED PROTEIN KINASE 1.1。SNF1 (Sucrose non-fermenting-1) 蛋白激酶在葡萄糖胁迫反应中具有重要的作用,在酵母产孢过程中也是必不可少的[43]。该蛋白最早在酿酒酵母中被鉴定到,研究证明SNF1在真核生物中具有高度保守性[44]。在哺乳动物中,AMPK是SNF1同源蛋白质,SNF1/AMPK蛋白激酶家族是一个高度保守的激酶家族,在细胞胁迫反应中起重要作用[45]。在拟南芥中,SNF1-related kinase 1 (SnRK1) 是SNF的同源蛋白质,研究证明SnRK1参与很多应激反应,比如胁迫反应[46]。在大麦中,SnRK1α和SnRK1β在花药中表达,反义SnRK1的表达能够引起花粉发育异常和大麦的雄性不 育[47]。此外,SnRK1在转录和代谢过程中也发挥着重要作用[48],能够调控能量稳态[46]。在酿酒酵母中,研究发现SNF1激酶在减数分裂起始和孢子形成过程中必不可少[49]。本文鉴定到的GRMZM2G180704_P01在拟南芥中的同源蛋白质是SNF1-RELATED PROTEIN KINASE 1.1 (AKIN10),研究显示,AKIN10可以和FUSCA3 (FUS3) 结合调控拟南芥侧生器官的发育和时期过渡[50]。因为鉴定到的GRMZM2G 180704_P01与拟南芥AT3G01090.2、水稻 (OSK1,Os05g0530500) 氨基酸序列具有高度的相似性,推测GRMZM2G180704_P01鉴定到的T170磷酸化位点具有保守性 (图7)。
表3 磷酸化的玉米早期花药中的激酶和磷酸化酶
Table 3 Phosphorylated kinases and phosphatases in maize early anther
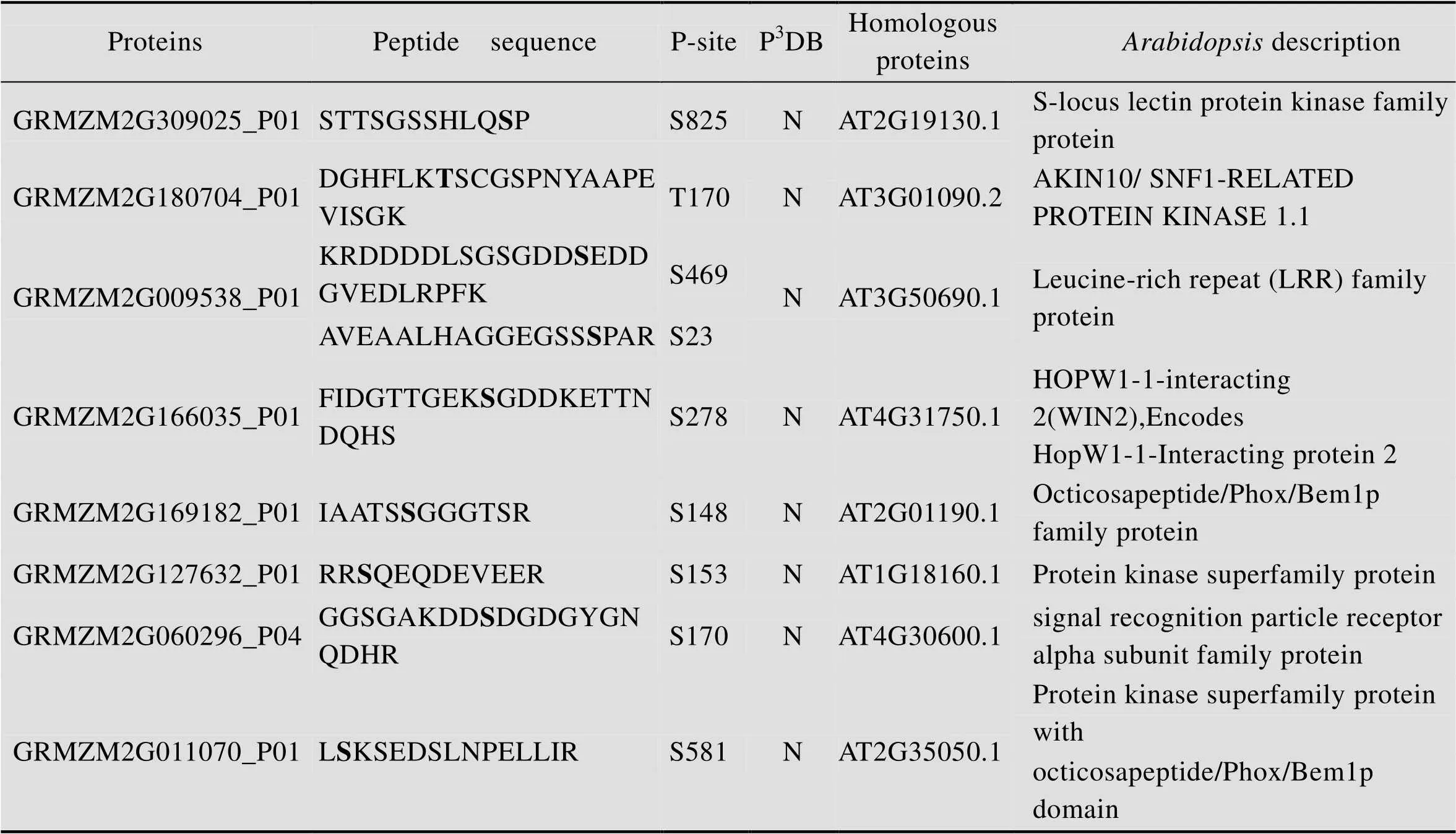
a) Bold amino acids indicated that these amino acids were phosphorylated in identified phosphoproteins. b) P-site means phosphorylation site. c) “N” means that phosphorylation site found in this study was not recorded in P3DB.
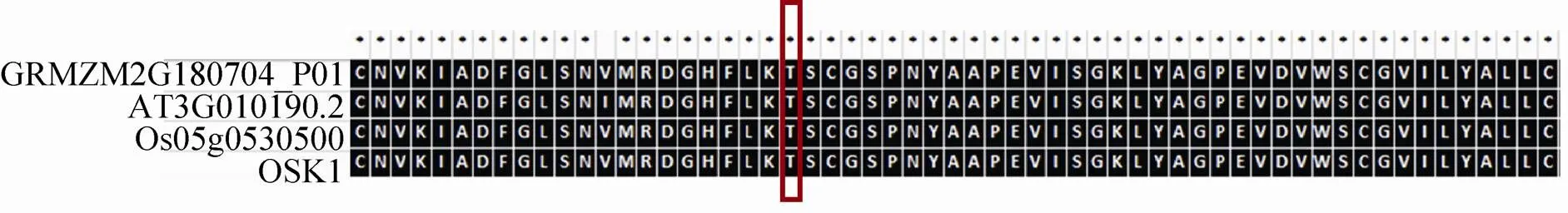
图7 玉米早期花药中鉴定到的磷酸化蛋白质与水稻和拟南芥中的同源蛋白质序列比对
2.6 磷酸化的转录因子
转录因子是动植物体内调控基因表达的重要元件,在调控转录,维持基因组稳定性方面具有重要意义。拟南芥和水稻等模式植物中报道和鉴定到的转录因子主要包括锌指家族、bZIP家族、bHLH家族以及MYC家族等[51-54]。GRMZM2G119640_P05中鉴定到的磷酸化位点是S142,在拟南芥中的同源蛋白质是AT3G21810.1,是C-X8-C-X5-C-X3-H类型的锌指蛋白质 (表4)。CCCH锌指基序广泛存在于有机体中,从人类到酵母,CCCH类型的锌指蛋白质可以与DNA、RNA和蛋白质结合发挥作 用[55]。该蛋白发生磷酸化,推测其在玉米早期花药发育过程中发挥作用。GRMZM2G010960_P01鉴定到的磷酸化位点是S767,在拟南芥中的同源蛋白质是AT1G10200.1,是GATA类型的锌指蛋白质 (表4)。GRMZM2G097726_P02鉴定到的磷酸化位点是T204,在拟南芥中的同源蛋白质是AT4G22140.1,是PHD-finger家族蛋白质。H2A、H2B、H3和H4都属于组蛋白,组蛋白修饰是一种重要的翻译后修饰,在转录的激活和沉默过程中发挥重要作用[56]。研究显示,植物的同源结构域 (PHD-finger) 可以作为一种识别组蛋白的特殊的结构域[57]。此外,PHD-finger在染色质相关的蛋白质中也存在,可以识别染色质修饰并招募其他因子行使功 能[58]。在拟南芥中,MMD1可以编码PHD-finger蛋白质,MMD1在雄性减数分裂过程中发挥重要作用[59]。因此,推测鉴定到的同源基因GRMZM2G097726_P02在玉米花药减数分裂过程中发挥作用。GRMZM2G175280_P01鉴定到磷酸化位点是S219,在拟南芥中的同源蛋白质是AT4G38900.1,是bZIP转录因子家族蛋白质;GRMZM2G178182_P01鉴定到的磷酸化位点是S259、S261、T273和S288,在拟南芥中的同源蛋白质是AT5G50915.1,是bHLH DNA-binding 超家族蛋白质 (表4)。转录因子在调控细胞过程和有机体的组分中发挥着重要的功能。bZIP和bHLH转录因子家族广泛存在于真核生物中,研究显示bZIP转录因子可以调控花发育[60]。在动物中,bHLH蛋白在感应信号,调控细胞周期,以及其他一些重要过程中发挥作用[61]。在植物中,bHLH蛋白参与很多生物过程,比如信号过程、胁迫过程和花发育过程[60]。水稻中已有研究显示,bHLH转录因子UDT1、TDR、TIP2参与花药发育过程,能够影响雄性育性[62-64]。因此,推测鉴定到的GRMZM2G178182_P01在玉米早期花药发育过程中发挥作用。转录因子的磷酸化的鉴定和研究为进一步理解转录调控的机制提供了理论支持,并为我们后续的遗传学研究提供了线索。
2.7 细胞周期和染色质相关的磷酸化蛋白质
通过与拟南芥同源蛋白质氨基酸序列比对,发现了一些磷酸化蛋白质与细胞周期以及染色质相关。GRMZM5G866734_P01鉴定到的磷酸化位点是S2和T5,在拟南芥中的同源蛋白质是AT1G19880.1 ,是RCC1家族蛋白质 (表4)。RCC1是拟南芥有丝分裂调控染色体浓缩的关键基因[65]。GRMZM2G036765_P02鉴定到的磷酸化位点是S3,在拟南芥中的同源蛋白质是AT3G09840.1,是细胞分裂周期蛋白48 (CDC48) (表4)。在酿酒酵母中,CDC48在纺锤体分离时发挥重要作用;在拟南芥中,CDC48在细胞分裂和细胞生长过程中具有重要功能[66]。GRMZM2G117100_P02鉴定到的磷酸化位点是S139,GRMZM2G139617_P01鉴定到的磷酸化位点是S479、S11、S367。GRMZM2G117100_P02和GRMZM2G139617_P01在拟南芥中的同源蛋白质均是AT3G48710.1;GRMZM2G375984_P01鉴定到的磷酸化位点是S379,在拟南芥中的同源蛋白质是AT5G55660.1;这3种蛋白都含有DEK结构域 (表4),在拟南芥中,含有DEK结构域的蛋白质可以调控染色质的结构和功 能[23]。因此,通过分析推测鉴定到的玉米早期花药磷酸化蛋白质参与了调控染色质的结构和功能以及细胞分裂过程。
3 结论
本文聚焦在玉米早期花药蛋白质组和磷酸化蛋白质组的研究上,利用TiO2亲和层析的方法富集磷酸化肽段,借助Nano UHPLC-MS/MS质谱手段,获得了1 032个蛋白质和210个磷酸化蛋白质,结合生物信息学分析,进一步对鉴定的蛋白质进行了功能预测,对磷酸化的14-3-3蛋白质、激酶、磷酸酶、转录因子、细胞周期和染色质相关的蛋白质进行了重点分析以及14-3-3磷酸化位点、AKIN10磷酸化位点的保守性分析。研究结果不仅能够丰富植物蛋白磷酸化数据库 (P3DB),同时对于推动玉米花药发育和翻译后修饰过程的研究提供了一定的信息基础。
REFERENCES
[1] Scott RJ, Spielman M, Dickinson HG. Stamen structure and function. Plant Cell, 2004, 16(Suppl 1): S46–S60.
[2] Sarah Hake JLB. Handbook of Maize: Genetics and Genomics. New York: Springer, 2009.
[3] Chang F, Wang YX, Wang SS, et al. Molecular control of microsporogenesis in. Curr Opin Plant Biol, 2011, 14(1): 66–73.
[4] Moon J, Skibbe D, Timofejeva L, et al. Regulation of cell divisions and differentiation by MALE STERILITY32 is required for anther development in maize. Plant J, 2013, 76(4): 592–602.
[5] Wang DX, Skibbe DS, Walbot V. Maize(), a putative β-1, 3-galactosyltransferase, modulates cell division, expansion, and differentiation during early maize anther development. Plant Reprod, 2013, 26(4): 329–338.
[6] Ma J, Morrow DJ, Fernandes J, et al. Comparative profiling of the sense and antisense transcriptome of maize lines. Genome Biol, 2006, 7(3): R22.
[7] Ma J, Skibbe DS, Fernandes J, et al. Male reproductive development: gene expression profiling of maize anther and pollen ontogeny. Genome Biol, 2008, 9(12): R181.
[8] Ma J, Duncan D, Morrow DJ, et al. Transcriptome profiling of maize anthers using genetic ablation to analyze pre-meiotic and tapetal cell types. Plant J, 2007, 50(4): 637–648.
[9] Silva-Sanchez C, Li HY, Chen SX. Recent advances and challenges in plant phosphoproteomics. Proteomics, 2015, 15(5/6): 1127–1141.
[10] Kersten B, Agrawal GK, Iwahashi H, et al. Plant phosphoproteomics: a long road ahead. Proteomics, 2006, 6(20): 5517–5528.
[11] Pandey A, Mann M. Proteomics to study genes and genomes. Nature, 2000, 405(6788): 837–846.
[12] Ye JY, Zhang ZB, Long HF, et al. Proteomic and phosphoproteomic analyses reveal extensive phosphorylation of regulatory proteins in developing rice anthers. Plant J, 2015, 84(3): 527–544.
[13] Guan YF, Meng XZ, Khanna R, et al. Phosphorylation of a WRKY transcription factor by MAPKs is required for pollen development and function in. PLoS Genet, 2014, 10(5): e1004384.
[14] Kersten B, Agrawal GK, Durek P, et al. Plant phosphoproteomics: an update. Proteomics, 2009, 9(4): 964–988.
[15] Batalha IL, Lowe CR, Roque ACA. Platforms for enrichment of phosphorylated proteins and peptides in proteomics. Trends Biotechnol, 2012, 30(2): 100–110.
[16] Mann M, Jensen ON. Proteomic analysis of post-translational modifications. Nat Biotechnol, 2003, 21(3): 255–261.
[17] Zhang H, Egger RL, Kelliher T, et al. Transcriptomes and proteomes define gene expression progression in pre-meiotic maize anthers. G3 (Bethesda), 2014, 4(6): 993–1010.
[18] Thingholm TE, Jørgensen TJD, Jensen ON, et al. Highly selective enrichment of phosphorylated peptides using titanium dioxide. Nat Protoc, 2006, 1(4): 1929–1935.
[19] Palumbo AM, Reid GE. Evaluation of gas-phase rearrangement and competing fragmentation reactions on protein phosphorylation site assignment using collision induced dissociation-MS/MS and MS3. Anal Chem, 2008, 80(24): 9735–9747.
[20] Usadel B, Nagel A, Steinhauser D, et al. PageMan: an interactive ontology tool to generate, display, and annotate overview graphs for profiling experiments. BMC Bioinf, 2006, 7(1): 535.
[21] Bindea G, Mlecnik B, Hackl H, et al. ClueGO: a cytoscape plug-in to decipher functionally grouped gene ontology and pathway annotation networks. Bioinformatics, 2009, 25(8): 1091–1093.
[22] Ho L, Crabtree GR. Chromatin remodelling during development. Nature, 2010, 463(7280): 474–484.
[23] Waidmann S, Kusenda B, Mayerhofer J, et al. A DEK domain-containing protein modulates chromatin structure and function in. Plant Cell, 2014, 26(11): 4328–4344.
[24] Pendle AF, Clark GP, Boon R, et al. Proteomic analysis of thenucleolus suggests novel nucleolar functions. Mol Biol Cell, 2005, 16(1): 260–269.
[25] Ma H. Molecular genetic analyses of microsporogenesis and microgametogenesis in flowering plants. Annu Rev Plant Biol, 2005, 56(1): 393–434.
[26] Yang KZ, Jiang M, Wang M, et al. Phosphorylation of serine 186 of bHLH transcription factor SPEECHLESS promotes stomatal development in. Mol Plant, 2015, 8(5): 783–795.
[27] de Boer AH, van Kleeff PJM, Gao J. Plant 14-3-3 proteins as spiders in a web of phosphorylation. Protoplasma, 2013, 250(2): 425–440.
[28] Denison FC, Paul AL, Zupanska AK, et al. 14-3-3 proteins in plant physiology. Semin Cell Dev Biol, 2011, 22(7): 720–727.
[29] Cotelle V, Meek SEM, Provan F, et al. 14-3-3s regulate global cleavage of their diverse binding partners in sugar-starvedcells. EMBO J, 2000, 19(12): 2869–2876.
[30] Camoni L, Harper JF, Palmgren MG. 14-3-3 proteins activate a plant calcium-dependent protein kinase (CDPK). FEBS Lett, 1998, 430(3): 381–384.
[31] Ikeda Y, Koizumi N, Kusano T, et al. Specific binding of a 14-3-3 protein to autophosphorylated WPK4, an SNF1-related wheat protein kinase, and to WPK4-phosphorylated nitrate reductase. J Biol Chem, 2000, 275(41): 31695–31700.
[32] Oh CS, Pedley KF, Martin GB. Tomato 14-3-3 protein 7 positively regulates immunity-associated programmed cell death by enhancing protein abundance and signaling ability of MAPKKKα. Plant Cell, 2010, 22(1): 260–272.
[33] Pertl H, Himly M, Gehwolf R, et al. Molecular and physiological characterisation of a 14-3-3 protein from lily pollen grains regulating the activity of the plasma membrane H+ ATPase during pollen grain germination and tube growth. Planta, 2001, 213(1): 132–141.
[34] Mishra NS, Tuteja R, Tuteja N. Signaling through MAP kinase networks in plants. Arch Biochem Biophys, 2006, 452(1): 55–68.
[35] Hadiarto T, Nanmori T, Matsuoka D, et al. Activation ofMAPK kinase kinase (AtMEKK1) and induction of AtMEKK1-AtMEK1 pathway by wounding. Planta, 2006, 223(4): 708–713.
[36] Stein JC, Howlett B, Boyes DC, et al. Molecular cloning of a putative receptor protein kinase gene encoded at the self-incompatibility locus of. Proc Natl Acad Sci USA, 1991, 88(19): 8816–8820.
[37] Gómez-Gómez L, Bauer Z, Boller T. Both the extracellular leucine-rich repeat domain and the kinase activity of FLS2 are required for flagellin binding and signaling in. Plant Cell, 2001, 13(5): 1155–1163.
[38] Hord CLH, Chen CB, Deyoung BJ, et al. The BAM1/BAM2 receptor-like kinases are important regulators ofearly anther development. Plant Cell, 2006, 18(7): 1667–1680.
[39] Albrecht C, Russinova E, Hecht V, et al. TheSOMATIC EMBRYOGENESIS RECEPTOR-LIKE KINASES1 and 2 control male sporogenesis. Plant Cell, 2005, 17(12): 3337–3349.
[40] Colcombet J, Boisson-Dernier A, Ros-Palau R, et al.SOMATIC EMBRYOGENESIS RECEPTOR KINASES1 and 2 are essential for tapetum development and microspore maturation. Plant Cell, 2005, 17(12): 3350–3361.
[41] Acevedo FG, Gamboa A, Paéz-Valencia J, et al. FLOR1, a putative interaction partner of the floral homeotic protein AGAMOUS, is a plant-specific intracellular LRR. Plant Sci, 2004, 167(2): 225–231.
[42] Cascales-Miñana B, Muñoz-Bertomeu J, Flores-Tornero M, et al. The phosphorylated pathway of serine biosynthesis is essential both for male gametophyte and embryo development and for root growth in. Plant Cell, 2013, 25(6): 2084–2101.
[43] Jiang R, Carlson M. Glucose regulates protein interactions within the yeast SNF1 protein kinase complex. Genes Dev, 1996, 10(24): 3105–3115.
[44] Celenza JL, Carlson M. A yeast gene that is essential for release from glucose repression encodes a protein kinase. Science, 1986, 233(4769): 1175–1180.
[45] Corton JM, Gillespie JG, Hardie DG. Role of the AMP-activated protein kinase in the cellular stress response. Curr Biol, 1994, 4(4): 315–324.
[46] Emanuelle S, Hossain MI, Moller IE, et al. SnRK1 fromis an atypical AMPK. Plant J, 2015, 82(2): 183–192.
[47] Zhang YH, Shewry PR, Jones H, et al. Expression of antisense SnRK1 protein kinase sequence causes abnormal pollen development and male sterility in transgenic barley. Plant J, 2001, 28(4): 431–441.
[48] Polge C, Thomas M. SNF1/AMPK/SnRK1 kinases, global regulators at the heart of energy control? Trends Plant Sci, 2007, 12(1): 20–28.
[49] Honigberg SM, Lee RH. Snf1 kinase connects nutritional pathways controlling meiosis in. Mol Cell Biol, 1998, 18(8): 4548–4555.
[50] Tsai AYL, Gazzarrini S. AKIN10 and FUSCA3 interact to control lateral organ development and phase transitions in. Plant J, 2012, 69(5): 809–821.
[51] Liu JX, Howell SH. bZIP28 and NF-Y transcription factors are activated by ER stress and assemble into a transcriptional complex to regulate stress response genes in. Plant Cell, 2010, 22(3): 782–796.
[52] Jensen RB, Lacour T, Albrethsen J, et al. FYVE zinc-finger proteins in the plant model: identification of PtdIns3P-binding residues by comparison of classic and variant FYVE domains. Biochem J, 2001, 359(1): 165–173.
[53] Zhu EG, You CJ, Wang SS, et al. The DYT1-interacting proteins bHLH010, bHLH089 and bHLH091 are redundantly required foranther development and transcriptome. Plant J, 2015, 83(6): 976–990.
[54] Zhang F, Yao J, Ke JY, et al. Structural basis of JAZ repression of MYC transcription factors in jasmonate signalling. Nature, 2015, 525(7568): 269–273.
[55] Carrick DM, Lai WS, Blackshear PJ. The tandem CCCH zinc finger protein tristetraprolin and its relevance to cytokine mRNA turnover and arthritis. Arthritis Res Ther, 2004, 6(6): 248–264.
[56] Kouzarides T. Chromatin modifications and their function. Cell, 2007, 128(4): 693–705.
[57] Sanchez R, Zhou MM. The PHD finger: a versatile epigenome reader. Trends Biochem Sci, 2011, 36(7): 364–372.
[58] Aasland R, Gibson TJ, Stewart AF. The PHD finger: implications for chromatin-mediated transcriptional regulation. Trends Biochem Sci, 1995, 20(2): 56–59.
[59] Yang XH, Makaroff CA, Ma H. Thegene encodes a PHD-Finger protein that is required for male meiosis. Plant Cell, 2003, 15(6): 1281–1295.
[60] Nijhawan A, Jain M, Tyagi AK, et al. Genomic survey and gene expression analysis of the basic leucine zipper transcription factor family in rice. Plant Physiol, 2008, 146(2): 333–350.
[61] Ledent V, Vervoort M. The basic helix-loop-helix protein family: comparative genomics and phylogenetic analysis. Genome Res, 2001, 11(5): 754–770.
[62] Fu ZZ, Yu J, Cheng XW, et al. The rice basic helix-loop-helix transcription factor TDR INTERACTING PROTEIN2 is a central switch in early anther development. Plant Cell, 2014, 26(4): 1512–1524.
[63] Jung KH, Han MJ, Lee YS, et al. Riceis a major regulator of early tapetum development. Plant Cell, 2005, 17(10): 2705–2722.
[64] Li N, Zhang DS, Liu HS, et al. The riceretardation gene is required for tapetum degradation and anther development. Plant Cell, 2006, 18(11): 2999–3014.
[65] Kühn K, Carrie C, Giraud E, et al. The RCC1 family protein RUG3 is required for splicing of nad2 and complex I biogenesis in mitochondria of. Plant J, 2011, 67(6): 1067–1080.
[66] Feiler HS, Desprez T, Santoni V, et al. The higher plantencodes a functional CDC48 homologue which is highly expressed in dividing and expanding cells. EMBO J, 1995, 14(22): 5626–5637.
Global proteomic and phosphoproteomic analysis of the premature maize anther
Zhimin Zhang*, Juanying Ye*, Haifei Long, Yue Hong, and Pingli Lu
Institute of Plant Biology, School of Life Sciences, Fudan University, Shanghai 200438, China
Reversible phosphorylation plays a crucial role in regulating protein activities and functions. Sexual reproduction directly affects yield of most agricultural crops. As the male reproductive organ, anther generates microspores (pollen), delivering gametes (sperms) to complete double fertilization in higher plants. Here, we took the advantage of Nano UHPLC-MS/MS to analyze maize (, B73) early anthers at proteomic and phosphoproteomic levels, to explore the protein and phosphorylation modification regulatory networks controlling maize anther development. Our proteomic analysis identified 3 016 unique peptides, belonging to 1 032 maize proteins. MapMan analysis revealed variously potential proteins associated with maize anther development, such as receptor-like kinases (GRMZM2G082823_P01 and GRMZM5G805485_P01). Using phospho-peptides enriched by TiO2affinity chromatography, our phosphoproteomic analysis detected 257 phospho-peptides from 210 phosphoproteins, discovering 223 phosphosites. Compared to the 86 maize phosphoproteins collected in the Plant Protein Phosphorylation Data Base (P3DB), we found that 203 phosphoproteins and 218 phosphosites were not revealed before. Further bioinformatics analysis revealed that phosphorylation of 14-3-3 proteins, kinases, phosphatases, transcription factors, cell cycle and chromatin structure related proteins might play important roles in regulating normal anther development in maize. Our findings not only enlarged the maize phosphoproteome data, but also provided information for analyzing the molecular mechanism controlling maize anther development at genetic and biochemical levels.
maize anther, proteome, phosphorylation, kinase, regulatory network
October 23, 2015; Accepted: December 25, 2015
农业生物技术
张智敏, 叶娟英, 龙海飞, 等. 玉米早期花药蛋白质组和磷酸化蛋白质组分析. 生物工程学报, 2016, 32(7): 937–955.
Zhang ZM, Ye JY, Long HF, et al. Global proteomic and phosphoproteomic analysis of the premature maize anther. Chin J Biotech, 2016, 32(7): 937–955.
Supported by: Shanghai Pujiang Talent Program to P.L. (No. 14PJ1400900).
Corresponding author: Pingli Lu. Tel: +86-21-51630535; E-mail: pinglilu@fudan.edu.cn
*These authors contributed equally to this study.
上海市浦江人才计划 (No. 14PJ1400900) 资助。
(本文责编 陈宏宇)

