小麦矮秆种质SN224的鉴定及农艺性状QTL分析
王 鑫马莹雪杨 阳王丹峰殷慧娟王洪刚,*
1山东农业大学农学院, 山东泰安 271018;2国家小麦改良中心泰安分中心, 山东泰安271018
小麦矮秆种质SN224的鉴定及农艺性状QTL分析
王 鑫1,2马莹雪1,2杨 阳1,2王丹峰1殷慧娟1王洪刚1,2,*
1山东农业大学农学院, 山东泰安 271018;2国家小麦改良中心泰安分中心, 山东泰安271018
SN224是从六倍体小黑麦与普通小麦杂种后代选育的矮秆小麦种质, 为对其有效利用提供参考依据, 本研究对其进行了细胞学和主要农艺性状的鉴定, 对矮秆性状的遗传特点进行了分析。结果表明, SN224平均株高68.6 cm,株型较紧凑, 纺锤穗、有芒、白粒, 千粒重42.0 g左右, 中抗条锈病和白粉病, 后期不早衰, 综合农艺性状较好; SN224根尖细胞染色体数目为42条, 花粉母细胞减数分裂MI可观察到21个二价体, 为1BL·1RS易位系; SN224/辉县红杂种F1株高介于双亲之间, F2群体的株高分离表现连续变异。利用已知主效矮秆基因Rht-B1b、Rht-D1b和Rht8以及1RS的特异分子标记检测证明, SN224不含有3个矮秆主效基因, 1RS对SN224矮秆性状的表达没有影响。利用SN224/辉县红 F2群体, 构建了含有 134个标记的分子标记连锁遗传图谱, 总长 1332.1 cM。采用加性-完备区间作图法(ICIM-ADD)进行 QTL分析, 检测到 2个降低株高的主效 QTL QPh1B和 QPh4B, 分别位于 1B染色体Xwmc719-Xgwm18和4B染色体Xgwm368-Xmag4284标记区间, 它们可分别解释株高变异的20.0%和10.2%; 检测到分别控制穗长、单株穗数和每穗小穗数的7个QTL; 在4B染色体KSUM062-Xmag4284标记区间同时检测到降低株高、增加穗长和单株穗数的QTL。
普通小麦-黑麦易位系; 农艺性状; 细胞学鉴定; QTL定位; 矮秆基因
小麦是世界性的重要粮食作物, 不断提高其产量水平对粮食安全至关重要。自20世纪60年代第一次“绿色革命”以来, 矮秆和半矮秆基因在小麦育种中得到广泛利用, 极大地提高了小麦的抗倒伏性及其单位面积的产量水平[1-3]。目前在已经发现的25个矮秆基因Rht中, 11个来自自然突变, 14个来自物理和化学诱变[4]。但是, 在小麦育种中应用较多的矮秆基因主要集中在来自“农林10号”的Rht-B1b (Rht1)和Rht-D1b (Rht2)以及“赤小麦(Akagomugi)”的Rht8 和 Rht9[5-6], 矮源比较单一。研究结果表明, 世界上70%以上的小麦品种的矮秆基因来自“农林10号”[7];在中国主要麦区的推广小麦品种和种质系中,Rht-B1b和Rht-D1b的平均分布频率分别达到24.3% 和46.9%[8]。鉴定和发现新的矮秆基因, 对丰富小麦矮秆性状的遗传多样性, 提高小麦品种的抗倒伏能力具有重要意义。
贾继增等[9]曾将我国小麦育种中的主要矮秆亲本分为四类。随着小麦育种工作的发展和SSR分子标记技术在分子标记辅助育种中的应用, 一批新的矮秆种质材料被发现和鉴定, 丰富了小麦育种的矮秆基因资源。石涛等[10]分析了山农 31504-1矮秆基因的遗传特点, 将其携带的主效矮秆基因定位在2AS上。宗浩等[11]对矮秆种质山农495分析证明, 其矮秆性状由位于染色体4BS上的1对隐性主效基因控制, 且为赤霉酸不敏感型。韩静然等[12]的鉴定结果表明, 冬小麦新种质N0238D具有矮秆、大穗、抗病、中早熟的特点, 其矮秆性状由染色体4B的1对主效隐性基因控制, 同时存在微效基因的影响, 并筛选到 1个与矮秆基因紧密连锁的 SSR标记Xgwm251。武军等[13]在普通小麦品种7182与华山新麦草(Psathyrostachys huashanica)杂交后代中选育出矮秆种质 B62, 并对其农艺性状特点和外源遗传物质进行了鉴定检测。杨恩年等[14]鉴定分析表明, 小麦种质 SW3243具有矮秆、多花、大穗、高产、迟播早熟等特点, 并用其育成一批高产新品系。闫美等[15]分析山农矮330的综合农艺性状及矮秆性状的遗传特点, 发现其分蘖成穗力强、每穗小穗数和穗粒数多、结实性好、综合抗病性较好, 其矮秆性状受1对不完全显性基因控制。付颖等[16]分析证明, 矮秆种质 N0381D为赤霉酸不敏感型, 矮秆性状由位于染色体2DL上的1对主效隐性基因控制, SSR标记Xwmc503与矮秆基因连锁。王刚等[17]对百农矮抗58的矮秆基因进行分子标记检测, 证明其携带Rht-D1b。欧俊梅等[18]对新创糯小麦种质 11-805分析发现, 其矮秆性状由 1对隐性主效基因控制, 并受一些微效基因的影响。张明等[19]鉴定结果表明,矮秆种质山农 342-9为赤霉酸不敏感型, 其矮秆性状受6B染色体上的1对隐性主效基因控制。昝凯等[20]鉴定证明, 从农家二棱大麦与普通小麦7182杂种后代选育的矮秆种质WB29的矮秆基因可能位于大麦染色体2HS上。崔淑佳等[21]分析发现, 2个小偃麦矮秆种质山农31505-1和山农31505-2是不同的矮秆小偃麦易位系, 其降秆效应可达 16%~23%, 其中山农31505-1的偃麦草染色体片段位于2D染色体。杨秋平等[22]对山农 11069-5鉴定分析证明, 其矮秆性状受 1对不完全显性主效基因控制, 对赤霉酸反应不敏感, 矮秆基因位于染色体4BS。
SN224是从六倍体小黑麦(AABBRR, 2n = 6x)和普通小麦(Triticum aestivum L. AABBDD, 2n = 6x)杂交后代中选育出来的矮秆种质材料。本研究鉴定SN224的主要农艺性状, 并分析其染色体构成及矮秆性状的遗传特点, 为其有效利用提供参考依据。
1 材料与方法
1.1 供试材料
矮秆小麦种质 SN224, 由本实验室利用从国际玉米小麦改良中心(CIMMYT)引进的六倍体小黑麦(引进号为 20068021)与普通小麦品系 SN4076和SN909复合杂交, 从其杂种后代选育而成。利用SN224与辉县红杂交获得F1, 再经自交获得F2分离群体(223个单株)。小麦品种农林10号、赤小麦、烟农15和辉县红及普通黑麦(Secale cereale)由国家小麦改良中心山东(泰安)分中心保存。
1.2 农艺性状调查
2013年10月6日和2014年10月7日将供试材料种植于山东农业大学农学实验站, 种植行长1.2 m, 株距6 cm, 行距30 cm, 其中种植SN224、辉县红及其杂种F1各2行, 按照正常田间管理。参考《农作物品种区域试验技术规程——小麦》(NY/T 1301-2007)[23]调查农艺性状, 从亲本及 F1材料随机选取10个单株统计其性状平均值。株高为植株从地面至穗顶端(不包括芒)的长度(cm), 于小麦成熟期测量; 株高构成指数(IL)为穗下节间和倒二节间长度之和与株高的比值; 芒形和穗形均采用五级标准。2014年6月和2015年6月收获后, 选取样本株的主茎测量各节间长度、穗长、每穗小穗数及每穗穗粒数。用SPSS 17.0 (Chicago, USA)软件统计分析数据。
1.3 抗病性调查
以辉县红种植感染行并作为感病对照, 于小麦拔节期人工注射接种条锈病菌CYR32和CYR33混合菌种(中国农业科学院植物保护研究所刘太国提供), 待辉县红充分发病后, 参照《小麦抗病虫性评价技术规范——第 1部分: 小麦抗条锈病评价技术规范》(NY/T 1443.1-2007)[24], 按反应型0~4分级标准记载反应型。在田间自然发病条件下调查成株期白粉病抗性, 以感病品种辉县红为感染行, 借助感染行诱发感染, 当辉县红充分发病后按照盛宝钦[25]提出的反应型0~4分级标准调查记载。
1.4 细胞学鉴定
参照He等[26]描述的方法分析根尖细胞(root tip cell, RTC)染色体数目及花粉母细胞(pollen mother cell, PMC)减数分裂染色体构型。参照 Bao等[27]和Tang等[28]描述的方法进行基因组原位杂交(genomic in situ hybridization, GISH)和荧光原位杂交(fluorescent in situ hybridization, FISH)。荧光探针pTa-535 和pSc119.2由生工生物工程(上海)股份有限公司合成。
1.5 矮秆基因的分子标记检测
按照杨松杰等[8]和Ahmad等[6]报道的矮秆基因Rht-B1b、Rht-D1b和Rht8的特异分子标记及方法检测矮秆基因, 以含有Rht-B1b、Rht-D1b的农林10号,含Rht-D1b的辉县红和Rht8的赤小麦作为对照。用于检测 1RS的特异分子标记 SCM7364序列为 F:5'-AGGCGGAGATGAAGTTCTTCT-3', R: 5'-CGCCT GGACGAACTTG-3'。
1.6 PCR反应
选用冻干叶片并参照 Stein等[29]提出的方法提取植物总 DNA。用于构建连锁遗传图谱的 SSR、EST-SSR和STS引物共计2552对, 均由生工生物工程(上海)股份有限公司合成。
参照Li等[30]的PCR方法, 采用Bio-Rad T100 Thermal PCR仪(Bio-Rad Laboratories, Inc., Singapore)。反应体系10 μL, 含100 ng模板DNA、10 pmol μL-1上下游引物各 1 μL、5 μL 2×Power Taq PCR MasterMix (Bioteke公司, 北京)。采用 touchdown PCR, 94℃预变性3 min, touchdown程序包含15个循环, 每循环94℃变性45 s, 65℃退火50 s, 72℃延伸55 s, 每循环减少1℃, 其后是30个循环, 每循环94℃变性40 s, 50℃退火40 s, 72℃延伸40 s, 最后72℃完全延伸5 min。PCR扩增产物经8%非变性聚丙烯酰胺凝胶电泳检测, 银染, 照相。
1.7 遗传连锁图谱构建及QTL分析
用 JoinMap v3.0 (Biometris, Wageningen, Netherlands, http://www.joinmap.nl/)构建连锁遗传图谱, 用Kosambi作图函数计算遗传距离[31], 设定LOD值为2.0。以QTL IciMapping v4.0[32]软件分析QTL, 采用加性-完备区间作图法(additive-inclusive composite interval mapping, ICIM-ADD)[33], 设定 LOD 值为2.5。
2 结果与分析
2.1 SN224的主要农艺性状
田间表型调查和考种结果表明(表1), SN224株型较紧凑, 纺锤穗, 有芒, 白粒; 株高67.6~70.1 cm,平均68.6 cm, IL为0.49; 千粒重42.0 g; 中抗条锈病和白粉病; 综合农艺性状较好, 后期不早衰。
2.2 SN224的细胞学鉴定
对 SN224的根尖细胞(RTC)有丝分裂和花粉母细胞减数分裂中期I (PMC MI)的染色体鉴定分析表明, SN224的根尖细胞染色体数目为42条, PMC MI的染色体构型为2n = 21II, 没有发现单价体和多价体。以488-5-dUTP标记的黑麦基因组DNA为探针,普通小麦品种烟农 15总基因组 DNA为封阻, 对SN224进行GISH分析发现, SN224 RTC的42条染色体中有 2条染色体为小麦-黑麦易位染色体(图1-a), PMC MI的21个二价体中有一个二价体具黑麦染色质信号(图1-b), 说明SN224为小麦-黑麦染色体易位系。进一步以人工合成的荧光探针 pTa-535 和pSc119.2对SN224进行FISH分析证明, SN224中的黑麦染色体片段均为1R短臂(图1-c), SN224是一个1BL·1RS易位系, 且具有较高的细胞学稳定性。
2.3 SN224矮秆性状的遗传特点及QTL分析
2.3.1 SN224矮秆性状的遗传特点 SN224 (株高68.6 cm)与辉县红(株高131.4 cm)杂交的F1平均株高 90.0 cm, 表明 SN224对 F1的降秆效应达到49%。将F1自交获得F2群体, 对223株F2分析表明,其株高呈现连续变异的正态分布特点(图2), 株高变异范围为54~131 cm, 标准差13.9, 偏度和峰度分别为-0.39和-0.21, 均小于1.0[34], 在群体中可发现双向超亲分离, 说明SN224的矮秆性状符合数量性状遗传的分离特点。
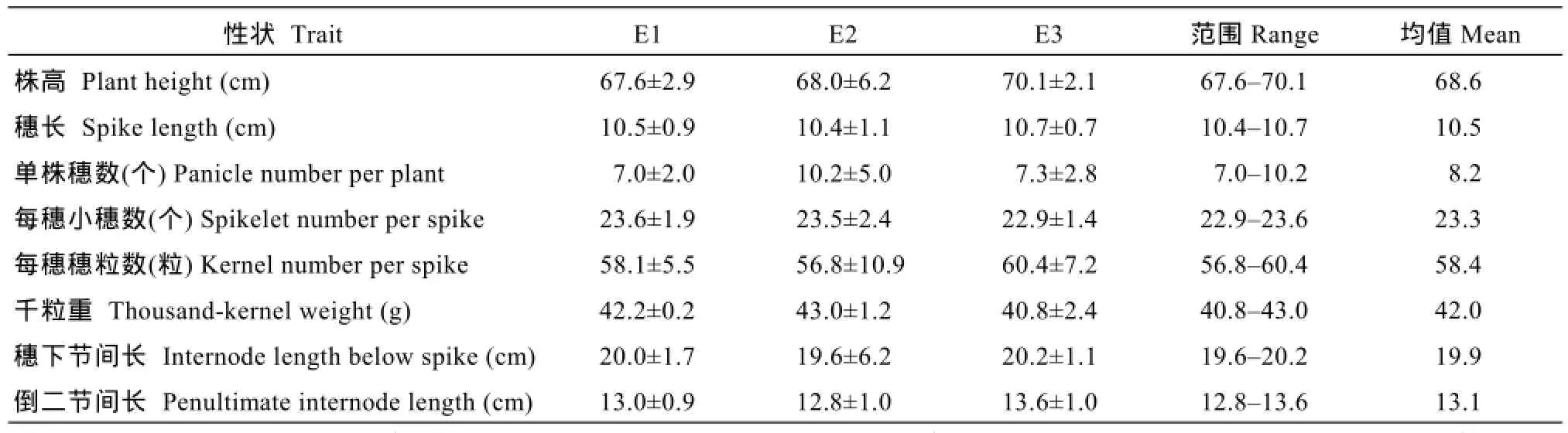
表1 SN224的主要农艺性状特点Table 1 Main agronomic traits of SN224

图1 SN224的细胞学鉴定Fig. 1 Cytological identification of SN224
2.3.2 SN224中矮秆基因的检测 为了明确SN224中是否存在已知的矮秆主效基因, 利用引物对NH-BF.2/MR1 (Rht-B1b基因特异扩增产物约400 bp)和 NH-BF.2/WR1.2 (Rht-B1a基因特异扩增产物约400 bp)分别进行了检测, 在同一材料中出现两对引物的扩增产物互补, 可以相互验证。同时, 利用引物对DF/MR2 (Rht-D1b特异扩增产物254 bp)和分子标记Xgwm261 (Rht8特异扩增产物192 bp)进行了检测。
检测结果表明, 在农林10号中可以扩增出Rht-B1b的400 bp特异条带和Rht-D1b的254 bp特异条带, 说明农林10号中携带Rht-B1b和Rht-D1b基因;在辉县红中可以扩增出Rht-D1b的254 bp特异条带,而没有出现Rht-B1b的400 bp特异条带, 说明辉县红只携带 Rht-D1b基因; 在 SN224中未扩增出Rht-B1b特异的400 bp条带、Rht-D1b特异的254 bp条带和Rht8特异的192 bp条带, 说明SN224不携带Rht-B1b、Rht-D1b和Rht8矮秆基因(图 3)。采用定位在1RS上的特异SSR标记SCM7364对SN224和F2群体检测表明, SCM7364与矮秆性状不存在连锁关系, 说明1RS对SN224矮秆性状的表达没有影响。
2.3.3 连锁遗传图谱构建和 QTL分析 利用2552对 SSR引物在 SN224和辉县红中筛选出 229对多态性引物, 进一步利用SN224/辉县红F2群体构建了包含134个标记, 全长1332.1 cM的连锁遗传图谱(图4)。该图谱共有19个连锁群, 涉及15条染色体(2A、2D、3D、4D、5D、7D除外), 标记间平均距离9.94 cM。

图2 SN224/辉县红F2群体株高的频率分布Fig. 2 Frequency distribution of plant height in SN224/Huixianghong F2population
利用构建的分子标记连锁遗传图谱在1B、4B、6AL和6D染色体上检测到4个控制株高的QTL (图4和表 2), 可解释 7.0%~20.0%的表型变异。在 4A 和4B染色体上分别检测到QEl4A.1、QEl4A.2、QEl4B三个控制穗长的QTL, 在3A、6AL和4B染色体上分别检测到QEs3A、QEs6A、QEs4B三个控制单株穗数的QTL, 在3BL染色体上检测到1个控制每穗小穗数的QTL QKn3B。
在控制株高的QTL中, 表型效应最大的是位于1B染色体Xwmc719-Xgwm18 (区间长度5.4 cM)标记区间的QPh1B, 该QTL的LOD值为7.2, 可解释表型变异的 20.0%, 是主效降秆 QTL; 在 4B染色体Xgwm368-Xmag4284 (区间长度7.1 cM)标记区间内的 QPh4B, 可降低株高约 5.8 cm, 能解释表型变异的10.2%, 为主效QTL。这2个QTL均来自SN224,它们累计可解释株高变异的30.2%。其余2个QTL,即QPh6D和QPh6A均来自辉县红的第六部分同源群, 可分别解释表型变异的7%, 降秆效应较小。
此外, 在区间 KSUM062-Xmag4284 (区间长度8.3 cM)内存在3个QTL (QPh4B、QEl4B和QEs4B),其中QPh4B和QEl4B的距离为2.0 cM, QPh4B和QEs4B的距离为3.0 cM; QPh4B在SN224中为主效降秆QTL, 而QEl4B可以增加SN224的穗长0.5 cm,可解释穗长变异的 10.2%, QEs4B可以增加 SN224的单株穗数2.0个, 可解释表型变异的15.4%, 说明该标记区间对于降低株高、增加穗长和单株穗数具有正效影响。在6AL的CWM79-Xcinau119 (区间长度13.6 cM)区间内检测到两个微效QTL (QPh6A和QEs6A), 对SN224的株高和单株穗数都具有正效应,QPh6A可以提高株高0.2 cm, QEs6A可以增加单株穗数0.9个。
3 讨论
在现代小麦育种中, 无论是纯系品种的选育还是杂种优势的利用, 通过矮秆基因降低株高、提高产量已成为重要的育种策略之一。在小麦生产中,高秆小麦品种在高肥水栽培条件下经常容易倒伏而减产, 而矮秆和半矮秆品种提高了品种的抗倒伏能力和收获指数, 从而获得高产。不断挖掘新的矮秆基因, 创造新的矮秆种质材料, 对于小麦品种株高性状的遗传改良具有重要意义。本研究鉴定的小麦矮秆种质SN224是从六倍体小黑麦与普通小麦杂交后代选育的1BL·1RS易位系, 平均株高68.6 cm, 具有矮秆、大穗、株型较紧凑、中抗条锈病和白粉病、综合农艺性状良好、后期不早衰等优良特点。初步杂交利用结果表明, SN224对杂种的降秆效应明显,杂种后代株高分离类型丰富, 对其他农艺性状没有不良影响, 在小麦育种中具有利用价值。
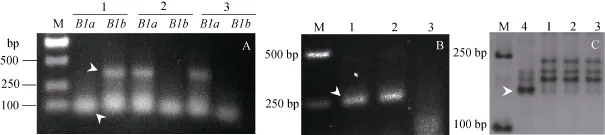
图3 SN224中矮秆位点Rht-B1 (A)、Rht-D1b (B)和Rht8 (C)的分子检测Fig. 3 Molecular detection of dwarfing genes Rht-B1 (A), Rht-D1b (B) and Rht8 (C) in SN224

图4 SN224/辉县红F2群体的连锁遗传图Fig. 4 Genetic linkage map of SN224/Huixianhong F2population

表2 SN224/辉县红F2QTL的检测结果Table 2 Detective results of QTL in SN224/Huixianhong F2population
小麦矮秆性状的遗传可分为由单基因控制的质量性状遗传和由多基因控制的数量性状遗传 2种类型, 但在生产上应用矮源的矮秆性状主要是单基因控制的质量性状, 且利用的矮秆基因主要是 Rht系列, 而对多基因控制的矮秆性状的研究较少。利用常用的主效矮秆基因Rht-B1b、Rht-D1b和Rht8的特异分子标记对SN224检测表明, SN224不含有上述矮秆基因。以SN224与辉县红杂交获得杂种F1, 其株高介于双亲之间, 在F2群体中株高的分离表现为连续变异的特点, 说明 SN224的株高性状的分离具有数量性状的特征, 可能受多基因控制。利用分子标记对SN224/辉县红F2群体进行QTL分析, 检测到4个与株高性状连锁的QTL, 其中2个降低株高的主效QTL分别位于小麦染色体1B和4B上, 均来自SN224, 它们累计可解释株高变异的30.2%。其中位于1B染色体 Xwmc719-Xgwm18区间的QPh1B,可解释20.0%的表型变异, 在4B染色体Xgwm368-Xmag4284区间内的 QPh4B, 可解释表型变异的10.2%。目前, 在1B染色体上还没有发现Rht类型基因的报道, 检测的矮秆QTL数量也较少。Liu等[35]曾在1B染色体Xgwm191.5-Xgwm191.6区间内检测到 1个主效降秆 QTL, 毕晓静[36]在 1B染色体Xgwm268-Xwmc419a区间内发现 1个微效降秆QTL。陈广凤等[37]利用SNP标记对株高相关性状进行关联分析, 在1B染色体上检测到与株高显著关联的SNP位点。本研究在1B染色体上检测到的QPh1B与上述QTL所在的标记区间不同, 可能是新的QTL。此外, 在小麦的4B染色体上已有定位的Rht-B1b基因,本研究利用与 Rht-B1b紧密连锁的分子标记在SN224中未检测出特异条带, 说明 SN224不含有Rht-B1b; 但在 SN224的 4B 染色体 Xgwm368-Xmag4284区间内检测到QPh4B, 它是一个能有效降低株高的主效QTL。这表明SN224可能是一个新的矮秆种质材料, 它能够有效降低其杂种后代的株高。
研究结果表明, Rht基因除降低株高外, 往往对其他农艺性状产生不良影响, 如减少穗粒数或降低千粒重等[38]。本研究在 4B染色体 KSUM062-Xmag4284区间检测到3个QTL (QPh4B、QEl4B和QEs4B), 携带这一标记区段的矮秆亲本SN224的穗长和单株穗数与高秆亲本辉县红差异显著(穗长8.6 cm, t = 12.0, t0.01,2= 2.9; 单株穗数5.9个, t = 7.4,t0.01,2= 2.9), 其中QPh4B为主效降秆QTL, QEl4B和QEs4B分别可以增加SN224的穗长0.5 cm和单株穗数2.0个, 说明该标记区间对降低株高、增加穗长和单株穗数具有正效影响, 因此该区段可能是一个重要的染色体区段, 值得进一步深入研究。
4 结论
矮秆种质SN224为1BL·1RS易位系, 平均株高68.6 cm, 综合农艺性状良好。其矮秆特性受多基因控制。利用F2分离群体在1B和4B染色体上检测到主效降秆QTL QPh1B和QPh4B, 可解释株高变异的30.2%; 在4B染色体KSUM062-Xmag4284区间同时检测到降低株高、增加穗长和单株穗数的 QTL。SN224可能是一个新的矮秆种质材料, 在小麦育种中具有利用价值。
References
[1] Peng J, Richards D E, Hartley N M, Murphy G P, Devos K M,Flintham J E, Beales J, Fish L J, Worland A J, Pelica F, Sudhakar D, Christou P, Snape J W, Gale M D, Harberd N P. “Green revolution” genes encode mutant gibberellin response modulators. Nature, 1999, 400: 256-261
[2] Hedden P. The genes of the green revolution. Trends Genet, 2003,19: 5-9
[3] Saville R J, Gosman N, Burt C J, Makepeace J, Steed A, Corbitt M, Chandler E, Brown J K M, Boulton M I, Nicholson P. The ‘Green Revolution’ dwarfing genes play a role in disease resistance in Triticum aestivum and Hordeum vulgare. J Exp Bot, 2011,63: 1271-1283
[4] 嵇怡, 旻缪珉, 陈学好. 植物矮生性状的分子遗传研究进展.分子植物育种, 2006, 4: 753-771
Ji Y, Miao M M, Chen X H. Progress on the molecular genetics of dwarf character in plants. Mol Plant Breed, 2006, 4: 753-771 (in Chinese with English abstract)
[5] Cadalen T, Sourdille P, Charmet G, Tixier M H, Gay G, Boeuf C,Bernard S, Leroy P, Bernard M. Molecular markers linked to genes affecting plant height in wheat using a doubledhaploid population. Theor Appl Genet, 1998, 96: 933-940
[6] Ahmad M, Sorrells M E. Distribution of microsatellite alleles linked to Rht8 dwarfing gene in wheat. Euphytica, 2002, 123:235-240
[7] Wang Z, Wu X, Ren Q, Chang X, Li R, Jing R. QTL mapping for developmental behavior of plant height in wheat (Triticum aestivum L.). Euphytica, 2010, 174: 447-458
[8] 杨松杰, 张晓科, 何中虎, 夏先春, 周阳. 用 STS标记检测矮秆基因Rht-B1b和Rht-D1b在中国小麦中的分布. 中国农业科学, 2006, 39: 1680-1688
Yang S J, Zhang X K, He Z H, Xia X C, Zhou Y. Distribution of dwarfing genes Rht-B1b and Rht-D1b in Chinese bread wheats detected by STS marker. Sci Agric Sin, 2006, 39: 1680-1688 (in Chinese with English abstract)
[9] 贾继增, 丁寿康, 李月华, 张辉. 中国小麦的主要矮秆基因及矮源的研究. 中国农业科学, 1992, 25(1): 1-5
Jia J Z, Ding S K, Li Y H, Zhang H. Studies of main dwarf genes and dwarf resources on Chinese wheat. Sci Agric Sin, 1992, 25(1):1-5 (in Chinese with English abstract)
[10] 石涛, 王洪刚, 何方, 邓世民, 高居荣. 小麦矮秆新基因的SSR标记. 山东农业科学, 2008, (8): 1-5
Shi T, Wang H G, He F, Deng S M, Gao J R. Identification of SSR markers linked to new wheat dwarf gene. Shandong Agric Sci, 2008, (8): 1-5 (in Chinese with English abstract)
[11] 宗浩, 崔法, 鲍印广, 赵春华, 王玉海, 杜斌, 王庆专, 王洪刚.小麦矮秆种质系山农 495矮秆基因的分子标记定位. 麦类作物学报, 2009, 29: 385-389 Zong H, Cui F, Bao Y G, Zhao C H, Wang Y H, Du B, Wang Q Z,Wang H G. Developing molecular markers for the Rht gene in dwarfing germplasm line Shannong 495. J Triticeae Crops, 2009,29: 385-389 (in Chinese with English abstract)
[12] 韩静然, 王长有, 赵宁娟, 吉万全, 赵毓, 刘雪利. 冬小麦新种质 N0238D矮秆性状的遗传分析. 西北农业学报, 2010,19(6): 60-63
Han J R, Wang C Y, Zhao N J, Ji W Q, Zhao Y, Liu X L. Genetic analysis of dwarf trait in new winter wheat germplasm N0238D. Acta Agric Boreali-Occident Sin, 2010, 19(6): 60-63 (in Chinese with English abstract)
[13] 武军, 马琳, 赵继新, 陈新宏, 刘淑会, 杨群慧. 普通小麦-华山新麦草矮秆种质 B62的分子细胞学鉴定. 西北农林科技大学学报(自然科学版), 2010, 38(12): 123-127
Wu J, Ma L, Zhao J X, Chen X H, Liu S H, Yang Q H. Molecular cytology on a dwarf germplasm derived from Triticum aestivum × Psathyrosatachys huashanica. J Northwest A&F Univ (Nat Sci Edn), 2010, 38(12): 123-127 (in Chinese with English abstract)
[14] 杨恩年, 李俊, 杨武云, 邹裕春. 矮秆大穗高产小麦育种亲本SW3243重要农艺性状特性及育种应用效果. 中国农学通报,2010, 26(12): 114-117
Yang E N, Li J, Yang W Y, Zou Y C. The agronomic traits and the utilization of dwarf, large spike and high yielding potential wheat breeding parent SW3243. Chin Agric Sci Bull, 2010, 26(12):114-117 (in Chinese with English abstract)
[15] 闫美, 刘如如, 于海涛, 达瓦, 格桑, 李安飞, 孔令让. 小麦矮秆种质山农矮 330的农艺性状与矮化特性研究. 山东农业科学, 2011, (8): 14-17
Yan M, Liu R R, Yu H T, Da W, Ge S, Li A F, Kong L R. Agronomic traits and dwarfing characteristics of wheat dwarf germplasm Shannong’ai 330. Shandong Agric Sci, 2011, (8): 14-17 (in Chinese with English abstract)
[16] 付颖, 吴金华, 王长有, 张保军, 吉万全. 小麦新种质N0381D矮秆基因的遗传与 SSR标记分析. 麦类作物学报, 2011, 31:411-415
Fu Y, Wu J H, Wang C Y, Zhang B J, Ji W Q. Genetic and SSR analysis of dwarf gene in new wheat germplasm N0381D. J Triticeae Crops, 2011, 31: 411-415 (in Chinese with English abstract)
[17] 王刚, 胡铁柱, 李小军, 董娜, 冯素伟, 李淦, 张立琳, 茹振钢.小麦新品种百农矮抗58及其亲本矮秆基因的检测. 河南农业科学, 2012, 41(9): 22-25
Wang G, Hu T Z, Li X J, Dong N, Feng S W, Li G, Zhang L L, Ru Z G. Detection of dwarfing genes in wheat variety AK58 and its parents. J Henan Agric Sci, 2012, 41(9): 22-25 (in Chinese with English abstract)
[18] 欧俊梅, 王治斌, 周强, 任勇, 李生荣, 陶军. 糯小麦矮源新种质11-805矮秆性状遗传研究. 中国农学通报, 2014, 30(24):152-155
Ou J M, Wang Z B, Zhou Q, Ren Y, Li S R, Tao J. Genetic rule of dwarf trait in new waxy dwarf wheat 11-805. Chin Agric Sci Bull,2014, 30(24): 152-155 (in Chinese with English abstract)
[19] 张明, 吴瑕, 张一铎, 张超, 牛祖彪, 崔淑佳, 杨秋平, 王洪刚.小麦矮秆种质系山农342-9矮秆基因的分子标记定位. 山东农业科学, 2014, 46(7): 7-10
Zhang M, Wu X, Zhang Y D, Zhang C, Niu Z B, Cui S J, Yang Q P, Wang H G. Molecular mapping of Rht gene in wheat dwarfing germplasm line Shannong 342-9. Shandong Agric Sci, 2014,46(7): 7-10 (in Chinese with English abstract)
[20] 昝凯, 郑青焕, 敬樊, 陈真真, 白宇浩, 刘洋, 王亮明, 刘淑会,王中华, 陈新宏. 小麦-大麦矮秆渗入系WB29的分子细胞学鉴定及其矮秆遗传特性分析. 农业生物技术学报, 2015, 23:1273-1281
Zan K, Zheng Q H, Jing F, Chen Z Z, Bai Y H, Liu Y, Wang L M,Liu S H, Wang Z H, Chen X H. Molecular cytogenetic identification of a wheat (Triticum aestivum L.)-barley (Hordeum vulgare ssp. distichon Hsü.) dwarf introgression line WB29 and its dwarf traits genetic analysis. J Agric Biol, 2015, 23: 1273-1281 (in Chinese with English abstract)
[21] 崔淑佳, 卢虹, 崔雨, 高居荣, 王洪刚, 李兴锋. 小麦-长穗偃麦草矮秆种质系的鉴定及遗传分析. 核农学报, 2015, 29:435-441
Cui S J, Lu H, Cui Y, Gao J R, Wang H G, Li X F. Identification and genetic analysis of dwarf wheat-Thinopyrum ponticum germplasms. J Nucl Agric Sci, 2015, 29: 435-441 (in Chinese with English abstract)
[22] 杨秋平, 杨阳, 王鑫, 马莹雪, 张明, 王洪刚. 小麦矮秆种质山农11069-5矮秆基因的遗传分析及分子定位. 分子植物育种,2015, 13: 71-76
Yang Q P, Yang Y, Wang X, Ma Y X, Zhang M, Wang H G. Genetic analysis and molecular mapping of the Rht gene in dwarf germplasm Shannong 11069-5. Mol Plant Breed, 2015, 13: 71-76 (in Chinese with English abstract)
[23] 中华人民共和国农业部. NY/T 1301-2007 农作物品种区域试验技术规程—小麦
Ministry of Agriculture of the People’s Republic of China. NY/T 1301-2007 Technical Procedures for Wheat Variety Regional Trials (in Chinese)
[24] 中华人民共和国农业部. NY/T 1443.1-2007小麦抗病虫性评价技术规范——第1部分: 小麦抗条锈病评价技术规范, 2007
Ministry of Agriculture of the People`s Republic of China. NY/T 1443.1—2007 Rules for resistance evaluation of wheat to diseases and insect pests Part1: Rule for resistance evaluation ofwheat to yellow rust (Puccinia striiformis West. f. sp. tritici Eriks. Et Henn.), 2007 (in Chinese)
[25] 盛宝钦. 用反应型记载小麦苗期白粉病. 植物保护, 1988, (1):49
Sheng B Q. Scoring powdery mildew with infection type at wheat seedling stage. Plant Prot, 1988, (1): 49 (in Chinese)
[26] He F, Xu J, Qi X, Bao Y, Li X, Zhao F, Wang H. Molecular cytogenetic characterization of two partial wheat Elytrigia elongata amphiploids resistant to powdery mildew. Plant Breed, 2013, 132:533-557
[27] Bao Y, Li X, Liu S, Cui F, Wang H. Molecular cytogenetic characterization of a new wheat-Thinopyrum partial amphiploid resistant to powdery mildew and stripe rust. Cyto Geno Res, 2009,126:390-395
[28] Tang Z, Yang Z, Fu S. Oligonucleotides replacing the roles of repetitive sequences pAs1, pSc119.2, pTa-535, pTa71, CCS1, and pAWRC.1 for FISH analysis, J Appl Genet, 2014, 55: 313-318
[29] Stein N, Herren G, Keller B. A new DNA extraction method for high-throughout marker analysis in a large genome species such as Triticum aestivum. Plant Breed, 2001, 120: 354-356
[30] Li J, Cui F, Ding A, Zhao C, Wang X, Wang L, Bao Y, Qi X, Li X,Gao J, Feng D, Wang H. QTL detection of seven quality traits in wheat using two related recombinant inbred line populations. Euphytica, 2002, 183: 207-226
[31] Kosambi D D. The estimation of map distances from recombination values. Ann Eugent, 1944, 12: 172-175
[32] Meng L, Li H H, Zhang L Y, Wang J K. QTL IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations. Crop J, 2015,3: 269-283
[33] Li H, Ribaut J M, Li Z, Wang J. Inclusive composite interval mapping (ICIM) for digenic epistasis of quantitative traits in biparental populations. Theor Appl Genet, 2008, 116: 243-260
[34] Cao G, Zhu J, He C, Gao Y, Yan J, Wu P. Impact of epistasis and QTL × environment interaction on the developmental behavior of plant height in rice (Oryza sativa L.). Theor Appl Genet, 2001,103: 153-160
[35] Liu D C, Gao M Q, Guan R X, Li R Z, Cao S H, Guo X L, Zhang A M, Mapping quantitative trait loci for plant height in wheat (Triticum aestivum L.) using a F2:3population. Acta Genet Sin,2002, 29: 706-711
[36] 毕晓静. 小麦重要农艺性状的遗传分析与 QTL定位. 西北农林科技大学硕士学位论文. 陕西杨凌, 2013
Bi X J. Genetic Analysis and QTL of Agronomic Traits in Wheat. MS Thesis of Dissertation of Northwest A&F University, Yangling, China, 2013
[37] 陈广凤, 陈建省, 田纪春. 小麦株高相关性状与SNP标记全基因组关联分析. 作物学报, 2015, 41: 1500-1509
Chen G F, Chen J S, Tian J C. Genome-wide association analysis between SNP markers and plant height related traits in wheat. Acta Agron Sin, 2015, 41: 1500-1509 (in Chinese with English abstract)
[38] Wang Y, Chen L, Du Y, Yang Z, Condon A G, Hu Y G. Genetic effect of dwarfing gene Rht13 compared with Rht-D1b on plant height and some agronomic traits in common wheat (Triticum aestivum L.). Field Crops Res, 2014, 162: 39-47
Identification of Dwarfing Wheat Germplasm SN224 and Analysis of QTLs for Its Agronomic Traits
WANG Xin1,2, MA Ying-Xue1,2, YANG Yang1,2, WANG Dan-Feng1, YIN Hui-Juan1, and WANG Hong-Gang1,2,*1College of Agronomy, Shandong Agricultural University, Tai’an 271018, China;2Shandong Subcentre of National Wheat Improvement Center,Tai’an 271018, China
SN224 is a dwarfing wheat line derived from the cross between hexaploid triticale (AABBRR, 2n = 6x) and common wheat (AABBDD, 2n = 6x). We evaluated its cytologic characteristic and main agronomic traits, and analyzed the genetic basis of dwarfing trait in order to use the germplasm in wheat breeding program. This white-grain wheat had compact plant type, spindle-shaped panicle and moderate resistance to powdery mildew (Blumeria graminis f. sp. tritici, Bgt) and stripe rust (Puccinia striiformis f. sp. tritici, Pst). There were 42 chromosomes in root tip cells, showing 21 bivalents in pollen mother cells. FISH confirmed that SN224 was a 1BL·1RS translocation line. The plant height was between two parents of F1from a cross between SN224 and Huixianhong and distributed continuously and normally in F2population. The detection of specific molecular marker for genes Rht-B1b, Rht-D1b, and Rht8 indicated that this line had none of the three dwarfing genes. In the meantime, the introduction of 1RS had no obvious effect on plant height. The F2population was used to construct a genetic linkage map containing 134 SSR markers which covered a total length of 1332.1 cM. Two major dwarfing QTLs on chromosomes 1B and 4B were detected by additive-inclusive composite interval mapping (ICIM-ADD). QPh1B and QPh4B, located in the Xwmc719-Xgwm18 and Xgwm368-Xmag4284 intervals, explained 20.0% and 10.2% of phenotypic variation, respectively. Seven QTLs controlling ear length, panicle number per plant, and kernel number per spike were detected. The QTL in KSUM062-Xmag4284 interval contributed to decrease plant height, increase ear length and panicle number per plant.
Common wheat-rye translocation line; Agronomic traits; Cytological identification; QTL mapping; Dwarfing gene
10.3724/SP.J.1006.2016.01134
本研究由“十二五”国家科技支撑计划项目(2013BAD01B02-8)资助。
This study was supported by the National Key Technology R&D Program of China (2013BAD01B02-8).
*
(Corresponding author): 王洪刚, E-mail: hgwang@sdau.edu.cn, Tel: 0538-8242141
联系方式: E-mail: 627407093@qq.com
Received(
): 2015-12-15; Accepted(接受日期): 2016-05-09; Published online(网络出版日期): 2016-05-23. URL: http://www.cnki.net/kcms/detail/11.1809.S.20160523.0853.010.html

