麻竹DlSCL6基因amiRNA前体合成及表达载体构建
陈东亮孙化雨李利超赵韩生高志民(1北京市农林科学研究院北京农业生物技术研究中心北京100097 国际竹藤中心国家林业局竹藤科学与技术重点实验室北京10010)
麻竹DlSCL6基因amiRNA前体合成及表达载体构建
陈东亮1,2孙化雨2李利超2赵韩生2高志民2
(1北京市农林科学研究院北京农业生物技术研究中心北京100097 2国际竹藤中心国家林业局竹藤科学与技术重点实验室北京100102)
Web MicroRNA Designer(WMD3)是一个专门应用于植物人工miRNA设计和前体序列设计的专业网站,其中利用重叠PCR法合成amiRNA前体序列,涉及多个PCR和纯化回收过程,步骤繁琐,周期较长。本研究利用WMD3设计了麻竹转录因子DlSCL6基因的人工miRNA序列和其前体序列,采用改进的重叠PCR方法,快速克隆获得了该前体序列,并以改造的pCMABIA1301载体为框架,构建了该前体的植物表达载体,为进一步研究利用amiRNA调控SCL6基因的表达奠定了基础。
麻竹;人工miRNA;重叠PCR;表达载体构建
microRNA(miRNA)是一类生物内源的小分子非编码RNA,广泛存在于动物、植物和微生物中。miRNA调控是基因表达转录后层面的重要调控方式之一,研究表明miRNA在植物的生长发育、物质代谢、信号转导以及抗逆等多种生理生化过程中发挥重要功能[1-3]。人工 miRNA (artificial miRNA,amiRNA)是通过模拟天然miRNA结构特点、作用机制,设计出一段与目标基因高度匹配的序列,利用分子生物学技术将其转入植物体内并使其表达,而达到对该基因抑制调控的目的。amiRNA的有效性已经在拟南芥 (Arabidopsis thaliana)、杨树 (Populus trichocarp)、水稻 (Oryza sativa)等十几种植物的数十个基因中得到验证[4-7]。
2009年,Weigel等发布了 WMD3软件平台(http://wmd3.weigelworld.org/cgi-bin/webapp.cgi),利用这个平台可以设计目标基因的amiRNA以及基于平台提供的若干前体框架的amiRNA表达前体及其合成引物。WMD3实现了amiRNA设计合成的自动化,大大方便了amiRNA设计合成。然而,利用WMD3平台合成amiRNA前体基因的重叠PCR法,不仅需要以其推荐的载体,比如RS300、NW55等为模板,而且PCR合成过程需要多次PCR和回收纯化,过程较为繁琐。本研究利用WMD3平台设计了麻竹(Dendrocalamus latiflorus)DlSCL6基因的amiRNA及基因RS300的表达前体,对原有的重叠PCR合成方法进行了改进。改进的重叠PCR方法,不需要其特定的载体作为模板,并且仅需1次重叠PCR和纯化回收过程,即可获得完整的amiRNA前体序列,大大简化了操作流程和缩短了合成周期。同时,利用pCAMBIA1301载体为框架,构建了DlSCL6基因人工miRNA的植物表达载体,为进一步研究利用amiRNA技术调控DlSCL6基因表达奠定了基础。
1 实验方法
1.1 amiRNA设计方法
利用WMD3在线程序Designer功能设计麻竹DlSCL6基因 (Genbank:KC182562)[8]的 amiRNA,默认以amiRNA优劣排序。选择最优的amiRNA序列,利用psRNAtarget在线程序进行靶位点验证。以RS300载体 (以拟南芥miR319a前体为框架)为模板,利用WMD3程序Oligo功能设计WMD3推荐的方法合成amiRNA前体所需的4条重叠引物。根据重叠引物和RS300模板序列,推导出含有amiRNA 和amiRNA∗的完整前体序列。分别在该amiRNA前体序列的5′端添加BamHⅠ和ClaⅠ2个酶切位点(GGATCCATCGAT),在3′端添加XbaⅠ酶切位点(TCTAGA),含有酶切位点的完整序列命名为preamiR-scl6,并利用mfold在线软件分析pre-amiR-scl6的二级折叠结构。
1.2 amiRNA合成
将设计的pre-amiR-scl6序列分割成10条短片度重叠引物 (P1—P10),每对相邻引物有10 bp左右的重叠碱基 (图1),引物尽量不超过60 nt,且避免不同引物末端连续3个以上碱基重复。改良的重叠PCR法,反应体系如下:引物P1和P10各0.8 μL,引物P2-P8各0.2 μL,PrimeSTAR HS高保真聚合酶 (TAKARA)0.2 μL,5×buffer 4 μL,dNTP 1.6 μL,MQW补足20 μL。通过设置温度梯度,对退火温度进行优化,反应条件为:预变性98℃ 5 min;变性98℃ 10 s,退火30~55℃ 15 s,延伸72℃30 s,35个循环;延伸72℃ 10 min。PCR扩增产物经2%琼脂糖电泳检测,符合预期大小的片段进行回收加A并连接pGEM T-easy载体,阳性克隆送华大基因测序。

图1 组装PCR引物示意图
1.3 amiRNA植物表达构建
以改造的pCAMBIA1301为框架构建表达载体,将测序正确的带有 pre-amiR-scl6的 T载体以及pCAMBIA1301质粒同时用BamHⅠ和XbaⅠ进行双酶切,回收线性化的表达载体和pre-amiR-scl6片段,利用T4连接酶过夜连接 (图2),并转化大肠杆菌DH5α。获得的阳性克隆提取质粒,用BamHⅠ和XbaⅠ进行双酶切验证。
2 结果与分析
2.1 DlSCL6基因amiRNA前体序列设计
利用WMD3设计DlSCL6基因的amiRNA,共得到57条候选的miRNA序列。选择最优的amiRNA(序列为:TATGGCATTAAACTCGAACGG),利用psRNAtarget在线软件进行评价,结果显示,Expectation(E)为1.0,Target Accessibility(UPE)为16.944,显示该amiRNA与靶基因结合位点结合的可信度较高。将amiRNA提交WMD3在线程序,设计出4条以RS300为模板的重叠PCR引物,根据RS300模板序列和引物序列,推导完整的前体序列,并在两端添加酶切位点,命名为pre-amiR-scl6。preamiR-scl6完整序列及其 miRNA和miRNA∗位置见图3。
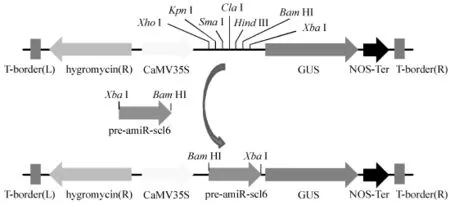
图2 amiRNA表达载体构建示意图

图3 pre-amiR-scl6序列
利用mfold软件分析合成的pre-amiR-scl6序列的二级结构,结果表明,除替换的 amiRNA及amiRNA∗序列外,其它都与拟南芥amiR159a前体结构类似 (图4),具有典型的 miRNA前体发卡环结构。

图4 pre-amiR-slc6折叠后的颈环结构
2.2 重叠PCR合成pre-amiR-scl6序列
根据pre-amiR-scl6序列,设计出的10条重叠PCR引物 (表1),除P10外,均在60 nt以内。利用TAKARA高保真酶,进行重叠PCR扩增。首先摸索合适的退火温度,设置温度范围30~55℃,依次每隔5℃为1个设定温度,PCR结果检测如图5所示,在30℃时可得到特异性较好且大小符合预期的条带 (约0.4 kb)。回收目的条带,连接至pGEM T-easy载体,并测序。测序结果比对显示,该序列与设计的pre-amiR-scl6序列完全一致 (396 bp),表明成功获得了DlSCL6基因的amiRNA的前体pre-amiR-scl6的序列。

表1 pre-amiR-scl6合成用到的重叠引物
2.3 pre-amiR-scl6植物表达载体构建与检测
为了研究amiRNA的功能,进行了pre-amiR-scl6表达载体的构建。将经BamHⅠ (GGATCC )和XbaⅠ (TCTAGA )双酶切后回收的pCAMBIA1301 和 pre-amiR-scl6片段过夜连接,转化大肠杆菌DH5α,在含有潮霉素的LB培养基平板上进行筛选。获得的阳性克隆提取质粒,经BamHⅠ和XbaⅠ双酶切检测,5个克隆中的2个在预期位置出现条带(改造的pCMABIA1301载体约9 kb,前体序列preamiR-scl6约0.4 kb)(图6),表明植物表达载体构建成功。

图5 pre-amiR-scl6 PCR合成电泳检测图
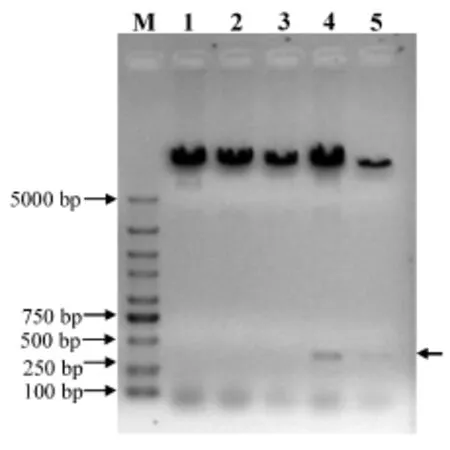
图6 植物表达载体酶切检测图
3 讨论
miRNA在植物生长发育中的调控作用越来越受到研究者的关注,但植物中并非所有基因都受到miRNA的调控。对于非miRNA调控的基因,可以参考miRNA的作用机理设计开发amiRNA,实现对其精准控制。相对于其它基因沉默技术,amiRNA具有较高的准确性和较低的脱靶率,因此在基因沉默研究中,amiRNA的应用越来越广泛。自首例amiRNA在拟南芥中应用[9]发表以来,越来越多的amiRNA研究工作得到了开展[10-11]。尤其是WMD3网站的开放为设计开发amiRNA提供了便利,WMD3可以根据靶基因序列,设计出所有符合要求的amiRNA,并根据序列的优劣进行排序。研究人员可以根据建议或个人需求选择合适的amiRNA,并选择提供的4种载体中的一种为模板设计重叠PCR引物,用于合成相应的amiRNA前体。然而,利用网站推荐的方法,操作过程比较繁琐,且耗时较长。
为了提高工作效率,同时降低成本,本研究对该方法进行了改进。首先根据WMD3设计的重叠引物和模板序列,推导出amiRNA的前体序列,将并其分解成末端具有10碱基重叠的10条短片度引物,奇数引物为正向序列,偶数引物为反向互补序列。为了降低引物的合成成本,单条引物长度尽量控制在60 nt以内。在进行重叠PCR时,2—8号引物只作为模板,加入量要少,而1和10号引物既是模板,又是扩增引物,适当加大加入量。通过对重叠PCR扩增时退火温度的研究,发现较低温度比较容易扩增出目的条带。利用改进的重叠PCR法,只需进行一次PCR反应和一次回收和测序,便可以获得设计的amiRNA前体序列,大大简化了原有的操作程序,省去了寻找或购买模板载体的麻烦,大大缩短了amiRNA前体合成的时间周期。
SCL6是植物特有转录因子家族GRAS家族的一员,在维持植物顶端分生组织正常功能中发挥重要作用。对拟南芥研究表明,SCL6-Ⅱ,SCL6-Ⅲ和SCL6-Ⅳ的突变导致侧芽数量较少,初生根变短等症状[12-13]。本研究构建了麻竹DlSCL6基因amiRNA的植物表达载体,为进一步验证amiRNA活性和研究麻竹中SCL6的表达调控奠定了基础。
[1] Chen X.microRNA biogenesis and function in plants[J].FEBS Letters,2005,579(26):5923-5931.
[2] Carra A,Mica E,Gambino G,et al.Cloning and characterization of small non-coding RNAs from grape[J].Plant Journal,2009,59(5):750-763.
[3] Seifert H J,Thiel G,Schmitt R.Transcriptional regulation of Arabidopsis MIR168a and argonaute1 homeostasis in abscisic acid and abiotic stress responses.[J].Plant Physiology,2012,158(3):1279-1292.
[4] Wonkeun P,Jixian Z,Jung-Youn L.Highly efficient gene silencing using perfect complementary artificial miRNA targeting AP1 or heteromeric artificial miRNA targeting AP1 and CAL genes[J].Plant Cell Reports,2009,28(3):469-480.
[5] Rui S,Yang C,Lu S,et al.Specific down-regulation of PAL genes by artificial microRNAs in Populus trichocarpa [J].Planta,2010,232(6):1281-1288.
[6] Butardo V M,Fitzgerald M A,Bird A R,et al.Impact of down-regulation of starch branching enzyme IIb in rice by artificial microRNA-and hairpin RNA-mediated RNA silencing.[J].Journal of Experimental Botany,2011,62 (14):267-275.
[7] Tiwari M,Sharma D,Trivedi P K.Artificial microRNA mediated gene silencing in plants:progress and perspectives[J].Plant Molecular Biology,2014,86(1/2):1-18.
[8] 杨丽,陈东亮,彭镇华,等.麻竹转录因子DlSCL6的基因克隆及在拟南芥中异位表达[J].林业科学,2014,50(11):52-57.
[9] Schwab R,Ossowski S,Riester M,et al.Highly specific gene silencing by artificial microRNAs in Arabidopsis[J].Plant Cell,2006,18(5):1121-1233.
[10] Ossowski S,Schwab R,Weigel D.Gene silencing in plants using artificial microRNAs and other small RNAs [J].Plant J,2008,53(4):674-690.
[11] Warthmann N,Chen H,Ossowski S,et al.Highly specific gene silencing by artificial miRNAs in rice[J].PLoS ONE,2008,3(3):e1829.
[12] Lee M H,Kim B,Song S K,et al.Large-scale analysis of the GRAS gene family in Arabidopsis thaliana[J].Plant Mol Biol,2008,67(6):659-670.
[13] Wang L,Mai Y X,Zhang Y C,et al.MicroRNA171ctargeted SCL6-Ⅱ,SCL6-Ⅲ,and SCL6-Ⅳ genes regulate shoot branching in Arabidopsis[J].Mol Plant,2010,3(5):794-806.
Synthesis and Expression Vector Construction of amiRNA Precursor for DlSCL6 in Dendrocalamus latiflorus
Chen Dongliang1,2Sun Huayu2Li Lichao2Zhao Hansheng2Gao Zhimin2
(1 Beijing Key Laboratory of Agricultural Genetic Resources and Biotechnology,Beijing Agro-Biotechnology Research Center,Beijing Academy of Agriculture and Forestry Sciences,Beijing 100097,China 2 International Center for Bamboo and Rattan,Key Laboratory on the Science and Technology of Bamboo and Rattan,Beijing 100102,China)
Web MicroRNA Designer(WMD3)is a professional website specially used for sequence design of plant artificial miRNA and its precursor.Overlap PCR method was used for the synthesis of amiRNA precursor sequence,which involved multiple PCR purifications and recovery processes with complicated steps and a longer period.In this study,sequences of plant artificial miRNA and its precursor for DlSCL6 in Dendrocalamus latiflorus were designed according to WMD3 and an improved method of overlapping PCR was used for quickly cloning of the precursor sequence.Based on the frame of pCMABIA1301 vector,the expression vector of amiRNA precursor for DlSCL6 was constructed,which laid a foundation for further study in the expression regulation of SCL6 using amiRNA.
Dendrocalamus latiflorus,amiRNA,overlap PCR,construction of expression vector
10.13640/j.cnki.wbr.2016.02.001
国家林业公益性行业科研专项经费项目 (编号:201504106);“十二五”国家科技支撑计划专题 (编号:2012BAD23B0504)。
陈东亮,男,博士,助理研究员,研究方向为花卉分子育种。E-mail:dlchen1984@126.com。
高志民,男,研究员,博士生导师,研究方向为竹藤生长发育的分子基础。E-mail:gaozhimin@icbr.ac.cn。

