CRISPR/Cas9技术的应用性研究
舒磊磊,甲芝莲,吴勇浒,索 伦,贾丽玲*
(1.上海交通大学生命科学技术学院,上海 200240;2.上海交通大学系统生物医学研究院,上海 200240;3.上海交通大学医学院附属第九人民医院,上海 200011)
CRISPR/Cas9技术的应用性研究
舒磊磊1,甲芝莲2,吴勇浒2,索伦3*,贾丽玲2*
(1.上海交通大学生命科学技术学院,上海 200240;2.上海交通大学系统生物医学研究院,上海 200240;3.上海交通大学医学院附属第九人民医院,上海 200011)
摘要:旨在探索CRISPR/Cas9技术的新应用,本研究通过在小鼠遗传操作(Genetic manipulation)中使用该技术,创建了一种在活体组织内研究基因功能的方法。通过胚胎电转、免疫组化和PCR分析等手段,结果发现,针对DCX基因进行编辑,再现了DCX下调导致神经元迁移障碍的表型,说明该技术能够用于小鼠遗传操作中下调基因表达;其次,该技术还成功应用于Pcdhα基因簇的编辑并研究了基因簇恒定区的功能;最后,通过基因型鉴定发现,胚胎电转中使用CRISPR/Cas9会导致目的基因的敲除、反转和重复事件的发生。综上所述,在小鼠胚胎电转中使用CRISPR/Cas9技术,能够成功编辑目的基因,从而实现活体组织内研究基因的在体功能。
关键词:CRISPR/Cas9;遗传操作;基因敲除;基因反转;基因重复
CRISPR/Cas9是一种RNA指导的DNA内切酶,依靠RNA-DNA的互补识别并切割特异基因序列从而达到精确的基因组编辑和基因修饰的目的。该技术自面世以来已经改变了生物领域的科研面貌,并迅速发展为实验室的一种常规试验手段。其已成功应用于细菌、人类细胞、斑马鱼、小鼠、猪、食蟹猴以及多种植物的基因组精确修饰,是最具有临床与应用前景的基因治疗技术之一[1]。该技术之所以被评为具有革命性的原因,在于其仅仅依靠一种蛋白(Cas9),通过一段特殊设计的与目的基因互补的寡聚核苷酸和一段常见的RNA组合,就能实现对目的基因组的精准定位和精确修饰。在哺乳动物中,以猪为例,约有3万个基因。然而,有些基因的功能却只有在其发育到特定时期在特定的组织中才会发挥其相应的功能。因此,倘若能够创建一种针对动物特定组织的、快速而有效的基因编辑技术,那么将对研究基因在特定组织中的功能提供非常有效的平台。为此,本研究选择小鼠为模型,以两种脑组织中高度表达且功能明确的基因DCX(Doublecortin)和Pcdhα为例,对CRISPR/Cas9遗传操作技术在小鼠脑组织中基因编辑的有效性进行研究。
DCX是高度表达于哺乳动物大脑皮质并在脑发育过程中起很重要作用的蛋白分子[2-4],对神经元的发育具有重要作用。而Pcdhα基因簇属于原钙粘蛋白(Protocadherin,Pcdh)家族,是钙粘蛋白(Cadherin)超家族里最大的亚家族,它们在神经元发生以及神经发育的过程中具有非常重要的作用,比如神经元生存[5-7]、树突发育[8-9]、突触形成[10-12]、自我回避[13]和轴突投射[14-15]等。
J.Li等发现,使用CRISPR和一对sgRNA转染体外培养的HEK293T细胞,非同源末端修复的结果主要有3种:敲除、反转和重复[16]。为鉴定CRISPR/Cas9在活体组织中是否同样可以达到基因编辑的目的,本研究首先分别针对DCX和Pcdha基因设计成对的sgRNA,并采用胚胎电转技术将其与Cas9表达质粒一同共转于小鼠大脑中。研究CRISPR/Cas9遗传操作技术对DCX和Pcdha基因编辑后对基因表达及神经元迁移的影响。并与PcdhaRNAi后的研究结果进行对比。进一步对目的基因PCR后测序,分析该技术是否能对小鼠活体组织中目的基因进行精确修饰,并产生精确的敲除、反转和重复事件。
1材料与方法
1.1材料
1.1.1试验材料、感受态细胞和表达载体试验中所用小鼠均为C57BL/6品系,饲养于SPF级别动物房中,动物房恒温恒湿,昼夜节律12 h/12 h;感受态细胞为DH5α菌株,为本实验室制备;sgRNA质粒载体为pGL3,由南京大学模式动物研究所赠送,pGEM-T Easy载体购自Promega公司。
1.1.2试剂与试剂盒PCR所用2× PCR mix购于Biotake公司,高保真酶KOD-plus购于Toyobo公司,T4 DNA连接酶、Bsa1内切酶购于NEB公司,染色剂fast green和DAPI(4′,6-二脒基-2-苯基吲哚)购于Sigma公司,封片剂购于DAKO公司,酵母提取物和胰蛋白胨购于OXOID公司,DNA Marker购于Invitrogen公司,多聚甲醛(PFA)购于Thermo公司,NaOH、EDTA、Tris-HCl、Na2HPO4、KH2PO4和Agar购于Sigma公司,NaCl、KCl、氨苄、IPTG、 X-Gel和戊巴比妥钠购于国药集团;质粒小量提取试剂盒、PCR 纯化试剂盒和DNA 胶回收试剂盒购于Axygen公司,质粒中量提取试剂盒和质粒大量提取试剂盒均购于Qiagen公司。
1.1.3仪器Nanodrop2000分光光度计购于Thermo公司,电转仪购于Bio-Rad公司,PCR仪购于Bio-Rad和Applied Biosystems公司,低温离心机购于Eppendorf公司,振动切片机购于Leica公司,荧光显微镜和体视显微镜购于Zeiss公司,激光扫描共聚焦显微镜购于Nikon公司。
1.1.4产品及服务试验中使用的各种引物(包括基因型鉴定引物、构建sgRNA质粒寡核苷酸等)均由上海生工生物工程股份有限公司合成;试验中所有需测序样本均送至上海桑尼生物科技有限公司或者上海生工生物工程股份有限公司测序。
1.2方法
1.2.1表型观察试验小鼠胚胎电转时间为胚胎期14.5 d,取脑时间为胚胎期19.5 d(E14.5~E19.5),在体视显微镜下观察取脑,使用荧光显微镜观察小鼠脑部荧光,使用激光扫描共聚焦显微镜观察脑片并拍照。
1.2.2遗传操作本研究以胚胎电转探索神经发育相关基因的功能为手段,故胚胎电转的部位位于小鼠脑部。质粒注射部位位于小鼠大脑脑室,电转使用38 V电压,电击5次,每次5 ms,两次电击间隔50 ms;每次注射的质粒大约为1 μL,各成分浓度:sgRNA 0.8 μg·μL-1,Cas9 1 μg·μL-1,fastgreen 0.000 3%。因为质粒带有负电荷,因此在外加电场的作用下会向正极移动,当把正极电仪放在小鼠大脑皮层外侧,负极电仪放在对侧,电转时质粒会向正极电仪所在的大脑皮层移动。
1.2.3脑片获取小鼠大脑固定时间大约为12 h,之后使用1× PBS浸泡清洗约12 h。清洗完后用震荡切片机切片,每片厚度50 μm,将需要观察的切片收集于24孔板中,孔中有预先放好的1×PBS,将要观察的脑片进行DAPI核染色,并置于水平脱色摇床常温孵育30 min,孵育过程避光。孵育完后,使用1×PBS清洗3次,每次5 min,清洗完将脑片转移到载玻片上并封片。此脑片即可使用激光扫描共聚焦显微镜观察并拍照。
1.2.4PCR分析对于需要进行基因型鉴定的小鼠大脑,需在荧光显微镜下观察荧光,并用手术刀取出荧光部分,放入PCR管中裂解。裂解时使用裂解液,其成分:NaOH 25 mmol·L-1,EDTA 0.2 mmol·L-1,体积为50 μL,裂解时以92 ℃裂解20 min,之后加入50 μL中和液中和并混匀,中和液成分:Tris-HCl 40 mmol·L-1,裂解液和中和液的溶剂均为去离子水。裂解之后进行PCR,PCR反应的组分和用量:5 μL 2 × PCR mix,0.5 μL Primer1(5 mmol·L-1),0.5 μL Primer2(5 mmol·L-1),0.5~1 μL模板,并用去离子补至10 μL。PCR反应条件:94 ℃ 3 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s/500 bp,72 ℃ 1 min, 36个循环。
将符合预期的PCR产物条带进行胶回收,胶回收产物经过T-A克隆插入pGEM-T Easy载体,之后转化感受态细胞,涂板于蓝白斑筛选平板。最后挑出白色菌落进行质粒小量提取,送公司测序。
1.2.5基因敲除表达质粒的构建胚胎电转中所用的sgRNA基因敲除质粒均为独立构建。试验所用质粒载体为pGL3,自带U6启动子,使用Bsa1酶切,该酶在U6启动子后面具有2个酶切位点且形成不同粘性末端。购买的用于构建sgRNA质粒的上游(F)和下游(R)引物序列首先均配成20 mmol·L-1的工作液,之后配置退火溶液,溶液各成分体积:F:5 μL,R:5 μL,NEBuffer 5 μL,并用去离子水补至50 μL。将上述溶液置于沸水中自然冷却至室温进行退火,退火后形成双链核苷酸两端有黏性末端序列,用T4连接酶与已酶切的pGL3载体相连接,测序,确认序列正确性。
1.2.6统计和分析试验中用ImageJ对小鼠脑片中荧光细胞进行计数,数据来自至少3组独立重复试验,然后取其平均数。计数采用双盲法,被计数的脑片也隐去其名称等相关信息。计数之后用t检验对两组不同数据进行比较,并以95%为置信区间。对于被计数的脑片中的细胞,我们采用梯形分割分区计数的方法,将小鼠大脑皮层从内到外分为10层,依次标记为10、9、8、7、6、5、4、3、2、1。最外层(第1层)区间置于大脑皮层的最外层,最内层(第10层)与切片内圈切线重合,然后记出每个区间的荧光细胞数量。
1.2.7构建sgRNA质粒的寡聚核苷酸序列和PCR分析的引物序列 试验中所用与目的基因互补的sgRNA的核苷酸序列见表1。基因型鉴定引物序列如表2所示。

表1 构建sgRNA的核苷酸序列
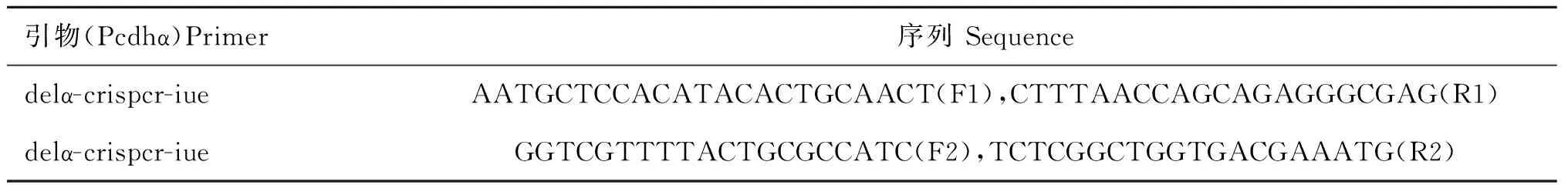
表2 PCR分析的引物序列
预期目的片段长度:敲除:F1+R1,280 bp;反转(上):F1+F2,365 bp;反转(下):R1+R2,415 bp;重复:F2+R2,506 bp
The predicted fragment sizes:deletion:F1+R1,280 bp;inversion(sense):F1+F2,365 bp;inversion(antisense):R1+R2,415 bp;duplication:F2+R2,506 bp
2结果
2.1CRISPR/Cas9转染后可重现目的基因RNAi的表型
本研究选取了功能明确的DCX基因来验证CRISPR/Cas9用于遗传操作的可行性。首先,试验组共转融合了eGFP蛋白的Cas9质粒以及作用于DCX编码区的两对sgRNA:DCX1和DCX2,对照组则转染了该Cas9基因以及sgRNA空载体,试验结果如图1a所示,对照组中被GFP标记的神经元大多数都迁移至外层,而试验组的神经元只有部分迁移到应到的位置。为了更加精确地了解神经元迁移状况,我们把脑片分成10层并进行统计。从统计结果(图1b)发现,转染DCX1 sgRNA的脑片在第2、4、7、9层与对照组相比有显著差异,转染了DCX2 sgRNA的脑片在第2、3、7、8层与对照组相比有显著差异。此结果与shRNA下调DCX基因表达后的结果一致。因此,推测在胚胎电转中使用CRISPR/Cas9能够使目的基因的表达量下降。
其次,使用该方法研究了Pcdhα基因簇恒定区的功能。在试验中使用胚胎电转的技术,针对Pcdhα基因簇的恒定区设计3个sgRNA:αcon1、αcon2和αcon3,用CRISPR/Cas9的手段,分别转染2个或3个sgRNA,使之能高效并特异地敲除Pcdhα基因簇恒定区,对照组则转染了该Cas9基因以及sgRNA空载体。试验结果如图2a所示,在敲除Pcdhα基因簇的恒定区之后,神经元的迁移会发生明显障碍,大部分神经元不能迁移至正常区域。统计结果(图2b)表明,与对照组相比,转染了特异性识别Pcdhα基因簇恒定区sgRNA的脑片神经元迁移明显滞后。这说明Pcdhα基因簇恒定区对神经元的正常迁移至关重要。

a.图中绿色是被GFP标记的神经元细胞;标尺:100 μm;b.横坐标:纵切大脑皮层由外到内分成10层,每个数字表示不同的层数;纵坐标:该层GFP阳性细胞占总GFP阳性细胞的比例;*.P<0.05,**.P<0.01。图2同a.The green in the figure are neuron cells labeled by GFP;Bar:100 μm;b.Abscissa:Longitudinal cerebral cortex is divided into 10 layers from outside to inside,each number means one different layer;Ordinate:The layer of GFP positive cells accounted for the proportion of total GFP positive cells;*.P<0.05,**.P<0.01.The same as Figure 2图1 下调DCX导致神经元迁移障碍Fig.1 Down-regulated DCX lead to neuron migration defects

图2 破坏Pcdhα基因簇恒定区导致神经元迁移障碍Fig.2 Deleting Pcdhα cluster constant exons lead to neuron migration defects
2.2CRISPR/Cas9可导致目的基因片段的敲除、反转和重复为了探索CRISPR/Cas9在局部组织遗传操作中的有效性,本研究在胚胎电转中共转一对sgRNA并分别位于Pcdhα基因簇的两端,然后获取有荧光细胞的部分脑部组织裂解之后进行PCR鉴定。鉴定结果表明,胚胎电转中应用CRISPR/Cas9可实现对目的基因的精确编辑并导致目的基因发生精确的敲除、反转和重复。发生敲除、反转和重复的示意图如图3所示。

图3 发生敲除、反转和重复的示意图Fig.3 Schematic diagram of deletion,inversion and duplication
PCR电泳结果显示(图4a,c,e左)条带大小与预期相符,将DNA进行回收,之后经过T-A克隆,连到pGEM-T Easy载体中进行测序。测序结果的接头处如图所示(图4a,c,e右),测序峰图(图4b,d,f))显示的是获得不同结果的接头处序列。
由图4可见,在遗传操作中使用CRISPR/Cas9会导致目的基因的敲除、反转和重复,之后我们对基因敲除之后的修复结果进行统计,发现其精确修复的比例高达70.8%(表3)。

表3 Pcdhα基因簇敲除、反转和重复3种事件精确修复的比例
3讨论



a,c,e左图表示基因型鉴定的电泳图;a,c,e右图表示测序结果的接头处序列;b,d,f.测序结果中存在的所有不同的情况的测序峰图;expected表示接头处预期精确修复的序列,precise表示接头处修复精确的条带a,c,e left.Electrophoresis identification of genotype;a,c,e right.Joint sequence of sequencing results;b,d,f.All different conditions in the sequencing results;expected.Precisely repaired of the joint;precise.DNA that is precisely repaired图4 胚胎电转中共转1对sgRNA敲除Pcdhα基因簇导致敲除、反转和重复事件及其基因型鉴定Fig.4 Contransfection of a pair of sgRNAs to delete Pcdhα cluster lead to deletion,inversion and duplication and the genotyping
CRISPR/Cas9是非常可靠而且简便的研究基因功能的工具,自其研发以来已经迅速成为一种强大且实用的方法。本研究中,在小鼠胚胎电转时使用CRISPR/Cas9研究在体基因的功能,并且得到了较好的结果。我们首先通过已知功能的基因DCX验证了该方法的可行性。研究表明,DCX在大脑皮质的发育中起很重要的作用[2,3,17],其缺陷会抑制大脑皮质中神经元迁移,也会抑制神经元从多极神经元向双极神经元的转变[4,18]。我们推测认为,胚胎电转时应用CRISPR/Cas9可以将DCX破坏,并产生相应表型。于是我们设计试验转染不同的sgRNA来破坏目的基因。结果表明,DCX被破坏之后的试验组神经元迁移明显障碍,证明了我们猜想的正确性。之后我们使用该方法研究Pcdhα基因恒定区的功能。Pcdhα基因簇对神经发育具有重要作用,在培养的海马神经元中的试验表明,敲除其Pcdhα基因簇或者过表达Pcdhα胞内段从而进行显性抑制均导致树突简化,且树突棘的宽度和长度降低,并且敲除Pcdhα基因簇会导致树突分枝和树突棘减少[8,19]等。以往在研究Pcdhα基因簇时,研究人员会将其全部敲除,从而使该基因完全不表达[8,19]。我们在试验中仅仅敲除了Pcdhα基因簇的恒定区,而Pcdhα基因簇的恒定区编码蛋白质的胞内段。试验结果表明,在敲除Pcdhα基因簇的恒定区之后,神经元的迁移会发生明显障碍,这说明Pcdhα基因簇的恒定区,即编码蛋白质的胞内段对神经元的正常迁移至关重要。J.Li等[16]提出,在体外培养的HEK293T细胞中使用CRISPR/Cas9敲除目的基因,非同源末端修复的结果有3种:敲除、反转和重复。与细胞系不同,神经元细胞不再分裂增殖,这样存在两种可能:一是可能因质粒在细胞中的维持时间更长而导致Cas9更彻底地切割,二是由于染色体构象与细胞系不同而导致Cas9工作效率的不同。我们想进一步探索在体外培养的细胞中发生的敲除、反转和重复事件在活体组织中会不会同样发生,于是设计试验在胚胎电转中使用CRISPR/Cas9共转一对sgRNA将小鼠整个Pcdhα基因簇敲除,并进行基因型鉴定,发现该技术可导致目的基因的敲除、反转和重复事件的发生,并且统计结果表明,精确修复的比例超过70%。然而,该手段与使用shRNA造成的基因表达下调的表型相比仍然具有一定的差距,因此如何提高该手段的效率仍然需要进一步的研究。同时,因为修复结果中存在目的基因的敲除、反转和重复等3种事件,因而无法确定某种事件在所有事件中发生的比例,也即不能确定出现的表型中哪种事件占据主导地位。
4结论
本研究证明了在遗传操作中使用CRISPR/Cas9能够产生目的基因的敲除、反转和重复事件,从而可以在活体组织内研究在体基因的功能。
参考文献(References):
[1]刘志国.CRISPR/Cas9系统介导基因组编辑的研究进展[J].畜牧兽医学报,2014,45(10):1567-1583.
LIU Z G.Research progress on CRISPR/Cas9 mediated genome editing[J].ActaVeterinariaetZootechnicaSinica,2014,45(10):1567-1583.(in Chinese)
[2]SAPIR T,ELBAUM M,REINER O.Reduction of microtubule catastrophe events by LIS1,platelet-activating factor acetylhydrolase subunit[J].EMBOJ,1997,16(23):6977-6984.
[3]GLEESON J G,LIN P T,FLANAGAN L A,et al.Doublecortin is a microtubule-associated protein and is expressed widely by migrating neurons[J].Neuron,1999,23(2):257-271.
[4]LOTURCO J.Doublecortin and a tale of two serines[J].Neuron,2004,41(2):175-177.
[5]CHEN W V,ALVAREZ F J,LEFEBVRE J L,et al.Functional significance of isoform diversification in the protocadherin gamma gene cluster[J].Neuron,2012,75(3):402-409.
[6]WANG X,WEINER J A,LEVI S,et al.Gamma protocadherins are required for survival of spinal interneurons[J].Neuron,2002,36(5):843-854.
[7]PRASAD T,WANG X,GRAY P A,et al.A differential developmental pattern of spinal interneuron apoptosis during synaptogenesis:insights from genetic analyses of the protocadherin-gamma gene cluster[J].Development,2008,135(24):4153-4164.
[8]SUO L,LU H,YING G,et al.Protocadherin clusters and cell adhesion kinase regulate dendrite complexity through Rho GTPase[J].JMolCellBiol,2012,4(6):362-376.
[9]LEDDEROSE J,DIETER S,SCHWARZ M K.Maturation of postnatally generated olfactory bulb granule cells depends on functional gamma-protocadherin expression[J].SciRep,2013,3:1514.
[10]WEINER J A,WANG X,TAPIA J C,et al.Gamma protocadherins are required for synaptic development in the spinal cord[J].ProcNatlAcadSciUSA,2005,102(1):8-14.
[11]GARRETT A M,WEINER J A.Control of CNS synapse development by {gamma}-protocadherin-mediated astrocyte-neuron contact[J].JNeurosci,2009,29(38):11723-11731.
[12]JUNGHANS D,HEIDENREICH M,HACK I,et al.Postsynaptic and differential localization to neuronal subtypes of protocadherin beta16 in the mammalian central nervous system[J].EurJNeurosci,2008,27(3):559-571.
[13]LEFEBVRE J L,KOSTADINOV D,CHEN W V,et al.Protocadherins mediate dendritic self-avoidance in the mammalian nervous system[J].Nature,2012,488(7412):517-521.
[14]KATORI S,HAMADA S,NOGUCHI Y,et al.Protocadherin-alpha family is required for serotonergic projections to appropriately innervate target brain areas[J].JNeurosci,2009,29(29):9137-9147.
[15]HASEGAWA S,HAMADA S,KUMODE Y,et al.The protocadherin-alpha family is involved in axonal coalescence of olfactory sensory neurons into glomeruli of the olfactory bulb in mouse[J].MolCellNeurosci,2008,38(1):66-79.
[16]LI J,SHOU J,GUO Y,et al.Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9[J].JMolCellBiol,2015,7(4):284-298.
[17]HORESH D,SAPIR T,FRANCIS F,et al.Doublecortin,a stabilizer of microtubules[J].HumMolGenet,1999,8(9):1599-1610.
[18]BAI J,RAMOS R L,ACKMAN J B,et al.RNAi reveals doublecortin is required for radial migration in rat neocortex[J].Nat Neurosci,2003,6(12):1277-1283.
[19]CHEN W V,MANIATIS T.Clustered protocadherins[J].Development,2013,140(16):3297-3302.
(编辑郭云雁)
doi:10.11843/j.issn.0366-6964.2016.07.003
收稿日期:2016-01-21
基金项目:国家自然科学基金(31200825)
作者简介:舒磊磊(1990-),男,安徽六安人,硕士生,主要从事分子和遗传生物学方面的研究,E-mail:leilshu@163.com *通信作者:索伦,副研究员,硕士生导师,主要从事基因在体功能研究,E-mail:suoyunfei@126.com;贾丽玲,实验师,E-mail:jllmao@126.com
中图分类号:S813.3
文献标志码:A
文章编号:0366-6964(2016)07-1316-08
The Application Research of CRISPR/Cas9
SHU Lei-lei1,JIA Zhi-lian2,WU Yong-hu2,SUO Lun3*,JIA Li-ling2*
(1.SchoolofLifeSciencesandBiotechnology,ShanghaiJiaoTongUniversity,Shanghai200240,China;2.InstituteofSystemsBiomedicine,ShanghaiJiaoTongUniversity,Shanghai200240,China;3.ShanghaiNinthPeople’sHospital,ShanghaiJiaoTongUniversitySchoolofMedicine,Shanghai200011,China)
Abstract:To investigate new application of CRISPR/Cas9 technology,we established a method to study genes function in living organism by the application of CRISPR/Cas9 in genetic manipulation.By using in vivo electroporation,immunohistochemistry and PCR analyzing,we first validated the function of DCX and the results indicated that CRISPR/Cas9 could be used in down-regulated gene expression in mouse genetic manipulation.Then this method was also used to study the function of constant exons of Pcdhα cluster.To further clarify the mechanism that led to the phenotype of neuron migration defects by DCX,we designed experiments to identify the genotype of target genes.The results indicated that CRISPR/Cas9 could lead to deletion,inversion and duplication of target genes.In conclusion,CRISPR/Cas9 technology could be used in mouse genetic manipulation and edit the target genes efficiently,which might be an ideal method to research gene function in living organism.
Key words:CRISPR/Cas9;genetic manipulation;deletion;inversion;duplication

