微生物合成2,3-丁二醇的代谢工程
童颖佳,邬文嘉,彭 辉,刘陆罡,黄 和,纪晓俊
微生物合成2,3-丁二醇的代谢工程
童颖佳,邬文嘉,彭辉,刘陆罡,黄和,纪晓俊
(南京工业大学生物与制药工程学院,材料化学工程国家重点实验室,江苏 南京 211816)
摘要:2,3-丁二醇 (2,3-BD) 是一种重要的微生物代谢产物,广泛应用于食品、医药、化工等多个领域。微生物合成2,3-BD的效率不高一直制约着其生物制造工业化进程,应用代谢工程的理论和方法优化微生物的代谢途径有望解决这一问题。本文全面总结了近年来微生物合成2,3-BD研究过程中的菌株改造和构建技术,包括过表达合成途径中的关键酶编码基因、敲除旁路代谢途径关键酶编码基因、应用辅因子工程手段对天然菌株代谢网络进行重新设计和合理改造,以及利用合成生物学技术在模式菌株中构建全新的代谢途径,实现2,3-BD的高效生物合成。最后,本文对未来的研究方向进行了展望,提出了进一步利用先进的合成生物学方法构建高效细胞工厂的指导性建议。
关键词:2,3-丁二醇;代谢;生化工程;辅因子调控;全局转录调控;合成生物学
引 言
随着石油资源的日益短缺和过度消耗,能源安全、环境污染成为全世界普遍关注的问题。通过生物学方法对微生物进行改造,实现大规模的物质加工与转化,能在发展经济的同时兼顾生态环境的保护,实现能源结构的绿色转型,是实现可持续发展战略、推动节能减排的一种有效的手段。2,3-丁二醇 (2,3-BD) 是一种重要的生物基化学品,广泛应用于食品、医药、化工等领域[1],可用于制备油墨、香水香薰、增湿剂、软化剂、炸药、燃料添加剂、增塑剂和药物手性载体等工业产品[2-3]。同时,通过特定的化学反应,2,3-BD可以衍生出多种重要的化学品,例如甲乙酮、1,3-丁二烯、乙偶姻、双乙酰等[4-5]。最早关于2,3-BD的研究报道可以追溯到20世纪初期。在1906年,Harden和Walpole首次报道了利用肺炎克雷伯氏菌 (Klebsiella pneumoniae) 发酵生产2,3-BD的研究[2]。由于第二次世界大战时对于1,3-丁二烯的大量需求,2,3-BD的研究得到了蓬勃的发展。但是,到了20世纪50年代,由于石油资源的大量开采和价格优势,以石油为原料生产1,3-丁二烯更加便宜,导致生物法生产2,3-BD逐渐被化学法所取代[6]。20世纪 70年代末,伴随着石油危机的爆发、价格上涨以及环境污染等问题,生物法合成2,3-BD又重新引起了社会各界的关注[7]。特别是21世纪以来,生物法生产2,3-BD的研究达到了新的高潮。然而,从自然界中筛选得到的生产2,3-BD的天然菌株有着生产强度和光学纯度低,对廉价的生物质可再生资源(包括淀粉,木质纤维素,糖蜜等)利用效率低等缺陷,且在发酵过程中生成了较多的副产物,导致2,3-BD的转化率较低,下游分离困难,限制了2,3-BD的工业化生产。随着代谢工程和合成生物学的兴起,对2,3-BD生产菌株和代谢途径改造的研究逐渐深入,改造手段和方法不断创新,2,3-BD的生产水平得到了质的飞跃。本文结合本课题组的前期研究工作,综述了利用代谢工程的原理和方法对天然微生物代谢网络进行重新设计和合理改造,以及利用合成生物学技术在模式菌株中构建2,3-BD合成途径的最新进展,并对未来的重点研究方向进行了展望。

表1 2,3-丁二醇天然生产菌株及研究水平Table 1 Biotechnological production of 2,3-butanediol using natural strains
1 生物合成 2,3-丁二醇的微生物及其代谢途径
在自然界中,很多微生物都能够以单糖为底物通过微生物代谢积累2,3-BD(表1)。在微生物典型的2,3-BD合成途径中,糖类物质在α-乙酰乳酸合成酶 (α-acetolactate synthase, ALS)、α-乙酰乳酸脱羧酶 (α-acetolactate decarboxylase, ALDC)、2,3-丁二醇脱氢酶(2,3-butanediol dehydrogenase,2,3-BDH;又称乙偶姻还原酶,acetoin reductase,AR)的作用下经丙酮酸、α-乙酰乳酸、乙偶姻并最终转化为2,3-BD。此过程伴随着副产物乙酸、乳酸、乙醇、丁二酸、甲酸等的生成[4](图1)。这种合成途径在克雷伯氏菌属 (Klebsiella)、类芽孢杆菌属(Paenibacillus)、肠杆菌属 (Enterobacter) 等菌属中广泛存在。几种典型微生物2,3-BD代谢途径关键酶编码基因的分布情况如图2所示。通常,编码这3种关键酶的基因位于同一操纵子 (budABC) 内,如土生克雷伯氏菌 (Klebsiella terrigena)、K. pneumoniae和产气肠杆菌 (Enterobacter aerogens),受到LysR型转录调控因子AlsR(由budR/alsR编码)的调控[4,17-18]。而产酸克雷伯氏菌 (Klebsiella oxytoca) 不仅拥有该操纵子,还存在着一种双乙酰/乙偶姻还原酶基因 dar。该基因远离操纵子budABC,属于短链脱氢酶/还原酶 (SDR) 家族,且与budC属于同一个同源组[17]。在其他产2,3-BD的典型菌株中,例如芽孢杆菌属 (Bacillus) 和沙雷氏菌属 (Serratia),编码ALS和ALDC的基因处于同一个操纵子alsSD中,同样受到AlsR的调控,可编码2,3-BDH的基因位于alsSD操纵子之外,表达水平并没有受到影响[19-20]。在枯草芽孢杆菌(Bacillus subtilis) 中,AlsR是通过连接到alsR基因与alsS基因之间的顺式作用区域来正向调节alsSD操纵子的转录的[19]。而在乳酸乳球菌 (Lactococcus lactis) 中,2,3-BD途径的相关基因并没有组成操纵子[17]。
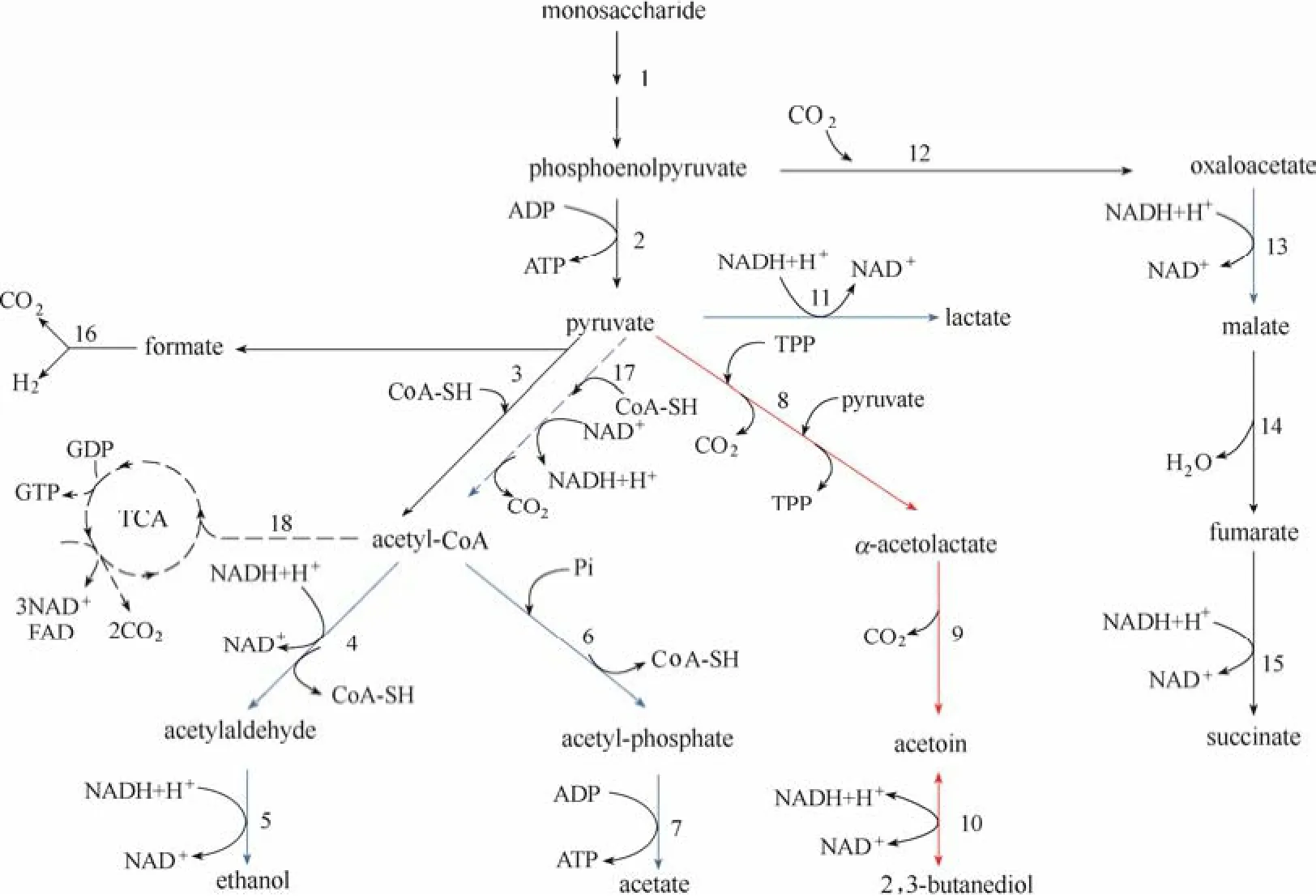
图1 混合酸-2,3-丁二醇代谢途径[2](虚线表示只在有氧条件下活跃的途径;红线表示在以往研究中被过表达的途径;蓝线表示在以往研究中被敲除的途径)Fig.1 Mixed acid-2,3-BD pathway[2](dashed lines represent the pathways that are active only under the aerobic conditions; red lines represent the pathways that once overexpressed; blue lines represent the pathways that once knocked out)
TCA—tricarboxylic acids cycle; 1—Embden-Meyerhof and pentose phosphate pathway enzymes; 2—pyruvate kinase; 3—pyruvate-formate lyase; 4—acetaldehyde dehydrogenase; 5—ethanol dehydrogenase; 6—phospho-transacetylase; 7—acetate kinase; 8—α-acetolactate synthase; 9—α-acetolactate decarboxylase; 10—2,3-butanediol dehydrogenase (acetoin reductase); 11—lactate dehydrogenase; 12—phosphoenolpyruvate decarboxylase; 13—malate dehydrogenase; 14—fumarase; 15—succinate dehydrogenase; 16—formate-hydrogen lyase complex; 17—pyruvate dehydrogenase multi-enzyme complex; 18—citroyl synthetase

图2 几种典型微生物2,3-丁二醇代谢途径关键酶编码基因分布情况[17]Fig.2 Genetic organization of genes involved in 2,3-BD production from representative natural producers[17]
TA—gene encoding the transcriptional activator; ALDC—gene encoding α-acetolactate decarboxylase; ALS—gene encoding α-acetolactate synthase; BDH—gene encoding 2,3-BD dehydrogenase/acetoin (diacetyl) reductase
目前,用来发酵生产2,3-BD的主要是细菌类,其中,具有较高潜力的生产菌株主要包括克雷伯氏菌属[8-9]、肠杆菌属[10-11]、芽孢杆菌属[12-14]、类芽孢杆菌属[15]以及沙雷氏菌属[16]等。目前,克雷伯氏菌属和类芽孢杆菌属被认为是最有可能进行工业化生产的菌种[2]。在真菌中,虽然一些酵母也具有生物合成2,3-BD的能力,但由于其生产的2,3-BD产量、纯度均较低,限制了其应用。但最近有很多研究通过代谢工程以及合成生物学技术,使得利用酵母生产2,3-BD变成了可能[21-22]。
2 微生物合成 2,3-丁二醇的代谢途径改造策略
通过传统诱变育种如物理诱变或者化学诱变等方式选育高产菌株一直是国内外提高菌种产量、性能的主要手段。但这种方式存在随机性和不确定性,需要经过大量的筛选才能获得想要的性状。利用基于全基因组的系统代谢工程和辅酶调控策略对关键代谢节点进行合理的遗传改造,通过多途径优化的方法阻断各种竞争支路来强化 2,3-BD生物合成代谢途径有望快速获得遗传性质稳定、发酵水平和产物光学纯度显著提高的细胞工厂。
2.1 利用代谢工程手段改造产 2,3-丁二醇的野生菌株
2.1.1 合成支路和底物利用途径的强化 从丙酮酸到2,3-BD的转化需要3种关键酶(ALS,ALDC,2,3-BDH)的联合作用,提高这 3种关键酶的表达能显著提高2,3-BD的产量,其中2,3-BDH由于其可逆的催化效果成为了研究者关注的重点。Guo 等[23]通过在 K. pneumoniae中过表达 ALS和2,3-BDH,构建了一株高产2,3-BD的重组菌,在以葡萄糖为碳源的补料发酵中,重组菌2,3-BD的产量与原始菌相比提高了 12%。Zhang[24]在粘质沙雷氏菌 (Serratia marcescens) H30中过表达了2,3-BDH的编码基因budC,使重组菌中2,3-BDH的活性提高了21倍。5 L罐发酵结果显示,过表达budC基因能在显著提高2,3-BD产量(增加了78.5%)的同时降低发酵液中乙偶姻以及副产物乙醇、乳酸、丁二酸的含量(分别下降了86.1%、45.3%、27.2%和51.9%)。Cho等[9]在一株代谢具有温度选择性的K. oxytoca M1中过表达了2,3-BDH的编码基因,使得K. oxytoca M1中乙偶姻的产量减少了43%,同时2,3-BD的产量也有了显著的提高,产量、产率以及生产强度分别达到了142.5 g·L-1,0.42 g·g-1和1.47 g·L-1·h-1,这是K. oxytoca中2,3-BD发酵生产的最高产量。Bai等[25]从S. marcescens MG1中鉴定了一种由slaC基因编码的meso-2,3-BDH,并发现失活slaC基因能导致(3R)-乙偶姻的大量积累,而在敲除slaC基因的S. marcescens MG1重组菌中过表达来自B. subtilis 168的2,3-BDH编码基因bdhA能促使(3R)-乙偶姻向(R, R)-2,3-BD的转化。在补料发酵时,重组菌在48 h内生成了89.81 g·L-1的(R, R)-2,3-BD,生产强度为 1.91 g·L-1·h-1。这些结果为高产 2,3-BD基因工程菌的构建提供了新的思路。
除了过表达2,3-BD的合成支路,通过代谢工程技术还能使工程菌具备利用廉价原料生产 2,3-BD的能力。Zheng等[26]通过在K. pneumoniae KG1中过表达内源的α-淀粉酶编码基因malS,使重组菌能直接利用淀粉而无须液化和糖化步骤。这是在克雷伯氏菌中通过过表达淀粉酶编码基因简化发酵过程的第一次尝试,但是在重组菌中淀粉酶的活性只有少量的提高,而2,3-BD极低的产量 (3.8 g·L-1) 以及转化率 (0.19 g·g-1) 则说明内源的淀粉酶编码基因并不能满足降解淀粉来高产 2,3-BD的需要。随后,Tsvetanova等[27]在K. pneumoniae G31-A中外源表达了来自于地衣芽孢杆菌 (Bacillus licheniformis) 的α-淀粉酶编码基因,通过对该α-淀粉酶编码基因的过表达,在K. pneumoniae中检测到了很高的淀粉酶活性。在以土豆淀粉为底物的同步糖化发酵实验中,获得2,3-BD 53.8 g·L-1。这是第一次在K. pneumoniae中成功表达外源淀粉酶编码基因,并第一次成功地以淀粉为底物发酵高产2,3-BD。Ji等[28]通过在K. oxytoca中过表达突变的环磷酸腺苷受体蛋白 CRP(in)的编码基因 crp(in),解除了葡萄糖对木糖的阻遏作用,使得突变株能够同时利用木糖和葡萄糖发酵生产2,3-BD,从而提高了菌株的生长速度以及2,3-BD的生产强度。Li等[11]在一株重组阴沟肠杆菌 (Enterobacter cloacae) SDM的基础上,通过失活葡萄糖转运蛋白编码基因 ptsG以及过表达半乳糖透性酶编码基因 galP的方法消除了碳代谢抑制,使其可以同时利用葡萄糖和木糖。接着,为了进一步提高(R, R)-2,3-BD的产量,又敲除了副产物合成编码基因ldh和frdA。在以葡萄糖和木糖为底物的补料发酵实验中,重组菌在 44 h内生产了152 g·L-1(R, R)-2,3-BD (纯度 > 97.7%),生产强度高达3.5 g·L-1·h-1,对葡萄糖和木糖的产率达理论产率的97.7%。这是到目前为止,以葡萄糖和木糖为底物微生物发酵生产 2,3-BD的最高产量和生产强度。由于碳代谢阻遏,甘蔗糖蜜中的混合糖(蔗糖,果糖)并不能像葡萄糖一样被有效利用。因此Jung等[10]敲除了E. aerogens中代谢阻遏/活性蛋白Cra的编码基因来提高果糖的利用效率,但是突变株中蔗糖的消耗速度却因此受到了影响。通过反转录和RT-PCR研究了cra缺失菌株中与蔗糖利用相关的6个关键基因(scrA,scrB,scrR,scrK,cra和crp)的转录水平后,发现scrA和scrB的转录水平分别下降了32%和24.9%。由于Cra是通过直接连接到scrAB操纵子的启动子区域来调节其表达的,而该启动子同时还受到ScrR(由scrR基因编码)的负调节作用,因此Jung等通过敲除scrR基因以及过表达scrAB操纵子的手段恢复了cra缺失菌株对蔗糖的利用。随后,他们还通过过表达ptsG基因提高了菌株对葡萄糖的利用效率。在补料发酵中,重组菌以甘蔗糖蜜为原料54 h生产了140 g·L-1的2,3-BD。该研究是利用代谢工程手段提高菌株对廉价碳源利用效率的一次成功的尝试。
到目前为止,以合成支路和底物利用途径的强化手段提高2,3-BD产量的研究成果汇总于表2。
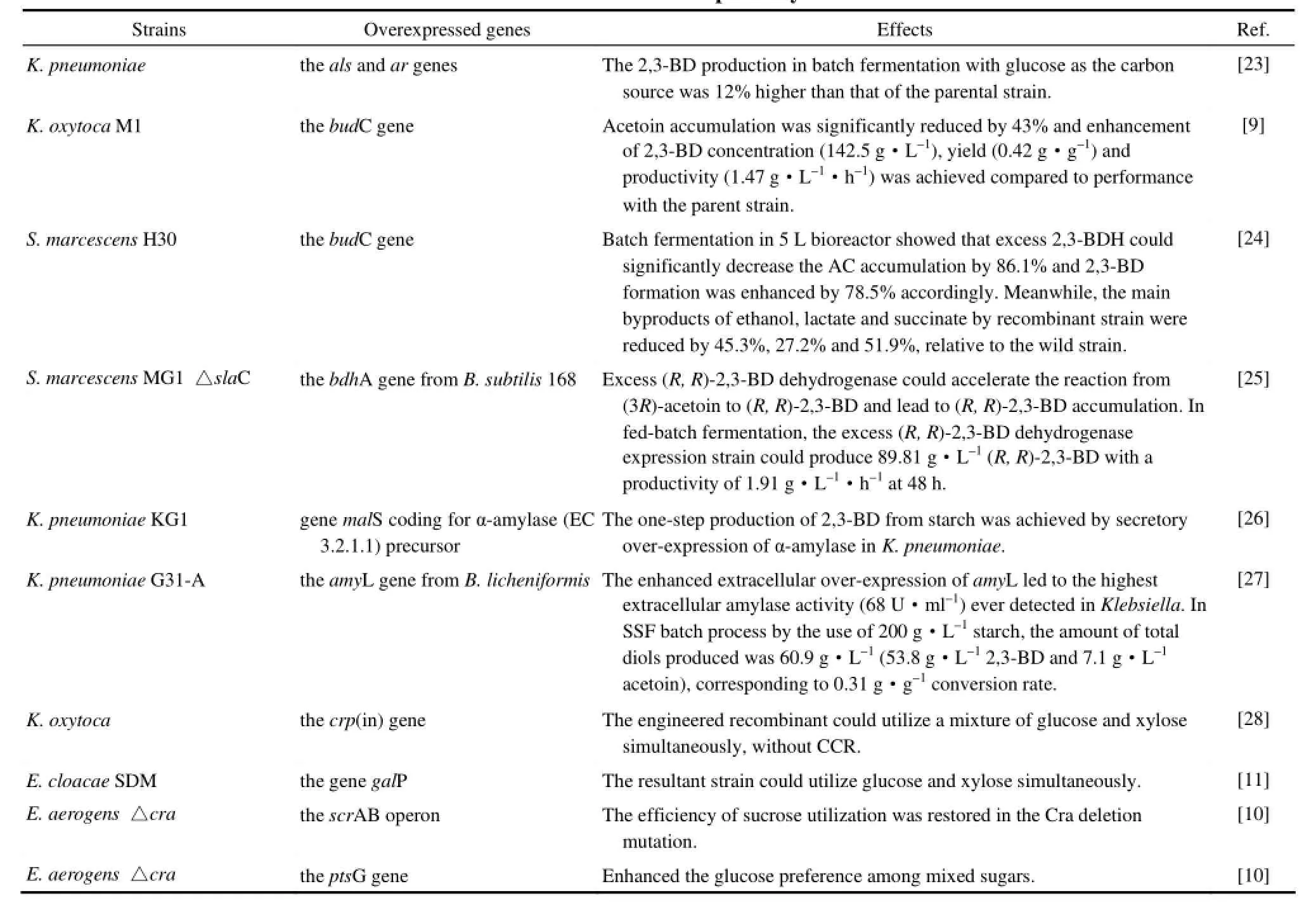
表3 2,3-丁二醇天然生产菌株合成支路和底物利用途径的强化及研究水平Table 2 Metabolic engineering for 2,3-butanediol overproduction by enhancements of 2,3-butanediol branches and substrate utilization pathways
2.1.2 旁路基因的敲除 随着代谢工程技术的兴起和不断进步,利用基因工程手段改造菌种以提高目标产物的产量并减少副产物的积累成为了最近研究的热点,混合酸发酵过程中副产物乙酸、乙醇、乳酸或丁二酸等相关基因的敲除成为了研究者普遍选择的改造策略。Jantama等[29]敲除了 K. oxytoca KMS005中的乙醇脱氢酶E编码基因adhE,乙酸激酶-磷酸转乙酰酶编码基因ackA-pta,以及乳酸脱氢酶编码基因ldhA,显著降低了副产物浓度。Ji等[30]通过同源重组的方法敲除了突变株 K. oxytoca ME-UD-3中的乙醛脱氢酶编码基因aldA,阻断了副产物乙醇的生成,并且使另一种副产物乙偶姻的产量也显著下降。利用突变株进行补料分批发酵,2,3-BD的产量可达130 g·L-1。Qi等[31]敲除了B. licheniformis WX-02的meso-2,3-BDH编码基因,从而阻断了meso-2,3-BD的合成,突变株能够专一积累30.76 g·L-1的(R,R)-2,3-BD。Rados等[32]在敲除了丙酮酸脱氢酶复合体E1亚基编码基因aceE,丙酮酸:醌氧化还原酶编码基因pqo,ldhA和苹果酸脱氢酶编码基因mdh的谷氨酸棒杆菌(Corynebacterium glutamicum) 中克隆并表达了来自于L. lactis的2,3-BD合成途径,在消除副产物的同时提高了2,3-BD的产量和生产强度,在微氧条件下2,3-BD的产量为6.3 g·L-1。Zhang等[16]发现利用S. marcescens H30发酵生产2,3-BD时,该菌会产生一种活性物质 (serrawettin W1),形成大量的泡沫,容易造成染菌且致使发酵过程中需要添加大量消泡剂。为了避免这一现象,他们敲除了编码serrawettin W1合成的关键酶基因swrW。重组菌发酵过程中产生的泡沫明显减少,并且2,3-BD的产量也得到了提高。最终,利用蔗糖批次流加发酵57 h后,2,3-BD的产量达到了152 g·L-1,这一结果是目前文献报道中2,3-BD的最高产量。
以往2,3-BD的生产都集中在糖类发酵方向,但生物柴油衍生的粗甘油也可以作为 2,3-BD发酵生产的一个廉价来源。可是在尝试以粗甘油作为原料发酵2,3-BD时,1,3-丙二醇的大量积累以及2,3-BD的低产量成为阻碍甘油发酵生产 2,3-BD的主要原因。为了解决这一问题,Cho等[33]敲除了K. oxytoca M1中编码甘油脱水酶大亚基的 pduC基因以及ldhA基因。发酵结果显示,以粗甘油为碳源时,重组菌生产了131.5 g·L-1的2,3-BD,且在生产过程中并没有检测到1,3-丙二醇。
目前高产 2,3-BD的野生菌株,如 K. pneumoniae、K. oxytoca、S. marcescens、E. cloacae等大多具有致病性,限制了它们的工业化应用。科学家们已经证实K. pneumoniae致病的毒力因子主要包括菌毛、荚膜多糖、脂多糖、受体、铁载体等[34]。通过代谢工程手段消除或弱化该菌的毒性已经引起了国内外研究者的关注。脂多糖(lipopolysaccharides,LPS),又称内毒素,是糖-蛋白质-磷脂复合物,属于革兰阴性菌的细胞壁组分。脂多糖可改变宿主免疫系统,导致致病和生理变化,并干扰其他抗原的免疫应答,还可激活凝血和补体系统,有助于细菌对宿主的感染[34]。Jung等[35]敲除了K. pneumoniae KCTC 2242、K. oxytoca KCTC 1686和K. oxytoca ATCC 43863中与脂多糖合成有关的葡糖基转移酶的编码基因 wabG,并发现基因wabG敲除对菌体生长并没有影响,但重组菌丧失了合成外核脂多糖以及荚膜的能力,显著地降低了菌体的毒性。Huynh等[36]敲除了 K. pneumoniae KCTC 2242中编码1类菌毛亚基蛋白的fimA基因和编码葡糖基转移酶的wabG基因。构建而成的重组菌不能合成外核脂多糖,也没有菌毛,但菌体的生长以及其生产 2,3-BD的能力并没有受到影响。Kim等[37]以一株wabG基因缺失的K. pneumoniae SGSB100为基础,通过敲除ldhA与过表达ALDC编码基因budA、ALS编码基因budB相结合的方法促进了微酸条件 (pH 5.5) 下葡萄糖与2,3-BD的有效转化,与原始菌相比,重组菌2,3-BD的产量提高了40%。从基因水平上消除或者弱化致病菌的毒性能极大地拓宽这些菌株的工业应用范围,成为了一种构建基因工程菌的新的思路。
到目前为止,以敲除旁路基因的手段提高2,3-BD产量的研究成果汇总于表3。

表3 基因敲除改造2,3-丁二醇天然生产菌株及研究水平Table 3 Metabolic engineering for 2,3-butanediol overproduction by knocking out genes
2.1.3 辅因子代谢与全局转录调控 以细胞为基础的生物转化过程常包含大量复杂的反应且需要特异性辅因子的参与[39-40],其中辅因子的供给不足又常常是影响生物转化的限制因素。因此,氧化还原辅因子代谢工程成为优化生物转化的重要代谢工程策略。Fu等[12]为了获得更多的NADH,在B. subtilis中表达了一种由udhA基因编码的转氢酶,同时通过低溶氧控制以及加入还原性物质等手段,最终重组菌发酵生产了49.29 g·L-1的(R, R)-2,3-BD。Yang 等[41]在B. subtilis中过表达了甲酸脱氢酶编码基因fdh并敲除了NADH氧化酶YodC的编码基因,构建而成的重组菌的2,3-BD浓度提高了19.9%,乙偶姻的浓度下降了71.9%。随后他们又利用插入失活的方法敲除了ldhA,提高了2,3-BD产量的同时降低了副产物乙偶姻和乳酸的浓度。Zhang等[42]在多粘类芽孢杆菌 (Paenibacillus polymyxa) ZJ-9中外源表达了一个来自博伊丁氏假丝酵母 (Candida boidinii) 的 NAD+依赖型甲酸脱氢酶 (FDH) 编码基因,显著提高了胞内NADH/NAD+的比例。在批次以及补料发酵中,与原始菌相比2,3-BD的产量分别提高了10.2%和8.0%。Yang等[14]在解淀粉芽孢杆菌 (Bacillus amyloliquefaciens) 中共表达了NAD+-依赖型甘油醛-3-磷酸脱氢酶 (GAPDH) 和NADH-依赖型2,3-BDH,节省了发酵时间且2,3-BD产量提高了22.7%,而乙偶姻、乳酸和丁二酸的产量分别下降了80.8%,33.3%和39.5%,原因是因为GAPDH的过表达增加了NADH的供应。在补料发酵实验中,45 h获得了2,3-BD 132.9 g·L-1,生产强度达到了 2.95 g·L-1·h-1。这是首次在 B. amyloliquefaciens中进行基因改造的报道。Geckil 等[43-44]和Jeong等[45]分别报道了含有来自透明颤菌(Vitreoscilla) 血红蛋白基因 (vgb) 的 E. aerogenes 和K. oxytoca在代谢流分配上的改变,发现vgb基因在低溶氧水平下的表达产物透明颤菌血红蛋白(VHb) 能够有效提高E. aerogenes和K. oxytoca的2,3-BD的产量。推测是由于VHb能够有效改善发酵过程的溶氧水平,从而影响葡萄糖的分解代谢,通过调节胞内NAD+/NADH和ADP/ATP的比率实现对葡萄糖代谢途径中碳代谢流的合理分配。
传统的育种方法主要通过对单一基因的消除或过度表达进行修饰,但由于生物细胞中代谢途径的高度复杂性,目的表型往往在整体水平上达不到较高的表达。全局转录调控 (global transcription machinery engineering, gTME)[46]是从整体水平改变细胞基因组转录,获得有益细胞表型的一种全新的定向进化方法,能对微生物细胞进行目标性能全局强化,是在基因和细胞水平上改造微生物细胞的新途径。近年来,来源于 LysR家族的转录调控因子AlsR对2,3-BD生产的影响引起了国内外学者的注意。AlsR由budR/alsR编码,与2,3-BD合成操纵子反向转录,其表达受其自身的副调控,且受一些外部刺激物如群体感应信号分子、乙酸和厌氧环境的诱导[47]。Zhang等[48]发现,使用强度适中的启动子PbudA来控制转录调节蛋白ALsR的表达能显著提高 2,3-BD前体物质乙偶姻的积累。有研究表明,AlsR是通过结合到B. subtilis alsS基因启动子前的特定位点来直接刺激alsSD操纵子的转录,但它对bdhA基因表达的影响却是未知的。Oliveira等[19]构建了一株 alsR::spc缺失的 B. subtilis,通过测量budA-lacZ转录融合的表达、budA基因mRNA的稳态水平以及2,3-BDH的酶活水平来研究AlsR对bdhA基因表达的影响。结果显示,突变菌中budA基因的表达出现在稳定期前期,表达水平虽然降低了但却并没有消失。通过电泳迁移率变化分析,纯化的AlsR蛋白并没有结合到budA基因的启动子区域,说明budA基因的表达是间接受到AlsR的调控的。Lee等[18]通过构建AlsR编码基因alsR过表达型 (SGSB101) 和 alsR缺陷型 (SGSB102) K. pneumoniae,从转录水平考察了alsR基因、2,3-BD生物合成相关基因 (budB, budA, budC) 和酸生物合成相关基因(ldhA,乙酸激酶编码基因 ack)在发酵过程中的变化。通过观察发现,在对数期,过表达 alsR基因的菌株其转录水平提高了 8倍,2,3-BD的产量提高了2倍。在敲除alsR基因的菌株中并没有检测到alsR转录,而且其酸合成相关基因的转录水平下降至1/70。
而辅因子与全局转录调控相结合的改造手段也成为了提高2,3-BD产量的一种行之有效的方法。Yang等[49]为提高B. amyloliquefaciens中2,3-BD的产量采用了3种策略,首先通过过表达甘油脱氢酶和2,3-BDH建立了一个NADH/NAD+再生系统,提高2,3-BD产量的同时减少了副产物的积累;其次选择了一个强度适中的启动子 PbudA来控制转录调节蛋白ALsR的表达,提高了流向2,3-BD支路的通量;最后通过一个3阶段的溶氧控制策略以及一个2阶段的 pH控制策略,最终生产出了 2,3-BD 102.3 g·L-1,产率为 0.44 g·g-1,生产强度为 1.16 g·L-1·h-1。
到目前为止,以辅因子代谢与全局转录调控手段提高2,3-BD产量的研究成果汇总于表4。

表4 2,3-丁二醇天然生产菌株的辅因子代谢改造及研究水平Table 4 Cofactor engineering for 2,3-butanediol overproduction
2.2 利用合成生物学方法构建2,3-丁二醇人工代谢途径
代谢工程改造微生物具有从简单易得以及廉价的原材料中生产大量化学物质的潜力[50]。但单纯的对原始菌株的改造已经不能满足这一需求,因此研究的重点逐渐转向了通过在基因工程菌中表达外源基因来构建宿主细胞原本并不存在的代谢途径,进而生物合成出所需要的目标产物。随着合成生物学的发展,人们已经可以方便地设计和利用非天然的微生物进行 2,3-BD的生产,且在不能天然合成2,3-BD的菌株体内构建人工途径,一般是以合成单一手性构型的2,3-BD为目标的。利用合成生物学手段构建的具有详细生物学特征的生物学元件来构建代谢途径具有更好的可预测性,能够提高代谢工程改造的效率,同时也可以为构建复杂的生物学系统提供技术支持。
2.2.1 以大肠杆菌为宿主 随着对 2,3-BD生物合成研究的不断深入以及代谢工程与合成生物学的蓬勃发展,在对2,3-BD代谢途径逐渐清晰的情况下,为了适应不同的发酵生产条件,利用合成生物学方法在模式菌株中构建新的2,3-BD合成途径,对代谢途径进行重新设计和合理改造成为最近研究的热点。大肠杆菌 (Escherichia coli) 由于具有生长速度快、代谢途径和代谢机制较清楚及遗传操作简单等优点,已成为最常用的代谢工程改造的宿主菌[1,51],早在20世纪90年代,便有利用重组E. coli生产2,3-BD的报道[52]。到目前为止,以E. coli为宿主合成不同构型2,3-BD的研究成果汇总于表5。
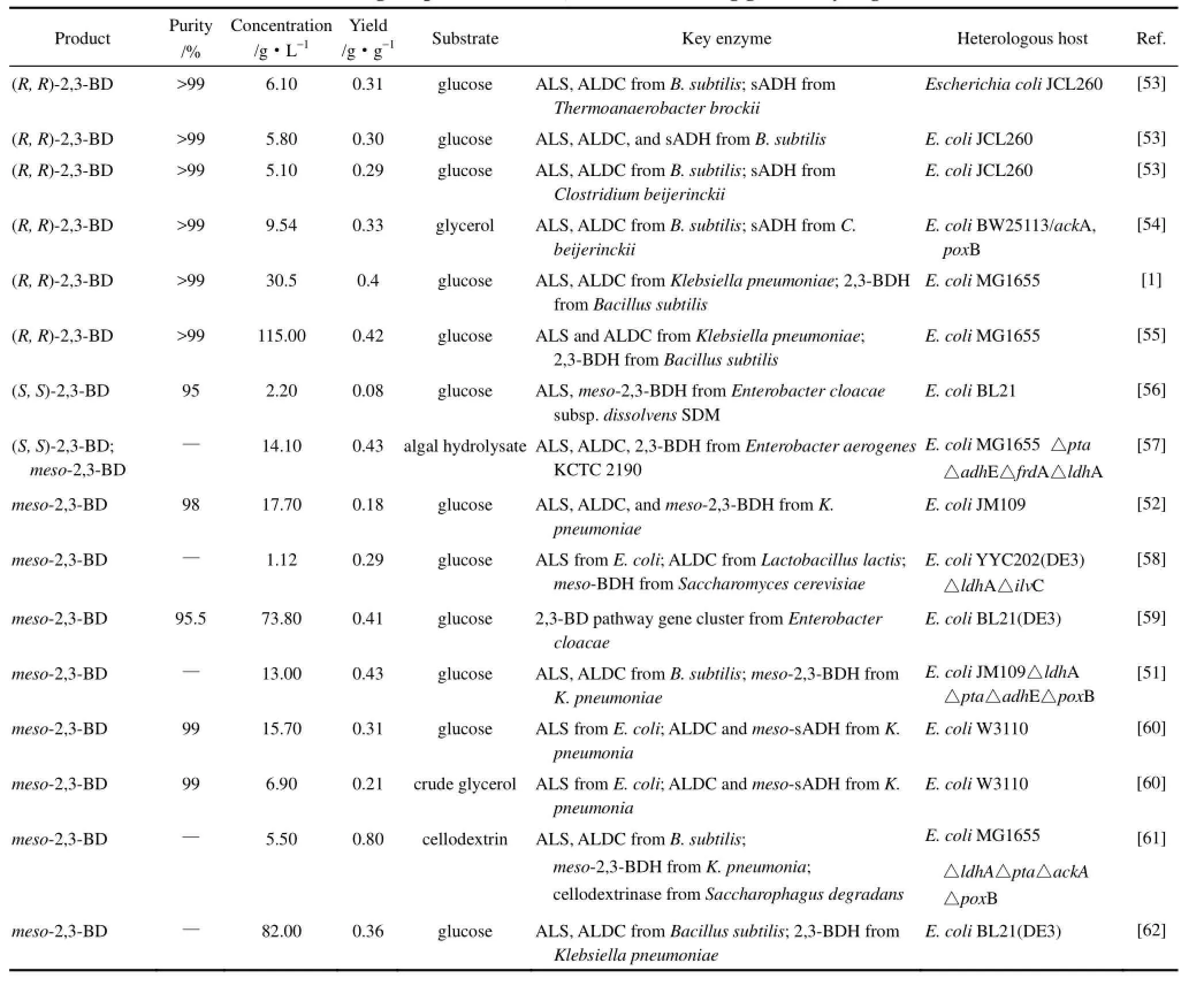
表5 大肠杆菌中2,3-丁二醇的研究水平Table 5 Biotechnological production of 2,3-butanediol using genetically engineered E. coli
Xu等[59]根据系统代谢工程的理念,从 B. subtilis、B. licheniformis、K. pneumoniae、S. marcescens和E. cloacae中克隆了2,3-BD生物合成的关键酶编码基因,分别以它们构建E. coli基因工程菌进行发酵测试,从中筛选产物产量最高的组合(来自E. cloacae),再通过对该基因组合进行不同强度启动子的优化,筛选得到其中产量最高的重组菌E. coli BL21/pET-RABC。该重组菌显示出优异的目标产物合成能力,经过一系列的条件优化,最终在批次发酵中获得24.6 g·L-1的meso-2,3-BD,补料发酵中获得73.8 g·L-1的meso-2,3-BD。Ji等[55]将来自于K. pneumoniae CICC 10011的budB、budA基因和来自于B. subtilis 168的(R, R)-2,3-BDH编码基因ydjL克隆到E. coli MG1655中,构建得到单一生产(R, R)-2,3-BD的工程菌,经一系列发酵条件优化,最终工程菌在补料发酵阶段(R, R)-2,3-BD的最高产量达到115 g·L-1,对映体纯度大于99%。这是到目前为止以 E. coli为宿主异源合成(R, R)-2,3-BD研究中所获得的最高产量。随后,Tong 等[1]在此基础上通过对重组菌进行启动子优化以及过表达关键酶基因等手段,使副产物乙偶姻的含量减少了20%。Li等[51]敲除了E. coli JM109菌株中混合酸途径副产物合成的相关基因 ldhA、pta(编码磷酸转乙酰酶)、adhE(编码乙醛脱氢酶及乙醇脱氢酶)、poxB(编码丙酮酸氧化酶),并在此基础上利用组成型启动子在E. coli中构建了一条适合在低溶氧条件下生产meso-2,3-BD的新途径。当溶氧降至 1%时,工程菌虽然生长缓慢却仍表现出较高的2,3-BD 得率,52 h发酵得到 meso-2,3-BD 13.0 g·L-1,相对于葡萄糖的得率达0.43 g·g-1,可以视作高纯度立体构型meso-2,3-BD工业化生产的一种新思路。目前生物制造(S,S)-2,3-BD大多采用的是生物催化或手性拆分的方法,底物多为乙偶姻、双乙酰或2,3-BD的混旋物[5],因此构建以葡萄糖等碳源为底物的完整的(S, S)-2,3-BD生物合成途径成为了科学家们努力的目标。Chu等[56]在E. coli中过表达了来自于E. cloacae subsp. dissolvens SDM的ALS编码基因和meso-2,3-BDH编码基因,利用双乙酰自发的非酶氧化脱羧过程成功构建了(S, S)-2,3-BD的代谢通路。通过优化发酵条件,得到了2.2 g·L-1的(S, S)-2,3-BD,对映体纯度达95%。这是第一次在模式菌株中以葡萄糖为碳源合成高纯度(S, S)-2,3-BD的报道。
影响胞内代谢生产的主要原因有胞内生产强度低、副产物竞争代谢流、代谢物或中间产物的积累会造成细胞毒害以及由于细胞膜的阻隔研究人员不能直接地监控代谢物等,这些因素限制了胞内代谢产量的进一步提高[62]。针对胞内代谢的缺陷,利用合成生物学的手段,无细胞代谢工程作为一种通过在体外实现催化蛋白的集合从而高效生产目标产物的技术应运而生。然而,高昂的辅因子和催化剂价格、辅因子和能量再生的限制以及较低的生产强度则阻碍了无细胞代谢工程的发展[63]。为了解决这一问题,Kay等[62]设计了一个从粗溶菌产物或细胞提取物中得到含有全部催化蛋白的无细胞系统来给高度活跃的外源途径提供能量。Kay等构建了一株E. coli基因工程菌来表达合成meso-2,3-BD所需的3种关键酶。随后他们证明了这株菌的溶菌产物在添加了葡萄糖以及仅催化量的辅因子NAD+和ATP之后,能够生产2,3-BD。在未经优化的情况下,批次发酵meso-2,3-BD的生产强度高达11.3 g·L-1·h-1,对葡萄糖的得率为0.36 g·g-1,为理论得率的71%。在30 h的补料发酵实验中,meso-2,3-BD的产量为82 g·L-1,生产强度为2.7 g·L-1·h-1。结果证明了在无细胞溶菌产物中强大的辅因子再生能力,并指出当生物转化的产量、生产强度或细胞毒性阻碍了全细胞发酵时,可以通过溶菌产物来加快原型代谢途径,实现分子转换。该方法可以视作2,3-BD高浓度生产的一种新思路。
随着对生物制造2,3-BD研究的日益深入,在设法提高产量和产率的同时,其制造过程中的经济性也逐渐成为关注与研究的焦点。为此,除了将合成生物学技术运用至2,3-BD途径构建之外,研究人员还试图开发一种廉价的碳水化合物材料来替代葡萄糖,并经过改造和优化提高菌体对这些原料的利用率。Shen等[54]在E. coli BW25113中表达了来自B. subtilis的 ALS和 ALDC,以及来自拜氏梭菌(Clostridium beijerinckii) 的次级醇脱氢酶ADH,在E. coli中构建了(R,R)-2,3-BD的人工合成途径,并尝试以甘油作为重组菌发酵的底物。该工程菌株以甘油为底物发酵时,(R,R)-2,3-BD的产量为 3.54 g·L-1。进一步敲除副产物乙酸合成途径的关键酶基因,(R,R)-2,3-BD的产量提高到了9.45 g·L-1,相对于甘油的转化率为0.33 g·(g甘油)-1,达到了理论转化率的68%。Shin等[61]将2,3-BD合成途径的引入与宿主菌副产物相关基因的敲除相结合,构建出一株专一生产meso-2,3-BD的E. coli基因工程菌,同时在该重组菌胞质中过表达来自Saccharophagus degradans的纤维素 3-糊精酶编码基因,使之能够以纤维素和半纤维素的部分水解产物纤维糊精为底物发酵生产 meso-2,3-BD。在发酵阶段,最终生成了meso-2,3-BD 5.5 g·L-1,乙偶姻2.7 g·L-1。这两种产物相对于纤维素糊精的总得率达0.8 g·g-1。海洋藻类生物由于其极快的生长速度、极大的生物量而成为生物燃料生产的一种潜在的能源。由于体内缺少木质素,它们并不需要经过严格的前处理过程,因此与其他的木质纤维素类生物质相比,海藻预处理过程的成本将要低许多。Mazumdar等[57]通过副产物基因敲除与2,3-BD合成通路整合等方法构建了一株能利用海藻水解液来发酵生产2,3-BD的重组E. coli,发酵中生成的2,3-BD与乙偶姻的总量为19 g·L-1,产率为0.43 g·g-1。这是第一次以海藻水解液发酵合成2,3-BD的报道。
2.2.2 以酿酒酵母为宿主 目前,高产2,3-BD的菌株,如K. pneumoniae,K. oxytoca,E. aerogens,S. marcescens等大多都是条件致病菌,而大规模工业化生产的安全性原则在很大程度上限制了这些菌株的应用。作为一种安全的微生物 (GRAS)[64],酿酒酵母 (Saccharomyces cerevisiae) 逐渐成为 2,3-BD生产的研究重点。在S. cerevisiae体内,糖酵解过程中产生的丙酮酸可以通过两条途径转换成2,3-BD[65-67]。一条途径是丙酮酸在ALS的催化作用下经过脱羧反应形成α-乙酰乳酸。在无氧条件下,α-乙酰乳酸在ALDC的催化下转化成乙偶姻。而当有O2存在时,α-乙酰乳酸会自发脱羧形成双乙酰,双乙酰在双乙酰还原酶 (DAR,又称乙偶姻脱氢酶)的作用下转变成乙偶姻。另一条途径是丙酮酸在丙酮酸脱羧酶 (PDC) 的催化下转化成乙醛,乙醛在PDC的催化下通过缩合反应转化成乙偶姻。最后,乙偶姻在2,3-BDH的作用下生成2,3-BD。但是,野生型的S. cerevisiae仅能生产微量的2,3-BD (0.4~2.0 g·L-1)[68],因此,更多的是尝试利用代谢工程以及合成生物学的手段构建含有外源 2,3-BD生产途径的S. cerevisiae基因工程菌。
因为S. cerevisiae在发酵时有着很强的积累乙醇的倾向,因此减少乙醇的生产并将代谢流转向2,3-BD对2,3-BD的生产至关重要。在S. cerevisiae中,丙酮酸在PDC的作用下经脱羧反应生成乙醛,乙醛再在乙醇脱氢酶 (ADH) 的作用下还原生成乙醇。由于乙醇途径和2,3-BD途径有着共同的中间代谢物,两者存在竞争机制,因此敲除ADH和PDC编码基因会使菌株生产乙醇的能力大大减弱,PDC的缺乏乃至消失会使得中间产物丙酮酸大量积累,从而最大限度地提高2,3-BD的产量。Ng等[69]敲除了S. cerevisiae中编码乙醇脱氢酶ADH1、ADH3和ADH5的3个基因,在微氧条件下,2,3-BD的产量提高了55倍。虽然重组菌与原始菌相比显著提高了产量,但2,3-BD的总体发酵水平还是不如常见的细菌,只有2.29 g·L-1。2013年,Kim等[22]通过构建PDC缺陷型 (pdc-) 菌株来进行葡萄糖的快速转化,并且使得副产物乙醇得到抑制。通过将 B. subtilis 2,3-BD合成途径的alsS 和alsD基因导入pdc-S. cerevisiae并过表达内源2,3-BDH的方法,在最优通气条件下分批补料发酵,最终2,3-BD的产量达到96.2 g·L-1。除了葡萄糖,纤维素糖类也能被用来生产 2,3-BD。Nan等[70]在一个 pdc-的 S. cerevisiae中过表达了来自B. subtilis的alsS、alsD基因,并引入了一条纤维二糖利用途径,使得重组菌能在微氧条件下以纤维二糖为底物合成2,3-BD。Kim等[71]以一株pdc-菌株 (SOS4) 为宿主,导入来自木糖发酵酵母的编码木糖同化酶的XYL1、XYL2 和XYL3基因,使得菌株得以消耗木糖,并以此将碳流向从乙醇转向了 2,3-BD。通过发酵补料,2,3-BD的产量达到43.6 g·L-1,(R, R)-2,3-BD对映体纯度达到 97%。这些结果表明,工程化的 S. cerevisiae菌株可以成为高效利用可再生资源生产2,3-BD的最适宿主菌株。
但是,生产2,3-BD的pdc-菌株却有着严重的生长缺陷问题。在酵母体内,乙醇发酵的主要功能就是再氧化糖酵解过程中产生的NADH,而乙醇途径的敲除会导致pdc-菌株体内NADH的大量积累,使得菌体内氧化还原失衡,从而阻碍细胞的生长[72]。除此之外,由于生成一分子的甘油就能氧化一分子的NADH,因此在微氧条件下,为了维持菌体内的氧化还原平衡,生产2,3-BD的pdc-菌株通常伴随着大量甘油的生成。Kim 等[64]在 pdc-的 S. cerevisiae SOS2的基础上构建了 5株有着不同 L. lactis NADH氧化酶表达强度的重组菌。NADH氧化酶的表达有效地降低了胞内 NADH/NAD+的比例。与原始菌相比,重组菌2,3-BD对葡萄糖的产率提高了 23.8%,而甘油对葡萄糖的产率则下降了65.3%,说明通过表达 NADH 氧化酶能改变NADH/NAD+的比例从而达到将流向甘油的代谢流引向2,3-BD的目的。在以上研究的基础上,Kim等[73]在酵母中过表达了来自B. subtilis的ALS、ALDC编码基因以及内源的2,3-BDH编码基因并敲除了与副产物乙醇、甘油生产相关的 5种乙醇脱氢酶(ADH1~ADH5)编码基因和2种甘油-3-磷酸盐脱氢酶(GPD1和 GPD2)编码基因。在随后的补料发酵中,重组菌 2,3-BD的产量最高可达 72.9 g·L-1,对葡萄糖的产率为0.41 g·g-1,生产强度为1.43 g·L-1·h-1。除此之外,由于葡萄糖会抑制pdc-菌株的呼吸作用导致其体内氧化还原失衡,因此pdc-菌株在葡萄糖为唯一碳源的培养基中无法生长[74],而且 pdc-菌株在生长过程中强烈依赖 C2物质(乙酸或乙醇)来合成生长所需的乙酰辅酶A。这些缺陷极大地限制了pdc-菌株的应用。有研究表明,MTH1基因编码序列的内部敲除会使pdc-菌株重新获得摄取葡萄糖的能力[75-76]。MTH1是一种能够阻碍己糖转运蛋白基因 (HXTs) 表达的葡萄糖敏感型转录因子。这种内部敲除能通过移除与蛋白降解相关的位点来增加MTH1的稳定性[75],维持胞内葡萄糖浓度至一个很低的水平并因此缓解葡萄糖的抑制。Lian等[21]选择S. cerevisiae作为宿主,通过敲除 PDC5-PDC6-PDC1编码基因使得从乙醇到2,3-BD的代谢流得到重新构建,但是得到的重组菌并不能在己糖作为单一碳源的培养基上生长。为了解决这一问题,他们通过过表达内部敲除的转录因子MTH1以及适应性进化等改造手段,成功地获得了一株能同时利用葡萄糖和半乳糖的工程酵母。随后(R, R)-2,3-BD生物合成途径的引入赋予了该菌株高产目标产物的能力,在以葡萄糖和半乳糖混合物为底物的补料发酵中,该重组酵母生产(R, R)-2,3-BD高达100 g·L-1以上。这是目前报道在S. cerevisiae中2,3-BD合成的最高产量,但仍然存在着发酵周期太长 (300 h) 的问题。
除了常规的利用质粒作为载体表达外源基因外,染色体由于其能自主复制、能确保外源基因在细胞世代中保持稳定等优点,具有质粒表达不可比拟的优势。Shi等[77]发明了一种一步高效无痕多拷贝的染色体整合技术 (Di-CRISPR),通过这种技术,成功地将一个18拷贝的、全长24 kb的木糖利用以及(R, R)-2,3-BD合成途径一步整合至S. cerevisiae染色体,获得了一株能以木糖为底物发酵生产(R, R)-2,3-BD的菌株,第一次实现了2,3-BD途径在S. cerevisiae染色体上的表达。虽然这种方法生产的2,3-BD产量并不高,但为构建一株稳定表达外源 2,3-BD合成途径的酵母工程菌提供了新的思路。
2.2.3 以蓝细菌为宿主 自从工业革命开始以来,人类大量使用化石燃料,这些过程把原本以有机碳形式存在的碳转化为二氧化碳并排放到空气中,从而造成资源的浪费以及环境问题的加剧。根据美国国家海洋和大气管理局 (NOAA) 发布的数据显示,2015年3月全球大气中的二氧化碳月平均浓度已经突破了全球变暖危险标志的400 μl·L-1关口。蓝藻能够通过光合作用直接将空气中的CO2转换为有机碳,既实现了资源的再利用,又缓解了日益严重的温室效应问题,并且由于其能在高盐和高温环境生长的特性也避免了其与陆地上的粮食作物竞争水源以及土地。但是除了高效的CO2固定能力,蓝藻并不能自发地积累有用的化学原料[78],而合成生物学的应用赋予了其生产人类所需化学品的能力[79-84]。
Oliver等[85]以细长聚球藻 (Synechococcus elongatus) PCC7942为宿主,在蓝藻中构建了2,3-BD的生物合成途径。选取B. subtilis的alsS基因,并在重组菌中分别表达了6种来自不同菌株的alsD基因,以及4种sADH基因。最终,含有alsS (B. subtilis),alsD(嗜水气单胞菌,Aeromonas hydrophila),adh (C. beijerinckii) 的重组菌表现了最佳的2,3-BD生产性能。利用CO2和光源为能源,生产得到的2,3-BD产量达到2.38 g·L-1。在之前工作的基础上,Oliver等[86]为了克服S. elongatus PCC7922中报告蛋白表达水平与生产所需蛋白表达水平的不一致,通过一个组合方法证明调节5′非翻译区可以产生一系列不同强度的蛋白质表达,克服了菌株中蛋白表达水平的不可预见性。
Savakis等[87]在集胞藻 (Synechocystis sp.) PCC6803中异源表达了来自乳酸菌和肠杆菌的ALS、ALDC和2,3-BDH编码基因。无论是质粒或是染色体表达这条人工构建的2,3-BD代谢途径,都可以在发酵液中检测到高达0.72 g·L-1(相当于8 mmol·L-1) 的四碳化合物,其中2,3-BD的浓度达0.43 g·L-1(相当于 4.7 mmol·L-1),成功实现了2,3-BD代谢途径在Synechocystis sp. PCC6803中的构建与组装。进一步研究发现,在限制二氧化碳供应的条件下,Synechocystis sp.能内源合成大量的ALS。而共表达一种可溶性转氢酶或者一种NADPH依赖型2,3-BDH能有效促进乙偶姻与2,3-BD的转化,从而生产出高纯度的meso-2,3-BD。
虽然目前蓝藻发酵生产 2,3-BD的产量均比较低,但是国内外研究学者的成果实现了将零成本的无机化合物 CO2转化为具有较高附加值的 2,3-BD的突破,并且符合目前社会广泛提倡的发展绿色低碳经济的趋势,所以该方法生产2,3-BD的前景不可估量。
3 总结与展望
步入 21世纪,社会的可持续发展正面临着前所未有的挑战,资源匮乏、能源紧缺和环境污染等现状带来的巨大压力,迫使传统的化工、材料和制药等工业制造领域的生产技术必须发生根本性的变革。以石化路线为主的化学制造存在原料紧缺、易污染环境等诸多缺陷,迫使人们寻找更有利于未来发展的工业制造方式。作为一种重要的化工原料,同时又是潜在的平台化合物,2,3-BD的应用前景广阔,因此近年来以其生物制造为主题的研究日益深入,并在菌种改良和底物优化等方面取得了显著成果。展望未来,应着重从以下几个方面开展后续的研究。
(1)在充分了解 2,3-BD生物合成途径和副产物代谢途径的基础上,结合辅因子调控与全局转录调控技术,通过对野生菌株进行基因改造来进一步提高2,3-BD的产量。
(2)目前高产 2,3-BD 的发酵菌株如 K. pneumoniae、K. oxytoca、S. marcescens、E. cloacae等均为class 2致病菌株,这在将来的大规模工业化生产中是非常不利的。如今,通过敲除 K. pneumoniae中致病因子相关基因来减轻或消除其致病性已经成为了现实,但还没有其他致病菌的类似报道。因此,对其他致病菌进行全基因组测序和注释,可以帮助研究者从基因角度了解菌株致病的机制和原因,为安全菌株的构建奠定坚实的基础。
(3)对于2,3-BD来说,虽然利用葡萄糖为底物发酵在产量、产率等方面均取得了较大的进展,但由于葡萄糖价格昂贵,降低了规模化生产的经济性。通过代谢工程以及合成生物学技术对菌株进行进一步改造以提高菌株对工业废弃物和非粮原料的利用效率是十分重要的。
(4)应当继续利用先进的合成生物学方法,例如人工合成基因线路技术、启动子工程技术以及基因组编辑技术等优化异源2,3-BD的代谢途径,实现胞内代谢流的最佳分配,快速获得遗传物质稳定、发酵水平和光学纯度提高的细胞工厂。
(5)需要深入优化蓝细菌作为新型宿主的研究,通过直接有效地利用CO2生产大宗化学品来实现能源的再生以及减缓温室效应。
References
[1] TONG Y J, JI X J, SHEN M Q, et al. Constructing a synthetic constitutive metabolic pathway in Escherichia coli for (R, R)-2,3-butanediol production [J]. Applied Microbiology & Biotechnology, 2016, 100(2): 637-647.
[2] JI X J, HUANG H, OUYANG P K. Microbial 2,3-butanediol production: a state-of-the-art review [J]. Biotechnology Advances, 2011, 29(3): 351-364.
[3] CELINSKA E, GRAJEK W. Biotechnological production of 2,3-butanediol-current state and prospects [J]. Biotechnology Advances, 2009, 27(6): 715-725.
[4] 纪晓俊, 聂志奎, 黎志勇, 等. 生物制造 2,3-丁二醇: 回顾与展望[J]. 化学进展, 2010, 22(12): 2450-2461.
JI X J, NIE Z K, LI Z Y, et al. Biotechnological production of 2,3-butanediol [J]. Progress in Chemistry, 2010, 22(12): 2450-2461.
[5] 沈梦秋, 纪晓俊, 聂志奎, 等. 生物制造不同立体构型2, 3-丁二醇:合成机理与实现方法 [J]. 催化学报, 2013, 34(2): 351-360.
SHEN M Q, JI X J, NIE Z K, et al. Biotechnological production of 2,3-butanediol stereoisomers: synthetic mechanism and realized methods [J]. Chinese Journal of Catalysis, 2013, 34(2): 351-360.
[6] ZENG A P, SABRA W. Microbial production of diols as platform chemicals: recent progresses [J]. Current Opinion in Biotechnology, 2011, 22(6): 749-757.
[7] 付晶, 王萌, 刘维喜, 等. 生物法制备 2,3-丁二醇的最新进展 [J].化学进展, 2012, 24(11): 2268-2276.
FU J, WANG M, LIU W X, et al. Latest advances of microbial production of 2,3-butanediol [J]. Progress in Chemistry, 2012, 24(11): 2268-2276.
[8] MA C, WANG A, QIN J, et al. Enhanced 2, 3-butanediol production by Klebsiella pneumoniae SDM [J]. Appl. Microbiol. Biotechnol., 2009, 82(1): 49-57.
[9] CHO S, KIM T, WOO H M, et al. Enhanced 2,3-butanediol production by optimizing fermentation conditions and engineering Klebsiella oxytoca M1 through overexpression of acetoin reductase [J]. Plos One, 2015, 10(9): e0138109.
[10] JUNG M Y, JUNG H M, LEE J, et al. Alleviation of carbon catabolite repression in Enterobacter aerogenes for efficient utilization of sugarcane molasses for 2,3-butanediol production [J]. Biotechnol. Biofuels, 2015, 8(1): 1-12.
[11] LI L X, LI K, WANG Y, et al. Metabolic engineering of Enterobacter cloacae for high-yield production of enantiopure (2R,3R)-2,3-butanediol from lignocellulose-derived sugars [J]. Metabolic Engineering, 2015, 28: 19-27.
[12] FU J, WANG Z, CHEN T, et al. NADH plays the vital role for chiral pure D-(-)-2,3-butanediol production in Bacillus subtilis under limited oxygen conditions [J]. Biotechnology and Bioengineering, 2014, 111(10): 2126-2131.
[13] LI L, ZHANG L, LI K, et al. A newly isolated Bacillus licheniformis strain thermophilically produces 2, 3-butanediol, a platform and fuel bio-chemical [J]. Biotechnol Biofuels, 2013, 6(1): 213-223.
[14] YANG T, RAO Z, ZHANG X, et al. Improved production of 2,3-butanediol in Bacillus amyloliquefaciens by over-expression of glyceraldehyde-3-phosphate dehydrogenase and 2,3-butanediol dehydrogenase [J]. Plos One, 2013, 8(10): e76149.
[15] HAESSLER T, SCHIEDER D, PFALLER R, et al. Enhanced fed-batch fermentation of 2, 3-butanediol by Paenibacillus polymyxa DSM 365 [J]. Bioresource Technology, 2012, 124: 237-244.
[16] ZHANG L, SUN J, HAO Y, et al. Microbial production of 2,3-butanediol by a surfactant (serrawettin)-deficient mutant of Serratia marcescens H30 [J]. J. Ind. Microbiol. Biotechnol., 2010, 37(8): 857-62.
[17] YANG T H, RATHNASINGH C, LEE H J, et al. Identification of acetoin reductases involved in 2, 3-butanediol pathway in Klebsiella oxytoca [J]. Journal of Biotechnology, 2014, 172: 59-66.
[18] LEE S, KIM B, JEONG D, et al. Observation of 2,3-butanediol biosynthesis in Lys regulator mutated Klebsiella pneumoniae at gene transcription level [J]. Journal of Biotechnology, 2013, 168(4): 520-526.
[19] OLIVEIRA R R D, NICHOLSON W L. The LysR-type transcriptional regulator (LTTR) AlsR indirectly regulates expression of the Bacillus subtilis bdhA gene encoding 2,3-butanediol dehydrogenase [J]. Appl. Microbiol. Biotechnol., 2013, 97(16): 7307-7316.
[20] FRAEDRICH C, MARCH A, FIEGE K, et al. The transcription factor AlsR binds and regulates the promoter of the alsSD operon responsible for acetoin formation in Bacillus subtilis [J]. Journal of Bacteriology, 2012, 194(5): 1100-1112.
[21] LIAN J, CHAO R, ZHAO H. Metabolic engineering of aSaccharomyces cerevisiae strain capable of simultaneously utilizing glucose and galactose to produce enantiopure (2R, 3R)-butanediol [J]. Metab. Eng., 2014, 23: 92-99.
[22] KIM S J, SEO S O, JIN Y S, et al. Production of 2, 3-butanediol by engineered Saccharomyces cerevisiae [J]. Bioresource Technology, 2013, 146: 274-281.
[23] GUO X W, ZHANG Y H, CAO C H, et al. Enhanced production of 2,3-butanediol by overexpressing acetolactate synthase and acetoin reductase in Klebsiella pneumoniae [J]. Biotechnology and Applied Biochemistry, 2014, 61(6): 707-715.
[24] ZHANG L. Enhanced 2,3-butanediol production by Serratia marcescens H30 with over-expression of 2,3-butanediol dehydrogenase [J]. Res. J. Biotechnol., 2015, 10(5): 75-80.
[25] BAI F, DAI L, FAN J, et al. Engineered Serratia marcescens for efficient (3R)-acetoin and (2R, 3R)-2, 3-butanediol production [J]. J. Ind. Microbiol. Biotechnol., 2015, 42(5): 779-786.
[26] ZHENG Y, ZHANG H, ZHAO L, et al. One-step production of 2,3-butanediol from starch by secretory over-expression of amylase in Klebsiella pneumoniae [J]. Journal of Chemical Technology and Biotechnology, 2008, 83(10): 1409-1412.
[27] TSVETANOVA F, PETROVA P, PETROV K. 2,3-Butanediol production from starch by engineered Klebsiella pneumoniae G31-A [J]. Appl. Microbiol. Biotechnol., 2014, 98(6): 2441-2451.
[28] JI X J, NIE Z K, HUANG H, et al. Elimination of carbon catabolite repression in Klebsiella oxytoca for efficient 2,3-butanediol production from glucose-xylose mixtures [J]. Appl. Microbiol. Biotechnol., 2011, 89(4): 1119-1125.
[29] JANTAMA K, POLYIAM P, KHUNNONKWAO P, et al. Efficient reduction of the formation of by-products and improvement of production yield of 2, 3-butanediol by a combined deletion of alcohol dehydrogenase, acetate kinase-phosphotransacetylase, and lactate dehydrogenase genes in metabolically engineered Klebsiella oxytoca in mineral salts medium [J]. Metab. Eng., 2015, 30: 16-26.
[30] JI X J, HUANG H, ZHU J G, et al. Engineering Klebsiella oxytoca for efficient 2,3-butanediol production through insertional inactivation of acetaldehyde dehydrogenase gene [J]. Appl. Microbiol. Biotechnol., 2010, 85(6): 1751-1758.
[31] QI G F, KANG Y F, LI L, et al. Deletion of meso-2, 3-butanediol dehydrogenase gene budC for enhanced D-2, 3-butanediol production in Bacillus licheniformis [J]. Biotechnol. Biofuels, 2014, 7(1): 754-760.
[32] RADOS D, CARVALHO A L, WIESCHALKA S, et al. Engineering Corynebacterium glutamicum for the production of 2, 3-butanediol [J]. Microb. Cell Fact., 2015, 14(1): 171.
[33] CHO S, KIM T, WOO H M, et al. High production of 2, 3-butanediol from biodiesel-derived crude glycerol by metabolically engineered Klebsiella oxytoca M1 [J]. Biotechnol. Biofuels, 2015, 8(1): 1-12.
[34] 康燕菲, 田平芳, 谭天伟. 肺炎克雷伯氏菌毒力因子的研究进展[J]. 微生物学报, 2015, 55(10): 1245-1252.
KANG Y F, TIAN P F, TAN T W. Research advances in the virulence factors of Klebsiella pneumoniae — a review [J]. Acta Microbiologica Sinica, 2015, 55 (10): 1245-1252.
[35] JUNG S G, JANG J H, KIM A Y, et al. Removal of pathogenic factors from 2,3-butanediol-producing Klebsiella species by inactivating virulence-related wabG gene [J]. Appl. Microbiol. Biotechnol., 2013, 97(5): 1997-2007.
[36] HUYNH D T, KIM A Y, SEOL I H, et al. Inactivation of the virulence factors from 2,3-butanediol-producing Klebsiella pneumoniae [J]. Appl. Microbiol. Biotechnol., 2015, 99(22): 9427-9438.
[37] KIM B, LEE S, JEONG D, et al. Redistribution of carbon flux toward 2,3-butanediol production in Klebsiella pneumoniae by metabolic engineering [J]. Plos One, 2014, 9(10): e105322.
[38] PARK J M, SONG H, LEE H J, et al. In silico aided metabolic engineering of Klebsiella oxytoca and fermentation optimization for enhanced 2,3-butanediol production [J]. J. Ind. Microbiol. Biotechnol., 2013, 40(9): 1057-1066.
[39] DUETZ W A, VAN BEILEN J B, WITHOLT B. Using proteins in their natural environment: potential and limitations of microbial whole-cell hydroxylations in applied biocatalysis [J]. Current Opinion in Biotechnology, 2001, 12(4): 419-425.
[40] DREPPER T, EGGERT T, HUMMEL W, et al. Novel biocatalysts for white biotechnology [J]. Biotechnology Journal, 2006, 1(7/8): 777-786.
[41] YANG T, RAO Z, HU G, et al. Metabolic engineering of Bacillus subtilis for redistributing the carbon flux to 2,3-butanediol by manipulating NADH levels [J]. Biotechnol. Biofuels, 2015, 8(1): 1-9.
[42] ZHANG L, XU Y Y, GAO J, et al. Introduction of the exogenous NADH coenzyme regeneration system and its influence on intracellular metabolic flux of Paenibacillus polymyxa [J]. Bioresource Technology, 2016, 201: 319-328.
[43] GECKIL H, GENCER S, KAHRAMAN H, et al. Genetic engineering of Enterobacter aerogenes with the Vitreoscilla hemoglobin gene: cell growth, survival, and antioxidant enzyme status under oxidative stress [J]. Research in Microbiology, 2003, 154(6): 425-431.
[44] GECKIL H, BARAK Z, CHIPMAN D M, et al. Enhanced production of acetoin and butanediol in recombinant Enterobacter aerogenes carrying Vitreoscilla hemoglobin gene [J]. Bioprocess and Biosystems Engineering, 2004, 26(5): 325-330.
[45] JEONG J W, PARK K M, CHUNG M, et al. Influence of Vitreoscilla hemoglobin gene expression on 2,3-butanediol production in Klebsiella oxytoca [J]. Biotechnology and Bioprocess Engineering, 2015, 20(1): 10-17.
[46] ALPER H, MOXLEY J, NEVOIGT E, et al. Engineering yeast transcription machinery for improved ethanol tolerance and production [J]. Science, 2006, 314(5805): 1565-1568.
[47] MOONS P, VAN HOUDT R, VIVIJS B, et al. Integrated regulation of acetoin fermentation by quorum sensing and pH in Serratia plymuthica RVH1 [J]. Applied and Environmental Microbiology, 2011, 77(10): 3422-3427.
[48] ZHANG X, ZHANG R Z, BAO T, et al. Moderate expression of the transcriptional regulator ALsR enhances acetoin production by Bacillus subtilis [J]. J. Ind. Microbiol. Biotechnol., 2013, 40(9): 1067-1076.
[49] YANG T W, RAO Z M, ZHANG X, et al. Enhanced 2, 3-butanediol production from biodiesel-derived glycerol by engineering of cofactor regeneration and manipulating carbon flux in Bacillus amyloliquefaciens [J]. Microbial Cell Factories, 2015, 14(1): 1-11.
[50] KEASLING J D. Manufacturing molecules through metabolic engineering [J]. Science, 2010, 330(6009): 1355-1358.
[51] LI Z J, JIAN J, WEI X X, et al. Microbial production of meso-2,3-butanediol by metabolically engineered Escherichia coli underlow oxygen condition [J]. Appl. Microbiol. Biotechnol., 2010, 87(6): 2001-2009.
[52] UI S, OKAJIMA Y, MIMURA A, et al. Molecular generation of an Escherichia coli strain producing only the meso-isomer of 2,3-butanediol [J]. Journal of Fermentation and Bioengineering, 1997, 84(3): 185-189.
[53] YAN Y, LEE C C, LIAO J C. Enantioselective synthesis of pure (R,R)-2,3-butanediol in Escherichia coli with stereospecific secondary alcohol dehydrogenases [J]. Organic & Biomolecular Chemistry, 2009, 7(19): 3914-3917.
[54] SHEN X, LIN Y, JAIN R, et al. Inhibition of acetate accumulation leads to enhanced production of (R, R)-2, 3-butanediol from glycerol in Escherichia coli [J]. J. Ind. Microbiol. Biotechnol., 2012, 39(11): 1725-1729.
[55] JI X J, LIU L G, SHEN M Q, et al. Constructing a synthetic metabolic pathway in Escherichia coli to produce the enantiomerically pure (R, R)-2,3-butanediol [J]. Biotechnology and Bioengineering, 2015, 112(5): 1056-1059.
[56] CHU H, XIN B, LIU P, et al. Metabolic engineering of Escherichia coli for production of (2S,3S)-butane-2,3-diol from glucose [J]. Biotechnol. Biofuels, 2015, 8(1): 1-11.
[57] MAZUMDAR S, LEE J, OH M K. Microbial production of 2,3-butanediol from seaweed hydrolysate using metabolically engineered Escherichia coli [J]. Bioresour. Technol., 2013, 136: 329-336.
[58] NIELSEN D R, YOON S H, YUAN C J, et al. Metabolic engineering of acetoin and meso-2,3-butanediol biosynthesis in E. coli [J]. Biotechnology Journal, 2010, 5(3): 274-284.
[59] XU Y, CHU H, GAO C, et al. Systematic metabolic engineering of Escherichia coli for high-yield production of fuel bio-chemical 2,3-butanediol [J]. Metabolic Engineering, 2014, 23: 22-33.
[60] LEE S, KIM B, PARK K, et al. Synthesis of pure meso-2,3-butanediol from crude glycerol using an engineered metabolic pathway in Escherichia coli [J]. Applied Biochemistry and Biotechnology, 2012, 166(7): 1801-1813.
[61] SHIN H D, YOON S H, WU J, et al. High-yield production of meso-2,3-butanediol from cellodextrin by engineered E. coli biocatalysts [J]. Bioresource Technology, 2012, 118: 367-373.
[62] KAY J E, JEWETT M C. Lysate of engineered Escherichia coli supports high-level conversion of glucose to 2,3-butanediol [J]. Metabolic Engineering, 2015, 32: 133-142.
[63] DUDLEY Q M, KARIM A S, JEWETT M C. Cell-free metabolic engineering: biomanufacturing beyond the cell [J]. Biotechnology Journal, 2015, 10(1): 69-82.
[64] KIM J W, SEO S O, ZHANG G C, et al. Expression of Lactococcus lactis NADH oxidase increases 2,3-butanediol production in Pdc-deficient Saccharomyces cerevisiae [J]. Bioresource Technology, 2015, 191: 512-519.
[65] ROMANO P, SUZZI G. Origin and production of acetoin during wine yeast fermentation [J]. Applied and Environmental Microbiology, 1996, 62(2): 309-315.
[66] CHEN G C, JORDAN F. Brewers-yeast pyruvate decarboxylase produces acetoin from acetaldehyde—a novel tool to study the mechanism of steps subsequent to carbon-dioxide loss [J]. Biochemistry, 1984, 23(16): 3576-3582.
[67] EHSANI M, FERNANDEZ M R, BIOSCA J A, et al. Reversal of coenzyme specificity of 2,3-butanediol dehydrogenase from Saccharomyces cerevisae and in vivo functional analysis [J]. Biotechnology and Bioengineering, 2009, 104(2): 381-389.
[68] EHSANI M, FERNANDEZ M R, BIOSCA J A, et al. Engineering of 2,3-butanediol dehydrogenase to reduce acetoin formation by glycerol-overproducing, low-alcohol Saccharomyces cerevisiae [J]. Appl. Environ. Microbiol., 2009, 75(10): 3196-3205.
[69] NG C Y, JUNG M Y, LEE J, et al. Production of 2, 3-butanediol in Saccharomyces cerevisiae by in silico aided metabolic engineering [J]. Microb. Cell Fact., 2012, 11(20): 115-120.
[70] NAN H, SEO S O, OH E J, et al. 2, 3-Butanediol production from cellobiose by engineered Saccharomyces cerevisiae [J]. Appl. Microbiol. Biotechnol., 2014, 98(12): 5757-5764.
[71] KIM S J, SEO S O, PARK Y C, et al. Production of 2, 3-butanediol from xylose by engineered Saccharomyces cerevisiae [J]. J. Biotechnol., 2014, 192: 376-382.
[72] FLIKWEERT M T, DE SWAAF M, VAN DIJKEN J P, et al. Growth requirements of pyruvate-decarboxylase-negative Saccharomyces cerevisiae [J]. FEMS Microbiology Letters, 1999, 174(1): 73-79.
[73] KIM S, HAHN J S. Efficient production of 2,3-butanediol in Saccharomyces cerevisiae by eliminating ethanol and glycerol production and redox rebalancing [J]. Metab. Eng., 2015, 31: 94-101.
[74] CAMBON B, MONTEIL V, REMIZE F, et al. Effects of GPD1 overexpression in Saccharomyces cerevisiae commercial wine yeast strains lacking ALD6 genes [J]. Applied and Environmental Microbiology, 2006, 72(7): 4688-4694.
[75] OUD B, FLORES C L, GANCEDO C, et al. An internal deletion in MTH1 enables growth on glucose of pyruvate-decarboxylase negative, non-fermentative Saccharomyces cerevisiae [J]. Microbial Cell Factories, 2012, 11(5): 54-55.
[76] VAN MARIS A J A, GEERTMAN J M A, VERMEULEN A, et al. Directed evolution of pyruvate decarboxylase-negative Saccharomyces cerevisiae, yielding a C-2-independent, glucosetolerant, and pyruvate-hyperproducing yeast [J]. Applied and Environmental Microbiology, 2004, 70(1): 159-166.
[77] SHI S, LIANG Y, ZHANG M M, et al. A highly efficient single-step, markerless strategy for multi-copy chromosomal integration of large biochemical pathways in Saccharomyces cerevisiae [J]. Metab. Eng., 2015, 33: 19-27.
[78] MACHADO I M P, ATSUMI S. Cyanobacterial biofuel production [J]. Journal of Biotechnology, 2012, 162(1): 50-56.
[79] ATSUMI S, HIGASHIDE W, LIAO J C. Direct photosynthetic recycling of carbon dioxide to isobutyraldehyde [J]. Nature Biotechnology, 2009, 27(12): 1177-U142.
[80] LAN E I, LIAO J C. ATP drives direct photosynthetic production of 1-butanol in cyanobacteria [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(16): 6018-6023.
[81] TAKAHAMA K, MATSUOKA M, NAGAHAMA K, et al. Construction and analysis of a recombinant cyanobacterium expressing a chromosomally inserted gene for an ethylene-forming enzyme at the psbAI locus [J]. Journal of Bioscience and Bioengineering, 2003, 95(3): 302-305.
[82] ZHOU J, ZHANG H, ZHANG Y, et al. Designing and creating a modularized synthetic pathway in cyanobacterium Synechocystisenables production of acetone from carbon dioxide [J]. Metabolic Engineering, 2012, 14(4): 394-400.
[83] LIU X, SHENG J, CURTISS R Ⅲ. Fatty acid production in genetically modified cyanobacteria [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(17): 6899-6904.
[84] TAN X, YAO L, GAO Q, et al. Photosynthesis driven conversion of carbon dioxide to fatty alcohols and hydrocarbons in cyanobacteria [J]. Metabolic Engineering, 2011, 13(2): 169-176.
[85] OLIVER J W, MACHADO I M, YONEDA H, et al. Cyanobacterial conversion of carbon dioxide to 2, 3-butanediol [J]. Proc. Natl. Acad. Sci. U. S. A., 2013, 110(4): 1249-1254.
[86] OLIVER J W K, MACHADO I M P, YONEDA H, et al. Combinatorial optimization of cyanobacterial 2,3-butanediol production [J]. Metabolic Engineering, 2014, 22: 76-82.
[87] SAVAKIS P E, ANGERMAYR S A, HELLINGWERF K J. Synthesis of 2,3-butanediol by Synechocystis sp. PCC6803 via heterologous expression of a catabolic pathway from lactic acid- and enterobacteria [J]. Metab. Eng., 2013, 20: 121-130.
2016-02-26收到初稿,2016-04-23收到修改稿。
联系人:纪晓俊。第一作者:童颖佳(1992—),女,博士研究生。
Received date: 2016-02-26.
中图分类号:O 622. 3; TQ 923
文献标志码:A
文章编号:0438—1157(2016)07—2656—16
DOI:10.11949/j.issn.0438-1157.20160209
基金项目:国家重点基础研究发展计划项目(2011CBA00800);国家自然科学基金项目(21376002,21476111);国家高技术研究发展计划项目(2011AA02A207);江苏高校优势学科建设工程资助项目。
Corresponding author:JI Xiaojun, associate professor, xiaojunji@njtech. edu.cn supported by the National Basic Research Program of China (2011CBA00800), the National Natural Science Foundation of China (21376002, 21476111), the National High Technology Research and Development Program of China (2011AA02A207) and the Priority Academic Program Development of Jiangsu Higher Education Institutions.
Metabolic engineering for efficient microbial production of 2,3-butanediol
TONG Yingjia, WU Wenjia, PENG Hui, LIU Lugang, HUANG He, JI Xiaojun
(State Key Laboratory of Materials-Oriented Chemical Engineering, College of Biotechnology and Pharmaceutical Engineering, Nanjing Tech University, Nanjing 211816, Jiangsu, China)
Abstract:2,3-butanediol (2,3-BD), which is considered as an important microbial metabolite, has been widely used in many fields such as food, medicine, chemical, and so on. Microbial 2,3-BD production has a history of more than 100 years, but the low efficiency of microbial 2,3-BD accumulation has constrained its process in biological manufacturing industrialization. Optimization of microbial metabolic pathway with the theory and method of metabolic engineering is expected to solve this problem. The objective of this paper is to review the state-of-the-art strain transformation and construction strategies in microbial synthesis of 2,3-BD, including overexpressing genes encoding for key enzymes in the 2,3-BD metabolic pathway, knocking out the metabolic bypass way genes, and using the methods of cofactor engineering in redesigning and reasonable transformation of the natural strains’ metabolic network. Besides that, the using of synthetic biology in constructing brand new 2,3-BD pathways in model strains, such as Escherichia coli, Saccharomyces cerevisiae and Cyanobacteria, in order to enhance the yield or chiral 2,3-BD production in microorganisms is also introduced in this review. Finally, the future research direction is prospected, and the guidelines to develop high-efficiency microbial cell factories byadvanced synthetic biology methods to achieve the optimal allocation of the intracellular metabolic flow are also proposed.
Key words:2,3-butanediol;metabolism;biochemical engineering;cofactors regulation;global transcription machinery engineering;synthetic biology

