柳树β微管蛋白基因家族的克隆和序列分析
睢金凯,饶国栋,2,张建国,2*
(1 国家林业局林木培育重点实验室,中国林业科学研究院林业研究所,北京 100091;2 南京林业大学南方现代林业协同创新中心,南京 210037)
柳树β微管蛋白基因家族的克隆和序列分析
睢金凯1,饶国栋1,2,张建国1,2*
(1 国家林业局林木培育重点实验室,中国林业科学研究院林业研究所,北京 100091;2 南京林业大学南方现代林业协同创新中心,南京 210037)
摘要:该研究克隆鉴定了旱柳和龙爪柳β微管蛋白基因,并对其进行了序列相似性、系统发育、染色体定位以及表达模式的分析。结果显示,2种柳树β微管蛋白基因家族各有20个成员,家族内部成员间核酸和氨基酸序列相似性分别在74.0%和86.6%以上,种间同源蛋白氨基酸序列相似性在85.8%以上,柳树与其它植物β微管蛋白间的氨基酸序列相似性在81.5%以上。系统发育分析显示,柳树β微管蛋白家族被分为4个亚组,结合杨树β微管蛋白基因染色体定位,推测柳树β微管蛋白基因家族经历了杨柳科全基因组重复事件和串联重复事件,而柳树TUB11和TUB12可能来源于区段重复或者转座。基因表达模式分析发现,该家族成员的表达具有一定的组织特异性,并且部分重复基因对在所检测组织中表达差异较大。柳树β微管蛋白基因家族成员序列的高度相似性、成员数量的进化扩张、以及表达模式的多样性可能赋予了细胞分裂与生长更高的灵活性,这对多年生木本植物的生长发育习性意义重大。
关键词:柳树;β微管蛋白;系统发育;序列相似性;表达模式
微管(MT)是由α微管蛋白(TUA)和β微管蛋白(TUB)构成的异质二聚体,它是真核生物细胞骨架的重要组成部分,在细胞有丝分裂[1]和减数分裂[2]中具有重要作用,植物微管骨架参与冷信号的传导[3],参与细胞的形态建成[4],并在植物细胞壁形成过程中引导纤维素微纤维的沉积[5]。因此,研究微管蛋白可能会有助于揭示植物的形态建成或植物外部形态形成的分子机制。
早期发现的动物、植物、原生生物和真菌中的TUA和TUB蛋白氨基酸序列相似性高达88%以上[6-12]。到目前为止,很多植物的微管蛋白基因已被克隆或鉴定,如拟南芥的6个TUA[13]和9个TUB基因[14],毛果杨(PopulustrichocarpaTorr. & Gray)的8个TUA和20个TUB基因[15],美洲山杨(PopulustremuloidesMichx.)的8个TUA和20个TUB基因[15],玉米(ZeamaysL.)的6个可编码TUA基因[16]和8个有功能的TUB基因[17],水稻(OryzasativaL.)中有4个TUA基因[18]和8个TUB基因[19]等。
本研究以中国特有的旱柳(SalixmatsudanaKoidz.)及其变种龙爪柳[S.matsudanavar.tortusoa(Vilm.)Rehd.][20]为材料,克隆了2种柳树的TUB基因,并对克隆的基因进行了组织特异性表达分析,进行了核酸和氨基酸序列相似性和系统发育分析以及染色体定位,为木本植物微管蛋白的研究提供了一定的理论基础。
1材料和方法
1.1材料
基因克隆以旱柳与龙爪柳水培苗叶片作为RNA提取材料;实时定量PCR实验所用的一年生枝条材料于成年大树上采集,成树生长于北京植物园。
簸箕柳(S.suchowensisW. C. Cheng ex G. Zhu)TUB基因通过本地Blast获得,其基因组数据下载于http://115.29.234.170/willow/;毛果杨TUB基因下载于毛果杨全基因组数据库(https://phytozome.jgi.doe.gov /pz/portal.html);拟南芥TUB基因下载于拟南芥全基因组数据库(http://www.arabidopsis.org/);水稻、玉米、高粱(SorghumbicolorL.)、二穗短柄草(Brachypodiumdistachyum(L.) Beauv.)和狗尾草(Setariaviridis(L.) Beauv.)等植物TUB基因通过Blast搜索Genbank数据库获得。
1.2方法
1.2.1柳树TUB基因的克隆以毛果杨β微管蛋白氨基酸序列为探针Blast搜索Genbank数据库,筛选获得20个钻天柳(Choseniaarbutifolia(Pall.) A. Skv.)TUB基因。由于TUB基因在各物种间的保守性很高,旱柳、龙爪柳与钻天柳又同属于杨柳科柳属植物,因此本研究以钻天柳TUB基因为模板,利用NCBI的在线引物设计工具(http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome)设计引物,克隆旱柳与龙爪柳TUB基因。利用EASYspin植物RNA快速提取试剂盒(北京艾德莱生物科技有限公司)和SuperRT cDNA第一链合成试剂盒(北京康为世纪生物科技有限公司)进行旱柳与龙爪柳总RNA提取和cDNA第一链合成,PCR克隆采用2×Es Taq MasterMix(北京康为世纪生物科技有限公司)进行。所用引物如表1所示。克隆所获得的CDNA片段由北京中科希林生物科技有限责任公司测序。
1.2.2TUB基因家族序列比对利用Bioedit软件将克隆获得的旱柳和龙爪柳TUB基因编码区(CDS)翻译成氨基酸序列,与钻天柳、簸箕柳、毛果杨、拟南芥、水稻、玉米、高粱、二穗短柄草和狗尾草等TUB基因进行物种间和物种内部核酸和氨基酸序列比对,分析TUB基因家族在同一物种不同成员间的序列相似性,以及在不同物种中的保守性。
1.2.3TUB蛋白的系统发育分析利用ClustalW将柳树与其它植物的TUB氨基酸序列进行序列比对,使用Mega 5.0软件构建系统发育树,选用邻接法(Neighbor-Joining, NJ),分支可信度检测自展值(bootstrap)为1 000次重复,氨基酸替换模型选用JTT(Jones-Taylor-Thornton)模型[21],缺失位点处理方法为完全删除。
1.2.4TUB基因的染色体定位利用基因家族全基因组定位工具WGMapping(http://bioinformatics.psb.ugent.be/plaza/versions/plaza_v3_dicots/genome_mapping/index)分析TUB基因在其染色体上的定位。
1.2.5柳树TUB基因家族的实时定量PCR将旱柳(Sm)与龙爪柳(Smt)一年生枝条分基部(S1)、中段(S2)、上部伸长区(S3)和茎尖(S4)4个部位分别提取RNA,利用SYBR®Fast qPCR Mix试剂盒(Takara)进行实时定量PCR,分析TUB基因在2种柳树一年生枝条不同部位的表达模式。

表1 柳树β微管蛋白基因引物
2结果与分析
2.1TUB基因家族序列相似性
克隆获得旱柳和龙爪柳TUB基因各20个,家族成员开放读码框(ORF)长度范围为1 335~1 353 bp,核酸序列相似性分别为74.1%~99.5%和74.0%~99.9%。预测的蛋白质氨基酸序列长度为445~451。旱柳中SmTUB2和SmTUB11基因编码的氨基酸序列相同,SmTUB7和SmTUB12编码的氨基酸序列相同,其它成员间的氨基酸序列相似性为86.6%~98.8%。龙爪柳中SmtTUB7和SmtTUB12基因编码的氨基酸序列相同,其它成员间的氨基酸序列相似性为87.0%~99.5%。虽然柳树TUB蛋白保守性很高,但其C末端也像其它植物一样是多变的。旱柳SmTUB4、SmTUB6、SmTUB10、SmTUB14-15、SmTUB17、SmTUB19-20与龙爪柳直系同源蛋白的氨基酸序列完全一致,其它直系同源蛋白的氨基酸序列相似性为98.0%~99.7%;旁系同源蛋白相似性为85.8%~99.7%。
将柳树TUB与其它被子植物β微管蛋白进行氨基酸序列比对,发现TUB在被子植物中非常保守。其中,旱柳SmTUB4、SmTUB14和SmTUB19与钻天柳直系同源蛋白的氨基酸序列完全一致;SmTUB14与毛果杨直系同源蛋白的氨基酸序列完全一致;而簸箕柳的TUB家族成员在搜索过程中只获得了12条完整序列,没有发现与旱柳完全一致的TUB同源蛋白。旱柳与上述3种杨柳科植物的其它TUB直系同源蛋白间的序列相似性则依次为95.9%~99.7%、91.5%~99.7%和90.5%~99.7%,旁系同源蛋白的序列相似性依次为85.8%~99.3%、85.6%~99.1%和81.5%~98.8%。相比之下,旱柳与其它被子植物如拟南芥、水稻、玉米、高粱、二穗短柄草和狗尾草中TUB同源蛋白的氨基酸序列相似性稍低,依次为86.0%~97.1%、86.8%~97.7%、86.6%~97.3%、87.0%~97.3%、88.8%~96.8%和88.8%~96.6%。
由于和旱柳亲缘关系非常近,龙爪柳与其它植物TUB同源蛋白间的序列相似性与旱柳中情况基本相同。它与钻天柳间完全一致的直系同源蛋白同样为TUB4、TUB14和TUB19;与毛果杨除TUB14外还有TUB18也完全一致;与簸箕柳间同样没有发现一致的TUB直系同源蛋白。龙爪柳与以上3种植物的其它TUB直系同源蛋白间的序列相似性依次为95.9%~99.7%、90.9%~99.5%和90.5%~99.7%,旁系同源蛋白间的序列相似性依次为86.0%~99.3%、85.8%~99.3%和81.8%~98.8%。同旱柳一样,龙爪柳与拟南芥、水稻、玉米、高粱、二穗短柄草和狗尾草中TUB同源蛋白的氨基酸序列相似性也低于杨柳科植物,序列相似性数据依次为86.2%~97.3%、87.0%~97.7%、86.8%~97.3%、87.2%~97.5%、89.0%~96.8%和 89.0%~96.6%。
2.2TUB蛋白系统发育分析
TUB蛋白家族在进化过程中经历了显著的扩张,其中包括双子叶植物全基因组复制事件、杨柳科植物全基因组复制事件以及串联重复,最终使得杨柳科的4个物种旱柳、龙爪柳、钻天柳和毛果杨TUB基因进化为现在的各20个。
系统发育分析结果显示,TUB蛋白家族可分为四大类(图1),这与Oakley等[15]的研究一致。第一类包括Class I与Class I-like,其成员最多,占全部分析序列的41.13%(51/124)。这一分支中包含SmTUB、SmtTUB与CaTUB各8个,约占各自成员总数的一半,而PtTUB则正好有一半聚类在此分支中。由于拓扑结构不紧密,3种柳树各有2个TUB蛋白(SmTUB9~10、SmtTUB9~10与CaTUB9~10)被划分在Class I-like中,毛果杨中有4个TUB蛋白(PtTUB9~12)归于此类。此外,Class I中包含有2个主要由单子叶植物TUB蛋白组成的小分支,其中包括12个单子叶TUB和1个拟南芥TUB(AtTUB6)。
第二分支Class Ⅱ成员数仅次于Class I,所占比例为33.87%(42/124)。其中大部分为双子叶植物TUB蛋白,包括6个旱柳(SmTUB1、SmTUB2/11、SmTUB3~6)、7个龙爪柳(SmtTUB1~6和SmtTUB11)、7个钻天柳(CaTUB1~6和CaTUB11)、6个杨树(PtTUB1~6)、5个簸箕柳(SsTUB1和SsTUB3~6)与3个拟南芥TUB(AtTUB2、AtTUB7 和AtTUB8);单子叶植物TUB只有玉米(ZmTUB1)、高粱(SbTUB2)、水稻(OsTUB2)和二穗短柄草(BdTUB2)各1个,4个单子叶植物TUB独成一支。
第三和第四分支成员较少,分别为16和19个,拓扑结构也比较简单,其中单、双子叶植物TUB都是分开聚类,且Class Ⅲ与Class Ⅳ中各有2个拟南芥TUB自成一支,分别为AtTUB1、AtTUB5和AtTUB4、AtTUB9。Class Ⅲ中包括10个杨柳科TUB,旱柳、龙爪柳、钻天柳、簸箕柳和毛果杨蛋白各2个,分别是SmTUB19~20、SmtTUB19~20、CaTUB19~20、SsTUB19~20和PtTUB19~20,这些蛋白的一个共同特点是都在其序列第39位上比其它TUB蛋白多了3个氨基酸残基,而其它蛋白在39位及其附近也是比较多变的。本分支的4个单子叶植物TUB分别为OsTUB8、SbTUB6、ZmTUB3和ZmTUB4。Class Ⅳ中包括2个旱柳TUB(SmTUB7/12和SmTUB8)、2个龙爪柳TUB(SmtTUB7/12和SmtTUB8)、2个钻天柳TUB(CaTUB7/12和CaSmTUB8)、2个毛果杨TUB(PtTUB7和PtTUB8)和1个簸箕柳TUB(SsTUB8),共9个杨柳科TUB蛋白。另外还包括3个水稻、3个高粱、1个玉米和1个二穗短柄草共8个单子叶植物TUB蛋白。
2.3TUB基因家族染色体定位分析
TUB基因染色体定位显示,20个PtTUB基因分布在超过半数的染色体上(10/19)。其中1号染色体上分布的TUB基因最多(6个);其次是9号染色体,分布有3个;3、6和16号染色体各分布有2个,而其余染色体(2、5、11、12和15号)上各有1个。毛果杨全基因组测序[22]表明杨树至少经历了三次全基因组重复事件,以及后来的多区段重复、串联重复和转座。对比最近的一次杨柳科全基因组复制事件后杨树染色体连锁图谱,发现20个杨树TUB基因都经历了本次复制事件,并且定位在重复区段的2个基因都被保留下来(图2)。其中,TUB3和TUB9、TUB4和TUB10、TUB13和TUB15以及TUB14和TUB16等4对基因还发生了串联重复,然而它们的进化历史并不相同。因为,系统发育分析显示前2对串联重复基因中TUB3和TUB4的关系较近,同样TUB9和TUB10也在同一个分支上,据此推测它们是先经历了串联重复事件,由一个祖先基因进化成为TUB3/4和TUB9/10,然后发生了杨柳科基因组重复事件使得2个基因加倍至4个:TUB3、TUB4、TUB9和TUB10。相反,后2对基因则是TUB13和TUB15进化关系较近,而TUB14和TUB16在同一分支,因此可以推测它们先由一个祖先基因经过基因组重复事件进化成TUB13/15和TUB14/16等2个基因,随后又发生串联重复造成基因加倍,形成4个基因。

标尺表示JTT模型下估算的单位遗传距离;SmTUB1~ SmTUB20,本研究克隆的20个旱柳TUB蛋白;SmtTUB1~ SmtTUB20,本研究克隆的20个龙爪柳TUB蛋白;SsTUB1~SsTUB6、SsTUB8~SsTUB10、SsTUB14和SsTUB8~SsTUB10,本地blast获得的14个簸箕柳TUB蛋白;CaTUB1~CaTUB20,20个钻天柳TUB蛋白;PtTUB1~PtTUB20,20个毛果杨TUB蛋白;AtTUB1~AtTUB9,9个拟南芥TUB蛋白;OsTUB1~ OsTUB8,8个水稻TUB蛋白;ZmTUB1~ ZmTUB7,7个玉米TUB蛋白;SbTUB1~ SbTUB6,6个高粱TUB蛋白;BdTUB1~ BdTUB5,5个二穗短柄草TUB蛋白;SvTUB1~SvTUB2,2个狗尾草TUB蛋白图1 植物TUB蛋白系统发育分析The bar means unit genetic distance estimated by JTT model; SmTUB1~ SmTUB20, 20 S. matsudana TUB proteins cloned in this study; SmtTUB1~ SmtTUB20, 20 S. matsudana var. tortusoa TUB proteins cloned in this study; SsTUB1~SsTUB6, SsTUB8~SsTUB10, SsTUB14 and SsTUB8~SsTUB10, 14 S.suchowensis TUB proteins obtained by local blast; CaTUB1~CaTUB20, 20 C. arbutifolia TUB proteins; PtTUB1~PtTUB20, 20 P. trichocarpa TUB proteins; AtTUB1~AtTUB9, nine A. thaliana TUB proteins; OsTUB1~ OsTUB8, eight O. sativa TUB proteins; ZmTUB1~ ZmTUB7, seven Z. mays TUB proteins; SbTUB1~ SbTUB6, six S. bicolor TUB proteins; BdTUB1~ BdTUB5, five B. distachyon TUB proteins; SvTUB1~SvTUB2, two S. viridis TUB proteinsFig. 1 Phylogenetic analysis of plant TUB proteins
共线性分析表明柳树与杨树染色体结构高度相似[23],据此推测柳树TUB基因家族同样经历了三次全基因组重复事件和串联重复事件,导致其家族成员剧增到20个。然而,柳树TUB11和TUB12基因(图2)的进化历史与杨树不同,杨树PtTUB11和PtTUB12共同聚类在Class I-like中,而柳树TUB11却与TUB1和TUB2共同聚类在Class Ⅱ中,TUB12与TUB7氨基酸序列相同,并与TUB8聚类在Class Ⅳ中。因此,与杨树不同,柳树TUB11和TUB12不是来源于同一个祖先基因,它们可能是由TUB1/TUB2和TUB7分别通过区段重复或转座产生。
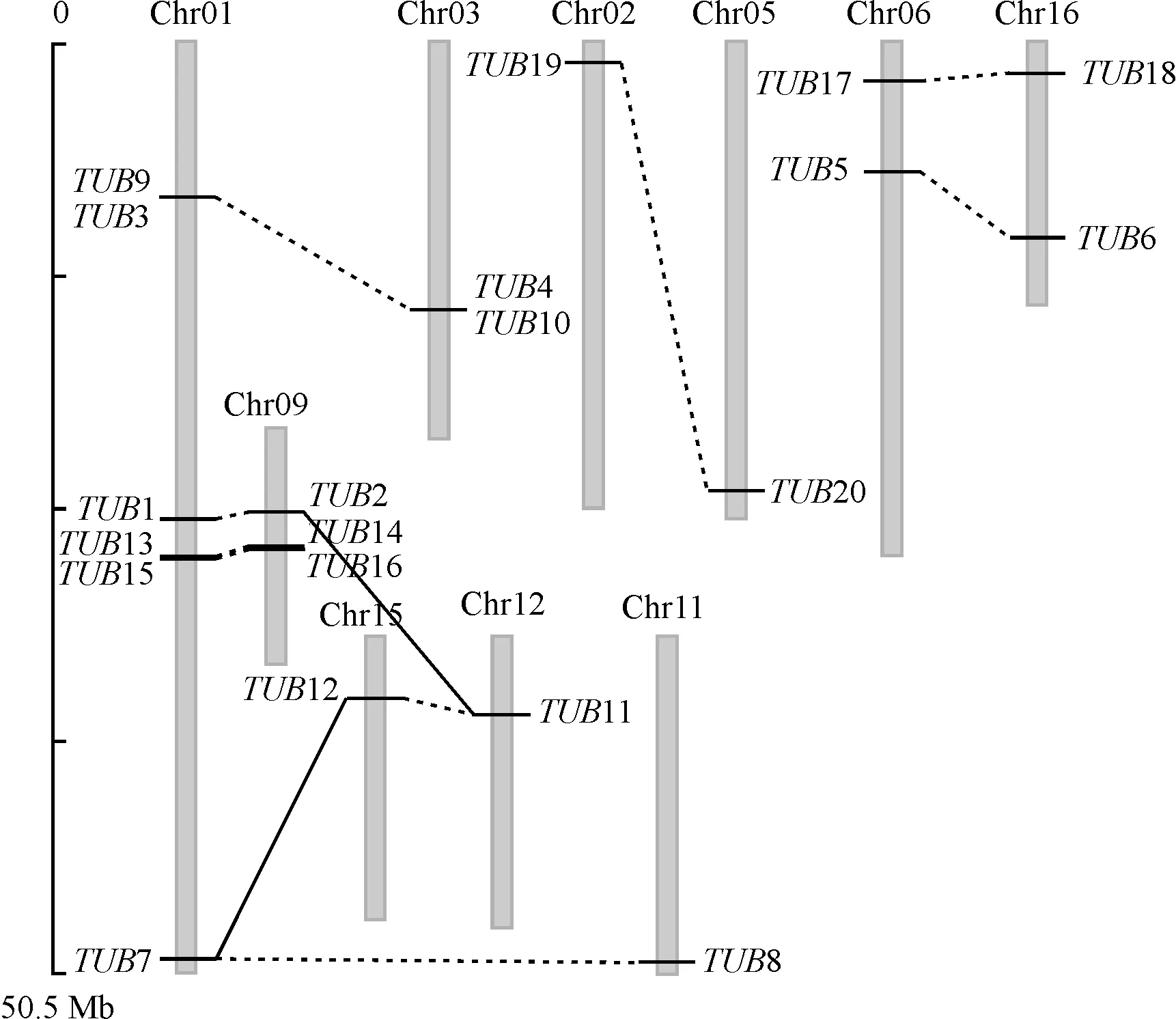
虚线连接的是TUBs复制基因,实线连接的是柳树TUB11和TUB12以及它们可能的重复基因。图2 TUB基因染色体定位与基因重复事件The duplicated paralogous pairs of TUBs are connected with dotted lines. TUB11 and TUB12 from Salix are connected with their potential duplicated paralogous with solid linesFig. 2 Chromosomal location and gene duplication events of TUB genes
2.4柳树TUB基因表达模式
近半数(9个)柳树TUB基因在所检测组织中不表达,其中大部分是重复基因。Class I中只检测到TUB13和TUB18在旱柳和龙爪柳枝条基部(S1)、中部(S2)、伸长区(S3)和顶端(S4)4个组织中表达(图3),且相对表达水平都很低(最高相对表达量为0.007 121)。在旱柳中,TUB13和TUB18在枝条的顶端表达水平最高,基部次之,中部和伸长区较低;而在龙爪柳中,SmtTUB13在枝条伸长区表达水平最高,其次是顶端分生区、基部和中部,SmtTUB18枝条顶端和伸长区表达水平都比较高,其次是基部和中部。作为TUB13的重复基因,TUB14、TUB15和TUB16均不在上述组织中表达;TUB17作为TUB18的重复基因也不表达。Class I-like中的2个基因TUB9和TUB10也不在上述组织中表达,而它们的重复基因TUB3和TUB4(Class Ⅱ)在上述4个组织中都有表达。
Class Ⅱ中的TUB11是上述4个柳树组织中的主要表达基因(图3),相对表达水平均在0.25以上,在旱柳枝条伸长区的表达水平最高(相对表达量为9.05),而在龙爪柳相应组织中的表达水平明显低于旱柳(相对表达量为1.80),该基因在2种柳树的顶端中表达水平也比较高且表达水平相当,而在枝条中部和基部表达水平较低。TUB1和TUB2作为TUB11的重复基因是除TUB11外表达量较高的基因,TUB3和TUB4次之。而旱柳TUB1~4在的枝条中部和顶端表达水平均高于龙爪柳,伸长区表达水平与龙爪柳相当;TUB2在龙爪柳枝条基部表达水平高于旱柳。TUB5和TUB6在两种柳树的上述4个组织中均未表达。

Sm. 旱柳;Smt. 龙爪柳;S1. 枝条基部;S2. 枝条中部;S3. 枝条伸长区;S4. 枝条顶端图3 柳树TUB基因表达模式Sm. S. matsudana; Smt. S. matsudana var. tortusoa; S1. Basal region of branch; S2. Middle region of branch; S3. Elongation region of branch; S4. Apical region of branchFig. 3 The expression profile of TUB in willows
Class Ⅲ中只有TUB19表达(图3),该基因在2种柳树的枝条顶端表达水平最高,在龙爪柳枝条伸长区表达水平也比较高,在其它组织中表达水平较低。未检测到其重复基因TUB20在上述组织中表达。Class Ⅳ中的3个基因(由于TUB7和TUB12核酸序列相似性非常高,导致无合适引物可以将2个基因的表达水平分开测定)在上述组织中均有表达,其中TUB8在旱柳枝条伸长区的表达水平明显高于旱柳和龙爪柳其它枝条组织。TUB7/12在旱柳枝条基部、中部、伸长区和顶端分生区的表达水平均高于龙爪柳的相应组织,其中以在旱柳枝条伸长区的表达水平最高,中部、顶端分生区和基部依次次之;该基因在龙爪柳枝条各组织中表达水平高低次序依次为伸长区、中部、基部和顶端分生区。
3讨论
通过克隆获得旱柳和龙爪柳TUB基因各20个,与毛果杨和美洲山杨的TUB基因家族[15]成员数相等,且其家族内部成员间核酸和氨基酸序列相似性也与杨树相当,甚至高于杨树,在分子水平一定程度上说明旱柳和龙爪柳亲缘关系很近,印证了中国植物志中龙爪柳是旱柳的变种一说[20]。旱柳和龙爪柳TUB蛋白家族成员的氨基酸序列高度保守,其C末端与其它植物一样也是多变的,有研究表明TUB蛋白C末端调节微管的组装[24],决定微管多聚体的聚合效率[25],可能构成马达蛋白的结合表面[26],并且还是很多微管结合蛋白的互作区域[27-33]。因此,可以推测柳树TUB蛋白的C末端也在其微管的组装,及与微管结合蛋白的互作中起重要作用。
系统发育和染色体定位分析显示,柳树TUB基因家族同杨树[22]一样经历了3次全基因组重复事件,以及后来的区段重复、串联重复和转座等基因重复事件。柳树与杨树TUB11和TUB12在进化上的区别暗示,在杨柳科植物基因组重复事件发生后杨属和柳属植物各自具有不同的进化历史。另外,由目前基因家族成员的数量判断,杨柳科植物TUB基因家族的进化扩张比草本植物更明显。比如,拟南芥中只有9个TUB基因[14]、玉米中目前只鉴定出8个TUB基因[17]、水稻中也只有8个TUB基因[19]等,而杨树和柳树都有20个TUB基因。这种基因扩张的不对等可能对木本植物多年生的生长习性意义重大。
柳树与杨树TUB基因的表达模式差异较大,杨树Class I和Class I-like亚组中的TUB9、TUB13、TUB15和TUB16是木质部中的主要表达基因[15],而柳树一年生枝条4个组织中未检测到TUB9、TUB15和TUB16的表达,且TUB13的表达水平也很低。相比之下,Class Ⅱ中的TUB11及其重复基因TUB1和TUB2在柳树中的表达量最高,因此柳树TUB11、TUB1和TUB2极有可能代替了杨树TUB9、TUB13、TUB15和TUB16的功能,调节柳树木质部的生长发育。但是,柳树TUB12并没有像TUB11一样在柳树一年生枝条中高表达,暗示重复事件产生的新基因进化命运是不同的。另外,TUB13的重复基因TUB14、TUB15和TUB16,TUB18的重复基因TUB17,TUB3和TUB4的重复基因TUB9和TUB10,以及TUB19的重复基因TUB20等基因在所检测组织中不表达,说明重复基因在进化过程中发生了功能缺失或功能分歧。
参考文献:
[1]RASMUSSEN C G, WRIGHT A J, M LLER S. The role of the cytoskeleton and associated proteins in determination of the plant cell division plane[J].ThePlantJournal, 2013, 75(2): 258-269.
[2]张平冬,康向阳。胡杨小孢子发生及微管骨架变化与异常研究[J]. 西北植物学报, 2013, 33(11): 2 166-2 171.
ZHANG D P, KANG X Y. Organization of microtubule and its abnormities during microsporogenesis inPopuluseuphratica[J].ActaBotanicaBoreali-OccidentaliaSinica, 2013, 33(11): 2 166-2 171.
[3]李永才,安黎哲,毕阳。微管骨架在植物适应低温胁迫中的功能研究进展[J]. 西北植物学报, 2006, 26(7): 1 500-1 504.
LI Y C, AN L Z, BI Y. Research advances about the role of microtubule cytoskeleton in plant acclimation to low-temperature stress[J].ActaBotanicaBoreali-OccidentaliaSinica, 2006, 26(7): 1 500-1 504.
[4]LEDBETTER M C, PORTER K R. A “microtubule” in plant cell fine structure[J].TheJournalofCellBiology, 1963, 19(1): 239-250.
[5]PAREDEZ A R, SOMERVILLE C R and EHRHARDT D W. Visualization of cellulose synthase demonstrates functional association with microtubules[J].Science, 2006, 312(5 779): 1 491-1 495.
[6]MAGES W, SALBAUM J M, HARPER J F,etal. Organization and structure ofVolvoxα-tubulin genes[J].MolecularandGeneralGenetics, 1988, 213(2/3): 449-458.
[7]HARPER J F, MAGES W. Organization and structure of Volvox β-tubulin genes[J].MolecularandGeneralGenetics, 1988, 213(2/3): 315-324.
[8]ROHEL E A, PAYNE A C, HALL L,etal. Isolation and characterization of α-tubulin genes fromSeptoriatriticiandRhynchosporiumsecalis, and comparative analysis of fungal α-tubulin sequences[J].CellMotilityandTheCytoskeleton, 1998, 41(3): 247-253.
[9]LIAUD M-F, BRINKMANN H, CERFF R. The β-tubulin gene family of pea: primary structures, genomic organization and intron-dependent evolution of genes[J].PlantMolecularBiology, 1992, 18(4): 639-651.
[10]PARKER S K, DETRICH H W. Evolution, organization, and expression of α-Tubulin genes in the antarctic FishNototheniacoriicepsadaptive expansion of a gene family by recent gene duplication, inversion, and divergence[J].JournalofBiologicalChemistry, 1998, 273(51): 34 358-34 369.
[11]SPITHILL T W, SAMARAS N. Genomic organization, chromosomal location and transcription of dispersed and repeated tubulin genes inLeishmaniamajor[J].MolecularandBiochemicalParasitology, 1987, 24(1): 23-37.
[12]ALEXANDRAKI D, RUDERMAN J V. Sequence heterogeneity, multiplicity, and genomic organization of alpha-and beta-tubulin genes in sea urchins[J].MolecularandCellularBiology, 1981, 1(12): 1 125-1 137.
[13]KOPCZAK S D, HAAS N A, HUSSEY P J,etal. The small genome ofArabidopsiscontains at least six expressed alpha-tubulin genes[J].ThePlantCellOnline, 1992, 4(5): 539-547.
[14]SNUSTAD D P, HAAS N A, KOPCZAK S D,etal. The small genome ofArabidopsiscontains at least nine expressed beta-tubulin genes[J].ThePlantCell, 1992, 4(5): 549-556.
[15]OAKLEY R V, WANG Y-S, RAMAKRISHNA W,etal. Differential expansion and expression of alpha- and beta-tubulin gene families inPopulus[J].PlantPhysiology, 2007, 145(3): 961-973.
[16]VILLEMUR R, JOYCE C M, HAAS N A,etal. α-Tubulin gene family of maize (ZeamaysL.): evidence for two ancient α-tubulin genes in plants[J].JournalofMolecularBiology, 1992, 227(1): 81-96.
[17]VILLEMUR R, HAAS N A, JOYCE C M,etal. Characterization of four new β-tubulin genes and their expression during male flower development in maize (ZeamaysL.)[J].PlantMolecularBiology, 1994, 24(2): 295-315.
[18]JEON J S, LEE S, JUNG K H,etal. Tissue-preferential expression of a rice α-tubulin gene,OsTubA1, mediated by the first intron[J].PlantPhysiology, 2000, 123(3): 1 005-1 014.
[19]YOSHIKAWA M, YANG G, KAWAGUCHI K,etal. Expression analyses of β-tubulin isotype genes in rice[J].PlantandCellPhysiology, 2003, 44(11): 1 202-1 207.
[20]王战,方振富。中国植物志[M]. 北京: 科学出版社, 1984: 133-134.
[21]JONES D T, TAYLOR W R, THORNTON J M. The rapid generation of mutation data matrices from protein sequences[J].ComputerApplicationsintheBiosciences, 1992, 8(3): 275-282.
[22]TUSKAN G A, DIFAZIO S, JANSSON S,etal. The genome of black cottonwood,Populustrichocarpa(Torr. & Gray)[J].Science, 2006, 313(5 793): 1 596-1 604.
[23]DAI X, HU Q, CAI Q,etal. The willow genome and divergent evolution from poplar after the common genome duplication[J].CellResearch, 2014, 24(10): 1 274-1 277.
[24]SERRANO L, TORRE J D L, MACCIONI R B,etal. Involvement of the carboxyl-terminal domain of tubulin in the regulation of its assembly[J].ProceedingsoftheNationalAcademyofSciences, 1984, 81(19): 5 989-5 993.
[25]SACKETT D L, BHATTACHARYYA B, WOLFF J. Tubulin subunit carboxyl termini determine polymerization efficiency[J].JournalofBiologicalChemistry, 1985, 260(260): 43-45.
[26]NOGALES E, WOLF S G, DOWNING K H. Structure of the alpha beta tubulin dimer by electron crystallography[J].Nature, 1998, 391(6 663): 89-106.
[27]CIFERRI C, PASQUALATO S, SCREPANTI E,etal. Implications for kinetochore-microtubule attachment from the structure of an engineered Ndc80 complex[J].Cell, 2008, 133(3): 427-439.
[28]WESTERMANN S, AVILA-SAKAR A, WANG H W,etal. Formation of a dynamic kinetochore-microtubule interface through assembly of the Dam1 ring complex[J].MolecularCell, 2005, 17(2): 277-290.
[29]MICHAEL N, SUSANNE B, MICHAL W,etal. Microtubules accelerate the kinase activity of Aurora-B by a reduction in dimensionality[J].PlosOne, 2014, 9(2): e86786.
[30]MARYA P K, SYED Z, FRAYLICH P E,etal. Kinesin and tau bind to distinct sites on microtubules[J].JournalofCellScience, 1994, 107(1): 339-344.
[31]BROUHARD G J, STEAR J H, NOETZEL T L,etal. XMAP215 is a processive microtubule polymerase[J].Cell, 2008, 132(1): 79-88.
[32]MARIJA Z, STEAR J H, HYMAN A A. EB1 recognizes the nucleotide state of tubulin in the microtubule lattice[J].PlosOne, 2009, 4(10): e7585.
[33]GUPTA K K, JOYCE M V, SLABBEKOORN A R,etal. Probing Interactions between CLIP-170, EB1, and Microtubules[J].JournalofMolecularBiology, 2010, 395(5): 1 049-1 062.
(编辑:宋亚珍)
Cloning and Sequence Analysis of β-Tubulin Gene Families in Willows
SUI Jinkai1, RAO Guodong1, 2, ZHANG Jianguo1, 2 *
(1 Research Institute of Forestry, Chinese Academy of Forestry, Key Laboratory of Tree Breeding and Cultivation, State Forestry Administration, Beijing 100091, China; 2 Collaborative Innovation Center of Sustainable Forestry in Southern China, Nanjing Forestry University, Nanjing 210037, China)
Abstract:In this study, β-tubulin genes of Salix matsudana Koidz. and S. matsudana var. tortusoa (Vilm.)Rehd. were cloned and identified. The sequence similarity, phylogeny, chromosomal localization and expression patterns were further analyzed. The result showed that each willow contained 20 β-tubulin genes, and each gene shared more than 74.0% and 86.6% nucleotide and amino acid sequence similarity with one another in each willow species. There was over 85.8% amino acid sequence similarity of the β-tubulin between the two willows. While the amino acid sequence similarity among Salix and other plants were larger than 81.5%. In the phylogenetic analysis, β-tubulin proteins formed four classes, and it was presumed that Salix β-tubulin gene families had undergone salicoid duplication and tandem duplication events combined with the study of chromosomal location of poplar β-tubulin. However, TUB11 and TUB12 were possibly derived from segmental duplication or replicative transposition. The expression pattern analysis showed a tissue specificity of β-tubulin genes in willows, and a section of duplicated-gene pairs showed different expression patterns in tested tissues. High degree of sequence similarity, evolutionary expansion and of members and expression pattern diversity of Salix β-tubulin gene families might confer flexibility in cell division and growth which is of important significance to the development and growth habit of perennial woody plants.
Key words:willow; β-tubulin; phylogenetic; sequence similarity; expression pattern
文章编号:1000-4025(2016)05-0902-08
doi:10.7606/j.issn.1000-4025.2016.05.0902
收稿日期:2015-10-12;修改稿收到日期:2016-04-25
基金项目:国家自然科学基金青年项目(31400569)
作者简介:睢金凯(1985-),男,博士,主要从事人工林定向培育研究。E-mail: suijinkai@163.com *通信作者:张建国,博士,研究员,博士生导师,主要从事森林培育研究。E-mail: rgd@caf.ac.cn
中图分类号:Q785;Q786
文献标志码:A

