除草剂草甘膦微生物降解技术研究进展
王军华+王易芬+陈蕾蕾+周庆新+杜方岭
摘要:草甘膦是目前世界上使用量最大的除草剂品种之一,造成日益严重的环境污染和生态危害,并随着食物链在哺乳动物体内富集,进而危害人类健康;微生物降解是清除环境中草甘膦的最主要途径,是降低草甘膦危害的一种有效方法。对近几年筛选得到的草甘膦高效降解菌、主要降解途径和降解产物、草甘膦的原位修复研究进行了总结,探讨了微生物降解用于缓解草甘膦造成的环境污染的难题。
关键词:草甘膦;微生物降解;降解途径;原位修复
中图分类号: S482.4;S182
文献标志码: A
文章编号:1002-1302(2016)04-0008-04
草甘膦(glyphosate)是一种高效除草剂,被植物经茎叶吸收后,通过抑制植株5-烯醇丙酮莽草酸-3-磷酸合成酶(5-enolpyruvylshikimate-3-phosphate synthase,EPSPS)的活性来干扰5-烯醇式莽草酸-3-磷酸的合成,从而抑制植株合成芳香性氨基酸,最终导致植物叶片枯黄脱落,死亡[1-2],可控制世界上绝大多数危害性杂草,成为全世界除草剂使用量最大的种类之一[3]。
草甘膦由Monsanto公司开发应用以来,一直被认为对非靶标生物具有低毒性。对草甘膦及其制剂的研究显示,草甘膦未在任何动物的组织中富集,也未对人类健康产生危害[3]。然而40多年的应用,尤其是1983年高抗草甘膦基因研发后[4],抗草甘膦转基因作物被广泛种植,引起的草甘膦大规模应用,导致其在土壤中大量积累,已经对土壤化学过程和生态系统造成影响,而且给环境带来安全风险,威胁了人类健康[5]。调查研究发现,土壤中残留的草甘膦会破坏土壤微生态,影响微生物数量和土壤酶的活性,尤其会抑制放线菌数量和脲酶的活性[6-7],而且残留的草甘膦会随地表径流进入地表水,影响水质,造成水生生态的失衡[8-9]。不仅如此,更有研究发现,草甘膦会影响孕期动物的胚胎发育,导致形态异变[10-12]。例如,干扰海胆胚胎细胞的细胞周期[10],增加非洲爪蛙的胚胎和鸡胚变异的可能性[11]等。同时,Clair等对小鼠研究发现,高剂量的草甘膦(约10 000 mg/kg)对小鼠睾丸的支持细胞有急性细胞毒性,诱导细胞坏死和凋亡;而低剂量的草甘膦(1 mg/kg,相当于摄食草甘膦处理的玉米后,人尿液中草甘膦的含量),虽然没有细胞毒性,但是会使睾丸酮的分泌降低35%,干扰睾丸的内分泌[12]。Poulsen等还发现,草甘膦能够穿透人类胎盘障碍进入胎儿室,影响胎儿正常发育[13]。由此可见,土壤中富集的草甘膦不仅对环境造成影响,破坏生态平衡,并能通过食物链的富集进入哺乳动物体内,进而威胁人类健康,草甘膦的安全问题应引起人们的足够重视。
1 草甘膦的生物降解菌
土壤中的草甘膦易发生水解、光化学降解、生物降解等,而生物降解是草甘膦降解的最主要途径[14-15]。在自然环境中,尤其是长期施用草甘膦的土壤中,存在种类繁多的能耐受或降解草甘膦的微生物菌群[16],这有利于寻找高效降解菌,对缓解草甘膦造成的环境危害具有重要意义。
在纯培养条件下,科学家们一直没能分离出可利用草甘膦的细菌,直至1983年,Moore等筛选出假单胞菌PG2982,是能利用草甘膦作为唯一磷源的细菌[17]。同位素标记法研究发现,通过C—P键的断裂,PG2982能将草甘膦代谢成肌氨酸[18],虽然PG2982降解草甘膦延滞期较长,但是为草甘膦降解菌的筛选奠定了基础。此后,陆续分离得到有草甘膦降解活性的假单胞菌[19-23]、产碱杆菌[23]、黄杆菌[24]、节杆菌[25-26]和根瘤菌[27],而Krzysko等则发现土壤中的真菌也具有降解草甘膦的能力[28-29]。早期研究的降解菌多是以草甘膦为唯一磷源,随着研究的深入,发现许多微生物也可以利用草甘膦作为碳氮源,节杆菌GLP-1的变种[26]、产黄青霉菌[30]被发现能利用草甘膦作为唯一氮源,链霉菌[31]则是将草甘膦作为唯一碳氮源,而青霉T1[32]和曲霉B21[33]对草甘膦也都表现出明显降解特性,降解率都在50%以上,B21对草甘膦的降解率甚至可达97%。
近年来,草甘膦的研究也取得许多进展,Sviridov等分别从甲基膦酸和草甘膦污染的土壤中分离出无色杆菌MPS12和苍白杆菌GPK3,将MPS12在含草甘膦的培养基中培养,MPS12A同样具有降解草甘膦的能力,这2株菌都是以草甘膦为唯一磷源进行生长,将草甘膦分别降解成肌氨酸和AMPA[34]。Hadi等在无膦培养基中也筛选出苍白杆菌GDOS,能将草甘膦降解为AMPA,GDOS在含磷培养基中对草甘膦还有降解能力,并且是报道的第一个能在几天内将毫摩尔草甘膦完全降解的纯菌,这对于污染土壤修复和废水治理具有重要意义[35]。康纪婷等对采自农药厂污水处理池的活性污泥样本进行筛选,共分离27株解磷细菌,复筛后发现假单胞菌BR13、摩根氏菌BR57对草甘膦都有降解作用,降解率均在50%以上[36]。Selvi等发现的假单胞菌T5以草甘膦为唯一磷源,并且在该菌中首次分离了有C—P裂解活性的酶[37]。Moneke等对不同菌株进行草甘膦降解特性的研究,发现荧光假单胞菌和醋酸杆菌在含7.2~25 mg/mL草甘膦培养基中显著增长,表现出较强的草甘膦降解特性[38]。Kryuchkova等从根瘤菌中筛选出5株能耐受草甘膦的细菌,进一步筛选出阴沟肠杆菌K7,该菌以草甘膦为唯一磷源,将其降解为肌氨酸,不仅如此,这类细菌还能侵入向日葵和甜高粱的根部,促进根苗的生长,有益于草甘膦污染土壤的治理和修复[39]。
由Fan等分离出的蜡样芽孢杆菌CB4是以草甘膦为唯一碳源,对草甘膦降解率可达94.5%,是第一个报道的能降解草甘膦的蜡样芽孢杆菌[40]。其后,Zhan等分离出的蜡样芽孢杆菌HYC-7,则是以草甘膦为唯一氮源生长[41]。而韩丽珍等从长期使用草甘膦的土壤中筛选得到节杆菌HX-5,以草甘膦为唯一碳氮源,降解率达到74.8%[42]。叶明等也发现节杆菌A5能以草甘膦为碳氮源,72 h对草甘膦的降解率达94.2%[43]。多噬伯克霍尔德氏菌WS-FJ9是李冠喜等筛选出的高效解磷细菌,在生物降解农药残留功能的研究中发现,该菌在以草甘膦为唯一碳源、氮源或磷源培养基上均能正常生长,优化发酵条件后,试验条件下该菌对草甘膦的降解率最高可达72.8%[44]。汤明强等发现胶红酵母能以草甘膦为唯一碳氮源[45-46],而吴学华等发现的米曲霉A-F02,能以草甘膦为碳源,培养7 d对草甘膦的降解率为86.8%[47]。
2 微生物对草甘膦的降解途径
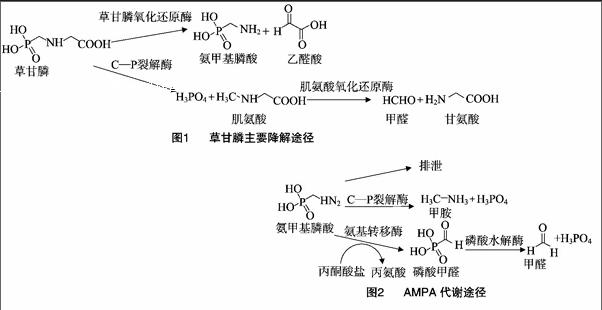
随着草甘膦降解菌研究的深入,菌株的降解途径也逐渐被阐明,微生物主要通过2条途径降解草甘膦,碳磷键(C—P键)断裂生成肌氨酸和碳氮键(C—N键)断裂生成氨甲基膦酸(AMPA)(图1)[48]。
2.1 肌氨酸途径
由Moore等筛选出的第1株草甘膦降解菌假单胞菌PG2982,是通过肌氨酸途径降解草甘膦[17],C—P键对化学反应和酶促反应有很高的稳定性,在微生物C—P裂解酶的作用下,草甘膦C—P键断裂,生成肌氨酸,为细菌生长提供磷源。此前,因为C—P裂解酶在胞外容易发生不可逆转的失活,这使得草甘膦特异性的C—P裂解酶一直是个谜[49]。最近,Selvi等从恶臭假单胞菌T5中分离出有活性的C—P裂解酶,从而揭开了该酶的神秘面纱。这是第1个纯化出的草甘膦特异性的C—P裂解酶,活性比无细胞萃取液提高600多倍,这对于草甘膦降解菌的深入研究有重要意义[37]。另一方面,生成的肌氨酸在肌氨酸氧化还原酶的作用下,快速代谢生成甲醛和甘氨酸,参与微生物的多个生理循环[18,50]。
2.2 AMAP途径
参与氨甲基膦酸(AMPA)途径的酶主要是草甘膦氧化还原酶(GOX),在该酶作用下,草甘膦C—N键断裂,生成氨甲基膦酸(AMPA)和乙醛酸[48]。截至目前,还没有发现单一微生物能通过该途径完全矿化草甘膦,大部分AMPA被微生物分泌出胞外进入环境,或被其他微生物吸收,发生C—P键断裂,最终矿化;乙醛酸则作为三羧酸循环的底物,参与微生物正常的代谢途径[19,24-25,48,51]。最近,Sviridov等发现磷酸水解酶可能参与了苍白杆菌GPK3的AMPA降解,并提出一个假设:AMPA首先在转氨酶的作用下,生成磷酸甲醛,进而作为磷酸水解酶的底物,发生水解,生成甲醛和磷酸(图2)[34],然而到目前为止,磷酸水解酶在AMPA代谢途径中的作用也仅限于假设,还没有研究提出直接的证据证明这一假设[23,52]。
研究还发现,1株降解菌还可能同时存在2种降解方式,例如GPK3,主要是经过AMPA途径降解草甘膦,同时还能检测到痕量的肌氨酸[34],表明GPK3也可通过C—P键断裂降解草甘膦,相同的现象也出现在假单胞菌LBr的降解中[19]。
而Dick等对筛选出的163株微生物进行筛选,发现筛选出的26株降解菌在磷饥饿的情况下能以草甘膦为唯一磷源将其降解,生成肌氨酸而不是AMPA[53]。进一步研究发现,肌氨酸途径在磷缺乏情况下才会发生,而环境中大都存在充足的磷源,能满足微生物的需求,因此草甘膦在环境中主要代谢生成AMPA[16],而肌氨酸途径只存在于实验室磷饥饿压力下筛选出来的降解菌中。这些表明,在实验室条件下,经过肌氨酸途径代谢的降解菌,是将草甘膦作为唯一磷源,诱导C—P裂解酶生成,而在自然环境下,环境中充足的磷源会负调节裂解酶,降解菌失去裂解草甘膦的能力;同时,不同于实验室培养,恶劣的自然环境也造成筛选菌株的不生长和降解能力的丧失,这都给草甘膦的实际应用提出了难题[19,26,54]。
可以看出,草甘膦降解菌的降解方式复杂多变,还需进行深入研究,而缺少准确、可靠的酶活性测定方法限制了对酶活性的研究。对草甘膦代谢途径和降解活性的研究,以同位素标记的草甘膦为底物,经代谢后,对降解产物进行NMR研究,是研究草甘膦代谢途径的一种有效方法[19,55],但该方法对设备要求高。或将草甘膦及其代谢产物衍生化[40,56],衍生化后经过不同检测手段,如高效液相色谱[56-57]、气相色谱[58-61]、薄层色谱[25,62]、毛细管电泳[63-64]等,对降解产物及降解途径进行分析,用于鉴别草甘膦氧化还原酶和C—P 裂解酶的活性;但试验中发现,发酵液中成分复杂,不同成分发生交叉,影响目标产物的辨识,干扰测定结果的准确性。因此,探寻精确、有效的草甘膦及其代谢产物的分析方法,对于解析草甘膦代谢方式,阐明微生物降解草甘膦的机理具有重要意义。
3 原位修复研究
目前,生物降解草甘膦还局限于实验室研究阶段,用于原位修复草甘膦污染的研究还很少,仅有少数土著微生物降解草甘膦的研究被报道[65-66]。而在分离的纯菌原位修复研究中,Shushkova等在被污染土壤中引入人苍白杆菌GPK3和无色杆菌Kg16,结果表明其对草甘膦的降解率比土著微生物菌群提高了数倍[67-68]。在引入降解菌的1~2周内,污染土壤的草甘膦含量、总体毒性水平、对植物的毒性水平已经恢复至正常土壤水平,而草甘膦浓度降低,使得土壤微生物活性恢复,表现为土著微生物的脱氢酶活性和生物量分别提高了12倍和1.6倍[67]。研究还显示降解能力跟土壤的垂直分布相关,只有土壤表层0~10 cm深的草甘膦可以被土著微生物和引入的微生物迅速降解,而10~30 cm深的土壤会因为没有降解菌GPK3,并且土著微生物的数量少而发生草甘膦蓄积,同时草甘膦会随着时间推移渗入土壤深处,雨水加速这一过程,影响草甘膦的降解[68]。
目前为止,还没有1株筛选得到的纯菌成功用于原位修复草甘膦污染的实例,主要原因在于,与其他微生物降解所遇到的难题相同,引入的微生物的生存竞争力不如土著微生物,同时恶劣的环境条件也不利于降解菌生存,使之在生存竞争压力下没有余力修复污染[54];而且,原位修复研究发现,土壤中充足的磷源抑制微生物对草甘膦的利用,实验室筛选得到的高效降解菌,在实际应用中却无法发挥作用[19,26]。
4 结语
草甘膦的使用,在造福人类的同时,也带来严重的环境和生态危害,微生物降解低毒、低耗,不会引入新污染,对于农药的治理是一种有效的手段,并且,环境残留的草甘膦主要通过微生物降解,这引导科学家们在草甘膦降解菌的筛选上进行努力,试图找到有效方法,缓解或消除草甘膦带来的环境危害。目前,已经发现多种微生物能以草甘膦为唯一的磷源、碳源或氮源,表现出极高的降解率,但目前的研究还仅仅处于实验室阶段,还没有在原位引入降解菌,成功修复受污染体系的应用实例。而且,在考虑生物修复有效性的同时,外源生物的引入是否会带来二次污染和生态危害,都需要考虑、研究和解决,这给生物修复环境污染的实际应用提出更多挑战。
参考文献:
[1]徐 杰,蒋世云,傅凤鸣,等. EPSP合酶的研究进展[J]. 生物技术通报,2014(6):40-50.
[2]向文胜,张文吉,王相晶,等. EPSP合成酶的特性及新抑制剂的研究进展[J]. 农药学学报,2000,2(2):1-8.
[3]Williams G M,Kroes R,Munro I C. Safety evaluation and risk assessment of the herbicide Roundup and its active ingredient,glyphosate,for humans[J]. Regulatory Toxicology and Pharmacology,2000,31(2):117-165.
[4]王 慧,闫晓红,徐 杰,等. 我国抗草甘膦基因的发掘现状[J]. 农业生物技术学报,2014,22(1):109-118.
[5]周垂帆,李 莹,张晓勇,等. 草甘膦毒性研究进展[J]. 生态环境学报,2013,22(10):1737-1743.
[6]刘 攀. 草甘膦对土壤微生态的影响及其抗性和降解真菌的研究[D]. 长春:吉林大学,2009.
[7]邓 晓,李雅琦. 草甘膦对土壤微生物影响的研究[J]. 农药,2005,44(2):59-62.
[8]范瑾煜. 水环境中低浓度草甘膦及制剂对鲫鱼的毒性效应研究[D]. 南京:南京大学,2013.
[9]Aparicio V C,de Geronimo E,Marino D A,et al. Environmental fate of glyphosate and aminomethylphosphonic acid in surface waters and soil of agricultural basins[J]. Chemosphere,2013,93(9): 1866-1873.
[10]Marc J,Mulner-Lorillon O,Boulben S,et al. Pesticide roundup provokes cell division dysfunction at the level of CDK1/cyclin B activation[J]. Chemical Research in Toxicology,2002,15(3):326-331.
[11]Paganelli A,Gnazzo V,Acosta H,et al. Glyphosate-Based herbicides produce teratogenic effects on vertebrates by impairing retinoic acid signaling[J]. Chemical Research in Toxicology,2010,23(10): 1586-1595.
[12]Clair E,Mesnage R,Travert C,et al. A glyphosate-based herbicide induces necrosis and apoptosis in mature rat testicular cells in vitro,and testosterone decrease at lower levels[J]. Toxicology in Vitro,2012,26(2):269-279.
[13]Poulsen M S,Rytting E,Mose T,et al. Modeling placental transport: Correlation of in vitro BeWo cell permeability and ex vivo human placental perfusion[J]. Toxicology in Vitro,2009,23(7):1380-1386.
[14]卢 信,赵炳梓,张佳宝,等. 除草剂草甘膦的性质及环境行为综述[J]. 土壤通报,2005,36(5):785-790.
[15]Araújo A F,Monteiro R R,Abarkeli R B. Effect of glyphosate on the microbial activity of two Brazilian soils[J]. Chemosphere,2003,52(5):799-804.
[16]Forlani G,Mangiagalli A,Nielsen E,et al. Degradation of the phosphonate herbicide glyphosate in soil: evidence for a possible involvement of unculturable microorganisms[J]. Soil Biology & Biochemistry,1999,31(7): 991-997.
[17]Moore J K,Braymer H D,Larson A D. Isolation of a Pseudomonas sp. which utilizes the phosphonate herbicide glyphosate[J]. Applied and Environmental Microbiology,1983,46(2): 316-320.
[18]Shinabarger D L,Braymer H D. Glyphosate catabolism by Pseudomonas sp. strain pg2982[J]. Journal of Bacteriology,1986,168(2): 702-707.
[19]Jacob G S,Garbow J R,Hallas L E,et al. Metabolism of glyphosate in Pseudomonas sp. strain LBr[J]. Applied and Environmental Microbiology,1988,54(12):2953-2958.
[20]Weidhase R,Albrecht B,Stock M,et al. Glyphosate utilization by Pseudomonas spec. GS[J]. Zentralblatt Fur Mikrobiologie,1990,145(6):433-438.
[21]Zboinska E,Lejczak B,Kafarski P. Organophonate utilization by the wild-type strain of Psrudomonas fluorrsens[J]. Applied and Environmental Microbiology,1992,58(9):2993-2999.
[22]Penalozavazquez A,Mena G L,Herreraestrella L,et al. Cloning and sequencing of the genes involved in glyphosate utilization by Pseudomonas pseydinallei[J]. Applied and Environmental Microbiology,1995,61(2):538-543.
[23]Talbot H W,Johnson L M,Munnecke D M. Glyphosate utilization by Pseudomonas sp. and Alcaligenes sp. isolated from environmental sources[J]. Current Microbiology,1984,10(5):255-259.
[24]Balthazor T M,Hallas L E. Glyphosate-degrading microorganisms from industrial activated-sludge[J]. Applied and Environmental Microbiology,1986,51(2):432-434.
[25]Pipke R,Amrhein N,Jacob G S,et al. Metabolism of glyphosate in an Arthrobacter sp. GLP-1[J]. European Journal of Biochemistry,1987,165(2):267-273.
[26]Pipke R A N,Characterization of a mutant of Arthrobacter sp. strain GLP-1 which utilizes the herbicide glyphosate as its sole source of phosphorus and nitrogen[J]. Applied and Environmental Microbiology,1988,54(11): 2868-2870.
[27]Liu C M,Mclean P A,Sookdeo C C,et al. Degradation of the herbicide glyphosate by members of the family rhizobiaceae[J]. Applied and Environmental Microbiology,1991,57(6): 1799-1804.
[28]Krzysko L T,Orlik A. The use of glyphosate as the sole source of phosphorus or carbon for the selection of soil-borne fungal strains capable to degrade this herbicide[J]. Chemosphere,1997,34(12): 2601-2605.
[29]Romero M C,Reinoso E H,Kiernan A M,et al. Biodegradation of glyphosate by wild yeasts[J]. Revista Mexicana de Micologia,2004,19: 45-50.
[30]Klimek M,Lejczak B,Kafarski P,et al. Metabolism of the phosphonate herbicide glyphosate by a non-nitrate-utilizing strain of Penicillium chrysogenum[J]. Pest Management Science,2001,57(9): 815-821.
[31]Obojska A,Lejczak B,Kubrak M. Degradation of phosphonates by streptomycete isolates[J]. Applied Microbiology and Biotechnology,1999,51(6): 872-876.
[32]潘 渠,杨志荣. 一株降解草甘磷的真菌分离鉴定[J]. 四川大学学报:自然科学版,2001,38(1):131-133.
[33]石成春,郭养浩,王大奈,等. 草甘膦曲霉生物降解的动力学研究[J]. 中国环境科学,2005,25(3):361-365.
[34]Sviridov A V,Shushkova T V,Zelenkova N F,et al. Distribution of glyphosate and methylphosphonate catabolism systems in soil bacteria Ochrobactrum anthropi and Achromobacter sp.[J]. Applied Microbiology and Biotechnology,2012,93(2): 787-796.
[35]Hadi F,Mousavi A,Noghabi K A,et al. New bacterial strain of the genus Ochrobactrum with glyphosate-degrading activity[J]. Journal of Environmental Science and Health. Part B,Pesticides,Food Contaminants,and Agricultural Wastes,2013,48(3): 208-213.
[36]康纪婷,吴 翔,甘炳成,等. 高效有机磷降解菌BR13和BR57的分离和鉴定[J]. 西南农业学报,2011,24(2):566-569.
[37]Selvi A A,Manonmani H K. Purification and characterization of carbon-phosphorus bond-cleavage enzyme from glyphosate degrading pseudomonas putida T5[J]. Preparative Biochemistry & Biotechnology,2015,45(4):380-397.
[38]Moneke A N,Okpala G N,Anyanwu C U. Biodegradation of glyphosate herbicide in vitro using bacterial isolates from four rice fields[J]. African Journal of Biotechnology,2010,9(26):4067-4074.
[39]Kryuchkova Y V,Burygin G L,Gogoleva N E,et al. Isolation and characterization of a glyphosate-degrading rhizosphere strain,Enterobacter cloacae K7[J]. Microbiological Research,2014,169(1):99-105.
[40]Fan J,Yang G,Zhao H,et al. Isolation,identification and characterization of a glyphosate-degrading bacterium,Bacillus cereus CB4,from soil[J]. Journal of General and Applied Microbiology,2012,58(4):263-271.
[41]Zhan T,Zhang K,Chen Y Y,et al. Improving glyphosate oxidation activity of glycine oxidase from bacillus cereus by directed evolution[J]. PLoS One,2013,8(11):e79175.
[42]韩丽珍,刘 飞,赵德刚. 1株草甘膦降解菌的分离鉴定及特性[J]. 贵州农业科学,2012,40(12):139-142,145.
[43]叶 明,陈九山,姚晓庆. 一株草甘膦降解菌分离鉴定及其降解特性研究[J]. 环境科学与技术,2009,32(3):39-41.
[44]李冠喜,吴小芹,叶建仁. 多噬伯克霍尔德氏菌WS-FJ9对草甘膦的降解特性[J]. 生态学报,2013,33(21):6885-6894.
[45]汤鸣强,孙丽花. 酵母菌S-2对草甘膦除草剂的降解特性[J]. 安徽农业科学,2010,38(4):1992-1994.
[46]汤鸣强,尤民生. 抗草甘膦酵母菌ZM-1的分离鉴定及其生长降解特性[J]. 微生物学通报,2010,37(9):1402-1409.
[47]Wu X H,Fu G M,Wan Y,et al. Isolation and identification of glyphosate-degraded strain Aspergillus oryzae sp. A-F02 and its degradation characteristics[J]. Plant Diseases and Pests,2010,1(5):54-57.
[48]Kishore G M,Barry G F. Glyphosate tolerant plants:WO92/00377 [P]. 1992-01-09.
[49]Kononova S V,Nesmeyanova M A. Phosphonates and their degradation by microorganisms[J]. Biochemistry,2002,67(2):184-195.
[50]Kishore G M,Jacob G S. Degradation of glyphosate by Pseudomonas sp. PG2982 via a sarcosine intermediate[J]. Journal of Biological Chemistry,1987,262(25):12164-12168.
[51]Avila L Z,Loo S H,Frost J W. Chemical and mutagenic analysis of aminomethylphosphonate biodegradation[J]. Journal of the American Chemical Society,1987,109(22):6758-6764.
[52]Balthazor T M,Hallas L E. Glyphosate-Degrading microorganisms from industrial activated sludge[J]. Applied and Environmental Microbiology,1986,51(2):432-434.
[53]Dick R E,Quinn J P. Glyphosate-Degrading isolates from environmentel-samples-occurrence and pathways of degradation[J]. Applied Microbiology and Biotechnology,1995,43(3):545-550.
[54]Bazot S,Lebeau T. Simultaneous mineralization of glyphosate and diuron by a consortium of three bacteria as free-and/or immobilized-cells formulations[J]. Applied Microbiology and Biotechnology,2008,77(6):1351-1358.
[55]Jacob G S,Schaefer J,Stejskal E O,et al. Solid-state NMR determination of glyphosate metabolism in a Pseudomonas sp.[J]. Journal of Biological Chemistry,1985,260(10): 5899-5905.
[56]Sviridov A V,Zelenkova N F,Vinokurova N G,et al. New approaches to identification and activity estimation of glyphosate degradation enzymes[J]. Biochemistry,2011,76(6): 720-725.
[57]Tomita M,Okuyama T,Watanabe S,et al. High-performance liquid
chromatographic determination of glyphosate and (aminomethyl) phosphonic acid in human serum after conversion into p-toluenesulphonyl derivatives[J]. Journal of Chromatography,1991,566(1): 239-243.
[58]Roy D N,Konar S K. Development of an analytical method for the determination of glyphosate and (aminomethyl) phosphonic acid residues in soils by nitrogen-selective gas chromatography[J]. Journal of Agricultural and Food Chemistry,1989,37(2): 441-443.
[59]Alferness P L,Iwata Y A. Phosphonic acid in Soil,plant and animal matrixes,and water by capillary gas chromatography with mass-selective detection[J]. Journal of Agricultural and Food Chemistry,1994,42(12): 2751-2759.
[60]Kudzin Z H,Gralak D K,Drabowicz J,et al. Novel approach for the simultaneous analysis of glyphosate and its metabolites[J]. Journal of Chromatography,2002,947(1): 129-141.
[61]王彦辉,李 欣,周小毛,等. 草甘膦铵盐在苎麻田的残留及消解动态[J]. 农药学学报,2010,12(2):201-206.
[62]Zelenkova N F,Vinokurova N G,Leontievskii A A. Determination of Amine-Containing phosphonic acids and amino acids as dansyl derivatives[J]. Journal of Analytical Chemistry,2010,65(11): 1143-1147.

