周岁细毛羊羊毛长度、产毛量与体质量的全基因组关联分析
狄 江,刘剑锋,徐新明,王 琼,拉扎特·艾尼瓦尔,于丽娟
(1. 新疆畜牧科学院, 乌鲁木齐 830000; 2.中国农业大学 动物科技学院, 北京 100193)
周岁细毛羊羊毛长度、产毛量与体质量的全基因组关联分析
狄江1,刘剑锋2,徐新明1,王琼1,拉扎特·艾尼瓦尔1,于丽娟1
(1. 新疆畜牧科学院, 乌鲁木齐830000; 2.中国农业大学 动物科技学院, 北京100193)
摘要为检测影响周岁细毛羊羊毛长度、毛量及体质量性状的基因组区域,利用OvineSNP50 BeadChip对365只中国美利奴(新疆型)个体基因分型,并采用single-locus回归方法进行全基因组关联分析。通过基因组水平的Permutations校正,检测到2个与羊毛长度及3个与毛量显著关联的SNPs。这些SNPs分别邻近5个已知基因(9.16 ~143.32 kb)。其中FIBIN、 HSD17B11及 PIAS1 3个基因参与羊毛生长、发育相关的生物学过程,且均包含于已知羊毛性状相关QTL区域内,为所检测到的关联结果提供进一步证据。对这些目标区域的进一步研究有助于揭示细毛羊羊毛长度、产量等性状的遗传机理。
关键词全基因组关联分析;羊毛;体质量;细毛羊
通过育种手段不断改善羊毛产量、羊毛长度、纤维直径及体质量性状是提高细毛羊综合品质和养殖效益的重要途径之一,详细了解这些经济性状的分子遗传机理,有助于加快育种进展、提高育种效率。过去20余年里,QTL方法已被普遍应用检测经济性状功能候选区域,然而截至目前在羊SheepQTLdb数据库中仅仅包括789个 QTLs,其中34个QTLs与羊毛性状连锁(http://www.animalgenome.org/cgi-bin/QTLdb/OA/index)。而且,由于QTL较大的置信区间,难以鉴别影响这些经济性状的特定基因。对于体质量性状,也仅发现MSTN(Myostatin)[1]、CLPG(Callipyge)[2]及MHC[3]等几个候选基因与绵羊生长及肌肉性状连锁。
随着分子技术快速发展,基于高通量SNP芯片技术的全基因组关联分析(GWAS)已成为探索经济性状相关候选功能区域的新的工具。目前,关于羊GWAS研究报道与牛、猪、鸡等畜种相比数量较少,且多数集中在疾病性状。通过GWAS研究已发现与小眼畸形[4]、软骨病[5]、副结核病[6]等羊病关联的基因或染色体区域。已有关于肉用羊及地方品种羊体质量及其相关性状的GWAS研究,并发现一些显著关联的染色体区域[7]。另外,在ASIP基因中检测到一个与被毛颜色变化关联的SNP[8]。最近,在中国美利奴(军垦型)中通过GWAS研究检测到一些与羊毛纤维直径、弯曲性状显著关联的SNP[9]。然而,针对细毛羊羊毛产量、体质量等重要经济性状的GWAS研究还未见报道。因此,本试验以中国美利奴周岁母羊(新疆型)为群体,通过利用Illumina OvineSNP50 BeadChip芯片,开展GWAS研究以检测与周岁羊毛长度、原毛量及体质量性状关联的潜在随机遗传变异,为进一步揭示细毛羊羊毛性状、生长性状的遗传机理并提高育种效率的研究提供借鉴。
1材料与方法
1.1动物来源与性状
试验羊只来源于新疆巩乃斯种羊场的中国美利奴(新疆型)核心育种群。该场羊群采取夏季放牧、冬季舍饲的饲养方式,全部羊群在基本一致的条件下饲养。本研究的365只周岁母羊(33只公羊后代)在剪毛后测定体质量、原毛量,采集体侧部毛样,室内测定羊毛长度和纤维直径。同时,采集耳组织样品于φ=75%乙醇保存待用。
1.2基因分型及质量控制
采用常规苯酚/氯仿方法提取基因组DNA。利用包括54 241个SNP位点的Illumina OvineSNP50 BeadChip芯片在Infinium II multisample assay (Illumina)平台上检测全部DNA样品基因型。通过iScan (Illumina) 扫描芯片结果,并利用GenomeStudio软件对原始数据进行分析,从而得到每个样品的SNP分型数据。
将全部SNP分型结果进行质控分析,去除不符合条件的SNP。去除条件为:杂交阳性率(call rate)<90%, 最小等位基因频率(MAF) <3%,显著偏离哈迪-温伯格平衡(HWE)(P<10-3,P为卡方显著性检验P值)。另外,不在羊常染色体上的SNP也被排除在进一步的关联分析之外。
1.3全基因组关联分析及统计推断
采用single-locus回归方法通过PLINK软件进行全基因组关联分析,模型如下:
y=μ+Xf+bm+Zs+e
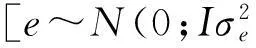
采用permutation方法对关联分析结果进行校正,通过permutation 10 000次获得3个性状的5%基因组水平显著P值阈值。
1.4基因注释
OvineSNP50 BeadChip中SNP位置主要基于羊早期基因组序列版本(Ovis_aries_v1.0)确定。首先利用SNP50 BeadChip基因组位置注释文件重新定位SNP在最近的基因组序列版本Ovis_aries_v3.1上的位置(http://www.livestockgenomics.csiro.au/sheep/oarv3/Oarv3.150kSNP. position.gff3);其次通过NCBI中的Ovisaries Annotation Release 100工具鉴定与显著关联SNP对应或邻近的羊基因(http://www.ncbi.nlm.nih.gov/projects /mapview/map_search.cgi?taxid=9940&build =100.0,基于 Ovis_aries_v3.1 注释);最后,利用绵羊数量性状位点数据库中已知数量性状位点对显著关联SNP进行比对、验证(Sheep QTLdb,http://www.animalgenome.org/cgi-bin/QTLdb/OA/index)。
2结果与分析
2.1表型及质控后的SNP分布
全部分析性状的统计结果描述见表1。分型结果经质控后,选择44 798个SNP用于进一步的关联分析。过滤后每条染色体上的SNP数量变化范围为672个(OAR26)~5 150个(OAR1),SNP间的距离变化范围为49.97 kb(OAR8)~65.59 kb (OAR26)。质控后的SNP在每条染色体上的分布见表2。
2.2全基因组关联分析
经过对中国美利奴周岁母羊毛长、毛量及体质量3个性状进行全基因组关联分析,采用permutation(10 000次)方法校正获得3个性状5%基因组水平显著P值阈值分别为:毛长1.31×10-6、毛量1.01×10-6、体质量8.76×10-7。采用校正后的P值作manhattan图,见图1。经基因组水平的permutation校正,共检测到5个SNP达到显著水平。其中与羊毛长度显著关联的SNP有2个,与毛量显著关联的SNP有3个,没有检测到与剪毛后体质量显著关联的SNP。这些显著关联的SNP分别位于5条不同的染色体,邻近已知羊基因的距离为9.16~143.32 kb。表3详细列示显著SNPs名称、位置及邻近基因等。
2.3显著SNP与已知数量性状位点比较
通过比较绵羊数量性状位点数据库,发现位于6、7和15号染色体上的显著关联SNP分别与已知影响羊毛长度、纤维直径的QTL区域吻合(表4)。其中15号染色体上的与羊毛长度显著关联的SNP也位于相同性状的已知QTL区域内,而6、7号染色体上与毛量显著关联的SNP则位于羊毛长度、纤维直径性状已知的QTL区域内。
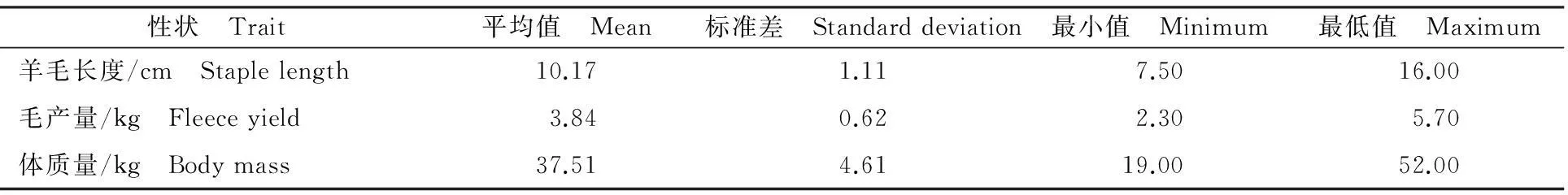
表1 周岁羊毛及体质量性状统计
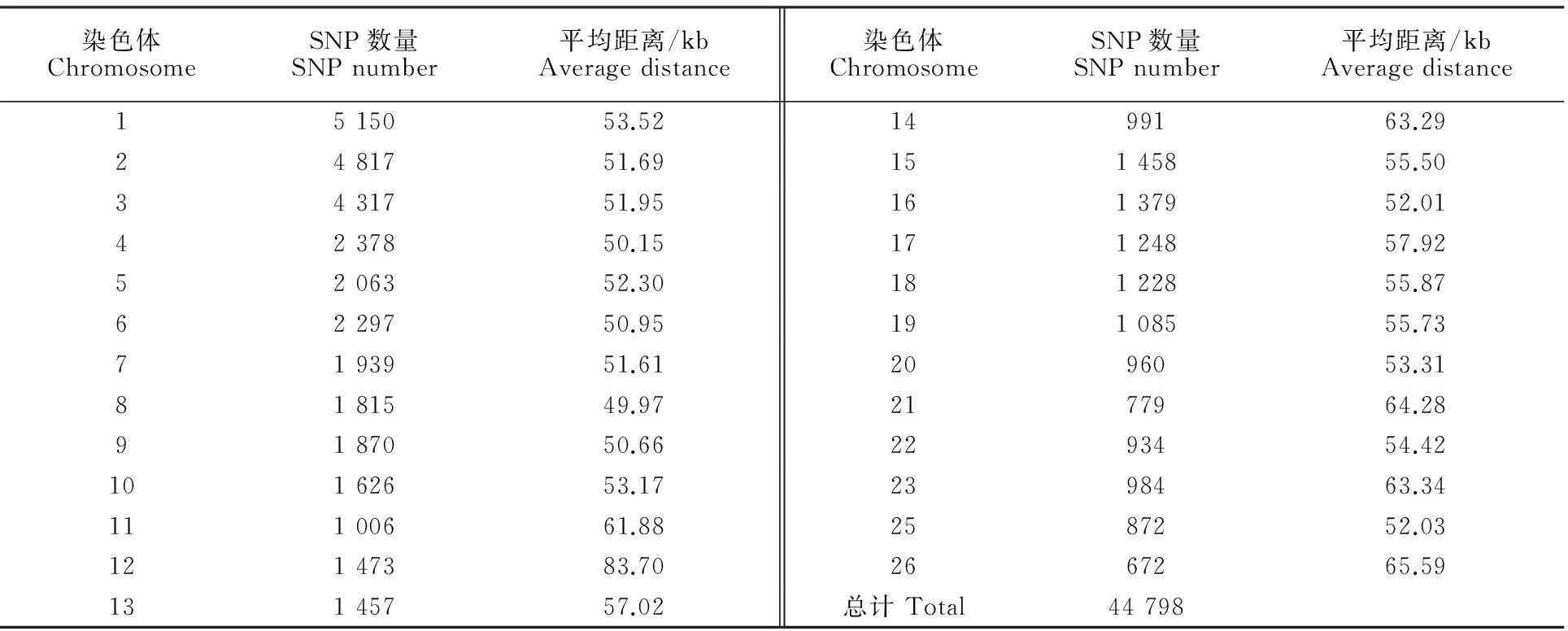
表2 质控后SNP在每条染色体的分布
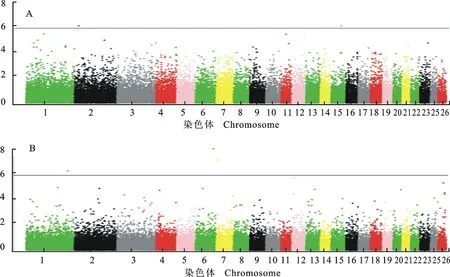
A.羊毛长度Staple length; B.毛量Fleece mass ;纵坐标为关联性检验的P的常用对数值Scale on y-axis is (P-value)common logarithm score of association analysis;蓝色线为5%基因组水平显著P值阈值线Blue line indicatesP-value threshold of genome-wise 5% significance level
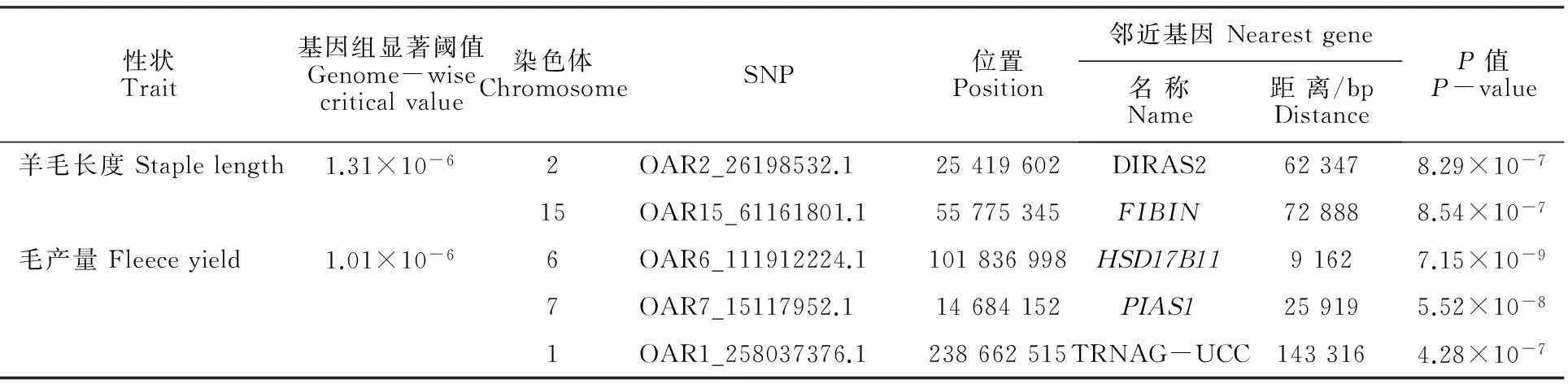
图1 羊毛性状全基因组关联分析曼哈顿图
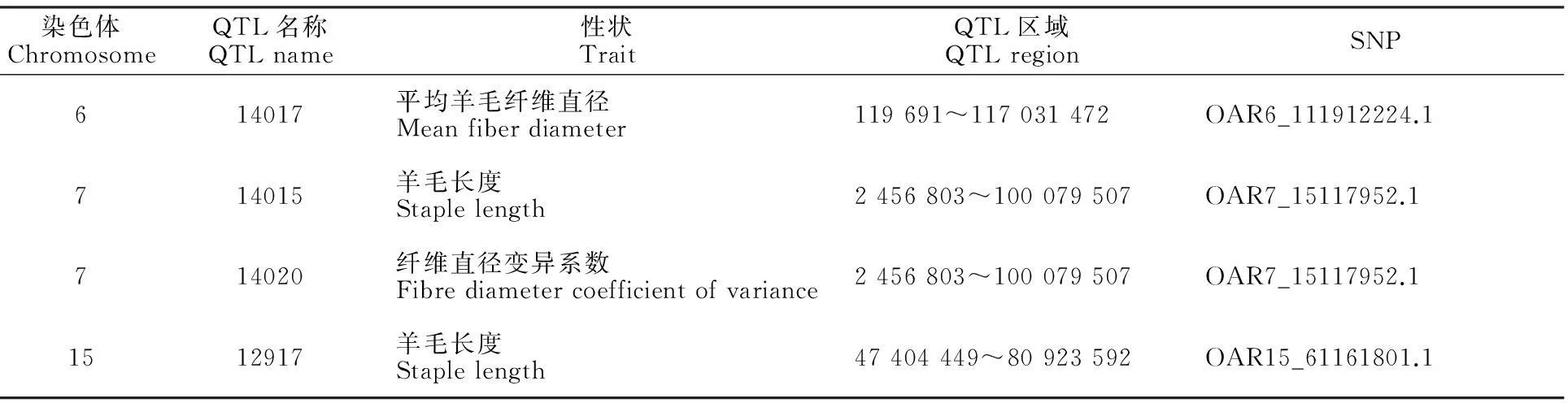
表4 与本研究显著关联SNPs相对应的已知QTLs
注:来源于 Sheep QTLdb. Available: http://www.animalgenome.org/cgi-bin/QTLdb/OA/index.
Note:Derived from Sheep QTLdb. Available:http://www.animalgenome.org/cgi-bin/QTLdb/OA/index.
3讨 论
毛囊发育及羊毛生长是一个复杂的生物学过程。在小鼠及人类中的研究表明,Wnt、TGFβ/BMP、 Sonic hedgehog、 FGF及NF-κB等信号通路及其这些信号通路间相互作用对毛囊发生、发育及毛发生长都有重要作用[10-12]。其中Wnt通路主要调控毛囊形态发生,并通过EDA/EDAR/NF-κB通路继续作用于毛发生长,Sonic hedgehog参与毛囊形态发生及随后的毛囊和毛发分化,NF-κB对Wnt通路有调控作用,Fgf通过Fgf7/Fgf10配体信号影响出生后形态发生期间毛发生长[10]。
本研究通过对周岁细毛羊羊毛性状的全基因组关联分析,在基因组水平经Permutation检验,获得与羊毛性状显著关联的5个SNPs,其中毛长2个、毛量3个。这5个SNPs分布于5条不同染色体,邻近5个已知的羊基因。与周岁羊羊毛长度显著关联的2个SNP中,OAR2_26198532.1邻近 DIRAS2基因 (DIRAS family, GTP-binding RAS-like 2),SNPOAR15_61161801.1 邻近FIBIN基因 (fin bud initiation factor homolog)。FIBIN是生长因子蛋白,在斑马鱼胸鳍芽及小鼠胚胎的前肢芽的触发有重要作用[13]。FIBIN通过作用于其下游的wnt及FGF信号诱发胸鳍芽[14],而这2个通路也影响羊毛生长。而且,在Sheep QTLdb中,OAR15 的47 404 449~80 923 592 bp区域内存在一个与羊毛长度关联的QTL,与FIBIN基因及其对应的SNPs所在区域及吻合,进一步提示该区域及FIBIN基因有必要作为影响羊毛纤维长度性状的重要候选者继续深入研究。DIRAS2属于small GTPaseRas家族,其成员在细胞形态发生和神经发生中有作用[15],但DIRAS2的详细功能还不清楚,是否影响毛发生长也未见报道。
与周岁毛量显著关联的3个SNPs中,OAR6_111912224.1邻近 HSD17B11基因 (hydroxysteroid (17-beta) dehydrogenase 11),OAR7_15117952.1邻近 PIAS1基因 (protein inhibitor of activated STAT 1),OAR1_258037376.1邻近TRNAG-UCC基因 (transfer RNA glycine (anticodon UCC))。 HSD17B11属于17β-hydroxysteroid脱氢酶(HSD17Bs),其能将3α-Diol(5α-androstan-3α,17β-diol)转化为雄酮,因此,在雄激素代谢中有重要作用[16]。研究表明,雄激素对毛发生长有重要但不同的影响,即可刺激胡须、腋窝等部位的毛发生长,也可抑制前额毛发的生长[17]。其以毛囊中的真皮乳头细胞作为靶细胞,通过胰岛素样生长因子I (IGF-I)促进毛发生长[18],或通过刺激TGF-β1表达抑制毛发生长[19]。尽管 HSD17B11是否直接影响毛发生长还未见相关研究报道,但本试验中检测到其与产毛量间显著的关联说明其有可能通过影响性激素代谢调控羊毛生长。 PIAS1是信号转导子和转录激活子1 (STAT1,Signal transducer and activatoroftranscription 1)的抑制因子,其能促进小泛素相关修饰物(Small ubiquitin-related modifier, SUMO)分子对靶蛋白的SUMO化修饰。SUMO化修饰能影响蛋白质间的相互作用,改变蛋白质于细胞内的位置,增加蛋白质的稳定性[20]。研究表明 PIAS1和SUMO化修饰在其他一些组织及系统中参与Wnt[21]、BMP[22]、Hedgehog[23]及NF-κB[24]等信号途径的调控,而这些通路也与毛发生长相关。
在Sheep QTLdb中,OAR6 的119 691~117 031 472 bp区域间存在一个与羊毛纤维直径关联的QTL,与OAR6_111912224所在区域吻合;OAR7 的2 456 803~100 079 507 bp区域间存在一个与羊毛长度关联的QTL,与OAR7_15117952.1所在区域吻合。研究表明,美利奴羊群中原毛量与纤维直径间(相关系数为0.36)、原毛量与羊毛长度间(相关系数为0.44)均存在较高程度的正遗传相关[25],这也进一步证明这2个基因与原毛量间可能的关联。对于TRNAG-UCC基因,未见有关其功能的文献报道。
从以上对几个相邻候选基因的功能描述看出,FIBIN、 HSD17B11、 PIAS1 3个基因在不同组织或物种中均参与与毛发发育和生长相关的信号通路,并且与其相关的SNP也位于已知的羊毛性状QTL区域内,进一步暗示这些基因可作为影响羊毛性状的候选基因。本研究中没有检测到与周岁羊体质量显著关联的SNP,同时也没有检测到与毛量、羊毛长度较高遗传相关相对应的基因多效性现象(未检测到同时与这2个性状显著关联的SNP),其原因可能与标记密度较低(排除18%SNP)、样本数量较少(365个个体)及每条染色体上SNP数量变化较大等因素有关。
4结 论
本研究通过全基因组关联分析,共检测到5个基因组水平上与周岁细毛羊羊毛长度、原毛量显著关联的SNP,且注释5个候选基因。这些候选基因中的3个有与毛发生长、发育相关的生物学功能,且均包含于已知羊毛性状QTL区域内。这些结论为本研究中的关联结果提供进一步的证据。对这些目标区域的进一步研究有助于揭示细毛羊羊毛长度、产量等性状的遗传机理。
参考文献Reference:
[1]CLOP A,MARCQ F,TAKEDA H,etal.A mutation creating a potential illegitimate microRNA target site in the myostatin gene affects muscularity in sheep[J].NatureGenetics,2006,38(7):813-818.
[2]SHAY T L,BERGHMANS S,SEGERS K,etal.Fine mapping and construction of a bovine contig spanning the ovine callipyge locus[J].MammalianGenome,2001,12:141-149.
[3]BOT J,KARLSSON L J E,GREEF J,etal.Association of the MHC with production traits in Merino ewes[J].LivestockProductionScience,2004,86(1-3):85-91.
[4]BECKER D,TETENS J,BRUNNER A,etal.Microphthalmia in texel sheep is associated with a missense mutation in the Paired-Like Homeodomain 3 (PITX3) gene[J].PLoSONE,2010,5(1):e8689.
[5]ZHAO X,DITTMER KE,BLAIR H T,etal.A novel nonsense mutation in the DMP1 gene identified by a genome-wide association study is responsible for inherited rickets in Corriedale sheep[J].PLoSONE,2011,6:e21739.
[6]PURDIE A C,PLAIN K M,BEGG D J,etal.Candidate gene and genome-wide association studies ofMycobacteriumaviumsubspparatuberculosisinfection in cattle and sheep:A review[J].ComparativeImmunologyMicrobiologyandInfectiousDiseases,2011,34:197-208.
[7]ZHANG L,LIU J,ZHAO F,etal.Genome-wide association studies for growth and meat production traits in sheep[J].PLoSONE,2013,8(6):e66569.
[8]LI M H ,TIIRIKKA T,KANTANEN J A.Genome-wide scan study identifies a single nucleotide substitution in ASIP associated with white versus non-white coat-colour variation in sheep(Ovisaries) [J].Heredity,2014,112:122-131.
[9]WANG Z,ZHANG H,YANG H,etal.Genome-wide association study for wool production traits in a Chinese Merino sheep population[J].PLoSONE,2014,9(9):e107101.
[10]RISHIKAYSH P,DEV K,DIAZ D,etal.Signaling involved in hair follicle morphogenesis and development[J].InternationalJournalofMolecularSciences,2014,15:1647-1670.
[11]MILLAR S E.Molecular mechanisms regulating hair follicle development[J].JournalofInvestigativeDermatology,2002,118:216-225.
[12]SCHMIDT-ULLRICH R,PAUS R.Molecular principles of hair follicle induction and morphogenesis[J].Bioessays,2005,27:247-261.
[13]LAKNER J,SEYER C,HERMSDORF T,etal.Characterization of the expression,promoteractivity and molecular architecture of fibin[J].BMCBiochemistry,2011,12:26.
[14]WAKAHARA T,KUSU N,YAMAUCHI H,etal.Fibin,a novel secreted lateral plate mesoderm signal,is essential for pectoral fin bud initiation in zebrafish[J].DevelopmentalBiology,2007,303(2):527-535.
[15]REIF A,NGUYEN T T,WEIFLOG L,etal.DIRAS2 is associated with adult ADHD,related traits,and co-morbid disorders[J].Neuropsychopharmacology,2011,36:2318-2327.
[16]BRERETON P,SUZUKI T,SASANO H,etal.Pan1b (17betaHSD11)-enzymatic activity and distribution in the lung[J].MolecularandCellularEndocrinology,2001,171(1/2):111-117.
[17]EBILING F J G.Hair follicles and associated glands are androgen targets[J].JournalofClinicalEndocrinology&Metabolism,1986,15:319-339.
[18]ITAMI S,KURATA S,TAKAYASU S.Androgen induction of follicular epithelial cell growth factor is mediated via insulin-like growth factor-I from dermal papilla cells[J].BiochemicalandBiophysicalResearchCommunications,1995,212:988-994.
[19]INUI S,FUKUZATOY,NAKAJIMA T,etal.Androgen-inducible TGF-β1 from balding dermal papilla cells inhibits epithelial cell growth:a clue to understanding paradoxical effects of androgen on human hair growth[J].FASEBJournal,2002,16:1967-1969.
[20]GEISS-FRIEDLANDER R,MELCHIOR F.Concepts in sumoylation:a decade on[J].NatureReviewsMolecularCellBiology,2007,8:947-956.
[21]YAMAMOTO H,IHARA M,MATSUURA Y,etal.Sumoylation is involved in beta-catenin-dependent activation of Tcf-4[J].EMBOJournal,2003,22:2047-2059.
[22]SHIMADA K,SUZUKI N,ONO Y,etal.Ubc9 promotes the stability of Smad4 and the nuclear accumulation of Smad1 inosteoblast-like Saos-2 cells[J].Bone,2008,42:886-893.
[23]COX B,BRISCOE J,ULLOA F.Sumoylation by Pias1 regulates the activity of the hedgehog dependent Gli transcription factors[J].PLoSONE,2010,5(8):e11996.
[24]HEO K,CHANG E,TAKEI Y,etal.Phosphorylation of protein inhibitor of activated STAT1 ( PIAS1) by MAPK-activated protein kinase-2 (MK2) inhibits endothelial inflammation via increasing both PIAS1 transrepression and SUMO E3 ligase activity[J].ArteriosclerosisThrombosisandVascularBiology,2013,33(2):321-329.
[25]SAFARI E,FOGARTY N M,GILMOUR A R.A review of genetic parameter estimates for wool,growth,meat and reproduction traits in sheep[J].LivestockProductionScience,2005,92(3):271-289.
Received 2015-02-16Returned2015-04-15
Foundation itemXinjiang High-Tech Research and Development Program (No.201211101); the National Key Technology Support Program of China (No.2011BAD28B05).
First authorDI Jiang, male, professor.Research area:animal genetics and breeding.E-mail:dijiang69@163.com
(责任编辑:成敏Responsible editor:CHENG Min)
Genome-wide Association Studies for Wool and Body Mass Traits in Yearling Fine Wool Sheep
DI Jiang1, LIU Jianfeng2,XU Xinming1,WANG Qiong1,Lazate·Ainiwaer1and YU Lijuan1
(1. Xinjiang Academy of Animal Sciences, Urumqi830000, China;2.College of Animal Science and Technology, China Agricultural University, Beijing100193, China)
AbstractFor identifying genomic regions and genes that play roles in staple length, greasy fleece yield and body mass of yearlingfine wool sheep, the genome-wide association studies (GWAS) were performed based on single-locus regression method using genotypes from the OvineSNP50 Bead Chip for these traits. Data consisted of 365 animals from the Chinese merino sheep breed. After we adjusted for multiple testing based on permutations, 2 and 3 genome-wise significant SNPs were identified for staple length, greasy fleece yield respectively. These SNPs were located close to known genes (9.16-143.32 kb). The three genes of FIBIN, HSD17B11, PIAS1 were highlighted, considering their functions of involving in the different signaling pathway related to hair/wool development and growth. The observation of QTL reports for wool staple length and fiber diameter, covering areas surrounding the three genes (SNPs) herein identified provided more evidence for these associations. Future studies targeting these areas could provide further knowledge to discover the genetic architecture underlying staple length and greasy fleece mass traits in Chinese Merino sheep.
Key wordsGWAS; Wool; Body mass;Fine wool sheep
收稿日期:2015-02-06修回日期:2015-04-15
基金项目:新疆高技术研究项目(201211101);国家科技支撑项目(2011BAD28B05)。
中图分类号S813.3
文献标志码A
文章编号1004-1389(2016)04-0496-06
网络出版日期:2016-04-02
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20160402.1111.006.html
第一作者:狄江,男,研究员,主要从事家畜遗传育种领域研究。Email: dijiang69@163.com

