水稻编码SAND结构域基因的序列与功能初步分析
陈志雄,戴双凤,夏昌选,谢海媚,李亚娟
(1.华南农业大学 农学院,广东 广州 510642;2.华南农业大学公共基础课实验教学中心,广东 广州 510642)
水稻编码SAND结构域基因的序列与功能初步分析
陈志雄1,戴双凤1,夏昌选1,谢海媚1,李亚娟2
(1.华南农业大学 农学院,广东 广州510642;2.华南农业大学公共基础课实验教学中心,广东 广州510642)
摘要:为研究水稻SAND结构域蛋白的序列和功能,利用Blast在水稻基因组中搜索编码SAND结构域的基因,然后构建增强表达载体并通过遗传转化研究基因功能。结果表明,水稻基因LOC_Os01g57240(命名为OsULT1)编码的氨基酸序列含有SAND结构域和ULT B-box共有序列,与其他植物ULT氨基酸序列相似性为49.3%~92.3%,具有较好的保守性;35S::OsULT1转基因株系部分颖花发育异常,出现内稃发育滞缓或退化、浆片过度发育或数目变多、雄蕊数目3~7枚、雌蕊2枚的现象;颖花内产生多余的小花,表明OsULT1对水稻花分生组织正常分化具有重要的调控作用。
关键词:水稻;花发育;SAND结构域;基因;序列
植物花的发育是按一定的步骤进行的,首先在光周期促进、春化促进、自动调控和赤霉素4种途径共同调控下[1],完成从营养生长向生殖生长的转换,这时一部分或全部的茎顶端分生组织形成花序分生组织原基。随后花序分生组织的周边区域发育形成花分生组织和花器官分生组织原基,最终形成花萼、花瓣、雄蕊和雌蕊四轮不同类型的花器官。拟南芥花器官分生组织基因主要有A类基因APETALA1(AP1)和AP2、B类基因AP3和PISTILLATA(PI)、C类功能基因AGAMOUS(AG),这3类基因单独或共同调控花器官原基的产生[2]。E类基因SEP1、SEP2和SEP3冗余作用决定花瓣、雄蕊和心皮以及花的有限发育[3-4]。除了AP2外,ABCE类基因编码植物特有的MADS-box蛋白,具有保守的MIKC结构域,能形成二聚体或高层次的复合体[5]。拟南芥MADS-box基因编码的蛋白所形成的复合体能够将叶片转变为花器官[6]。因此,花发育的四复合体模型认为AP1-AP1-X-X、AP1-AP3-PI-SEP、AP3-PI-AG-SEP和AG-AG-SEP-SEP MADS-box蛋白的复合体分别为目标基因特异性结合基因,决定了相应花器官的形成[4]。
水稻花形态特征特异,花发育过程中各分生组织的许多特征与双子叶植物明显不同,是研究单子叶植物花发育的模式材料。水稻花发育的基因调控研究已取得了一定进展,调控不同阶段花发育相关基因均有报道,如TETFL1的同源基因RCN1和RCN2、LFY的同源基因APO2/RFL、调控水稻枝梗和小穗分生组织发育相关的基因有SP1、DEP2、OsAPO1/SCM2、IPA1、TAW1、LAX、LOG1和参与调控小穗分生组织发育及小穗向小花分生组织过渡的基因有FZP、SNB和MSF1[7-8]。水稻基因组中存在与拟南芥同源的花器官分生组织特异基因,如A类基因OsMADS14和OMADS15、B类基因SPW1/OsMADS16、OsMADS2和OsMADS4、C类基因OsMADS3、OsMADS58和DL、E类基因LSH1/OsMADS1[9-10]。水稻花发育调控基因在功能上与拟南芥具有保守性,但也出现一定的分化。
植物花发育是一个涉及众多基因相互协调、共同调控的复杂过程。花发育过程的花分生组织形态特征异常往往导致花器官数目变异,如拟南芥wuschel(wus)突变体花分生组织变成有限性分化[11]、perianthia突变体花分生组织大小未变但花器官空间分布的变化[12]、花分生组织大小,如clavata1、2、3和wiggum突变体[13-14]。花分生组织大小对花器官数目的调控机理研究最清楚的是拟南芥的CLV信号途径[13]。水稻中存在着类似拟南芥的CLV信号途径,水稻FON1是CLV1的同源基因,控制了花分生组织的大小。FON4对分生组织的正常终止是必需的,FON4属于CLV3/ESR相关的基因家族[7-8]。拟南芥ultrapetala1(ult1)突变体花序分生组织变大,产生多余的花和花器官,花分生组织有限性缺失,AtULT1编码SADN结构域蛋白,通过WUS-AG空间反馈环,负调控WUS基因活性进而影响花分生组分化有限性,是茎端分生组织和花分生组织细胞积累的负调控因子[15-16]。本研究利用AtULT1序列搜索水稻基因组序列发现了一个同源基因ULTRAPETALA1(OsULT1),首先分析了OsULT1的氨基酸序列,并且构建OsULT1增强表达载体,通过农杆菌介导法转化栽培稻,观察转基因株系的花器官变异情况,旨在初步明确OsULT1对花器官发育的影响,充实水稻生殖发育的分子调控机制。
1材料和方法
1.1序列分析
利用拟南芥ULTRAPETALA1(AtULT1)基因序列在水稻基因组注释项目(http://rice.plantbiology.msu.edu/)进行Blast,选E-value 小于e-10的记录进行分析。并选用AtULT1氨基酸序列在 phytozome(http://www.phytozome.net)进行BlastP,获取同源基因。利用PROSITE(http://prosite.expasy.org/)预测蛋白质结构域。在水稻全长cDNA数据库KOME(http://cdna01.dna.affrc.go.jp/cDNA/)搜索到全长cDNA。序列比对和系统进化树构建分别采用ClustalX 1.83和MEGA 5.2软件。
1.2总RNA提取与RT-PCR
取水稻日本晴孕穗期幼穗,利用TRIzol法(Invitrogen公司)提取总RNA。采用SuperScript RTⅡ逆转录酶(Invitrogen公司)将RNA逆转录成第一链,离心管中加入5 μg总RNA和1 μL Oligo(dT)15(Promega公司),补充一定体积DEPC水到8 μL。在70 ℃水浴中加热5 min,后在冰上冷却,稍离心。然后依次加入4 μL 5× First Strand Buffer(Invitrogen公司)、2 μL 0.1 mol/L DTT(Invitrogen公司)、1.6 μL 2.5 mmol/L DNTP(TaKaRa公司)和1 μL Rasin(40 U/μL)(Promega公司),混匀后42 ℃反应2 min;加1 μL逆转录酶SuperScript RTⅡ,42 ℃反应1 h,70 ℃反应15 min,终止反应。
RT-PCR以逆转录的cDNA为模板,扩增体系为20 μL,内含2.0 μL cDNA、2.0 μL 10× PCR Buffer、0.8 μL 2.5 mmol/L dNTP、4.0 μL 引物(1.0 μmol/L)、0.2 μLTaq酶(5 U/μL)和11 μL ddH2O。PCR反应条件为94 ℃预变性5 min;94 ℃ 45 s,58 ℃ 45 s,72 ℃ 45 s,循环28次;72 ℃ 10 min(延伸)。OsULT1基因特异性引物为OsULT1-F(5′-TTATTTTGA GTTGTTGCGGTGTG-3′)和OsULT1-R(5′-CGGGGA ATCATTGGACCTG-3′)。潮霉素基因引物Hpt-F(5′-TCCGGAGCCTCCGCTCGAAGTAG-3′)和Hpt-R(5′-CTGAACTCACCGCGACGTCGTC-3′)。
1.3PCR产物回收和克隆及测序
利用0.8%琼脂糖电泳检测PCR扩增的产物,并割胶回收目的DNA片段,用于连接反应。连接反应体系为PCR产物5 μL,pUCm-T载体0.5 μL(25 ng),T4连接酶1 μL(4 U),10×T4连接酶缓冲液2 μL,ddH2O 11.5 μL,反应总体积20 μL,14 ℃过夜。取5~10 μL连接产物,加入到100 μL感受态DH-5α菌株的细胞悬浮液中,混匀,置冰上30 min,42 ℃热击90 s,冰上3~5 min。加入0.5 mL的LB培养液,37 ℃振荡培养1 h。取100 μL培养液涂布于LB培养平板上,37 ℃培养到蓝白斑区分明显。挑选白色菌落,37 ℃振荡培养12 h。用碱裂解法抽提质粒,送至生工生物工程(上海)股份有限公司进行双向测序
1.4转基因试验与表型分析
构建了35S::OsULT1增强表达转基因质粒,设计引物PCR扩增OsULT1 cDNA编码区序列,克隆到pUCm-T载体上,双酶切后,连接到pCAMBIA1301的多克隆位点。水稻遗传转化采用农杆菌介导法[17]。水稻稻穗破口时,选取幼穗,观察内外稃片数、浆片、花药、柱头和子房的数目,并选择含有不同数目花器官典型的小花进行拍照。
2结果与分析
2.1水稻ULTRAPETALA1的序列获取
以拟南芥ULTRAPETALA1(AtULT1)基因编码区序列Blast水稻基因组注释项目(TIGR),获得2个E-value 小于e-10的记录号,分别为LOC_Os01g57240和LOC_Os05g42290。PROSITE预测结果表明,LOC_Os01g57240编码的氨基酸序列含有SAND结构域;在水稻全长cDNA数据库搜索到与LOC_Os01g57240编码区相匹配的全长cDNA AK064375,编码区序列相似性达99%,仅有2个碱基的差异,表明LOC_Os01g57240基因能转录形成mRNA,将它命名为OsULT1。LOC_Os05g42290编码的氨基酸序列不具有SAND结构域,在水稻全长cDNA数据库发现了与它编码区相匹配的全长cDNA AK108614,但两者相似性较差,LOC_Os05g42290不作后续分析。
2.2水稻ULTRAPETALA1的序列比对与进化分析
利用OsULT1氨基酸序列在 Phytozome(http://www.phytozome.net)进行BlastP,结果发现双子叶植物拟南芥、苜蓿、大豆和单子叶植物玉米、二穗短柄草、谷子基因组有水稻OsULT1同源基因,具有较好的保守性,编码的氨基酸序列中含有SAND结构域和ULT B-box共有序列(图1);这8种植物ULT氨基酸序列相似性较高,为49.3%~92.3%。水稻OsULT1与GmULT序列相似性最低为54.6%,而与SiULT的相似性最高达87.6%。

OsULT1.LOC_Os01g57240,水稻;ZmULT.GRMZM2G082745,玉米;SiULT.Si002762m,谷子;BdULT.Bradi2g51790,
拟南芥AtULT1和AtULT2 SADN结构域碱基序列发生错义突变,导致花序和花分生组织变大而产生多余的花或花器官。将植物ULT蛋白SAND结构域序列与人类、绣丽隐杆线虫的ULT蛋白SAND结构域序列进行多序列比对,发现它们之间的保守性较差,相似性为9.9%~20.2%,而植物SAND结构域序列之间相似性则较高,相似性为47.2%~84.6%,但是2个重要的基序TPxxFE和KDWK均存在,二级结构也存在较大的差异(图2)。系统进化树分析表明,植物ULT蛋白与人类、绣丽隐杆线虫的ULT蛋白明显分成两大类,进化关系较远,而植物间则相对较近(图3)。
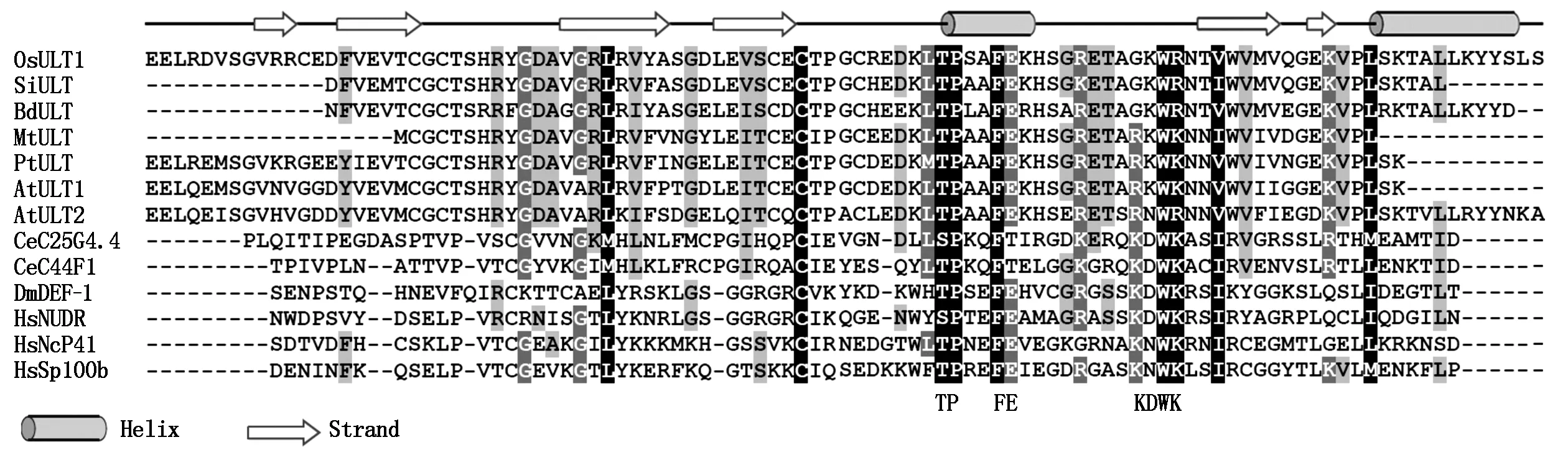
OsULT1.LOC_Os01g57240,水稻;SiULT.Si002762m,谷子;BdULT.Bradi2g51790,二穗短柄草;MtULT.AC229695_4,苜蓿;PtULT.
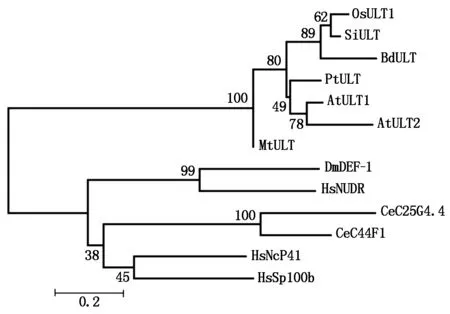
图3 水稻OsULT1与其他物种
2.3水稻ULTRAPETALA1基因的功能研究
据OsULT1基因全长cDNA AK064375克隆序列设计特异性引物扩增CDS序列并测序,结果表明,扩增条带的序列与预测的一样。将OsULT1 CDS插入pCAMBIA1301多克隆位点,构建了OsULT1超表达载体35S::OsULT1,通过农杆菌介导法进行遗传转化粳稻品种日本晴,并获得转基因株系,利用pCAMBIA1301载体携带的潮霉素基因设计特异性引物,进行PCR扩增检测,表明35S::OsULT1整合到日本晴基因组(图4-A)。RT-PCR检测结果表明转基因株系OsULT1基因的表达量大于对照日本晴(图4-B)。
转基因株系株营养生长期植株形态与野生型水稻类似,茎秆、叶片、叶鞘形态未表现出变异。抽穗期,幼穗顶端部分小穗表现出变异(图5)。正常水稻颖花由4轮花器官组成,从外到内依次为外稃、内稃各1片、1对浆片、6枚雄蕊和1枚雌蕊,颖花的基部有1对退化的护颖。转基因株系35S::OsULT1小穗表现花器官结构,如小穗轴上长一颖花和一稃片状结构(图5-A)、内稃退化成尖状结构(图5-B)、外稃包住内稃(图5-C)、外稃与内稃之间有浆片状结构(图5-C1)、内稃里长有雄蕊和雌蕊(图5-C2)、外稃包住另一完整的小花(图5-D、D1、D2)、颖花的内、外稃内长有类稃片结构1或2片(图5-E~F)等,有的花丝顶端发育出柱头(图5-G),有的内稃发育带缓或退化(图5-H~J)。转基因株系35S::OsULT1产生2对浆片的颖花(图5-K),浆片过度发育与花丝相粘连(图5-L)。转基因株系35S::OsULT1雌蕊异常表现为2枚雌蕊(图5-K)。转基因株系35S::OsULT1颖花内花药数变化较大,有3枚(图5-K)、4枚(图5-M)、5枚(图5-N)、6枚(图5-O)、7枚(图5-P);颖花内花丝花药粘连在一起(图5-Q)。这些变异性状在转基因株系后代均可观察到,表明OsULT1表达水平变异会影响水稻花分生组织正常分化。

A:1.DL2000 标准分子;2,3.日本晴;4~10.T0转基因株系;

图5 35S::OsULT1转基因株系花器官变异
3讨论与结论
水稻作为重要粮食作物之一,其生殖发育(花序)影响与稻谷产量紧密相关,研究水稻生殖发育机制具有重要的理论意义和实践价值。与双子叶模式植物相比,水稻等单子叶植物花序发育的机制研究明显落后于双子叶植物,相关突变体较少是原因之一。反向遗传学方法是研究水稻花发育的基因调控机制的有效方法[10]。拟南芥AtULT1在花序、花分生组织大小以及维系花分生组织有限性的负调控中发挥着重要的作用[15-16],拟南芥AtULT1和AtULT2表达水平下调导致侧生器官发育畸形和早期茎端分生组织分化停止[18],可见拟南芥ULT基因在花发育中的重要性。本研究结果表明,水稻基因组存在一个ULT基因OsULT1,其编码的蛋白质含有SAND结构和TPxxFE基序。DmDEAF-1、HsNUDR和HsGMEB1/2等SAND 结构蛋白与DNA特异性结合,SAND结构域通过KDWK基序调控这种相互作用,SAND结构域是AIRE反式激活、同质多聚化和核定位所必需的。三维结构模型分析SAND结构域是一新的DNA结合分子,参与基因转录调控[19]。拟南芥AtULT1与 ATX1 trxG因子相互作用,影响AGAMOUS组蛋白甲基化调控AGAMOUS的转录水平[20]。水稻OsULT1与拟南芥AtULT1的SAND结构序列相似为67.3%,预示着水稻OsULT1具有与AtULT1类似的功能,在花发育过程中发挥调控作用。
转基因株系35S::OsULT1少数颖花四轮花器官数目均表现变异,转基因株系后代表现类似的变异性状,表明OsULT1表达水平变异会影响水稻花分生组织正常分化。35S::OsULT1转基因株系大部分颖花的花器官未出现变异,这可能与转基因株系中OsULT1表达水平增加较小有关。单子叶水稻花发育模型与双子叶植物相似,水稻C类功能基因有OsMADS3和OsMADS58,SEP类功能基因LSH1/OsMADS1等,这些基因发生变异会导致花器官数目、同源异型转化和花分生组织分化有限性发生变化。水稻中也存在着类似拟南芥的CLV信号途径。水稻FON1和FON4分别是拟南芥CLV1和CLV3/ESRLRR同源基因,FON1 控制了花分生组织的大小,FON4对分生组织的正常终止是必需的,表型上的相似性暗示FON1与FON4 在控制分生组织发育的通路中共同起作用,因此,有必要进一步探讨ULT1与它们之间的调控关系。
参考文献:
[1]Jaeger K E,Pullen N,Lamzin S,et al.Interlocking feedback loops govern the dynamic behavior of the floral transition inArabidopsis[J].Plant Cell,2013,25(3):820-833.
[2]Stewart D,Graciet E,Wellmer F.Molecular and regulatory mechanisms controlling floral organ development[J].FEBS Journal,2016,doi:10.1111/febs.13640.
[3]Goto K,Kyozuka J,Bowman J L.Turning floral organs into leaves,leaves into floral organs[J].Curr Opin Genet Dev,2001,11(4):449-456.
[5]Immink R G,Gadella T W Jr,Ferrario S,et al.Analysis of MADS box protein-protein interactions in living plant cells[J].Proc Natl Acad Sci USA,2002,99(4):2416-2421.
[6]Honma T,Goto K.Complexes of MADS-box proteins are sufficient to convert leaves into floral organs[J].Nature,2001,409(6819):525-529.
[7]张亚芳,左示敏,陈宗祥,等.水稻花序发育的分子调控研究进展[J].江苏农业科学,2014,42(11):4-8.
[8]卢寰,时振英.水稻穗发育的分子生物学研究进展[J].植物生理学报,2013,49(2): 111-121.
[9]Zhang D B,Yuan Z.Molecular control of grass inflorescence development[J].Annu Rev Plant Biol,2014,65:553-578.
[10]于新,王建军,王才林.水稻花器官发育的分子机理[J].分子植物育种,2013,11(4):617-624.
[11]Laux T,Mayer K F,Berger J,et al.TheWUSCHELgene is required for shoot and floral meristem integrity inArabidopsis[J].Development,1996,122(1):87-96.
[12]Running M P,Meyerowitz E M.Mutations in thePERIANTHIAgene ofArabidopsisspecifically alter floral organ number and initiation pattern[J].Development,1996,122(4):1261-1269.
[13]Clark S E,Running M P,Meyerowitz E M.CLAVATA1,a regulator of meristem and flower development inArabidopsis[J].Development,1993,119(2):397-418.
[14]Running M P,Fletcher J C,Meyerowitz E M.TheWIGGUMgene is required for proper regulation of floral meristem size inArabidopsis[J].Development,1998,125(14):2545-2553.
[15]Carles C C,Lertpiriyapong K,Reville K,et al.TheULTRAPETALA1 gene functions early inArabidopsisdevelopment to restrict shoot apical meristem activity,and acts throughWUSCHELto regulate floral meristem determinacy[J].Genetics,2004,167(4):1893-1903.
[16]Engelhorn J,Moreau F,Fletcher J C,et al.ULTRAPETALA1 andLEAFYpathways function independently in specifying identity and determinacy at theArabidopsisfloral meristem[J].Ann Bot,2014,114(7):1497-1505
[17]Hiei Y,Ohta S,Komari T,et al.Efficient transformation of rice (OryzasativaL.) mediated byAgrobacteriumand sequence analysis of the boundaries of the T-DNA[J].The Plant Journal,1994,6(2):271-282.
[18]Monfared M M,Carles C C,Rossignol P,et al.TheULT1 andULT2 trxG genes play overlapping roles in Arabidopsis development and gene regulation[J].Mol Plant,2013,6(5):1564-1579.
[19]Bottomley M J,Collard M W,Huggenvik J I,et al.The SAND domain structure defines a novel DNA binding fold in transcriptional regulation[J].Nature Structural Biology,2001,8(7):626-633.
[20]Pires H R,Shemyakina E A,Fletcher J C.TheULTRAPETALA1 trxG factor contributes to patterning theArabidopsisadaxial-abaxial leaf polarity axis[J].Plant Signal Behav,2015,10(7):e1034422.
Preliminary Analysis on Sequence and Function of Rice Gene Encoding SAND-domain Protein
CHEN Zhixiong1,DAI Shuangfeng1,XIA Changxuan1,XIE Haimei1,LI Yajuan2
(1.College of Agronomy,South China Agricultural University,Guangzhou510642,China;2.Center of Experimental Teaching for Common Basic Courses,South China Agricultural University,Guangzhou510642,China)
Abstract:To study the sequence and function of rice SAND domain protein,the gene encoding SAND was identified by Blast against the rice genome,and then over-expression vector was constructed to study its gene function by genetic transformation.The results found that the rice genome contains a gene LOC_Os01g57240(named as OsULT1),which encoded amino acid sequence containing SAND domain and ULT B-box consensus sequence.The amino acid sequence similarity between OsULT1 and ULTs of other plants varied from 49.3% to 92.3%, they were highly conservative.The 35S::OsULT1 transgenic lines generated some mutant spikilets,which included under-developed or degenerated paleas,over-developed locidules,the increased number of locidules,the variant number of stamen,two pistils,or extra floret.The results indicates OsULT1 should play important roles in regulation of floral meristem identity in rice.
Key words:Rice(Oryza sativa L.);Floral development;SAND-domain;Gene;Sequence
doi:10.7668/hbnxb.2016.02.001
中图分类号:S511.03;Q78
文献标识码:A
文章编号:1000-7091(2016)02-0001-06
作者简介:陈志雄(1975-),男,福建莆田人,副研究员,博士,主要从事水稻分子遗传与种质创新研究。通讯作者:李亚娟(1979-),女,山东菏泽人,高级实验师,博士,主要从事水稻生殖遗传学研究。
基金项目:国家自然科学基金项目(31271688);广东省自然科学基金项目(S2011010005114)
收稿日期:2016-02-01

