苯胺降解菌群的驯化及培养条件的优化
张 淼,崔岱宗,张 昊,赵 敏
(东北林业大学,黑龙江哈尔滨 150040)
苯胺降解菌群的驯化及培养条件的优化
张淼,崔岱宗,张昊,*赵敏
(东北林业大学,黑龙江哈尔滨150040)
摘要:通过对长期受苯胺污染的土壤进行定向驯化,筛选出能够降解苯胺的高效菌群。经16S rDNA基因扩增测序对高效降解苯胺的混合菌群进行鉴定,初步鉴定该混合菌群分别为Serratia marcescens属、Bacillus属、Escherichia coli.属、Leclercia tamura属。通过单因素变量法对苯胺选择性培养基进行优化,研究表明该混合菌群最适碳源为可溶性淀粉,其最佳质量浓度为2 g/L;最适氮源为氯化铵,其最佳质量浓度为1 g/L;菌群最佳接种量为4%。优化后的苯胺降解率为95%,较未优化前提高了76.4%,对今后探究高效降解苯胺的方法具有重要的应用价值。
关键词:苯胺降解;菌株鉴定;培养基的优化
苯胺是指苯环上的1个氢原子被氨基取代而生成的化合物,广泛应用于染料、农药和医药等领域[1]。随着硝基苯催化氢法的诞生,苯胺产量迅速提高。据不完全统计,我国每年苯胺产量已高达万吨[2]。在生产和使用过程中,部分苯胺未经处理即排放到环境中[3]。这类物质化学性质稳定、难于降解,且具有致癌、致畸、致突变性,严重影响着赖以生存的环境及人类健康。因此,苯胺的无害化处理迫在眉睫[4]。目前,国内外研究者探索了大量处理苯胺废水的方法,主要包括物理化学法和生物法。其中,物理法主要包括吸附、萃取和超声波降解;化学法则主要利用氧化及电氧化技术[5-7]。较物理化学法、生物法反应条件温和,造价低廉,无明显的二次污染,因此采用生物法降解苯胺受到了广泛关注。
近年来,研究者已分离出多株可降解苯胺的菌群,它们分别属于假单胞菌属(Pseudomonas)、从毛单胞菌属(Comamonas)、不动杆菌属(Acinetobacter)、红球菌属(Rhodococcus)、弗拉托菌属(Frateuria)、莫拉克斯氏菌属(Moraxella)和诺卡氏菌属(Nocardia)[1,8-13]。筛选出的菌株降解苯胺的能力各不相同,在降解的过程中有时会出现初级阶段降解速率快、后阶段降解不理想的状态,或者是初级阶段降解速率慢、后阶段降解速率快的现象,这说明现有的技术仍存在不稳定因素[14]。因此,构建一个高效且稳定降解苯胺的菌株是解决生物降解苯胺的重要途径之一。
研究中,笔者从长期受苯胺污染的土壤中驯化并分离出对苯胺具有较强降解能力的菌群。通过16S rDNA序列分析技术对该混合菌群进行鉴定分析,确定混合菌群中所含有的菌株类型及其属性,并在此基础上优化了降解培养条件。
1材料与方法
1.1材料
研究中的环境样品取自于吉林省吉林市某染料厂附近的土壤。
1.2苯胺降解菌株的筛选
称取采集的土样约2 g,放入装有50 mL灭菌蒸馏水的锥形瓶中,加入6~8颗玻璃珠子。将锥形瓶置于37℃的培养箱中,以120 r/min转速振荡3 h,保证土壤与灭菌蒸馏水充分地混合。振荡结束后,将悬浊液静止放置1 h,使颗粒物质沉淀,收取上清液,即为接种的环境样品。
在超净实验台中,将50 mL灭菌处理的LB液体培养基分装到锥形瓶中,然后分别接种1 mL处理过的土壤上清液。置于37℃的恒温培养箱中,培养24 h。采用相同的方法,接种第1代富集菌群的菌液1 mL于新鲜的灭菌LB液体培养基中进行振荡培养,使样品中的菌群可以得到富集和活化。
分装苯胺(100 mg/L)筛选培养基50 mL于锥形瓶中,接种第2次活化菌液1 mL,置于37℃的恒温培养箱过夜进行筛选培养,测量其降解率。采用相同的方法,逐步提高筛选培养基中苯胺的质量浓度至1 000 mg/L,观察苯胺的降解效果,初步得到一个相对稳定的降解苯胺菌群。
将筛选后的混合菌群在LB固体筛选培养基(苯胺质量浓度为1 000 mg/L)中进行梯度稀释划线分离,且对长出的单一菌落进行划线纯培养。
保存选出的降解菌株,用甘油作为保护剂,置于- 40℃冰箱保存。
1.3降解菌株的鉴定
采用16S rDNA序列分析的方法对分离的好氧苯胺降解菌进行鉴定,菌株基因组DNA的提取方法按本研究组之前所描述的方法进行。以提取的基因组DNA为模板,采用细菌通用引物27F:5'- GAGTTTG ATCMTGGCTCAG- 3';1492R:5'- TACGGYTACCTTA CGACTT- 3',对细菌的16S rDNA序列进行扩增。PCR反应条件如下:94℃预变性3 min;94℃变性30 s,56℃退火30 s,72℃延伸90 s,30个循环;72℃保温10 min,4℃保存。将PCR产物用0.8%琼脂糖凝胶电泳(120 V)检测,并用凝胶成像扫描系统照相分析。将纯化后的PCR产物连接至pMD 18- T质粒,过夜后转化到大肠杆菌JM109感受态中,挑取确定为阳性克隆的菌液送到测序公司进行测序。采用Contig Express软件对所测得的序列进行拼接,并在NCBI数据库中进行同源性比对,下载相关序列,确定所获菌群的菌属。
1.4菌体对苯胺降解曲线的测定
将保存的菌种按接种量2%接种于LB液体培养基中,使菌体活化。
将活化后的菌群按接种量2%接种于苯胺筛选培养基中,置于37℃培养箱中培养,每2 h取样1次。
将取出的菌体样品,以转速10 000 r/min离心3 min。取上清液,做苯胺降解率的测定。
苯胺降解率的测定方法:在2.5 mL体系中,加入20 μL样品上清液,980 μL蒸馏水,8 μL KHSO4,调节pH值在1.5~2.0时,加入5 μL 5%的NaNO2,摇匀后静置3 min,再加入氨基磺酸铵50 μL,振荡后静置3 min,最后加入2%的N - 1 -萘基-乙二胺盐酸盐100 μL,加水稀释至2.5 mL,放置10 min,于紫外分光光度545 nm处测定吸光度(以蒸馏水做空白对照),并与标准质量浓度苯胺吸光度进行对比,进而求出降解率;每个样品做1个平行对照。
1.5培养基的优化
1.5.1最适碳源的测定
将50 mL碳源分别为葡萄糖、果糖、蔗糖、乳糖、可溶性淀粉和甘油的筛选培养基(苯胺质量浓度为1 000 mg/L)装入灭菌的锥形瓶中,将活化后的菌群按接种量2%分别加入各锥形瓶。每种碳源设置2个平行试验,置于37℃恒温培养箱中,振荡培养20 h后,取出3 mL菌液,以10 000 r/min转速离心3 min,取上清液,按照上述苯胺降解率的测定方法处理后,于545 nm处测定苯胺的剩余吸光度,计算苯胺的降解率。
1.5.2最适碳源添加量的测定
将50 mL碳源(可溶性淀粉)质量浓度分别为2,4,6,8,10,12 g/L的筛选培养基(苯胺质量浓度为1 000 mg/L)装入锥形瓶中,后续操作方法同1.5.1。
1.5.3最适氮源的测定
将50 mL氮源分别为氯化铵、硝酸钾、硫酸铵、硝酸铵、纯蛋白胨和酵母提取液的筛选培养基(苯胺质量浓度为1 000 mg/L)装入锥形瓶,后续操作方法同1.5.1。
1.5.4最适氮源添加量的测定
将50 mL氮源(氯化铵)质量浓度分别为1,2,3,4,5,6 g/L的筛选培养基(苯胺质量浓度为1 000 mg/L)装入锥形瓶中,后续操作方法同1.5.1。1.5.5最适接种量的测定
将50 mL质量浓度为1 000 mg/L的苯胺筛选培养基装入锥形瓶中,把已经活化的菌株按照1%,2%,3%,4%,5%,6%的接种量分别接种到各锥形瓶中。后续操作方法同1.5.1。
2结果与分析
2.1混合菌群中纯菌株的筛选
采用不断提高培养基中苯胺质量浓度的方法对土壤样品菌群进行驯化,最终得到可以稳定降解苯胺的混合菌群,并对该混合菌群进行划线分离,从而获得18个纯菌株。
2.2纯菌鉴定结果
纯菌株DNA基因组电泳见图1,16S rDNA的PCR扩增结果见图2。

图1 纯菌株DNA基因组电泳
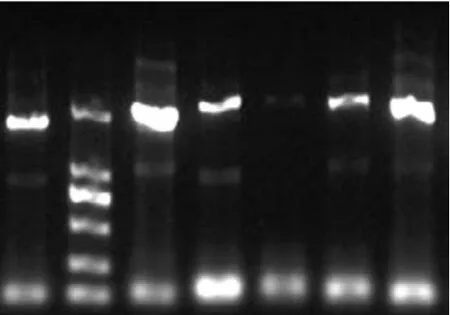
图2 16S rDNA的PCR扩增结果
测序结果表明,纯菌株扩增出的16S rDNA片段大小从1 504~1 514 bp不等,经Gen Bank数据库比对,发现其与沙雷氏菌(Serratia marcescens)、芽孢杆菌(Bacillus)、大肠杆菌(Escherichia coli.)、勒克氏菌(Leclercia tamura)同源性最高,相似度均高达99%以上。目前,已有报道表明芽孢杆菌属有降解苯胺及其派生物的能力。
2.3苯胺降解曲线的测定
苯胺降解率见图3。
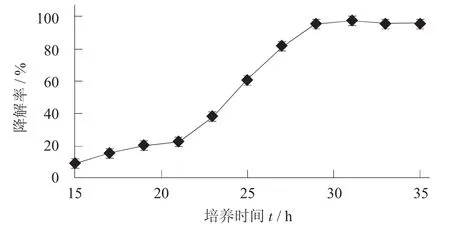
图3 苯胺降解率
由图3可知,混合菌群在苯胺质量浓度为1 000 mg/L的降解培养基中,前15 h降解效果不明显;从15 h开始,降解率有了明显上升;25~29 h时,降解效率提高较快;31 h时,降解率达顶峰,降解效果比较稳定。推测最初的15 h内混合菌群处于延滞期,生长缓慢,对苯胺的降解率很低;从15 h开始,菌群进入对数生长期,菌群生长量明显增加,对苯胺的降解率最快,其降解率高达95%;31 h后菌体生长处于稳定期,对苯胺的降解能力达到平稳状态。
2.4降解条件的优化
2.4.1最适碳源的测定
分别测定了以葡萄糖、果糖、蔗糖、乳糖、可溶性淀粉和甘油作为碳源时,混合菌株在培养基中对苯胺的降解情况。
不同碳源对苯胺降解的影响见图4。
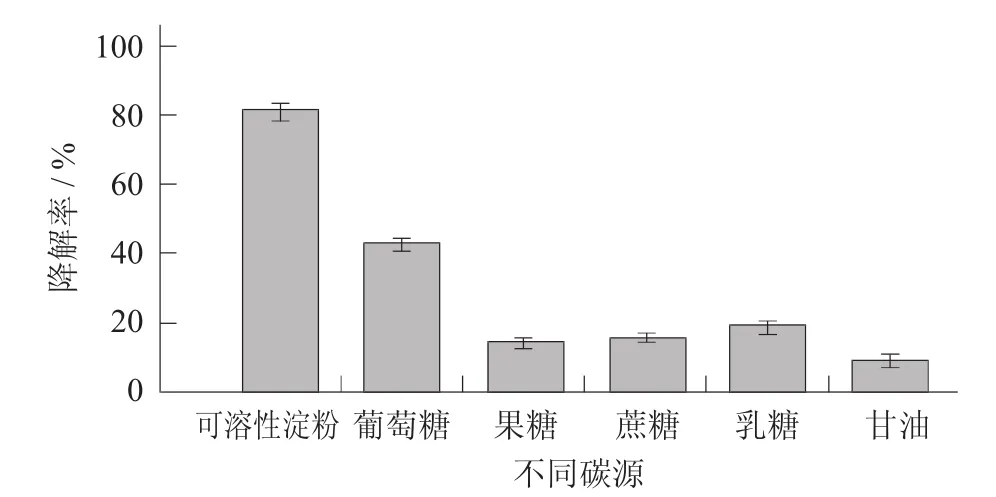
图4 不同碳源对苯胺降解的影响
由图4可知,混合菌株对苯胺降解的最适碳源为可溶性淀粉,在此碳源下约81.3%的苯胺在20 h内被降解,远远高于在其他碳源筛选培养基中的降解率。当以蔗糖、果糖、乳糖、甘油分别作为碳源时,混合菌群对苯胺均能进行降解,但降解效果并不明显,说明这些碳源不能被该菌群充分利用。
2.4.2最适碳源添加量的测定
测定了可溶性淀粉的质量浓度为2,4,6,8,10,12 g/L时,混合菌株对苯胺的降解情况。
不同碳源添加量对苯胺降解的影响见图5。

图5 不同碳源添加量对苯胺降解的影响
由图5可知,在前5组添加量条件下,混合菌群对苯胺的降解率均高于70%,说明可溶性淀粉的质量浓度对苯胺的降解效果并没有明显差异。其中,当可溶性淀粉的质量浓度分别为2 g/L和10 g/L时,降解率均已高于80%,但考虑成本,选择质量浓度为2 g/L的可溶性淀粉作为最适质量浓度。当可溶性淀粉质量浓度为12 g/L时,对苯胺的降解率降至为42%,推测该质量浓度抑制菌群对苯胺的降解能力。
2.4.3最适氮源的测定
研究中测定了在不同氮源的条件下,混合菌群对苯胺降解的效率。氮源分别为氯化铵、硝酸钾、硝酸铵、硫酸铵、纯蛋白胨和酵母提取液。
不同氮源对苯胺降解的影响见图6。
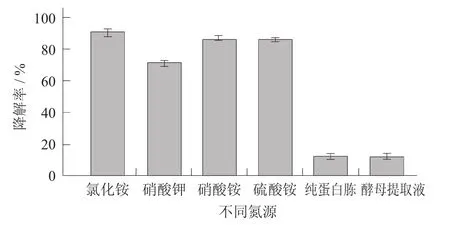
图6 不同氮源对苯胺降解的影响
由图6可知,氯化铵、硫酸铵和硝酸铵分别作为氮源时,混合菌株对苯胺的降解率均超过了80%,说明NH4+作为氮源时,混合菌群对苯胺降解率均很高。其中,氯化铵作为氮源时,苯胺的降解率为91%,对苯胺的降解效果最佳;但当纯蛋白胨和酵母提取液作为氮源时,混合菌群对苯胺的降解效果不很明显,较其他氮源显著下降,这可能是因为有机氮源结构复杂不易被利用。
2.4.4氮源添加量的测定
研究中已确定氯化铵为最佳氮源,因而继续探讨不同氮源质量浓度对苯胺降解的影响。氯化铵质量浓度分别为1,2,3,4,5,6 g/L。
不同氮源添加量对苯胺降解的影响见图7。
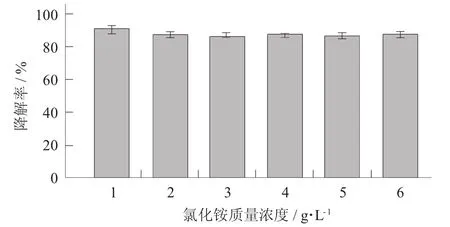
图7 不同氮源添加量对苯胺降解的影响
由图7可知,混合菌株在氯化铵质量浓度从1 g/L 到6 g/L对苯胺的降解效果无明显差异,均达到86%以上。其中,当氯化铵质量浓度为1 g/L时,混合菌群对苯胺的降解率略高于其他氯化铵质量浓度对苯胺的降解率,为91%。因此,选择质量浓度为1 g/L的氯化铵作为氮源。
2.4.5最适接种量的测定
研究中通过改变混合菌群的接种量,观察混合菌群对苯胺降解效果的影响。混合菌群的接种量分别为1%,2%,3%,4%,5%,6%。
混合菌群接种量对苯胺降解的影响见图8。
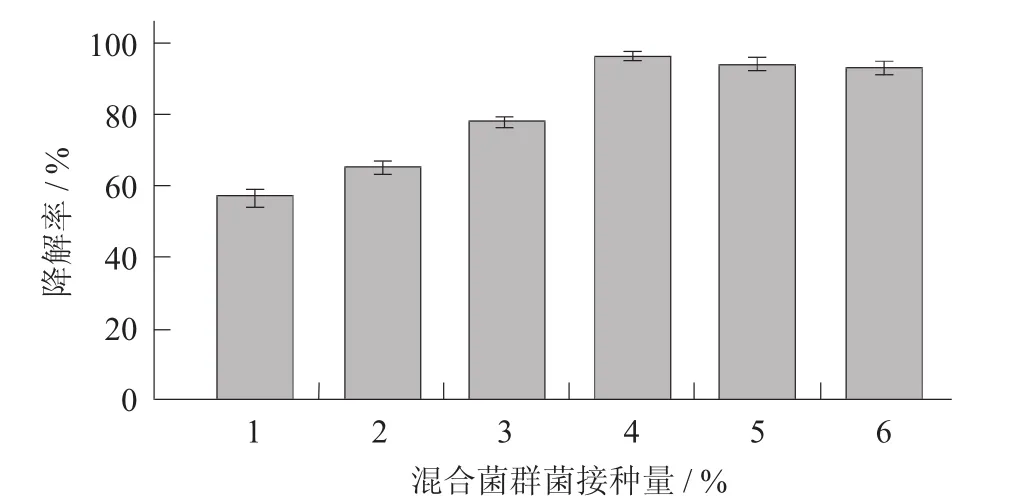
图8 混合菌群接种量对苯胺降解的影响
由图8可知,接种量由1%增加到4%时,随着接种量的逐步递增,混合菌群对苯胺降解率逐步提高;当混合菌群接种量达到6%后,苯胺的降解效果无明显变化。推测当接种量较少时,由于底物相对较多,从而延长了菌群的延滞期,导致苯胺的降解效果不高;随着接种量的提高,底物对菌群的抑制作用逐渐降低,使得延滞期缩短;但接种量过大时,菌群的数量已经达到最大,对苯胺的降解能力也达到最高,所以没有明显的提高。
3 结论
本研究在环境样品中驯化出在好氧条件下对苯胺进行降解的混合菌群,经过分子学鉴定后,该混合菌群属于沙雷氏菌属、芽孢杆菌属、大肠杆菌属、勒克氏菌属;该混合菌群可在好氧条件下较好地降解苯胺。理化因素对混合菌群的影响显示,该混合菌群在2 g/L的可溶性淀粉,1 g/L的氯化铵,混合菌群的接种量为4%时,混合菌群对苯胺的降解率高达95%,而最初的降解培养基在培养20 h时,苯胺降解率为21.15%,优化后降解率提高了76.4%。研究表明,该筛选的混合菌群可以高效地降解苯胺,具有潜在的应用价值。
参考文献:
[1]Bachofer R,Lingens F,Schafer W. Conversion of aniline into pyrocatechol by a Nocardia sp:incorporation of oxygen-18 [J] . FEBS Letters,1975,50:288-290.
[2]Dos Santos A B,Cervantes F J,Yaya-Beas R E,et al. Effect of redox mediator,AQDS,on the decolourisation of a reactive azo dye containing triazine group in a thermophilic anaerobic EGSB reactor [J] . Enzyme and Microbial Technology,2003,33(7):942-951.
[3]吴耀国,曹睿,慧林,等.反硝化条件下河岸渗滤过程中苯胺的降解[J] .地球科学,2006,31(2):273-278.
[4]Keith L H,Telliard W A. Priority pollutants I. A perspective view [J] . Environmental Science and Technology,1979,13(4):416-423.
[5]顾浩飞,安太成,文晟,等.超声光催化降解苯胺及其衍生物研究[J] .环境科学学报,2003,23(5):593-597.
[6]徐越群,刘秉涛.活性炭纤维处理含苯胺废水的试验研究[J] .人民黄河,2009,31(6):70-74.
[7]高宇,何刨龙,傅敏.电化学降解与声电化降解苯胺溶液的对比实验研究[J] .环境污染与防治,2004,26(1):54-56.
[8]Hinteregger C,Loidl M,Streichsbier F. Characterization of isofunctional ring - cleaving enzymes in aniline and 3 -chloroaniline degradation by Pseudomonas acidovorans CA28 [J] . Fems Microbiology Letters,1992,97:261-266.
[9]Parales R E,Ontl T A,Gibson D T. Cloning and sequence analysis of a catechol 2,3-dioxygenase gene from the nitrobenzene- degrading strain Comamonas sp. JS765 [ J] . The Journal of General and Applied Microbiology,1997,19:385-391.
[10]Kim S I,Leem S H,Choi J S,et al. Cloning and characterization of two cata genes in Acinetobacter lwoffii K24 [J] . Journal of Bacteriology,1997,179:5 226-5 231.
[11]Aoki K,Ohtsuka K,Shinke R,et al. Isolation of aniline assimilation bacteria and physiological characterization of aniline biodegradation in Rhodococcus erythropolis AN13 [J] . Agricultural and Biological Chemistry,1983,47:2 569-2 575.
[12]Murakumi S,Takashima A,Takemoto J,et al. Cloning and sequence analysis of two catecholdegrading gene clusters from the aniline - assimilating bacterium Frateuria species ANA-18 [J] . Gene,1999,226:189-198.
[13]Zeyer J,Wasserfallen A,Timmis K N. Microbial mineralization of ring-substituted anilines through an ortho-cleavage pathway [J] . Applied and Environmental Microbiology,1985,50:447-453.
[14]梁泉峰.生物降解菌株Delftia tsuruhatensis AD9中染色体编码的苯胺代谢基因簇的克隆和功能研究[D] .北京:中国农业科学院,2005.
Acclimation of Aniline Degradation Bacteria and the Optimization of Cultivated Conditions
ZHANG Miao,CUI Daizong,ZHANG Hao,*ZHAO Min
(Northeast Forestry University,Harbin,Heilongjiang 150040,China)
Abstract:Through special domestication,mixture strains which could efficiently degrade aniline are isolated from the soil which is endured the pollution of aniline. By analysis of the molecular biology of the 16S rDNA sequence,the strains belonged to Serratia marcescens,Bacillus,Escherichia coli.,Leclercia tamura,respectively. Base on the principle of a single variable factor,this study optimizes the degradation of medium carbon sources,nitrogen sources and the corresponding dosage,inoculum size. The experiments show that the best carbon source for degradation of aniline is starch,whose best concentration is 2 g/L;the growth best nitrogen source is ammonium chloride,whose optimal culture concentration is 1 g/L;the best vaccination is 4%. Optimized aniline degradation is 95%,with an increasing of 76.4%,exhibiting a remarkable degradation capability. This study is meaningful to seek for the approach on degradation of aniline efficiently.
Key words:aniline degradation;strains identification;optimization of culture medium
*通讯作者:赵敏(1964—),男,博士,教授,研究方向为环境微生物。
作者简介:张淼(1989—),女,硕士,研究方向为环境微生物。
收稿日期:2015- 12- 11
文章编号:1671- 9646(2016)03a- 0018- 04
中图分类号:X132
文献标志码:A
doi:10.16693/j.cnki.1671- 9646(X).2016.03.005

