马氏珠母贝SNP标记开发及家系遗传多态性分析
李耀国,刘文广,林坚士,何毛贤
(1.中国科学院南海海洋研究所 中国科学院热带海洋生物资源与生态重点实验室 广东省应用海洋生物学重点实验室,广东 广州 510301;2.中国科学院大学,北京 100049)
马氏珠母贝SNP标记开发及家系遗传多态性分析
李耀国1,2,刘文广1,林坚士1,何毛贤1
(1.中国科学院南海海洋研究所中国科学院热带海洋生物资源与生态重点实验室广东省应用海洋生物学重点实验室,广东广州510301;2.中国科学院大学,北京100049)
摘要:利用13个源自转录组序列的SNP标记对3个马氏珠母贝(Pinctada fucata)家系进行遗传多态性分析及聚类分析。结果显示,家系1#、3#、6#的平均期望杂合度(He)分别为0.307 8、0.318 9和0.382 7;平均观测杂合度(Ho)分别为0.342 2、0.341 0和0.394 2;平均多态信息含量(PIC)分别为0.243 5、0.247 9和0.297 7。结果表明,3个马氏珠母贝家系具低度或中度遗传多态性,为马氏珠母贝内SNP应用于遗传多态性分析等提供了基础。利用SNP标记对3个家系进行聚类分析,发现半同胞家系1#和3#在家系及个体聚类中均先聚在一起,然后再与亲缘关系较远的家系6#聚在一起。新开发的SNP标记能较准确地对家系及个体进行聚类,为SNP标记应用于马氏珠母贝遗传关系分析提供了研究基础。
关键词:马氏珠母贝;SNP;遗传多态性;聚类分析
马氏珠母贝(Pinctada fucata),又名合浦珠母贝,是世界上重要的海水珍珠贝,在中国广东、广西、海南沿海有着广泛分布。海洋贝类养殖过程中普遍存在育种混乱、寄生虫病及环境胁迫等问题,给经济生产带来重大损失(Hine etal,2000;Liu etal,2012)。通过常规的选择和杂交育种可对海水养殖品种进行遗传改良(汤娇雯等,2009)。如利用单对配对法构建生长快、个体大的马氏珠母贝家系及定向选择改良经济性状(何毛贤等,2006;何毛贤等,2007)。随着水产动物的遗传改良进入分子育种时代,利用分子标记进行辅助育种可以更准确地估计个体育种价值及加快育种速度(桂建芳等,2012;Taylor,2014)。随机扩增多态性DNA(杜民等,2013)及扩增片段长度多态性标记(秦溱等,2014)、微卫星(李小宁等,2009;孙立元等,2014)和单核苷酸多态性(SNP)标记(Jiang et al,2011)以及线粒体DNA标记(胡静等,2014)等已广泛应用于水产动物遗传育种研究。
马氏珠母贝中各种分子标记均得到开发及应用。在群体及家系的遗传多态性分析方面,王爱民等(2000)利用随机扩增多态DNA标记对马氏珠母贝天然群体及养殖群体的遗传多样性进行了分析及比较,发现天然群体的遗传多样性大于养殖群体。许成帅等(2013)采用微卫星标记对合浦珠母贝家系的遗传多态性进行了检测,发现总体上生长较快的家系遗传参数平均较大。此外,Huang等(2014)在马氏珠母贝内开发了一批SNP标记并对其群体遗传结构进行了分析。在性状关联分析方面,邓岳文等(2013)开展了马氏珠母贝生长性状与微卫星标记的关联分析,检测到与体质量、壳高、壳宽等显著相关的标记并确定了相应性状的优势基因型。随着下一代测序技术的发展,通过高通量测序在马氏珠母贝内鉴定了大量的SNP标记,构建了高密度遗传连锁图谱并定位了一批数量性状位点(Shietal,2014;Lietal,2014)。在各类分子标记中,SNP在基因组中因数量丰富且易于分型而成为理想的分子标记(Jonesetal,2013)。多种方法如四引物扩增受阻突变体系PCR(Ye et al,2001),高分辨率溶解曲线法(Yu et al,2011),简化基因组测序法(Houston etal,2012)等均被用于SNP标记分型分析。其中四引物扩增受阻突变体系PCR是一种相对简单、快速有效的SNP分型方法(Zhang et al,2013)。该方法对每个SNP位点设计四条引物,并在两条核苷酸特异性引物的3端倒数第3位引入错配碱基以提高引物末端碱基结合特异性。在长牡蛎(Crassostrea gigas Thunber g)(Kong etal,2014)等物种中已利用该方法进行SNP分型分析。
目前针对马氏珠母贝SNP标记的开发及应用还不是很深入,且尚未有关于SNP标记用于马氏珠母贝家系及个体聚类分析的相关报导。该研究基于转录组测序序列开发了13个新的SNP标记,并利用这些标记对3个马氏珠母贝家系进行了遗传多态性分析及聚类分析,为SNP标记应用于马氏珠母贝遗传育种研究提供了基础。
1材料与方法
1.1试验材料及DNA提取
该研究中马氏珠母贝家系于2012年4月采用单对配对的方式构建,亲本选自深圳大鹏澳海区的养殖群体。3个基础研究用家系(8月龄贝)分别为:1#(n= 49,个体编号1-49),3#(n= 49,个体编号50~98)和6#(n=56,个体编号99~154),其中1#和3#为具有共同父本的半同胞家系。对3个家系全部个体分别取闭壳肌用95%乙醇固定。采用E.Z.N.A软体动物DNA提取试剂盒(OMEGA)提取基因组DNA。DNA浓度及质量分别通过分光光度计及1%琼脂糖凝胶电泳检测,稀释至50 ng/μL,-20°C保存。
1.2 SNP分型分析
基于马氏珠母贝转录组测序数据(Huang et al,2013),采用SOAPsnp软件(Li et al,2009)筛选假定的SNP。SNP位点两端的序列长度不短于100 bp,以适于引物设计,序列中无不确定碱基,符合上述标准的转录组序列中SNP位点定义为假定的SNP位点(Huangetal,2014),用于进一步验证分析。利用四引物扩增受阻突变体系PCR在线引物设计程序(http://cedar.genetics.soton.ac.uk/ public_html/primer1.html)对59个假定SNP位点设计引物并进行分型分析。PCR反应体积为25μL,含12.5 μL Premix Ex Taq(Takara),每条引物各0.5 μL(10 pmol/μL),1 μL DNA(50 ng/μL),0.25μLTaq酶(5U/μL)和9.25μLH2O。程序为:94°C预变性5min,94°C30s,合适退火温度下1min,72°C 1min,35个循环;最后72°C延伸10min。PCR产物经3.5%琼脂糖凝胶电泳检测后做分型分析。将含有SNP位点的转录组序列所对应的氨基酸序列在NCBI蛋白质数据库中进行比对寻找同源性序列。将SNP引起的碱基变异与标准氨基酸密码子表进行比较确定是否引起了编码氨基酸的变异。
1.3遗传多态性分析
SNP位点的最小等位基因频率(MAF),观测等位基因数(Na),有效等位基因数(Ne),期望杂合度(He),观测杂合度(Ho),偏离哈代温伯格平衡(HW E)的概率均通过POPGENE version 1.32软件进行计算(Yeh etal,1999)。多态信息含量通过软件PIC-Calc version 0.6(Shao et al,2013)进行计算。
1.4家系及个体聚类分析
基于等位基因频率(Neiet al,1983),利用PowerMarkerversion 3.25(Liu etal,2005)软件对马氏珠母贝1#、3#和6#家系间及个体间遗传距离进行计算。基于非加权配对算数平均法,使用MEGA 5(Tamura etal,2011)分别绘制3个家系及个体聚类图。
2实验结果
2.1 SNP引物序列及变异类型
对59个假定SNP位点进行四引物扩增受阻突变体系PCR检测,结果显示13个位点具有多态性,其引物及特征见表1。在59个假定SNP中,C/T转变所占比率最高(33.8%)。其中SNP U101671-455可导致脯氨酸转变为谷氨酸,SNP U101773-373可导致丝氨酸转变为天冬酰胺。结果见表1。
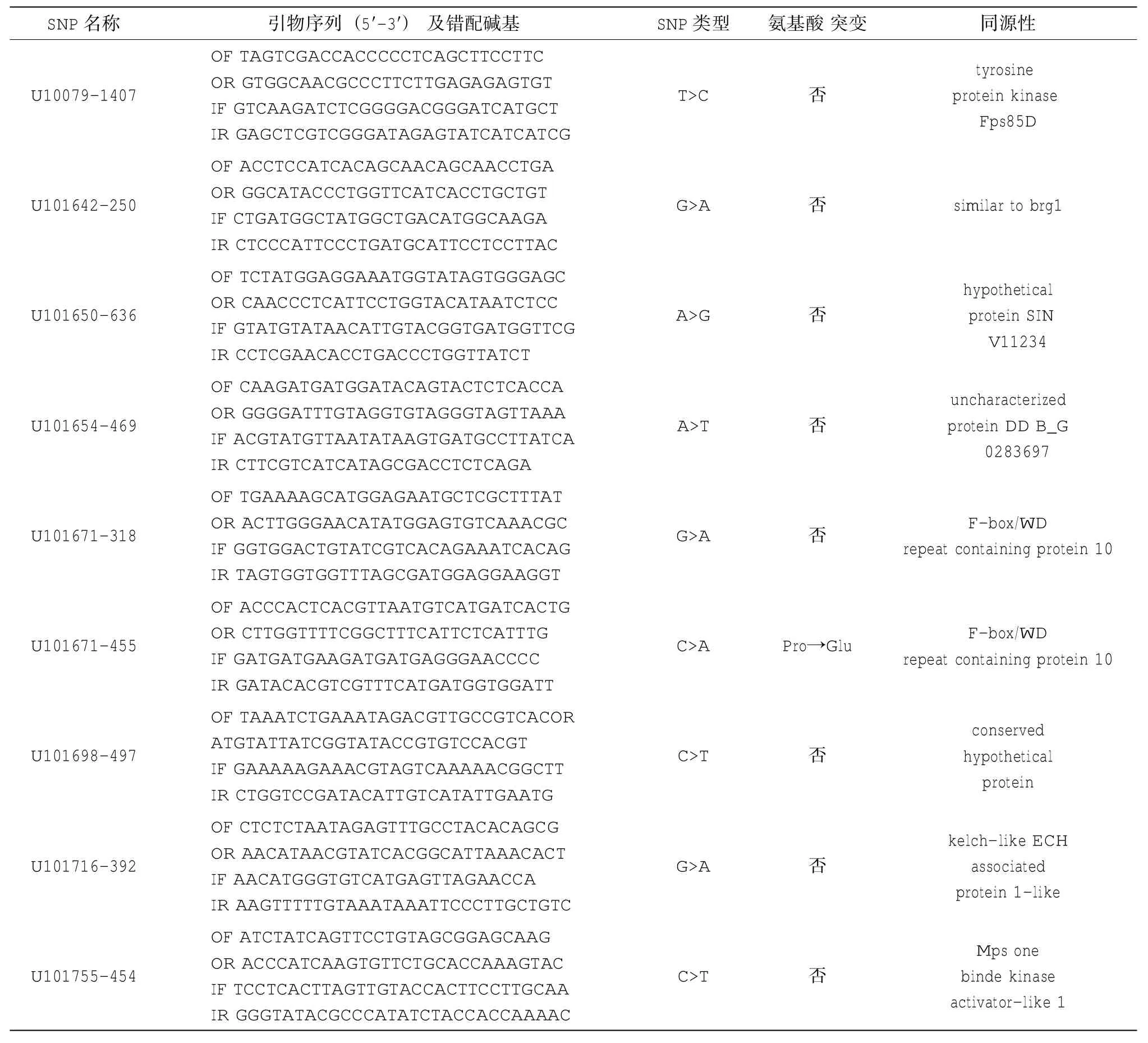
表1 马氏珠母贝13个SNP标记的特征及引物序列
2.2家系遗传多态性分析
利用经过分型验证的13个SNP标记对马氏珠母贝3个家系进行遗传多态性分析,四引物扩增受阻突变体系PCR如图1所示。结果显示家系1#、3#和6#的平均期望杂合度(He)分别为0.307 8、0.318 9和0.382 7;平均观测杂合度(Ho)分别为0.342 2、0.341 0和0.394 2。两个半同胞家系1#和 3#的平均PIC值分别为0.243 5和0.247 9,均属于较低的多态性(PIC<0.25);家系6#的平均PIC值分别为0.297 7,属于中度多态性(0.25 < PIC < 0.5)(Botstein,1980)。

续表1

表2 SNP标记对3个马氏珠母贝家系遗传多态性分析
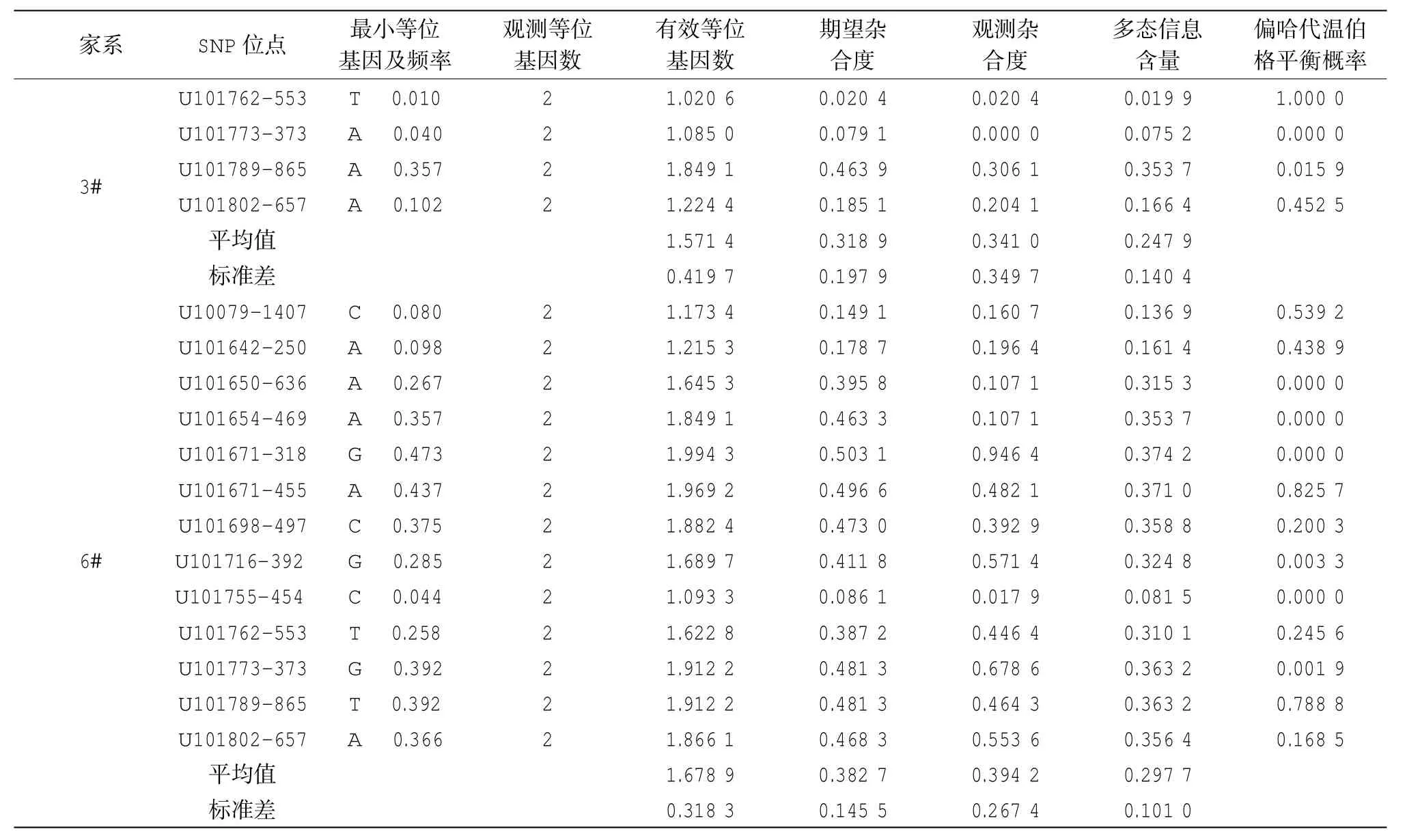
续表2
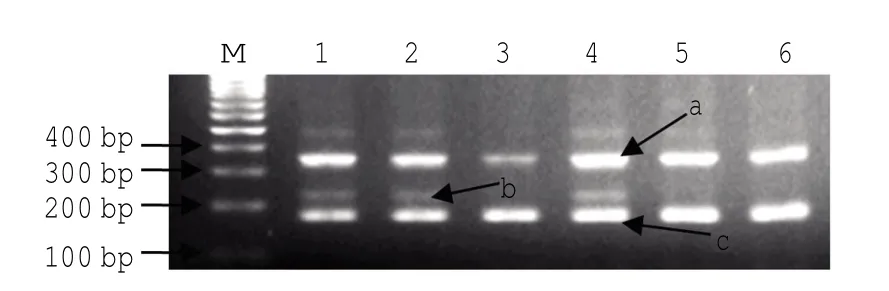
图1 马氏珠母贝SNP位点U101716-392四引物扩增受阻突变体系PCR电泳图(M泳道为100 bp DNA marker,1-6对应不同样品PCR扩增产物;a对应外扩增产物,大小为358 bp,b为等位基因G对应产物,大小为231 bp,c为等位基因A对应产物,大小为183 bp)
2.3马氏珠母贝家系及个体聚类分析
基于1#、3#和6#家系间及个体间的遗传矩阵对家系及个体进行聚类分析,结果如图2所示。其中a为半同胞家系1#和3#先聚在一起,6#则在另外分支。个体聚类分析中,来自家系1#和3#的个体总体上先聚在一起,再与家系6#的个体聚类。半同胞家系1#和3#的遗传距离最近(0.054 7),家系3#和6#的遗传距离最远(0.081 4)。
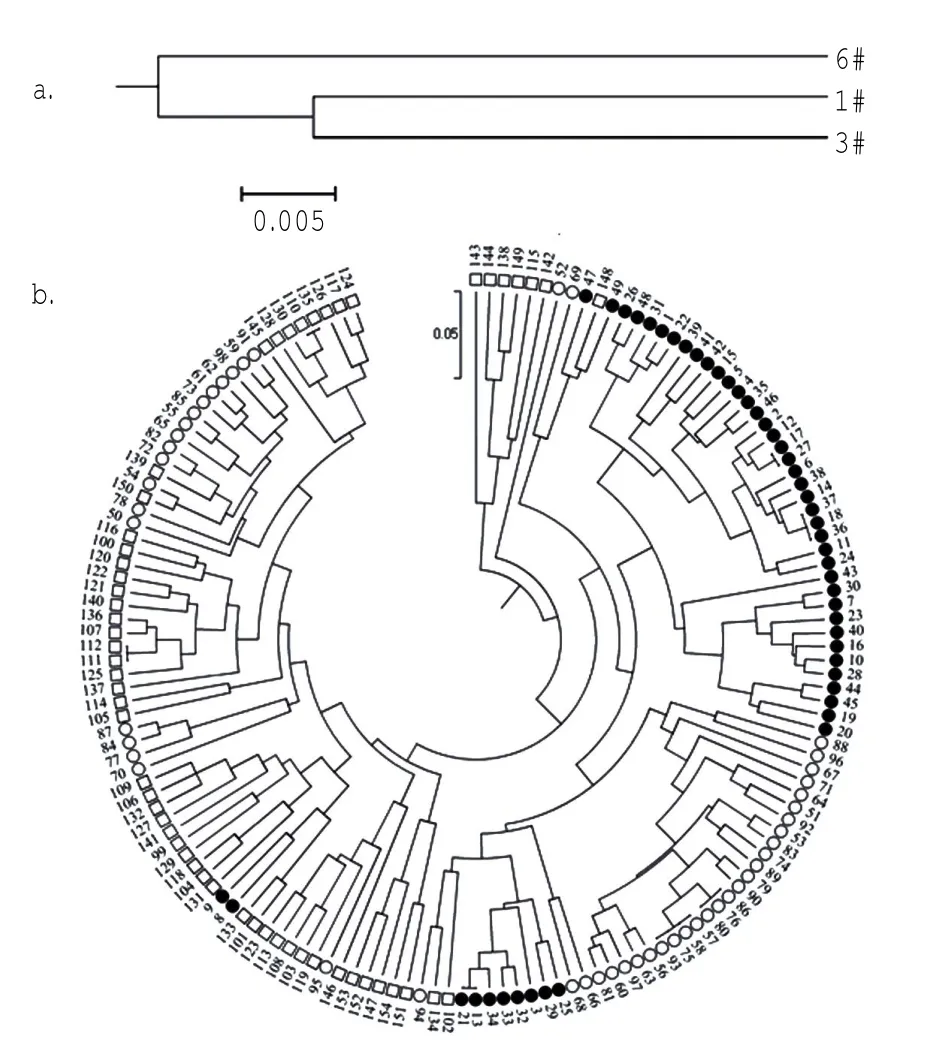
图2 马氏珠母贝家系及个体聚类分析a.马氏珠母贝3个家系聚类图;b.马氏珠母贝3个家系全部个体聚类图,●为1#内个体,○为3#内个体,□为6#内个体。
3讨论
3.1 SNP类型
3.2家系遗传多态性分析
利用SNP标记对马氏珠母贝3个家系进行分析,结果显示其平均PIC值分别为0.243 5、0.2479 和0.297 7。相对于许成帅等(2013)报导的利用微卫星标记分析合浦珠母贝家系的遗传多态性低(壳长速长与慢长家系PIC值分别为0.31和0.39)。该研究中马氏珠母贝遗传多态性处于较低或中等的原因可能有以下两个方面:一是与微卫星标记相比,SNP标记的多态信息含量较低(Kong et al,2014);二是家系构建过程中存在的对性状(基因型)的选择,可能降低了该研究中家系的遗传多态性。SNP标记相对于微卫星等DNA标记等位基因数少,但其具有较高比例的非同义突变(如在13 个SNP标记中存在两个非同义突变),可直接产生氨基酸变异,可能更利于开展性状关联分析及数量性状位点定位。家系1#和3#为半同胞家系,根据SNP标记遗传多态性分析结果可知这两个家系具有相近的遗传多态性。家系1#和3#的平均PIC值(0.243 5和0.247 9),平均He值(0.307 8和0.318 9)和平均Ho值(0.342 2和0.341 0)均是3个家系中最接近的,与亲缘关系一致。说明这些SNP标记可以用于家系遗传多态性分析。
3.3家系及个体聚类分析
分子标记中,扩增片段长度多态性(代悦等,2010)、微卫星标记(杜晓东等,2011)已被用于马氏珠母贝家系聚类分析,并能正确地对家系进行聚类。该研究首次尝试利用SNP标记对马氏珠母贝家系及个体分别进行聚类分析。根据结果可知同父异母的半同胞家系1#和3#先聚在一起,再与亲缘关系较远的6#聚类。同时从遗传距离计算的结果可知家系1#和3#的遗传距离最近(0.0547),家系6#则在另外的分支,与家系1#及3#的遗传距离分别为0.0814和0.0613。聚类结果与三个家系的亲缘关系相符合,表明可通过这些SNP标记对马氏珠母贝家系进行准确的聚类。对3个马氏珠母贝家系的全部个体进行聚类分析的结果显示,除小部分个分散分布于其它家系中外,大部分的个体均按照各自家系来源聚在一起。相对于其它分子标记如微卫星标记及扩增片段长度多态性标记,SNP主要由两个等位基因组成,代表的多态性相对较低(Kongetal,2014),要想更准确地对个体进行聚类可能需要开发更多的标记来增加聚类能力。
马氏珠母贝自古以来即是培育优良珍珠的母贝。由于过度捕捞及生存的海域环境受到破坏,其物种资源受到很大的破坏(Liu etal,2012)。人工成功繁殖马氏珠母贝以来,存在明显的育种混乱等问题并导致了马氏珠母贝种质衰退,育珠质量下降(何毛贤等,2006;王学颖等,2012)。为解决这些问题,有必要对马氏珠母贝提纯复壮,以保持其优良性状。传统的杂交、混合选育、家系构建等对马氏珠母贝优良性状的选择起到了明显的作用。而快速发展的基于基因组的分子标记技术则进一步加强了水产动物育种精度及速度。本研究结合传统的家系构建及快速发展的分子标记技术,利用13个转录组来源的SNP标记进行了家系遗传多态性分析。三个马氏珠母贝家系处于较低或中度的遗传多态性水平,可能与亲本的遗传背景有关。因而可有选择性地对构建的家系采用回交等方式恢复其遗传多态性,以利于马氏珠母贝种质资源的保护。
参考文献
Botstein D,W hite R L,Skolnick M,etal,1980.Construction of a genetic linkage map in man using restriction fragment length polymorphisms.Am JHum Genet,32 (3):314-331.
Cooper D N,Krawczak M,1989.Themutational spectrum of single basepair substitutions causing human genetic disease:patterns and predictions.Hum Genet,85 (1):55-74.
HayesB,Laerdahl JK,Lien S,etal,2007.An extensive resourceofsingle nucleotide polymorphism markers associated with Atlantic salmon (Salmo salar) expressed sequences.Aquaculture,265 (1-4) :82-90.
对大型植物的监测,在植被繁茂季节对沟道每1km布设1个调查点,如遇到生态条件突变则加测一点,共布设调查点9个。每个点根据实地特点设置 1~2个 5 m×5 m的乔灌样方,2~3 个 1 m×1 m 草本样方,0~2个1 m×1 m水生植物样方。调查植物种类与数量。
Hine P M,Thorne T,2000.A survey of some parasites and diseases of several species of bivalve mollusc in northern W estern Australia.Dis AquatOrgan,40 (1):67-78.
Houston R D,Davey JW,Bishop S C,et al,2012.Characterization of QTL-linked and genome-wide restriction site-associated DNA(RAD) markers in farmed Atlantic salmon.BMC Genomics,13:244.
Huang X D,Zhao M,Liu W G,etal,2013.Gigabase-scale transcriptome analysis on four speciesof pearl oysters.Mar Biotechnol(NY),15 (3):253-264.
Huang X D,W u S Z,Guan Y Y,et al,2014.Identification of sixteen single-nucleotide polymorphism markers in the pearl oyster,Pinctada fucata,for population genetic structure analysis.JGenet,93 (1):e1-e4.
Jiang G D,Li JQ,Li L,etal,2011.Development of 44 gene-based SNP markers in Zhikong scallop,Chlamys farreri.Conserv Genet Resour,3 (4):659-663.
Jones D B,Jerry D R,KhatkarM S,et al,2013.A high-density SNP genetic linkage map for thesilver-lipped pearl oyster,Pinctada maxima:avaluable resource for gene localisation andmarkerassisted selection.BMC Genomics,14:810.
Kong L F,Bai J,Li Q,2014.Comparative assessment of genomic SSR,EST–SSR and EST–SNP markers for evaluation of the genetic diversity of wild and cultured Pacific oyster,Crassostrea gigas Thunberg.Aquaculture,420-421 (supplement1):S85-S91.
LiR,Yu C,Li Y,etal,2009.SOAP2:an improved ultrafast tool for short read alignment.Bioinformatics,25 (15):1966-1967.
Li Y,He M,2014.Genetic Mapping and QTL analysisof growth-related traits in Pinctadafucata using restriction-site associated DNA sequencing.PLoSONE 9 (11):e111707.
LiuK,Muse SV,2005.PowerMarker:an integrated analysis environment forgeneticmarkeranalysis.Bioinformatics,21 (9):2128-2129.
Liu W G,Huang X D,Lin J S,et al,2012.Seawater acidification andelevatedtemperature affect gene expression patterns of the pearl oyster Pinctada fucata.PLoSONE,7 (3):e33679.
Nei M,Tajima F,Tateno Y,1983.Accuracy of estimated phylogenetic trees from molecular data.JMol Evol,19 (2):153-170.
Shao K,XiongMH,Nian X,etal,2013.Characterization ofmicrosatellite loci in Sinilabeo rendahli and cross-amplification in four other Chinese cyprinid species.Conserv GenetResour,5 (1):9-13.
Shi Y H,W ang S,Gu Z F,et al,2014.High-density single nucleotide polymorphisms linkage and quantitative trait locusmapping of the pearl oyster,Pinctada fucata martensii Dunker.Aquaculture,434:376-384.
Tamura K,Peterson D,Peterson N,et al,2011.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Mol Biol Evol,28 (10):2731-2739.
Taylor J F,2014.Implementation and accuracy of genomic selection.Aquaculture,420-421 (supplement1):S8-S14.
Yeh FC,Yang R,Boyle T,etal,1999.POPGENE version 1.32,the userfriendly shareware for population genetic analysis [M] .Canada:Molecular Biologyand Biotechnology Centre,UniversityofAlberta.
YeS,Dhillon S,KeX,et al,2001.An efficient procedure for genotyping singlenucleotide polymorphisms.Nucleic AcidsRes,29 (17):e88.
Yu H,He Y,W ang X,et al,2011.Polymorphism in a serine protease inhibitor gene and its association with disease resistance in the eastern oyster (Crassostrea virginica Gmelin) .Fish Shellfish Immunol,30 (3):757-762.
Zhang JY,W ang W J,Kong J,et al,2013.Construction of a genetic linkagemap in Fenneropenaeus chinensis using SNP markers.Russ JMar Biol,39 (2):136-142.
代悦,喻子牛,赵晓霞,等,2010.马氏珠母贝4个壳色选育系F1遗传结构的AFLP分析.基因组学与应用生物学,29(2):252-258.
邓岳文,高远镇,王学颖,等,2013.马氏珠母贝生长性状与ESTSSR标记的关联分析.农业生物技术学报,21(1):77-88.
杜民,尹绍武,刘艳红,等,2013.4种裸胸鳝的分子遗传多样性和亲缘关系的RAPD分析.海洋通报,32(3):321-327.
杜晓东,高远镇,邓岳文,等,2011.利用微卫星标记进行马氏珠母贝家系遗传结构分析与系谱认证.水产学报,35(12):1795-1804.
桂建芳,朱作言,2012.水产动物重要经济性状的分子基础及其遗传改良.科学通报,57(19):1719-1729.
何毛贤,管云雁,林岳光,等,2007.马氏珠母贝家系的生长比较.热带海洋学报,26(1):39-43.
何毛贤,史兼华,林岳光,等,2006.马氏珠母贝选育子一代生长特性研究.热带海洋学报,25(1):19-22.
胡静,侯新远,尹绍武,等,2014.基于mtDNA ND1基因序列研究不同地理群体波纹唇鱼的遗传多样性和遗传分化.海洋通报,33(2):163-168.
李小宁,张殿昌,朱彩艳,等,2009.合浦珠母贝微卫星DNA标记分离与分析.福建水产(1):48-54.
秦溱,王晓清,曾志南,等,2014.泥东风螺4个群体遗传多样性的AFLP分析.湖南农业大学学报(自然科学版),40(3):299-304.
孙立元,郭华阳,朱彩艳,等,2014.卵形鲳鲹育种群体遗传多样性分析.南方水产科学,10(2):67-71.
汤娇雯,张富,陈兆波,2009.中国海水养殖种类遗传育种进展与发展趋势.南方水产,5(4):77-84.
王爱民,邓凤姣,张锡元,等,2000.马氏珠母贝遗传多样性的RAPD分析.武汉大学学报(自然科学版),46(4):467-470.
王学颖,高远镇,杜晓东,等,2012.马氏珠母贝金黄壳色系F3和基础群体遗传结构比较.海洋通报,31(3):324-328.
许成帅,范嗣刚,黄桂菊,等,2013.合浦珠母贝家系遗传多样性与性状相关性.广东农业科学(12):156-158.
(本文编辑:袁泽轶)
Developm entof SNP m arkers in Pinctada fucata and its app lication for fam ily genetic analysis
LIYao-guo1,2,LIU W en-guang1,LIN Jian-shi1,HE Mao-xian1
(1.CASKey LaboratoryofTropicalMarine Bio-resourcesand Ecology,Guangdong ProvincialKey LaboratoryofApplied Marine Biology,South China Sea InstituteofOceanology,Chinese AcademyofSciences,Guangzhou 510301,Guangdong,China;2.UniversityofChinese AcademyofSciences,Beijing100039,China)
Abstract:A transcription databasewas tracked to identify and validate SNP markers.A totalof thirteen SNPswere used for genetic analysis in Pinctada fucata.For Pinctada fucata family 1#,3#and 6#,the average expected heterozygosities(He)were 0.307 8,0.318 9 and 0.382 7,respectively;the average observed heterozygosities(Ho)were 0.342 2,0.341 0 and 0.394 2,respectively;and the average polymorphism information contents(PIC)were 0.243 5,0.247 9 and 0.297 7,respectively.Thehalfsibling family 1#and 3#were firstly clustered together in the dendrogram,and then clusteredwith family 6#,which was in accordance with the genetic relationship.The developmentof novel SNPs in Pinctada fucata can provide genetic tools forgenetic diversity analysisand clusteringanalysis.
Keywords:Pinctada fucata;single nucleotide polymorphism;genetic diversity;clusteranalysis
通讯作者:何毛贤,博士,研究员,从事海洋贝类遗传育种研究。电子邮箱:hmx@scsio.ac.cn。
作者简介:李耀国(1986-),男,博士研究生,从事海洋贝类遗传育种研究。电子邮箱:yaoguolijkl@163.com。
基金项目:国家高技术研究发展计划(863计划)(2012AA10A410);广东省海洋渔业科技推广专项(A201301A03);广东省科技计划(2013B020308005)。
收稿日期:2015-01-08;
修订日期:2015-04-07
Doi:10.11840/j.issn.1001-6392.2016.01.013
中图分类号:S968.31
文献标识码:A
文章编号:1001-6932(2016)01-0096-07

