Illumina Miseq平台深度测定酸奶中微生物多样性
智楠楠,宗 凯,杨捷琳,姚 剑,*,魏兆军
(1.安徽出入境检验检疫局技术中心,安徽合肥 230032;2.上海出入境检验检疫局,上海 200135;3.合肥工业大学食品科学与工程学院,安徽合肥 230009)
Illumina Miseq平台深度测定酸奶中微生物多样性
智楠楠1,3,宗 凯1,杨捷琳2,姚 剑1,*,魏兆军3,*
(1.安徽出入境检验检疫局技术中心,安徽合肥 230032;2.上海出入境检验检疫局,上海 200135;3.合肥工业大学食品科学与工程学院,安徽合肥 230009)
目的:建立Illumina Miseq深度测序筛查酸奶中微生物的方法,分析酸奶中微生物的多样性。方法:以9种酸奶样品为研究对象,提取酸奶中细菌基因组DNA,应用高通量测序技术测定细菌的 16s rDNA v1-v3 变异区序列,得到9种样品中微生物群体分布和丰度、以及不同样品间的菌种差异与进化关系。结果:酸奶中微生物主要为厚壁菌门(Firmicutes),占据99.6%之高,其中厚壁菌门主要以链球菌属(Streptococcus、87.1%)、乳杆菌属(Lactobacillus、10.3%)、乳球菌属(Lactococcus、0.3%)组成,9种酸奶中有3种同时含有链球菌属和乳杆菌属,其余6种样品中链球菌属几乎占据全部(>97%)。结论:Illumina Miseq 深度测序技术可快速精确深入掌握酸奶中微生物多样性,结果对比显示不同酸奶菌种同质化程度高,其中链球菌属占绝对优势,同时发现部分酸奶标签标示不符。
Illumina Miseq,酸奶,微生物,多样性
乳酸菌(lactic acid becteria)是一类能在可利用的碳水化合物发酵过程中产生大量乳酸的细菌的总称[1]。乳酸菌添加到牛奶或羊奶中可发酵制得酸奶,具有改善腹泻、改善人体肠道菌群平衡、提高营养物质生物利用度、增强机体免疫和抗癌的独特功能[2-6]。但调查发现目前市场上销售的众多酸奶中,在乳酸菌的标示上问题较多[7],存在食品安全隐患,故对此类产品进行菌群鉴别尤为重要。

表1 样品及产地
鉴别酸奶乳酸菌的传统方法是梯度稀释平板培养,观察菌落形态、革兰氏染色、KOH实验、过氧化氢酶实验,测定气体的形成等理化方法[8],但由于微生物的多样性和复杂性,且有些具有不可培养性或培养后难以鉴别,故传统方法无法准确真实的捕捉微生物群落多样性[9]。近年来随着深度测序技术的发展,可从分子生物学角度出发来研究食品中的微生物群落组成,直接从样品中提取全基因组用于Illumina Miseq 高通量深度测序,可以有效克服传统方法中出现的问题[10-12]。
Illumina Miseq高通量深度测序技术在国外已广泛应用于农业、林业、牧业、矿井、医学、微生物等诸多领域,其中在医学微生物方面主要用于对一些基质复杂的样本如人体肠道,皮肤表层等进行菌群多样性分析,以协助判断治疗由于菌群失调引起的某些疾病,取得显著成果[13-14]。随着此技术的推广,近年来国内出现利用此技术针对土壤、含菌空气、植物果实、动物粪便等进行菌群鉴别[15-19],获得不同环境下相应微生物的种群分布情况,测序结果精确可靠。本研究将Illumina Miseq高通量测序技术应用于含菌食品中的微生物鉴别,旨在通过分析细菌16S rDNA的可变区序列获得9种酸奶中微生物组成,初步探讨目前市售酸奶中微生物多样性及分布情况,为消费者在选购酸奶方面提供理论依据及为生产厂家研制新型发酵酸奶提供参考。
1 材料与方法
1.1 材料与试剂
常见的9种酸奶 采购市场,见表1;溶菌酶 上海励瑞生物科技有限公司;蛋白酶k 上海励瑞生物科技有限公司;TE缓冲液 北京索莱宝科技有限公司;缓冲液GA 天根生化科技(北京)有限公司。
PTC-200PCR仪 美国BIO-RAD公司;Stratos冷冻高速离心机、ULT-1786-6V超低温冰箱 美国赛默飞世尔科技公司;Illumina Miseq测序仪 美国Illumina公司;细菌基因组DNA提取试剂盒(离心柱型) 天根生化科技(北京)有限公司;Simplicity超纯水仪 德国Merck Millipore公司。
1.2 实验方法
1.2.1 样品基因组DNA提取 取20 mL酸奶4000 r/min离心5 min,取全部上清12000 r/min离心10 min使菌体完全沉淀,向沉淀中加入200 μL缓冲液GA,振荡至菌体彻底悬浮,加入溶菌酶37 ℃处理40 min,之后按提取试剂盒步骤操作进行基因组DNA提取。
1.2.2 PCR扩增 针对细菌16s rDNA v1-v3区,选用特异性引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和534R(5′-ATTACCGCGGCTGCTGG-3′)进行扩增。 PCR条件是分别保持50 ℃ 2 min、95 ℃ 3 min,然后执行95 ℃ 20 S、58 ℃ 30 S、72 ℃ 2 min共30个循环,最后 4 ℃条件下保存。
1.2.3 Illuminna Miseq测序 将上述九种样品的PCR产物送去上海昂朴生物科技有限公司进行高通量测序。
2 结果分析
2.1 测序信息
本实验共测序样品9个,每个样本获得的原始测序reads数目(raw-Reads)和去除嵌合体后每个样本获得的测序reads(clean-Reads)数目见表2。
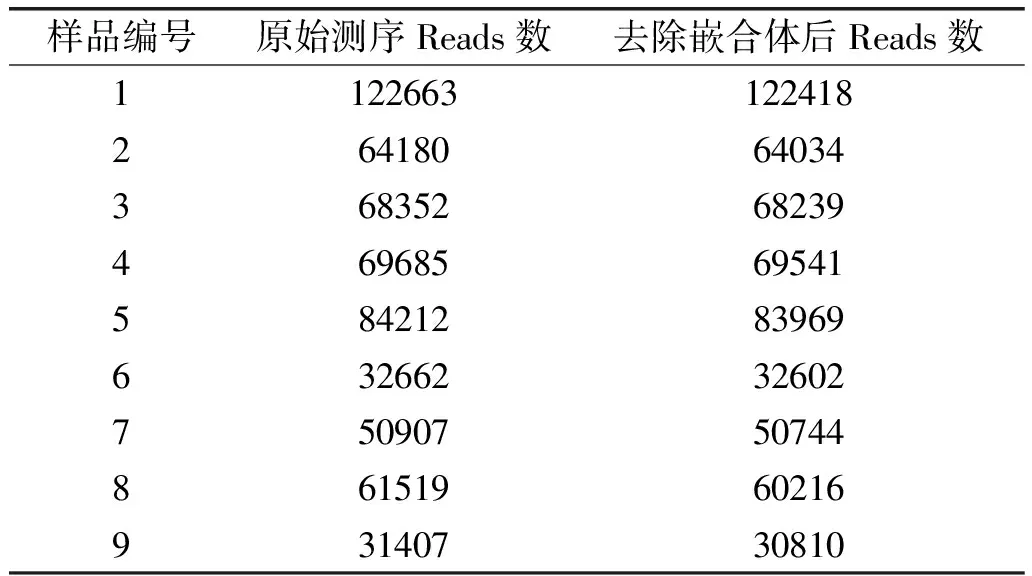
表2 样本reads分布统计
2.2 稀释曲线
采用对测序序列进行随机抽样的方法,以抽到的序列数与它们所能代表OTU的数目构建曲线,即稀释性曲线。当曲线趋于平坦时,说明测序数据量合理,此时再测更多的数据量对发现新OTU的边际贡献很小;反之则表明继续测序还可能产生较多新的OTU。因此,通过作稀释性曲线,可以得出样品的测序深度情况。由图1可知多数样本在序列数在接近40000时,observed_species/OTU已经接近饱和,说明再增加测序量对于发现新species/OTU的边际贡献很小。
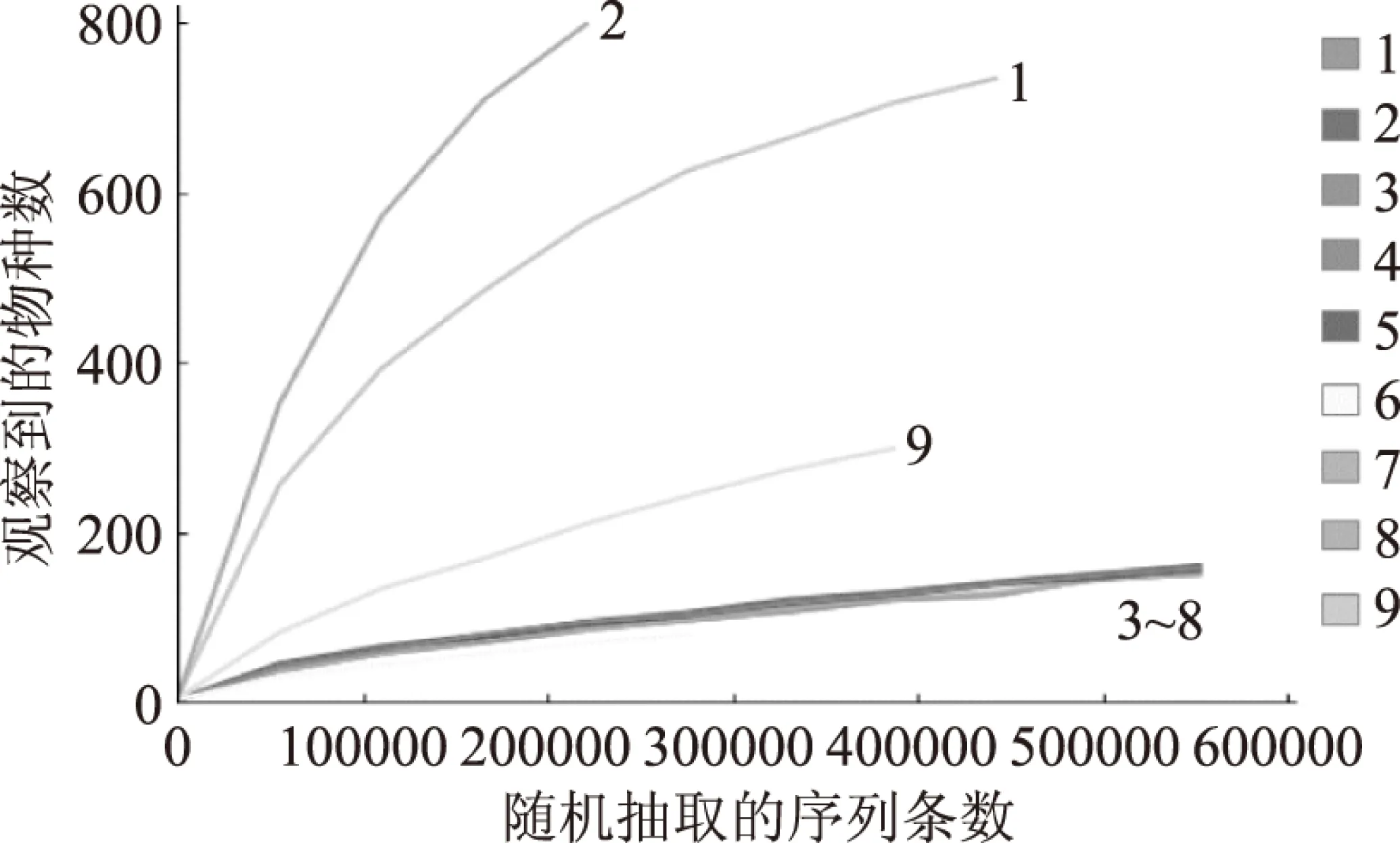
图1 样品稀释性曲线Fig.1 rarefaction curve analysis of nine samples注:横坐标为用于取样的序列数目,纵坐标为观测到的speceis/OTU。
2.3 alpha多样性指数统计
可通过多项指数来评价样品中微生物分布的alpha多样性。其中衡量菌群丰度(Community rich-ness)的指数有:菌种丰富度指数(chao1)和观测到的物种数(observed_species),其中chao1用来估计群落中的OTU数目。衡量菌群多样性(Community diversity)的指数有香农指数(shannon)和辛普森指数(simpson),其中香农指数用于衡量群落的异质性,辛普森指数代表随机取样的两个体属于不同种的概率,其值越大,代表样品物种分布越均匀。由表3以四个衡量指数详细列出了不同样品中微生物的丰富度和多样性分布情况,由香浓指数和辛普森指数可以看出 9号样品物种丰富度最高且物种分布最均匀。
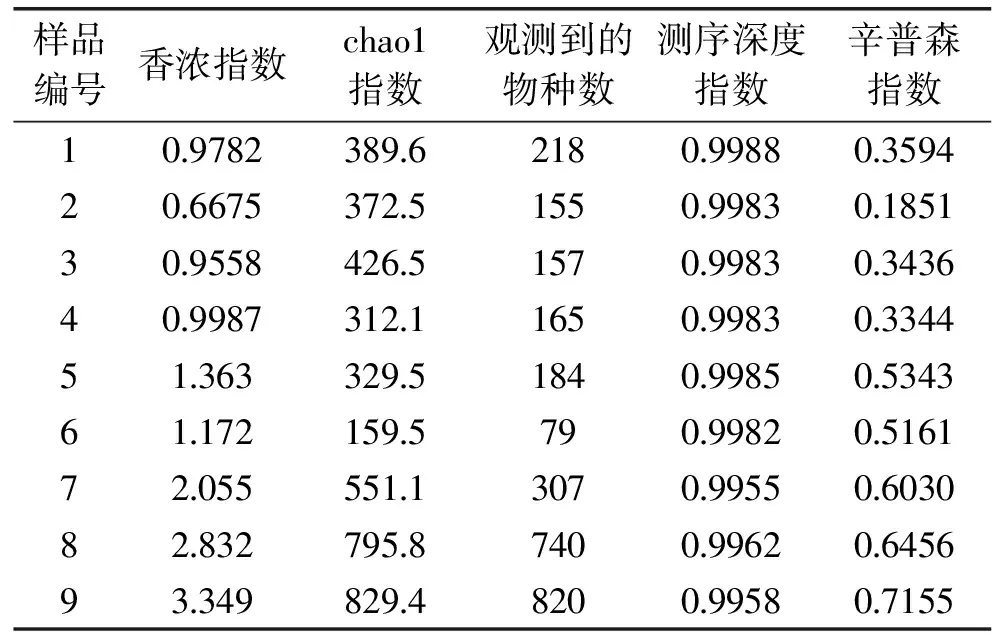
表3 alpha多样性指数统计
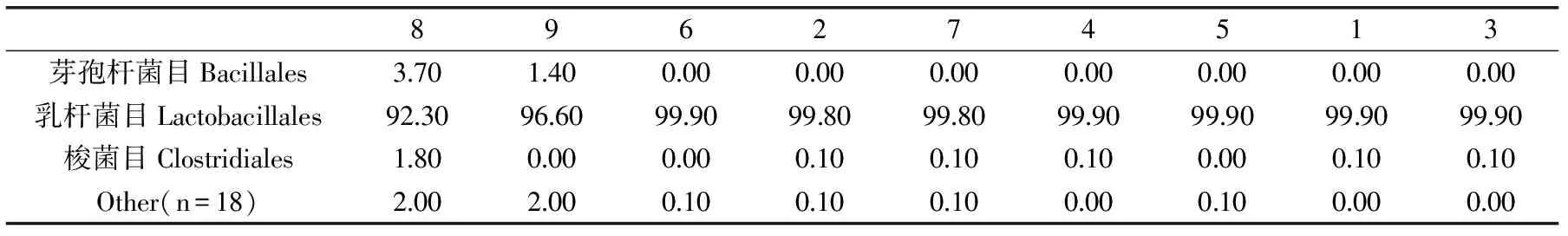
表4 目层面上各样品含菌百分比(%)
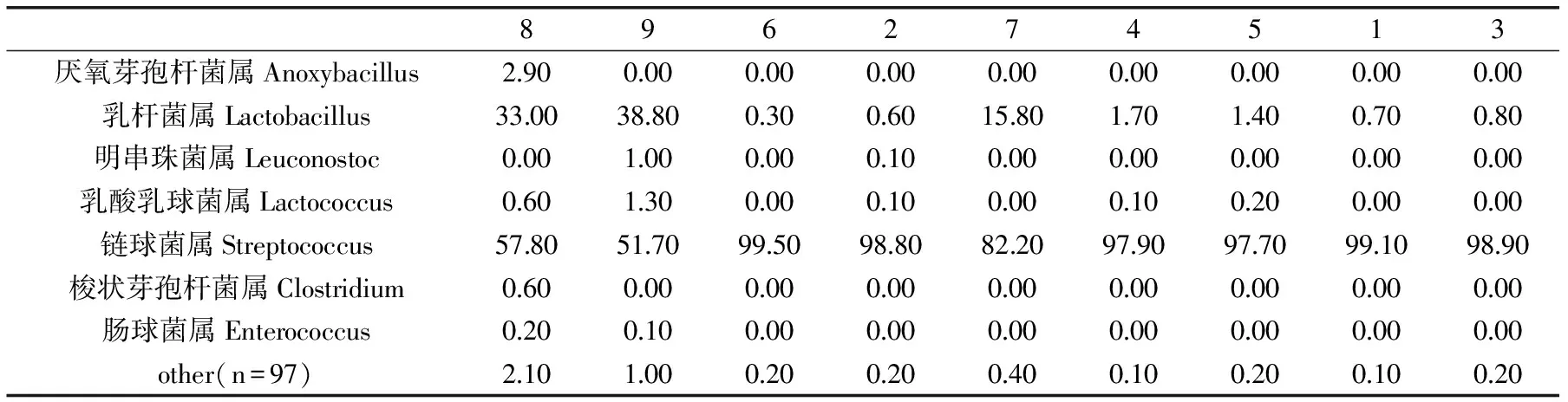
表5 属层面上各样品含菌百分比(%)
2.4 酸奶中微生物分布比较
所测序列片段通过物种注释共可分为7门13纲21目62科104属。在不同分类水平上相对丰度低于0.1%的合并为other。从门的分类水平来看主要为厚壁菌门(Firmicutes、99.6%),从纲的分类水平来看主要为芽孢杆菌纲(Bacill、99.3%)。从目的分类水平来看主要包括乳杆菌目(Lactobacillales、98.7%)、芽孢杆菌目(Bacillales、0.6%)、梭菌目(Clostridiales、0.2%)(表4)。由以上三个分类水平和alpha多样性指数统计综合分析可以看出市场上绝大多数酸奶中所含微生物基本都属于厚壁门芽孢杆菌纲乳杆菌目,不同样品间虽然物种均匀度相差较大,但相似度很高。
从属的分类水平来看主要包括链球菌属(Streptococcus、87.1%)、乳杆菌属(Lactobacillus、10.3%)、乳酸乳球菌属(Lactococcus、0.3%)、厌氧芽孢杆菌属(Anoxybacillus、0.3%)(表5)。
另由表6可以看出,链球菌属(Streptococcus)在每个样品中都占绝对菌群优势,乳杆菌属(Lactobacillus)除在7号、8号、9号样品中分别占15.8%、33.0%、38.8%外,在其余六种样品中含量极低。对于同一生产厂家的两种不同产品3号和6号,所含菌种分布情况极为相似。另外与标示菌种对比可以看出,部分产品标有双歧杆菌但未检出,部分产品检出未标示的菌种,由此看出酸奶产品在标签问题上存在不规范现象。

表6 九种样品中标示菌种与检测结果比较
2.5 物种分类学系统组成树和丰度分布曲线分析
本实验在前述分类学分析中,已经得到了每个OTU的丰度和对应的分类学信息,然后将测序得到的物种丰度信息回归至数据库的分类学系统关系树中,可以从整个分类系统上全面了解九种样品中所有微生物的进化关系和丰度差异,如图2所示分支为最为优势的为硬壁菌门(Firmicutes)分支中的链球菌属(Streptococcus),图中色柱的高低代表该样品在该处丰度的高低。同时由丰度分布曲线(图3)可以看出样品9物种的组成最丰富且均匀程度最高。
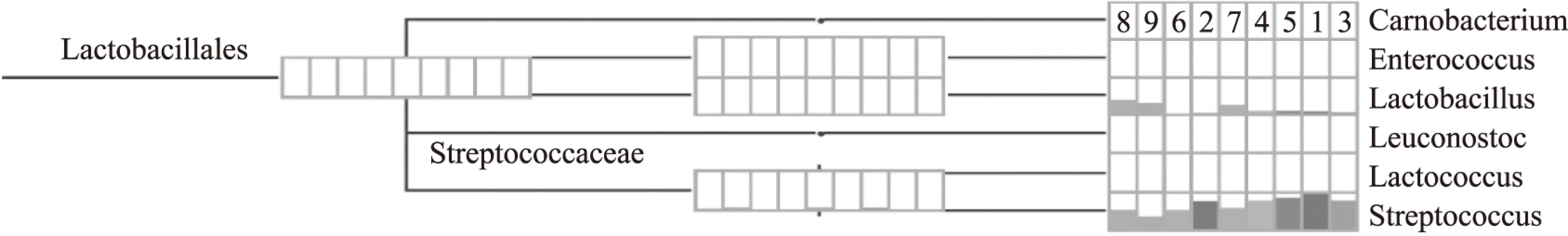
图2 各样品物种分类学系统组成树Fig.2 The taxonomic system Composition tree of each sample
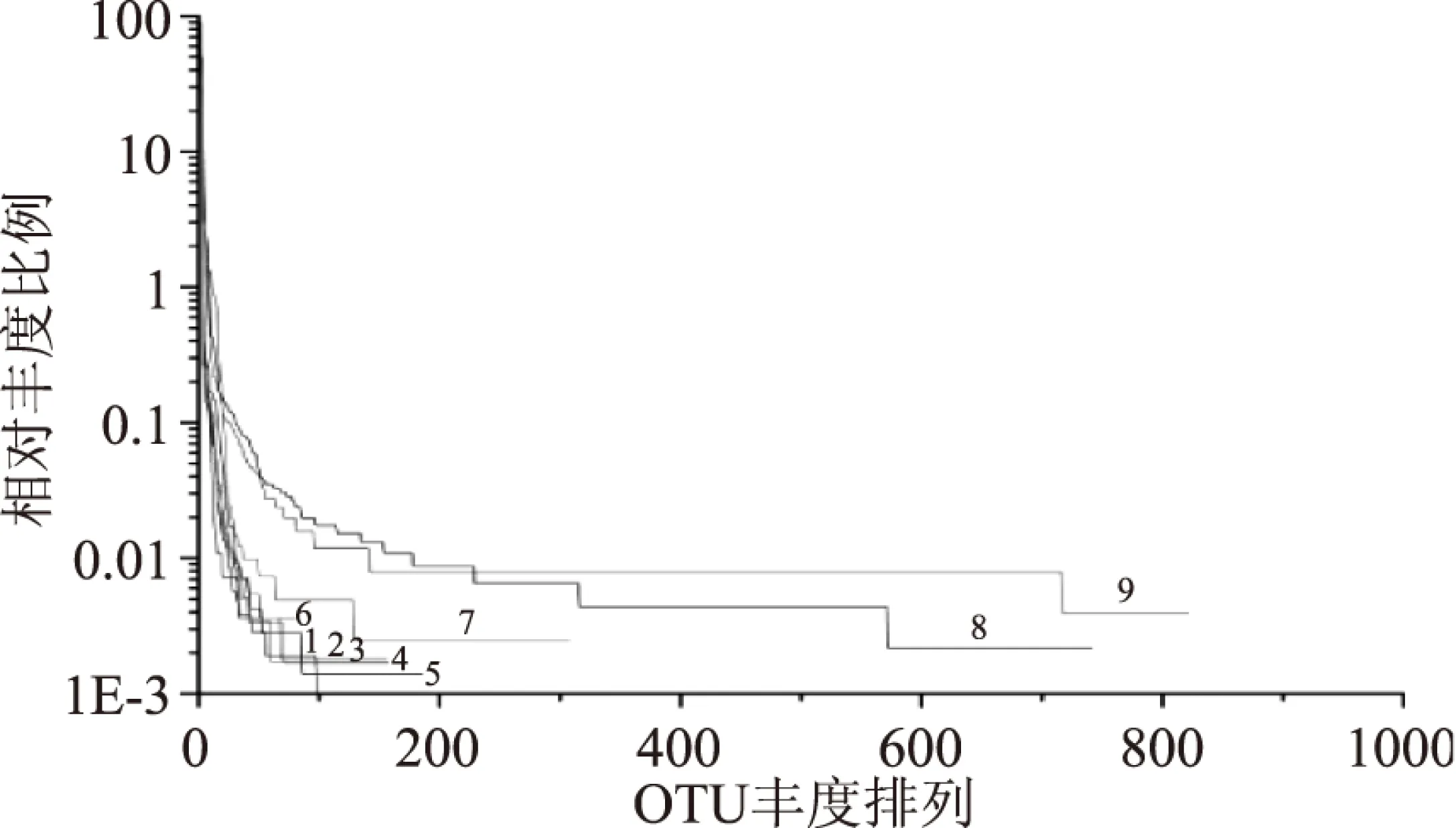
图3 丰度分布曲线Fig.3 Rank-Abundance curve
3 讨论
Illumina Miseq测序技术针对细菌16S rDNA可变区进行扩增测序并对原始数据进行质量控制,生成操作分类单元OTU,然后对微生物进行分类学分析以掌握其多样性及分布情况,使人们对复杂环境中微生物的认识由物种水平上升到基因层面,这一技术的出现解决了大多数微生物的不可培养性和基因组的多样性问题,受到很多研究者青睐[20]。
同类研究中,焦晶凯等[19]测定干酪中最为优势的菌群为硬壁菌门(Firmicutes),这与本研究结果一致,杨捷琳等[7]调查发现益生菌制品中存在标签不符的问题,R Temmerman等[21]发现调查时发现40%的乳制品标签出现错误,更值得注意的是国外一些调查研究表明,即使是从制造商处直接获得的商业菌株,部分也会出现实际含菌种类与标识不符的问题[22]。可见多数调查都显示益生菌食品在标签上存在问题,因为国内相关产品众多,新产品又层出不穷,甚至益生菌在医药和动物饲料行业也已广泛应用,故为保证食品药品的安全性,未来全面对益生菌产品中所含微生物展开全面深度调查尤为重要。
4 结论
本实验利用Illumina Miseq平台对九个酸奶样品进行基因组测序,共得到微生物有效序列582573条,通过物种注释得到7门13纲21目62科104属,揭示了常见市售酸奶中微生物的群落结构、丰度差异和进化关系。针对不同酸奶所含菌种相似度非常高而标签显示菌种各有不同的问题,也可能是添加的其它菌种在酸奶发酵、储藏过程中由于环境相对苛刻不易存活,针对这一问题未来应在生产时多注重温度控制或添加一些利于乳酸菌生长的物质,以保证乳酸菌的活性及多样化。与此同时,新型发酵菌剂的研究也至关重要。
[1]孟祥晨. 乳酸菌与乳品发酵剂[M]. 北京:科学出版社,2009.
[2]Plessas S,Bosnea L,Alexopoulos A,et al. Potential effects of probiotics in cheese and yogurt production:A review[J]. Engineering in Life Sciences,2012,12(4):433-440.
[3]El-Abbadi N H,Dao M C,Meydani S N. Yogurt:role in healthy and active aging.[J]. American Journal of Clinical Nutrition,2014,99(5 Suppl):1263S-1270S.
[4]Van Niel C W,Feudtner C,Garrison M M,et al. Lactobacillus therapy for acute infectious diarrhea in children:a meta-analysis.[J]. Pediatrics,2002,109(4):678-684.
[5]Borchers A T,Keen C L,Gershwin M E. The influence of yogurt/lactobacillus on the innate and acquired immune response[J]. Clinical Reviews in Allergy & Immunology,2002,22(3):207-230.
[6]Hong Y F,Lee Y D,Park J Y,et al. Immune Regulatory Effect of Newly Isolated Lactobacillus delbrueckii from Indian Traditional Yogurt.[J]. Journal of Microbiology & Biotechnology,2015,25(8).
[7]杨捷琳,袁辰刚,窦同海,等. 深度测序技术检测益生菌产品菌株组成及16SrDNA序列[J]. 食品科学,2013,34(20):241-245.
[8]Berta G,Chebenová,Brežná B,et al. Identification of lactic acid bacteria in Slovakian bryndza cheese[J]. Journal of Food & Nutrition Research,2009,48(2):65-71.
[9]Degnan P H,Ochman H. Illumina-based analysis of microbial community diversity.[J]. Isme Journal,2012,6(1):183-194.
[10]Chebeňová-Turcovská V,Ženišová K,Kuchta T,et al. Culture-independent detection of microorganisms in traditional Slovakian bryndza cheese[J]. International Journal of Food Microbiology,2011,150(1):73-78.
[11]陈泽斌,李冰,王定康,等. Illumina MiSeq高通量测序分
析核桃内生细菌多样性[J]. 江苏农业学报,2015(5):1129-1133.
[12]Xie G,Lan W,Gao Q,et al. Microbial community structure in fermentation process of Shaoxing rice wine by Illumina-based metagenomic sequencing[J]. Journal of the Science of Food & Agriculture,2013,93(12):3121-3125.
[13]Logares R,Sunagawa S,Salazar G,et al. Metagenomic 16S rDNA Illumina tags are a powerful alternative to amplicon sequencing to explore diversity and structure of microbial communities[J]. British Medical Journal,2014,2(4477):613-614.
[14]Fan W,Huo G,Li X,et al. Diversity of the intestinal microbiota in different patterns of feeding infants by Illumina high-throughput sequencing[J]. World Journal of Microbiology & Biotechnology,2013,29(12):2365-2372.
[15]Keshri J,Mankazana B B J,Momba M N B. Profile of bacterial communities in South African mine-water samples using Illumina next-generation sequencing platform[J]. Applied Microbiology & Biotechnology,2015,99(7):3233-3242.
[16]李燕明,佟训靓,许宏涛,等. Illumina高通量测序技术检测医院内空气中微生物[J]. 温州医科大学学报,2015(6):437-439.
[17]王立志,徐谊英. 圈养大熊猫粪便中微生物多样性的研究[J]. 四川动物,2016(1):17-23.
[18]王林,李冰,朱健. 高通量测序技术在人工湿地微生物多样性研究中的研究进展[J]. 中国农学通报,2016(5):10-15.
[19]焦晶凯,莫蓓红. Illumina MiSeq平台高覆盖率测定干酪中的细菌微生物多样性[J]. 中国酿造,2014,33(5):34-38.
[20]秦楠,栗东芳,杨瑞馥. 高通量测序技术及其在微生物学研究中的应用[J]. 微生物学报,2011,51(4):445-457.
[21]Temmerman R,Pot B,Huys G,et al. Identification and antibiotic susceptibility of bacterial isolates from probiotic products[J]. International Journal of Food Microbiology,2003,81(1):1-10.
[22]Yeung P S,Sanders M E,Kitts C L,et al. Species-Specific Identification of Commercial Probiotic Strains[J]. Journal of Dairy Science,2002,85(5):1039-1051.
Microbial diversity of yogurt depth detected by Illumina Miseq platform
ZHI Nan-nan1,3,ZONG Kai1,YANG Jie-lin2,YAO Jian1,*,WEI Zhao-jun3,*
(1.Technology center of Anhui Entry-exit Inspection and Quarantine Bureau,Hefei 230032,China; 2.Shanghai Entry and Exit Inspection and Quarantine Bureau,Shanghai 200135,China; 3.School of food science and Engineering,HeFei University of Technology,Hefei 230009,China)
Establishing a method based on Illumina Miseq deep sequencing to analyze the microbial diversity of yogurt. Took 9 kinds of yogurt samples as the research objects and extracted the whole genome of bacteria of yogurt. The V1-V3 variation region of 16S rDNA were detected by High throughput sequencing technology to obtain the distribution and abundance of microbial community in 9 samples as well as the species difference and evolution relationship of bacteria among these samples. The results showed that the main microbes of the yogurt were Firmicutes(99.6%)which mainly composed by Streptococcus(87.1%),Lactobacillus(10.3%),and Lactococcus(0.3%).There were 3 kinds of yogurt containing Streptococcus and Lactobacillus with a high number,Streptococcus(97%)accounted for almost all in the other six samples.Using Illumina Miseq deep sequencing technology can master the microbial diversity of yogurt quickly and accurately. The results showed that the bacteria of yogurt were highly similar and streptococcus took a dominant position. At the same time,it was found that some yogurt labels didn’t match the analysis results well.
Illumina Miseq;yogurt;microorganism;diversity
2016-05-09
智楠楠(1993-),女,硕士,研究方向:食品微生物,E-mail:zhinannan74@163.com。
*通讯作者:姚剑(1971-),女,博士,研究方向:食品微生物,E-mail:msyao@126.com。 魏兆军(1970-),男,教授,博士生导师,研究方向:食品加工技术,E-mail:weizhaojun@163.com。
长三角地区功能性微生态制剂关键检测技术及溯源体系的开发应用(1503062007);长三角地区微生态制剂关键检测技术及溯源体系的开发应用(14495810200)。
TS252.4
A
1002-0306(2016)24-0000-00
10.13386/j.issn1002-0306.2016.24.000

