蝴蝶兰液泡型ATP酶E亚基基因的克隆及序列分析
袁秀云,田云芳,梁 芳,蒋素华,许申平,王默霏,崔 波*(.郑州师范学院 生物工程研究所,河南 郑州 450044; .郑州师范学院 生命科学学院,河南 郑州 450044)
蝴蝶兰液泡型ATP酶E亚基基因的克隆及序列分析
袁秀云1,田云芳2,梁 芳1,蒋素华1,许申平1,王默霏1,崔 波1*
(1.郑州师范学院 生物工程研究所,河南 郑州 450044; 2.郑州师范学院 生命科学学院,河南 郑州 450044)
E亚基是液泡型ATP 酶(V-ATPase)多亚基复合体的重要组成部分,响应植物的非生物胁迫,对其基因的克隆及分析有助于阐释其逆境条件下的分子调节机制。根据蝴蝶兰低温诱导差异蛋白的序列设计简并引物,利用RT-PCR和RACE技术从蝴蝶兰叶片中克隆获得2条V-ATPase E亚基的cDNA序列,分别命名为PhVHA-Ea和PhVHA-Eb,GenBank登录号分别为KT758849和KT758850。PhVHA-Ea和PhVHA-Eb全长分别为960 bp和1 036 bp,二者均具有687 bp的开放阅读框,编码228个氨基酸,包含相同的vATP-synt_E保守域;生物信息学分析表明,2个序列编码蛋白具有相似的二级结构和相同的亲水性,预测的三级结构也相同。系统进化树分析显示,蝴蝶兰PhVHA-Ea和PhVHA-Eb与单子叶植物液泡型ATP 酶E亚基距离最近。
蝴蝶兰; 液泡型ATP 酶; E亚基; RACE; 序列分析
液泡型氢离子三磷酸腺苷酶(Vacuolar proton-ATPase,V-ATPase)是一类多亚基膜结合转运蛋白,广泛分布于真核细胞的内膜系统上[1-2]。在结构上,V-ATPase由膜外周球颈结构的V1和膜内部的V0两大亚复合体组成,其中V1部分功能是催化ATP水解,由A、B、C、D、E、F、G、H亚基组成,而V0部分功能是跨内膜形成质子通道,由a、c、c′、c″、d、e亚基组成[3-5]。V-ATPase利用水解ATP产生的能量将质子泵入细胞区室内,形成跨膜的质子电化学势,维持细胞内离子平衡,为其他各种离子及代谢产物的转运提供动力,维持机体正常的代谢平衡[6-7]。研究表明,V-ATPase结构复杂,功能多样[8-9],当植物处于高盐、干旱、低温或重金属等胁迫时,V-ATPase通过调节各亚基的表达和酶活性的变化使植物适应环境[10-12]。
V-ATPase E亚基是V1的重要组成部分[13],在V1和V0复合体的组装步骤中起关键作用[14-15]。同时V-ATPase E亚基对机体内新陈代谢、发育及响应逆境胁迫也起重要作用。例如,秀丽隐杆线虫(Caenorhabditiselegans)的V-ATPase E亚基对其pH值的内平衡及幼虫发育至关重要[16];拟南芥V-ATPase E亚基有3个异位表达的编码基因(AtVHA-E1、AtVHA-E2和AtVHA-E3),具有不同的特性和功能[17-18]。对小麦的研究表明,V-ATPase E亚基基因在拟南芥中超表达能促进盐胁迫下种子发芽、根及成苗的发育[19]。小麦V-ATPase各亚基在高盐和ABA胁迫下协同表达促进小麦对高盐的抗性[19]。兰科植物中还未见V-ATPase的研究报道。
蝴蝶兰(Phalaenopsisamabilis)以花型奇特、花色艳丽、花期长而著称,具有很高的观赏价值。但蝴蝶兰原产于热带及亚热带地区,适合生长在温湿的环境,在我国北方地区只能在现代化温室内种植,种植成本较高,因此研究蝴蝶兰的抗寒分子调控机制不但能够丰富植物的低温逆境调控理论,而且能为兰花的引种栽培和分子育种提供依据,最终达到兰花栽培节能、环保、高效的目的。在对蝴蝶兰低温诱导差异蛋白的研究中,发现有1个V-ATPase E亚基蛋白的上调表达[20],为深入研究该基因在蝴蝶兰低温胁迫中的作用,本研究以蝴蝶兰叶片为材料克隆了V-ATPase E亚基基因的全长,并对其序列进行分析,以期为开展该基因的功能及在低温胁迫中的分子调控机制研究奠定基础。
1 材料和方法
1.1 材料与处理
蝴蝶兰栽培品种满天红来自郑州师范学院生物工程研究所组培室,以生长在1/2MS基本培养基的生根苗为试验材料。将生长大小一致的瓶苗放在光照培养箱中,13 ℃/8 ℃的昼夜温度,12 h/12 h的光照条件,培养9 d 时取第2叶片组织,取材后迅速用液氮速冻,于-80 ℃冰箱备用。
1.2 方法
1.2.1 RNA的提取 叶片组织的总RNA用Trizol试剂(Invitrogen)提取。RNA的完整性用质量分数为1%的琼脂糖凝胶电泳鉴定,并用Q5000核酸蛋白分析仪(美国Quawell)测定其A260、A280值,计算RNA浓度和纯度。
1.2.2 蝴蝶兰PhVHA-E基因全长的克隆 采用M-MLV反转录酶(TaKaRa)以叶片总RNA为模板合成单链cDNA。根据蝴蝶兰低温诱导差异蛋白分析中鉴定的蛋白(gi:388495094和gi:115438974)序列及肽段序列DLIVQGLLR、LKEPAVLLR和IVCENTLDAR[20],设计1对简并引物V-ATP-F和 V-ATP-R(表1),用于扩增蝴蝶兰PhVHA-E基因的中间片段,预期长度为270 bp。PCR扩增体系为20 μL,含10×Buffer 2 μL,4种dNTP各150 μmol/L,每条引物0.5 μmol/L,cDNA模板1 000~2 000 ng,TaqDNA聚合酶1 U。PCR扩增程序为:95 ℃变性5 min;95 ℃ 40 s,54 ℃ 40 s,72 ℃ 1 min,35个循环;72 ℃延伸10 min。回收目的片段,连接到pMD19-T载体上,转入大肠杆菌DH5α菌株,进行克隆和测序。根据以上步骤所得的2条cDNA基因片段序列,设计2对3′端特异引物ATP-Ea-F1、ATP-Ea-F2,以及ATP-Eb-F1、ATP-Eb-F2;2对5′端特异引物ATP-Ea-R1、ATP-Ea-R2,以及ATP-Eb-R1、ATP-Eb-R2(表1),按SMARTTMRACE Amplification kit(Clontech)操作说明进行扩增,回收扩增产物,克隆到pMD19-T载体上,转入大肠杆菌DH5α菌株,鉴定后进行测序并与中间片段进行拼接。然后再根据拼接的序列设计1对引物ATP-ORFF和ATP-ORFR进行开放阅读框(ORF)的扩增并进行验证(表1)。引物和测序由上海立菲生物技术有限公司完成。

表1 引物序列
1.2.3 蝴蝶兰PhVHA-E基因序列的分析 利用NCBI的Open Reading Frame Finder 工具分析PhVHA-E基因的ORF;PhVHA-E基因的核苷酸和氨基酸序列分别通过Blastx和Blastp进行同源序列的搜索和比对;功能域分析用NCBI 的蛋白保守区数据库 (conserved domain database,CDD) 进行,采用ProtScale程序分析该蛋白质亲疏水性;蛋白质二级结构预测采用GOR Protein secondary structure prediction分析;利用phyre2对其三级结构进行预测;使用MEGA软件的NJ法构建系统进化树。
2 结果与分析
2.1 蝴蝶兰PhVHA-E基因全长的克隆
叶片总RNA提取后经1%琼脂糖凝胶电泳检测显示,28S和18S条带清晰,28S浓度约为18S的2倍(图1),核酸蛋白分析仪检测其OD260/OD280为2.04,质量浓度为778.9 ng/μL,能够满足基因克隆的要求。

图1 蝴蝶兰叶片总RNA提取结果
以蝴蝶兰叶片总RNA反转录的cDNA为模板,以简并引物V-ATP-F和V-ATP-R进行中间片段的PCR 扩增,如期得到1个270 bp 的保守片段(图2D),测序得到2条编码序列,2条核苷酸序列的一致性为95.93%,具有11个碱基的差异,编码的氨基酸序列一致性为93.26%,具有6个氨基酸的差异,都包含预期的LKEPAVLLR和IVCENTLDAR肽段,将2条片段分别命名为ATP-Ea和ATP-Eb。2条序列经Blastx分析,其氨基酸为vATP-synt-E多功能域,该功能域包含V-ATPase E亚基和古细菌V-ATPase E亚基。ATP-Ea与玉米(Zeamays,ACG31413.1)、荷花(Nelumbonucifera,XP_010251551.1)和高粱(Sorghumbicolor,XP_002456080.1)的V-ATPase E亚基有78%、75%和79%的一致性。ATP-Eb与晚香玉(Polianthestuberosa,AIN42254.1)、油棕(Elaeisguineensis,XP_010914312.1)和荷花(Nelumbonucifera,XP_010251551.1)的氨基酸分别具有75%、76%和73%的一致性。说明此2条中间片段为V-ATPase E亚基基因序列。

A:3′端RACE; B:5′端RACE; C:ORF扩增; D:中间片段的扩增;1:ATP-Ea的3′端片段; 2:ATP-Eb的3′端片段; 3:ATP-Ea的5′端片段; 4:ATP-Eb的5′端片段; 5:PhVHA-Ea的ORF片段; 6:PhVHA-Eb的ORF片段; 7:中间片段; M:Marker
根据ATP-Ea序列设计的3′端巢式特异引物ATP-Ea-F1和ATP-Ea-F2,采用RACE技术,得到ATP-Ea 3′端316 bp的片段(图2A-1),根据ATP-Eb序列设计的3′端巢式特异引物ATP-Eb-F1和ATP-Eb-F2,进行RACE试验,得到ATP-Eb 3′端393 bp的片段(图2A-2);同样采用RACE技术,利用ATP-Ea和ATP-Eb 5′端巢式特异引物ATP-Ea-R1和ATP-Ea-R2、ATP-Eb-R1和ATP-Eb-R2分别得到ATP-Ea和ATP-Eb 5′端536 bp和533 bp的片段(图2B-3,图2B-4)。将中间片段ATP-Ea和ATP-Eb分别与其3′端目的片段和5′端目的片段序列拼接,然后在开放阅读框区域设计引物,进行扩增,得到了预期687 bp的ORF片段(图2C),最终得到960 bp和1 036 bp,分别命名为PhVHA-Ea和PhVHA-Eb。通过ORF查找,PhVHA-Ea具有86 bp的5′ UTR和187 bp的3′ UTR,PhVHA-Eb具有89 bp 的5′ UTR和260 bp的3′ UTR,2条序列均有1个687 bp的ORF,编码228 个氨基酸;PhVHA-Ea和PhVHA-Eb的核苷酸序列一致性为89.02%,主要差异在编码区两端。在编码区有19个核苷酸碱基的差异(图3),氨基酸一致性为96.49%,有8个氨基酸的差异(图4)。通过Blastn分析,PhVHA-Ea的核苷酸序列与油棕(Elaeisguineensis,XM_010916010.1)、海枣(Phoenixdactylifera,XM_008800441.1 )和水稻(Oryzasativa,NM_001050302.1)V-ATPase E亚基分别具有80%、79%和79%的一致性。PhVHA-Eb的核苷酸序列经过Blastn分析得到了相似的结果。将PhVHA-Ea和PhVHA-Eb的序列提交GenBank,登录号分别为KT758849和KT758850。

图3 PhVHA-E基因核苷酸序列的比对

图4 PhVHA-E基因氨基酸序列的比对
2.2PhVHA-E基因的生物信息学分析
利用 NCBI 的蛋白质保守区数据库 (conserved domain database,CDD)对PhVHA-E基因进行编码蛋白质保守区预测,结果表明,PhVHA-Ea和PhVHA-Eb编码的蛋白质与登录号为pfam01991的蛋白质匹配,该蛋白质在16—225位氨基酸残基之间有1个vATP-synt_E功能保守域(图5)。预测结果表明,PhVHA-Ea和PhVHA-Eb编码的蛋白质分子质量均约为26.39 ku,等电点约为7。疏水性最大值为1.967,最小值为-3.567,氨基酸残基大部分在亲水区域。说明此2条基因序列编码的蛋白质为亲水性蛋白(图6C)。二级结构预测表明,PhVHA-Ea编码的蛋白质包括61.4%的α螺旋、13.6%的延伸链、7.02%的β转角、17.98%的随机卷曲(图6A)。PhVHA-Eb编码的蛋白质与PhVHA-Ea相似,同样包括α螺旋、延伸链、β转角和随机卷曲,分别占序列的59.66%、15.35%、4.82%和20.18%(图6B)。phyre2在线预测3级结构结果表明,PhVHA-Ea和PhVHA-Eb编码的蛋白具有相同的三级结构,都与蛋白模型c4dl0J具有95%(217个氨基酸)的覆盖度和100%的可信度(图6D),该蛋白质为V-ATPase E亚基蛋白,为一水解酶,是酵母液泡型ATP酶EGC头部异源三聚体外周茎轴复合体的晶体结构的一部分[21]。以上分析表明,PhVHA-Ea和PhVHA-Eb为同一功能的V-ATPase E亚基基因。

图5 PhVHA-E蛋白保守结构域
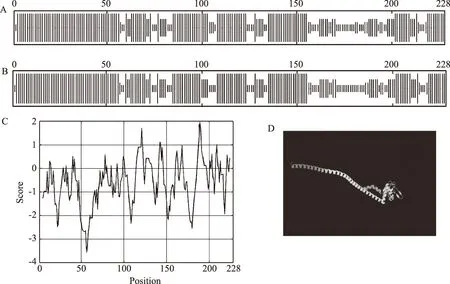
A:PhVHA-Ea二级结构预测; B:PhVHA-Eb二级结构预测; C:疏水性分析; D:三级结构预测图6 蝴蝶兰PhVHA-E基因预测蛋白的特征
为进一步分析蝴蝶兰PhVHA-E基因与其他植物的V-ATPase E亚基基因的进化关系,将其氨基酸序列提交至NCBI 在线进行Blastp搜索,结果表明,蝴蝶兰V-ATPase E亚基的氨基酸序列与其他植物该蛋白质的氨基酸序列覆盖率为99%以上,序列一致性为74%~83%,说明该基因的氨基酸序列相对保守。选择枇杷(Eriobotryajaponica)V-ATPase E1亚基(AIY85417.1)、葡萄(Vitisvinifera)V-ATPase E1亚基(XP_002270168)、水稻(Oryzasativa)V-ATPase E2亚基(OsVHA-E2,NP_001055857.1)和V-ATPase E亚基(NP_001043767.1)、蒺藜苜蓿(Medicagotruncatula)V-ATPase E1亚基(XP_003611036.1)、小麦(Triticumaestivum)V-ATPase E亚基(ABC70183.1)、大麦(Hordeumvulgare)V-ATPase E亚基(YLP,AAD10336.1)、 醉蝶花(Tarenayahassleriana)V-ATPase E1亚基(XP_010529628.1)和E3亚基(XP_010533611.1)、拟南芥(Arabidopsisthaliana)V-ATPase E1亚基(AtVHA-E1,NP_192853.1)、E2亚基(AtVHA-E2,NP_187468.1)、E3亚基(AtVHA-E3,NP_176602.1)等基因构建系统进化树(图7),结果显示,分析的9个物种14个基因被分为2个大的分支,在第1大分支中,包括拟南芥、醉蝶花、枇杷和蒺藜苜蓿等双子叶植物亚支和单子叶植物小麦、大麦、水稻和蝴蝶兰亚支。其中拟南芥的AtVHA-E1与醉蝶花V-ATPase E1亚基、拟南芥的AtVHA-E3与醉蝶花V-ATPase E3亚基、枇杷V-ATPase E1亚基与蒺藜苜蓿V-ATPase E1亚基分别有较近的亲缘关系;本研究中蝴蝶兰的2个V-ATPase E亚基关系最近,与单子叶植物小麦、大麦和水稻的V-ATPase E亚基聚在一起。在另外1个分支中,拟南芥AtVHA-E2和水稻OsVHA-E2关系最近,与葡萄V-ATPase E1亚基聚在一起。分析表明,蝴蝶兰的2个V-ATPase E亚基在进化上具有明显的种属特异性,与植物分类系统有较强的相关性;植物的V-ATPase E2亚基与V-ATPase E1亚基和E3亚基亲缘关系相对较远。
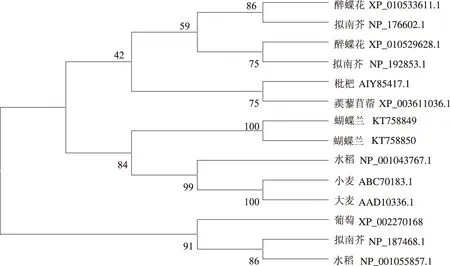
图7 蝴蝶兰PhVHA-E基因的系统进化分析
3 结论与讨论
本研究首次从兰科植物蝴蝶兰叶片中克隆了2条V-ATPase E亚基基因的全长cDNA序列,并对其进行了生物信息学分析,结果表明,该序列具有vATP-synt_E功能保守域,编码的蛋白质为一亲水性蛋白,具有水解酶活性;三级结构显示该蛋白质为酵母V-ATPase外周茎轴EGC复合体的一部分,研究结果与其他生物V-ATPase E亚基的特征一致[22-24]。
在拟南芥中,V-ATPase E亚基有3个亚型,各亚型间具有不同程度的功能特化,其中AtVHA-E1是胚胎发育表达的主要亚型,而AtVHA-E2是花粉特异表达亚型,在配子体发育中具有专一性但又不是必需的,AtVHA-E3主要在发育种子的胚乳及周围组织中表达[5]。对突变体研究表明,AtVHA-E3能够恢复AtVHA-El缺失的表型,而AtVHA-E2却不能[17-18]。目前对其他植物V-ATPase E亚基基因的研究相对较少,在不同生物中基因名称和亚型还没有一致的命名[5],例如,大麦的V-ATPase E亚基基因命名是TpP31[25],小麦的是TaVATE(ABC70183.1)[26]。本研究中系统进化树分析结果也显示了这一现象:例如醉蝶花和拟南芥的AtVHA-E1和AtVHA-E3分别显示较近的亲缘关系,与这2种植物在植物界的系统进化关系一致[27-28],而葡萄V-ATPase E1亚基(XP 002270168)与拟南芥AtVHA-E2(NP 187468.1)和水稻(NP_001055857.1)的OsVHA-E2关系较近,既没有体现物种之间的进化关系,也没有体现出V-ATPase E亚基的同源关系,说明了V-ATPase E亚基基因命名的混乱。本研究中蝴蝶兰的2个V-ATPase E亚基基因核苷酸序列一致性为89.02%,而氨基酸的一致性为96.49%,只有8个氨基酸的差异,由此推断这2个核苷酸序列是蝴蝶兰V-ATPase E亚基基因的同源基因,来源于不同亲本,但是由于没有显示与拟南芥的3个V-ATPase E亚基基因亚型更近的亲缘关系,为了慎重起见,以PhVHA-Ea和PhVHA-Eb命名。
由于V-ATPase通过调节细胞内的离子平衡参与物质的转运[29-30],涉及到多种信号的调节[22],参与多种胁迫生理调控[31-33]。V-ATPase E亚基基因在胁迫中的调控作用也有报道[20,26],然而其调控机制还是未知。蝴蝶兰作为热带和亚热带物种,对低温更为敏感。在低温胁迫下的研究发现了该基因蛋白质的上调表达[20],而其作用机制有待于进一步研究。显然,本试验结果为进一步研究V-ATPase E亚基基因功能及在低温胁迫下的响应机制奠定了基础。
[1] Jefferies K C,Cipriano D J,Forgac M.Function,structure and regulation of the vacuolar(H+)-ATPases[J].Arch Biochem Biophys,2008,476(1):33-42.
[2] Marshansky V,Rubinstein J L,Grüber G.Eukaryotic V-ATPase:Novel structural findings and functional insights[J].Biochim Biophys Acta,2014,1837(6):857-879.
[3] Cipriano D J,Wang Y,Bond S,etal.Structure and regulation of the vacuolar ATPases[J].Biochim Biophys Acta,2008,1777(7/8):599-604.
[4] Toei M,Saum R,Forgac M.Regulation and isoform function of the V-ATPases[J].Biochemistry,2010,49(23):4715-4723.
[5] Sze H,Schumacher K,Müller M L,etal.A simple nomenclature for a complex proton pump:VHA genes encode the vacuolar (H+)-ATPase [J].Trends Plant Sci,2002,7(4):157-161.
[6] Dettmer J,Hong-Hermesdorf A,Stierhof Y D,etal.Va-cuolar H+-ATPase activity is required for endocytic and secretory trafficking inArabidopsis[J].Plant Cell,2006,18(3):715-730.
[7] Zhang T,Zhou Q,Ogmundsdottir M H,etal.Mitf is a master regulator of the V-ATPase,forming a control mo-dule for cellular homeostasis with V-ATPase and TORC1[J].J Cell Sci,2015,128(15):2938-2950.
[8] Ma B,Xiang Y,An L.Structural bases of physiological functions and roles of the vacuolar (H+)-ATPase [J].Cell Signal,2011,23(8):1244-1256.
[9] Rawson S,Phillips C,Huss M,etal.Structure of the vacuolar H+-ATPase rotary motor reveals new mechanistic insights [J].Structure,2015,23(3):461-471.
[10] Dietz K J,Tavakoli N,Kluge C,etal.Significance of the V-type ATPase for the adaptation to stressful growth conditions and its regulation on the molecular and biochemical level[J].J Exp Bot,2001,52(363):1969-1980.
[11] Hanitzsch M,Schnitzer D,Seidel T,etal.Transcript le-vel regulation of the vacuolar (H+)-ATPase subunit isoforms VHA-a,VHA-E and VHA-G inArabidopsistha-liana[J].Mol Membr Biol,2007,24(5/6):507-518.
[12] 张玉红,安志刚.植物V-H+-ATP酶适应逆境的分子调控机理[J].草业科学,2013,30(2):245-252.
[13] Grüber G,Godovac-Zimmermann J,Link T A,etal.Expression,purification,and characterization of subunit E,an essential subunit of the vacuolar ATPase[J].Biochem Biophys Res Commun,2002,298(3):383-391.
[14] Arata Y,Baleja J D,Forgac M.Cysteine-directed cross-linking to subunit B suggests that subunit E forms part of the peripheral stalk of the vacuolar H+-ATPase[J].J Biol Chem,2002,277(5):3357-3363.
[15] Ohira M,Smardon A M,Charsky C M,etal.The E and G subunits of the yeast V-ATPase interact tightly and are both present at more than one copy per V1 complex[J].J Biol Chem,2006,281(32):22752-22760.
[16] Ji Y J,Choi K Y,Song H O,etal.VHA-8,the E subunit of V+-ATPase,is essential for pH homeostasis and larval development inC.elegans[J].FEBS Lett,2006,580(13):3161-3166.
[17] Strompen G,Dettmer J,Stierhof Y D,etal.Arabidopsisvacuolar H+-ATPase subunit E isoform 1 is required for Golgi organization and vacuole function in embryogenesis[J].Plant J,2005,41(1):125-132.
[18] Dettmer J,Liu T Y,Schumacher K.Functional analysis ofArabidopsisV-ATPase subunit VHA-E isoforms[J].Eur J Cell Biol,2010,89(2/3):152-156.
[19] Zhao Q,Zhao Y J,Zhao B C,etal.Cloning and functio-nal analysis of wheat V-H+-ATPase subunit genes[J].Plant Mol Biol,2009,69(1/2):33-46.
[20] Yuan X Y,Liang F,Jiang S H,etal.Differential protein expression inPhalaenopsisunder low temperature [J].Appl Biochem Biotechnol,2015,175(2):909-924.
[21] Oot R A,Huang L S,Berry E A,etal.Crystal structure of the yeast vacuolar ATPase heterotrimeric EGC(head) peripheral stalk complex[J].Structure,2012,20(11):1881-1892.
[22] Parra K J,Chan C Y,Chen J.Saccharomyces cerevisiae vacuolar H+-ATPase regulation by disassembly and reassembly:One structure and multiple signals[J].Eukaryot Cell,2014,13(6):706-714.
[23] Jones R P,Durose L J,Findlay J B,etal.Defined sites of interaction between subunits E(Vma4p),C(Vma5p),and G(Vma10p) within the stator structure of the vacuolar H+-ATPase [J].Biochemistry,2005,44(10):3933-3941.
[24] Féthière J,Venzke D,Diepholz M,etal.Building the stator of the yeast vacuolar-ATPase:Specific interaction between subunits E and G[J].J Biol Chem,2004,279(39):40670-40676.
[25] Dietz K J,Rudloff S,Ageorges A,etal.Subunit E of the vacuolar (H+)-ATPase ofHordeumvulgareL.:cDNA cloning,expression and immunological analysis[J].Plant J,1995,8(4):521-529.
[26] Zhang X H,Li B,Hu Y G,etal.The wheat E subunit of V-type H+-ATPase is involved in the plant response to osmotic stress[J].Int J Mol Sci,2014,15(9):16196-16210.
[27] Bhide A,Schliesky S,Reich M,etal.Analysis of the floral transcriptome ofTarenayahassleriana(Cleomaceae),a member of the sister group to theBrassicaceae:Towards understanding the base of morphological diversity inBrassicales[J].BMC Genomics,2014,15:140.
[28] Cheng S,van den Bergh E,Zeng P,etal.TheTarenayahasslerianagenome provides insight into reproductive trait and genome evolution of crucifers[J].Plant Cell,2013,25(8):2813-2830.
[29] Chan C Y,Parra K J.Yeast phosphofructokinase-1 su-bunit Pfk2p is necessary for pH homeostasis and glucose-dependent vacuolar ATPase reassembly[J].J Biol Chem,2014,289(28):19448-194457.
[30] Schumacher K,Krebs M.The V-ATPase:Small cargo,large effects[J].Curr Opin Plant Biol,2010,13(6): 724-730.
[31] He X,Huang X,Shen Y,etal.Wheat V-H+-ATPase subunit genes significantly affect salt tolerance inArabidopsisthaliana[J].PLoS One,2014,9(1):e86982.
[32] Mirzaei M,Pascovici D,Atwell B J,etal.Differential regulation of aquaporins,small GTPases and V-ATPases proteins in rice leaves subjected to drought stress and recovery[J].Proteomics,2012,2(6):864-877.
[33] Zhang M,Fang Y,Liang Z,etal.Enhanced expression of vacuolar H+-ATPase subunit E in the roots is associated with the adaptation ofBroussonetiapapyriferato salt stress[J].PLoS One,2012,7(10):e48183.
Cloning and Sequence Analysis of Vacuolar Proton-ATPase E Subunit Gene fromPhalaenopsisamabilis
YUAN Xiuyun1,TIAN Yunfang2,LIANG Fang1,JIANG Suhua1,XU Shenping1,WANG Mofei1,CUI Bo1*
(1.Institute of Bioengineering,Zhengzhou Normal University,Zhengzhou 450044,China; 2.College of Life Science,Zhengzhou Normal University,Zhengzhou 450044,China)
E subunit is an important part of the multisubunit complex of V-ATPase,which responds to abiotic stress in plant.Cloning and analysis of the gene encoding V-ATPase E subunit is helpful to elucidate its molecular regulation mechanism under stress.In this paper,two cDNA sequences of V-ATPase E subunit genes fromPhalaenopsisamabilisleaf were isolated by the methods of RT-PCR combined with RACE techniques using degenerate primers designed according to the sequence of the differentially expressed protein.They were named asPhVHA-EaandPhVHA-Eb,the GenBank accession numbers were KT758849 and KT758850 respectively.The two sequences length were 960 bp and 1 036 bp with the intact open reading frame of 687 bp,encoding a polypeptide of 228 amino acids with the same domain of vATP-synt_E.Bioinformatics analysis indicated that the two sequences had similar secondary structure,the same feature of hydrophilic protein and the same three-dimensional structure.The phylogenetic analysis revealedPhVHA-EaandPhVHA-Ebwere closed to the V-ATPase E subunits gene from monocots.
Phalaenopsisamabilis; V-ATPase; E subunit; RACE; sequence analysis
2016-01-20
河南省科技攻关项目(092102110128)
袁秀云(1970-),女,河南许昌人,教授,博士,主要从事植物生物技术研究。E-mail:yuanxiuyun@163.com
*通讯作者:崔 波(1962-),男,河南泌阳人,教授,博士,主要从事花卉育种研究。 E-mail:laocuibo@163.com
S682.31
A
1004-3268(2016)06-0104-07

