高分辨率熔解曲线分析法检测大肠埃希菌gyrA基因突变
高分辨率熔解曲线分析法检测大肠埃希菌gyrA基因突变

随着喹诺酮类药物在临床和畜牧业的广泛应用,大肠埃希菌对喹诺酮类药物已呈现相当高的耐药率[1-2]。gyrA基因突变造成的DNA旋转酶A亚单位(GyrA)结构改变是大肠埃希菌对喹诺酮类药物耐药的最重要机制[3-4]。目前对gyrA突变的研究方法有:扩增喹诺酮耐药决定区(quinolone resistance determining region,QRDR)后基因测序分析、限制性内切酶Hinf Ⅰ的酶切进行限制性片段长度多态性分析(restriction fragment length polymorphism,RFLP)、单链构象多态性 (single-strand conformation polymorphism, SSCP)分析等方法[5-8]。这些方法需有专门的设备,检测费用昂贵,操作烦琐,需要一定的技术和经验,并且都存在一定的检测局限性。高分辨率熔解曲线(high-resolution melting, HRM) 分析技术是近年来发展起来的分子生物学新技术。用新型饱和染料(LC Green、LC Green PLUS、SYTO 9和Eva Green等)代替传统熔解曲线分析中的非饱和染料SYBER GreenN对PCR的扩增产物进行熔解曲线分析,根椐溶解温度(melting temperature,Tm)值和熔解曲线的峰形来分析扩增的目的基因,有非常高的灵敏度,且不需要专门的检测设备,在一些加装了分析模块的常规定量聚合酶链反应(polymerase chain reaction, PCR)仪上也可以检测分析。目前HRM技术已被应用于基因分型、序列匹配、突变扫描等领域,有着广泛的应用前景。为了探索HRM技术检测大肠埃希氏菌的gyrA基因突变的可行性,我们采用序列分析法和HRM技术分别检测了70株临床分离的大肠埃希菌gyrA基因的突变情况。
材料和方法
一、菌株来源
70株大肠埃希菌均分离自2010年2月至2011年8月连云港市第一人民医院临床标本, 包括尿液、痰、血标本和其它标本,排除重复菌株。所有菌株均经西门子MicroScan WalkAway-96 Plus 全自动细菌鉴定药敏分析仪鉴定,其中环丙沙星敏感38株、耐药32 株。质控菌株为大肠埃希菌ATCC25922、铜绿假单胞菌ATCC27853,均购自国家卫计委临床检验中心。
二、 方法
1.大肠埃希菌gyrA基因QRDR的突变检测参照文献[8]设计引物,PCR扩增所有70株大肠埃希菌株gyrA基因的QRDR片段,引物序列为F: 5′-CTCCTATCTGGATTATGCG-3′、R:5′-GGTCGATAGAACCGAAGTTA-3′,由上海生工生物有限公司合成,扩增产物长度为284 bp,位于QRDR的第19~113位密码子。PCR反应混合体系Perfect ShotTMTaq购自宝生物工程大连有限公司。GeneAmp 2700 PCR扩增仪为美国PE公司产品。菌株DNA提取参考文献[9]稍作改动:挑取一营养琼脂37 ℃过夜培养的中等大小菌落于50 μL无菌蒸馏水中,95 ℃ 10 min裂解细菌,释放DNA,10 000×g离心10 min,取上清液作为DNA模板。PCR扩增条件参照文献[8],循环数为30次。将扩增产物送上海生工生物有限公司测序,测得序列用blast 软件与敏感株K011(Genbank登记号:CP002516.1)做序列比对,检测是否有突变、突变位点及碱基变化。

结果
一、gyrA基因测序结果
对70株大肠埃希菌gyrA基因进行普通PCR扩增并测序。结果显示共有43株(61.4%)gyrA基因发生突变,有27株(30.6%)为未突变的野生株,突变发生在83位密码子TCG→TTG、85位密码子GTT→GTC、87位密码子GAC→AAC及91位密码子CGT→CGC 4个位点,其中单一位点突变11株,多位点突变32株。依椐gyrA基因突变的不同位点数和类型将70株菌株分成野生(未发生突变)和Ⅰ(24株,2点突变)、Ⅱ(15株,1点突变)、Ⅲ(14株,3点突变)、Ⅳ(2株,4点突变)、Ⅴ(2株,2点突变)5个组,见图1、图2及表1。
图1 部分菌株gyrA基因普通PCR扩增电泳图
二、gyrA基因扩增产物的熔解曲线分析结果
gyrA基因突变株HRM与未发生突变的野生株有明显差异,可以准确区分野生株和突变株,准确率达98.6%(69/70); 43株gyrA基因突变株检出42株,敏感性为97.7%(42/43),其中只发生1个碱基突变的11株突变株(Ⅱ组)均被正确检出。检出的42株突变株按不同突变类型被正确分组。见图3、图4。
三、gyrA基因扩增产物的Tm检测结果
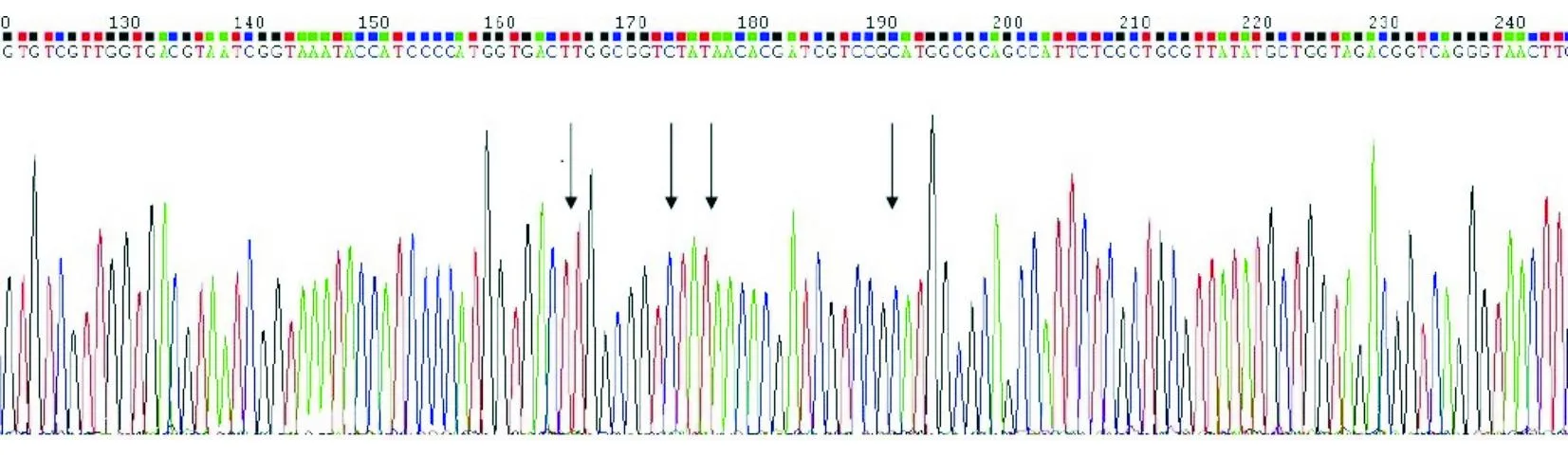
注:箭头示处为突变位点

图2 发生多点突变位的gyrA基因测序图

图3 70株大肠埃希菌溶解曲线标准化图形
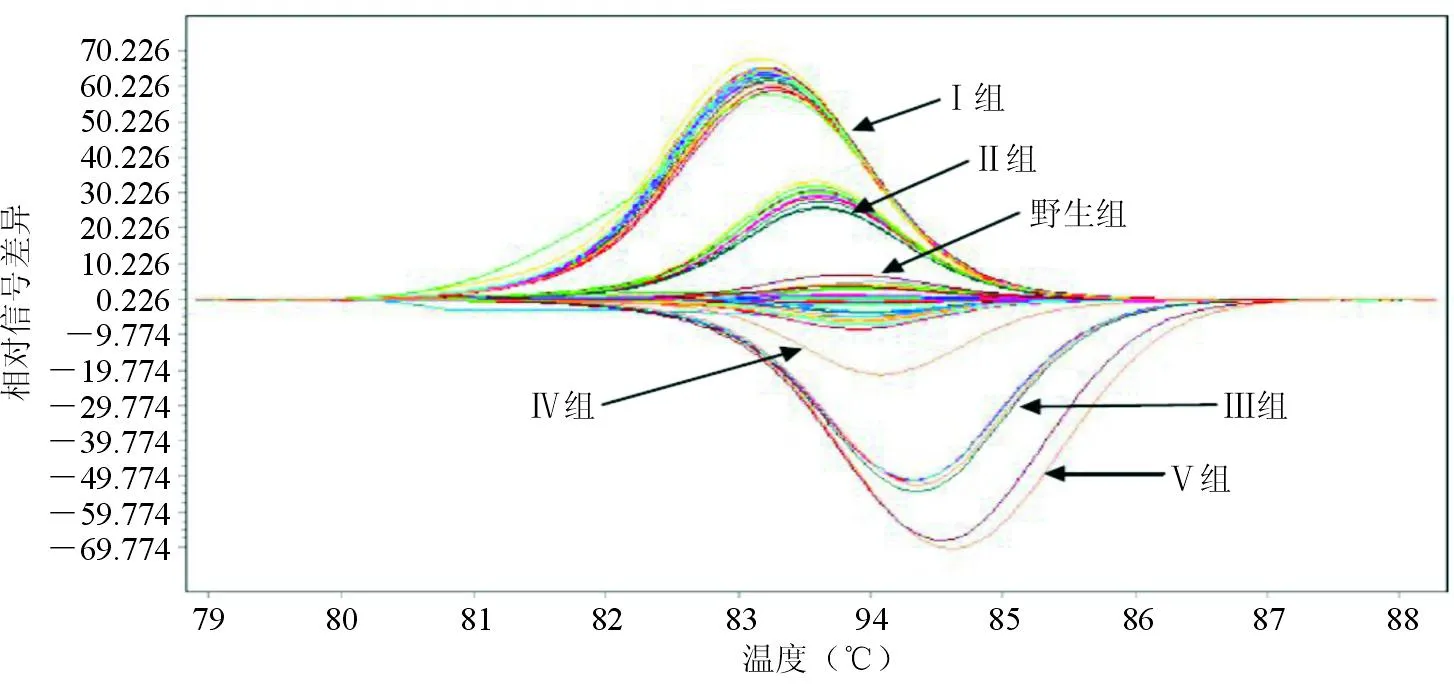
图4 70株大肠埃希菌溶解曲线标准化温度差异图形

图5 70株大肠埃希菌的Tm峰形图
讨论
促旋酶A亚单位编码基因gyrA突变而导致促旋酶结构改变是大肠埃希菌耐喹诺酮类药物的最重要机制,改变主要位于编码67~106氨基酸的密码子之间,这个区间通常称为QRDR。对应的gyrA基因最常见的突变为83位密码子TCG→TTG、87位密码子GAC→AAC、91位密码子CGT→CGC等[5,11]。研究表明突变的类型不同和突变点的多寡对于喹诺酮类药物的MIC值有明显影响[4]。
目前,研究gyrA突变的方法中以测序法最为可靠也最准确,能够确定突变的类型和位置,但是需有基因测序设备,且检测费用昂贵。RFLP法主要利用gyrA基因突变中最常见为的83位密码子碱基C→T突变的特点,但是突变如不是发生在83位密码子上就可能发生漏检。SSCP是依据点突变引起单链DNA分子立体构象的改变来实现电泳分离的。这种方法的不足是某些位置的点突变对单链DNA分子立体构象的改变不起作用或作用很小,再加上其它条件的影响,使聚丙烯酰胺凝胶电泳无法分辨造成漏检。这几种方法都需要一定的技术和经验,而且都是2个步骤的分析,操作较为烦琐且较易发生样本污染,所以都存在一定的局限性。
HRM分析法是用新型饱和染料代替传统熔解曲线分析中的非饱和染料对PCR的扩增产物进行熔解曲线分析[12]。该检测技术近年来已应用于临床微生物检验中的菌种鉴定、耐药机制检测等多个研究领域[13-16]。它主要根椐突变株发生碱基突变后DNA的GC含量与野生株不同而导致的Tm值和熔解曲线峰形的差异,通过基因扫描敏感地鉴别突变株和野生株的DNA。从已有的研究报道和前期的测序结果表明:大肠埃希菌gyrA基因最常见的突变位点均由碱基G或C突变为T或A。由于DNA的GC含量发生了改变,必然会导致DNA的Tm值和熔解曲线峰形发生改变,这就使HRM技术快速检测出大肠埃希菌gyrA基因突变成为可能。2007年,SLINGER 等[10]报道了将HRM技术应用于沙门氏菌gyrA基因的突变检测,成功检测出了临床分离的沙门氏菌gyrA突变株。但SLINGER等[10]研究发现沙门氏菌gyrA基因只是单位点的突变。然而本研究对临床分离的70株大肠埃希菌gyrA基因测序表明:大肠埃希菌的gyrA基因突变有单位点、双位点、多位点突变的情况存在,突变型远较沙门氏菌复杂。本研究运用HRM技术对临床分离的70株大肠埃希菌gyrA基因突变进行检测,与基因测序法相比正确率达到98.6%(69/70),对检测出的42株突变株的突变型能正确地鉴别分组。整个实验过程从挑取细菌单菌落制备模板DNA、试剂准备、荧光定量PCR扩增到熔解曲线分析可在1~2 h内完成,1次最多可完成96个样本检测,符合微生物快速诊断的发展以及大样本检测的要求。如果在检测过程中加入已知突变位点和突变型的菌株作参考菌株,将待检菌株的分析曲线或Tm值与参考菌株作比较可以直接检测出待检菌的突变点和突变类型,无需做后续的基因测序,成本低廉。且由于检测为封闭不开盖连续检测,避免了样本污染,扩增产物还可以用来做其它实验分析。本研究显示HRM技术检测大肠埃希菌gyrA基因突变具有简单、快速、准确优点,可以作为检测大肠埃希菌耐药机制的新选择。由于本研究检测的样本数不够多,因此利用HRM技术检测大肠埃希菌gyrA基因突变的可行性还需要加大样本量进一步验证。
由于HRM技术是基于突变菌株DNA中GC含量发生改变的原理,所以当细菌基因发生多位点突变,且多个突变的GC改变相互抵消导致发生突变后的DNA的GC含量与野生株相同的情况下,HRM技术就无法正确检测到这种突变。本研究第Ⅳ组菌株由于发生了4个位点突变, 突变的碱基改变相互抵消后DNA与野生株相比碱基改变为G→A 、T→C,无明显GC含量改变,Tm值(83.96)变化不大,造成有一株菌的突变没有被检测出来,这是HRM技术检测细菌基因多位点突变的方法学的局限性。
参考文献
[1]肖永红,沈萍,魏泽庆,等.Mohnarin 2011年度全国细菌耐药监测[J].中华医院感染学杂志,2012,22(22):4946-4952.
[2]赵倩,汪雅萍,应春妹,等.质粒介导aac(6′)-Ib基因检测与喹诺酮类耐药[J].检验医学,2013,28(3):199-202.
[3]BEARDEN DT,DANZIGER LH.Mechanism of action of and resistance to quinolones [J].Pharmacotherapy,2001,21(10 Pt 2):224S-232S.
[4]TRUONG QC,NGUYEN VAN JC,SHLAES D,et al.A novel,double mutation in DNA gyrase A ofEscherichiacoliconferring resistance to quinolone antibiotics[J].Antimicrob Agents Chemother,1997,41(1):85-90.
[5]崔生辉,李景云,马越,等.大肠埃希菌中氟喹诺酮耐药机制功能分析[J].中华微生物学和免疫学杂志,2008,28(4):338-342.
[6]赵旺胜,江淑芳,顾兵,等.南京地区鲍曼不动杆菌喹诺酮类药物耐药基因突变的研究[J].临床检验杂志,2007,25(2):90-92.
[7]肖永红,王其南.聚合酶链反应RFLP与SCCP检测伤寒杆菌DNA旋转酶基因变异[J].中国抗生素杂志,2000,25(4):286-288.
[8]蒋萍,陈恒屹,张坚磊,等.耐喹诺酮类大肠埃希菌耐药机制的研究[J].中华医院感染学杂志,2007,17(6):635-638.
[9]刘文恩,陈腊梅,李艳明,等.在中国大陆首次检出CTX-M-15型超广谱β-内酰胺酶基因[J].中国医师杂志,2007,9(10):1311-1314.
[10]SLINGER R, BELLFOY D, DESJARDINS M, et al.High-resolution melting assay for the detection ofgyrAmutations causing quinolone resistance inSalmonellaentericaserovars Typhi and Paratyphi[J].Diagnostic Microbiol Infecti Dis,2007,57(4):455-458.
[11]NIWA H,HOBO S,ANZAI T.A nuclectide mutation associated with fluorquinolone resistance observed ingyrAof in vitro obtained Rhodococcus equi mutants[J].Vet Microbiol,2006,115(1-3):264-268.
[12]REED GH, KENT JO, WITTWER CT.High-resolution DNA melting analysis for simple and efficient molecular diagnostics[J].Pharmacogenomics,2007,8(6):597-608.
[13]李刚,赵燕,魏取好,等.高分辨熔解曲线法检测临床常见KPC基因亚型[J].中华检验医学杂志,2011,34(6):520-522.
[14]CHENG JC, HUANG CL, LIN CC, et al. Rapid detection and identification of clinically important bacteria by high-resolution melting analysis after broad-range ribosomal RNA real-time PCR[J]. Clin Chem,2006,52(11):1997-2004.
[15]ONG DC, YAM WC, SIU GK, et al. Rapid detection of rifampicin- and Isoniazid-resistantMycobacteriumtuberculosisby high-resolution melting analysis[J]. J Clin Microbiol,2010,48(4):1047-1054.
[16]HJELMSØ MH, HANEN LH, BAELUM J, et al. High-resolution nelt analysis for rapid comparison of bacterial community compositions[J]. Appl Environ Microbiol,2014,80(12):3568-3575.
茆海丰,刘洪书
(连云港市第一人民医院检验科,江苏 连云港 222002)
摘要:目的探讨高分辨率溶解曲线(HRM)分析法在检测大肠埃希菌gyrA基因突变中的可行性。方法收集临床非重复分离的大肠埃希菌70株,常规聚合酶链反应(PCR)扩增大肠埃希菌的gyrA基因,采用序列分析法检测gyrA基因的变异情况。设计特异性引物,采用荧光定量PCR扩增这70株大肠埃希菌株的gyrA基因,通过对扩增产物进行HRM分析和熔解温度(Tm)值测定确定gyrA基因变异情况。将检测结果与序列分析法检测结果进行比较。结果基因测序显示70株大肠埃希菌中有43株gyrA基因发生了突变,突变类型分为5型。HRM分析法可以准确区分野生株和突变株,准确率达98.6%(69/70),正确检测出了43株突变菌株中的42株,且对不同的突变类型准确分型。Tm分析显示野生株和突变株之间以及各种突变型之间有明显差异。结论HRM技术具有准确性高、简单、快速、成本低廉、高通量等优点,可以作为检测大肠埃希氏菌gyrA基因突变耐药机制的新选择。
关键词:大肠埃希菌; gyrA基因; 高分辨率熔解曲线
The detection ofgyrAgene mutation inEscherichiacoliby high-resolution melting analysisMAOHaifeng,LIUHongshu.(TheFirstPeople′sHospitalofLianyungangCity,JiangsuLianyungang222002,China)
Abstract:ObjectiveTo investigate the feasibility of high-resolution melting (HRM) analysis to detect gyrA gene mutation in Escherichia coli. MethodsA total of 70 isolates of Escherichia coli were collected, and gyrA gene was amplified with conventional polymerase chain reaction(PCR). Gene mutation was detected by sequence analysis. Specific primer was designed. The gyrA gene was amplified by fluorescence quantitation PCR. The mutation of gyrA gene was detected by amplifying product HRM curve and melting temperature (Tm) analysis. The result was compared with that of gene sequence analysis. ResultsgyrA gene mutation sequence analysis showed that 43 of 70 isolates occurred gyrA gene mutation. In these mutations, 5 types were found. HRM analysis can accurately distinguish between wild and mutant isolates. The detection accuracy was 98.6% (69/70).The 42 of 43 mutant isolates were correctly detected, and different type gene mutations can be correctly typed. Tm analysis also showed the difference of wild and mutant isolates. ConclusionsHRM analysis is accurate, simple, rapid, cheap and high-throughput, and it and would be a novel choice in analyzing drug resistance mechanism of Escherichia coli.
Key words:Escherichia coli; gyrA gene; High-resolution melting
收稿日期:(2015-01-12)
作者简介:茆海丰,男,1969年生,硕士,主任技师,主要研究方向为细菌耐药机制。
基金项目:江苏省连云港市卫生局科研项目(11014)
中图分类号:(本文编辑:范基农)
文章编号:1673-8640(2015)11-1138-05Q503 1673-8640(2015)11-1143-04R446.1
文献标志码:ADOI:10.3969/j.issn.1673-8640.2015.11.018 ADOI:10.3969/j.issn.1673-8640.2015.11.019

