对虾传染性皮下及造血组织坏死病毒编码蛋白的相互作用研究*
陈沈雪 魏永伟 苗 亮 陈 炯
(宁波大学海洋学院 生物化学与分子生物学实验室 宁波 315211)
传染性皮下及造血组织坏死病毒(Infection hypodermal and hematopoietic necrosis virus, IHHNV)是感染对虾的一种重要病原, 首次发现于美国夏威夷地区的南美蓝对虾(Litopenaeus stylirostris), IHHNV感染可引起南美蓝对虾幼虾 90%的死亡(Tang et al,2000; Tang et al, 2001; Encinas-García et al, 2015)。IHHNV亦可感染南美白对虾(Litopenaeus vannamei),但不引起对虾的死亡, 而引起慢性矮小残缺综合征(Runt Deformity Syndrome, RDS)(Hsieh et al, 2006;Dhar et al, 2007, Lightner, 2011), 患病对虾生长缓慢,导致其产量和质量急剧下降, 给对虾养殖业造成巨大的经济损失(Galván-Alvarez et al, 2012; Silva et al,2014; Encinas-García et al, 2015)。
IHHNV是单链线性DNA病毒, 无囊膜, 为二十面体, 直径为 22—23 nm, 属于细小病毒科(Parvoviridae)、浓核病毒亚科(Densovirinae), 是目前已知最小的对虾病毒(Lightner, 1999; Mendoza-Cano et al, 2014; Silva et al, 2014; Shen et al, 2015)。IHHNV基因组全长约3.9—4 kb, 含有3个开放阅读框(Open Reading Frame, ORF), ORF1和ORF2分别编码非结构蛋白1和2 (nonstructural protein 1, NS1; nonstructural protein 2, NS2), ORF3编码病毒的衣壳蛋白(capsid protein, CP)。NS1、NS2和CP分别含666个氨基酸(75.77 kDa), 343个氨基酸(42.11 kDa)和329个氨基酸(37.48 kDa) (Shike et al, 2000; Vega-Heredia et al,2012)。Tang等(2003)研究认为NS1可能参与调控病毒转录及复制过程中的酶活性; Geng等(2012)研究发现 NS2可与对虾肌动蛋白相互作用, 并在病毒感染过程中起着重要作用; Hou等(2009)将CP进行原核表达后, 发现该重组蛋白能够自我组装成病毒样颗粒(virus-like particles, VLPs)。除上述报道外, 目前针对IHHNV编码蛋白的相关基础研究鲜有报道。病毒编码蛋白间的相互作用在病毒生命周期中发挥重要作用, 能够直接影响病毒复制和侵染, 因此, 研究病毒编码蛋白间的相互作用有助于深入理解病毒感染过程和病毒与宿主间的相互作用(Guoet al, 2001; Lianet al, 2014)。然而, 目前尚未有IHHNV编码蛋白间相互作用展开相关研究的报道。本研究构建了 IHHNV编码的三种蛋白NS1、NS2和CP的酵母双杂交载体,研究3个编码蛋白间的相互作用情况, 以期为深入研究IHHNV的组装机制和致病机理研究奠定基础。
1 材料与方法
1.1 材料
大肠杆菌TG1、酵母菌株AH109, 载体pGADT7、pGBKT7、pGADT7-T、pGBKT7-53、pGBKT7-lam由本实验室保存。SD/-Leu、SD/-Trp、SD/-Ade/-Leu、SD/-His/-Leu、SD/-Ade/-Trp、SD/-His/-Trp、SD/-Leu/-Trp、SD/-Ade/-His/-Leu/-Trp、Minimal SD Base、X-α-gal购自 Clontech公司; PCR相关试剂、pMD19-T Vector Cloning Kit、NdeI、BamHI等购自TaKaRa公司; GEL Extraction Kit和Plasmid Mini Kit I购自Omega公司; 引物由英潍捷基(上海)贸易有限公司合成; 序列由上海华大基因进行测定。
1.2 方法
1.2.1 PCR扩增IHHNV NS1、NS2、CP序列 根据本实验室克隆的 IHHNV的基因组全长序列(KP733862)设计引物(见表 1), 以本实验室提取的IHHNV Wenzhou株基因组为模板进行PCR扩增。反应条件为: 94℃预变性2 min, 94℃ 30 s, 60℃退火30 s, 72℃延伸 2 min, 共 35个循环, 最后 72℃延伸10 min。参照GEL Extraction Kit回收PCR产物,并将其克隆至pMD19-T载体。将重组载体命名为pMD19-NS1、pMD19-NS2、pMD19-CP。

表1 本研究用到的寡核苷酸引物Tab.1 The oligonucleotide primers used in this study
1.2.2 猎物载体及诱饵载体的构建及鉴定 参照Plasmid Mini Kit I精抽重组质粒, 用NdeI和BamHI进行双酶切, 并用同样的酶双酶切 pGADT7和pGBKT7。回收酶切产物并进行连接, 构建酵母猎物载体 pGADT7-NS1、pGADT7-NS2、pGADT7-CP,诱饵载体pGBKT7-NS1、pGBKT7-NS2、pGBKT7-CP,并转化大肠杆菌 TG1, 涂布于含有氨苄霉素(100 μg/mL)或卡那霉素(50 μg/mL)的LB平板。挑选单菌落经PCR初步验证, 再用NdeI和BamHI进行双酶切验证后,将阳性克隆送至上海华大基因科技有限公司进行测序。
1.2.3 猎物蛋白及诱饵蛋白对酵母 AH109的毒性检测 用灭菌的牙签挑取含有重组猎物载体或诱饵载体的酵母 AH109 阳性克隆至 50 mL SD/-Leu/Amp (20μg/ml)或 SD/-Trp/Kan (20 μg/mL)液体培养基中, 30℃, 230 r/min培养24 h后, 检测其OD值。
1.2.4 猎物蛋白及诱饵蛋白自激活检测 将AH109/pGADT7-NS1、AH109/pGADT7-NS2、AH109/pGADT7-CP 菌液涂布于 SD/-Leu/X-α-gal、SD/-His/-Leu/X-α-gal、SD/-Ade/-Leu/X-α-gal, AH109/pGBKT7-NS1、AH109/pGBKT7-NS2、AH09/pGBKT7-CP菌液涂布于 SD/-Trp/X-α-gal、SD/-His/-Trp/X-α-gal、SD/-Ade/-Trp/X-α-gal平板, 30℃倒置培养 3—5 d, 观察其生长情况和颜色变化。
1.2.5 猎物载体及诱饵载体共转化酵母 酵母猎物载体及诱饵载体精抽后, 根据 LiAc法共转化至酵母AH109中, 涂布于SD/-Leu/-Trp固体培养基, 30℃倒置培养 3—4 d。随机挑取 6个单菌落至SD/-Leu/-Trp液体培养基, 30℃, 230 r/min培养24 h。经菌液 PCR验证后, 点种至 SD/-Ade/-His/-Leu/-Trp/X-α-gal固体培养基, 倒置培养3—5 d, 并观察其生长及颜色变化。
1.2.6 IHHNV CP自身作用区域的鉴定 参照方法1.2.1和1.2.2, 根据IHHNV的CP序列设计引物(见表1), 以本实验室提取的IHHNV Wenzhou株基因组为模板进行PCR扩增。切胶回收目的片段, 并分别克隆至pMD19-T载体, 经Nde I和BamH I双酶切后, 连接到经相同酶切的pGBKT7载体, 构建CP缺失诱饵载体 pGBKT7-CP151(+)、pGBKT7-CP301(+)、pGBKT7-CP451(+)、pGBKT7-CP601(+)、pGBKT7-CP387(-)、pGBKT7-CP537(-)、pGBKT7-CP687(-)、pGBKT7-CP837(-)。随后, PCR和双酶切验证重组CP缺失诱饵载体, 经测序无误后, 再分别与猎物载体pGADT7-CP共转化至酵母 AH109中, 涂布于SD/-Leu/-Trp平板, 并进一步点种于 SD/-Ade/-His/-Leu/-Trp/X-α-gal平板, 观察其生长及颜色变化。
2 结果
2.1 IHHNV编码蛋白序列的扩增
以本实验室提取的IHHNV Wenzhou株基因组为模板进行PCR扩增NS1、NS2、CP序列, 得到约2 kb、1.1 kb、1 kb的片段(图1), 与预期目的片段大小相符合。
2.2 猎物载体及诱饵载体的鉴定
重组猎物载体及诱饵载体经 PCR及 Nde I和BamH I双酶切分析, 结果表明NS1、NS2、CP已成功插入到 pGADT7(图 2A)和 pGBKT7载体(图 2B)。测序结果显示, 目的片段已正确插入到猎物载体和诱饵载体, NS1、NS2、CP序列无任何碱基的缺失或突变。
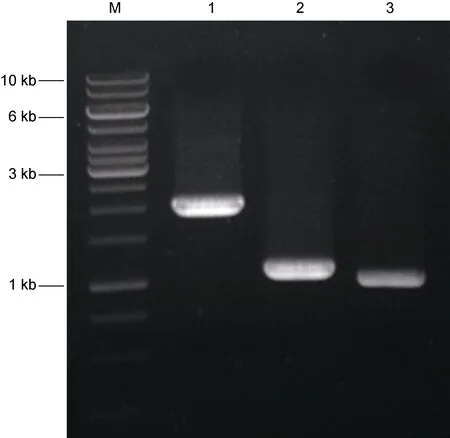
图1 IHHNV NS1、NS2、CP基因的PCR扩增Fig.1 PCR amplification of IHHNV NS1, NS2 and CP
2.3 猎物蛋白及诱饵蛋白对酵母AH109的毒性检测
挑取 AH109/pGADT7-NS1、AH109/pGADT7-NS2、AH109/pGADT7-CP单克隆于50 mL SD/-Leu/Amp (20 μg/mL)培养基中, pGBKT7-NS1、pGBKT7-NS2、pGBKT7-CP 于 SD/-Trp/Kan (20 μg/ml)培养基中, 30℃振荡培养24 h, 测得菌液OD600分别为0.83、1.02、1.16, 均大于 0.8, 表明猎物蛋白及诱饵蛋白对酵母AH109无毒性作用, 可用于进一步实验的展开。
2.4 猎物蛋白及诱饵蛋白的自激活检测
将 AH109/pGADT7-NS1、AH109/pGADT7-NS2、AH109/pGADT7-CP 涂布于 SD/-Leu/X-α-gal、SD/-His/-Leu/X-α-gal、SD/-Ade/-Leu/X-α-gal, 观察到其均能在 SD/-Leu/X-α-gal平板上生长但不显蓝(图 3A),而在 SD/-His/-Leu/X-α-gal 和 SD/-Ade/-Leu/X-α-gal平板均不能生长(图 3B和 3C); 将 AH109/pGBKT7-NS1、AH109/pGBKT7-NS2、AH109/pGBKT7-CP 涂布于 SD/-Trp/X-α-gal、SD/-His/-Trp/X-α-gal 和 SD/-Ade/-Trp/X-α-gal平板上, 观察到其能在 SD/-Trp/X-α-gal平板上生长但不显蓝(图 3D), 在 SD/-His/-Trp/X-α-gal 和 SD/-Ade/-Trp/X-α-gal 平板均不能生长(图3E和3F)。结果表明, 该重组猎物载体及诱饵载体表达的蛋白无自激活特性, 不能自激活 AH109报告基因ADE2、HIS3和MEL1的表达, 可应用该酵母双杂交系统进行IHHNV 编码的NS1、NS2和CP蛋白间相互作用研究。
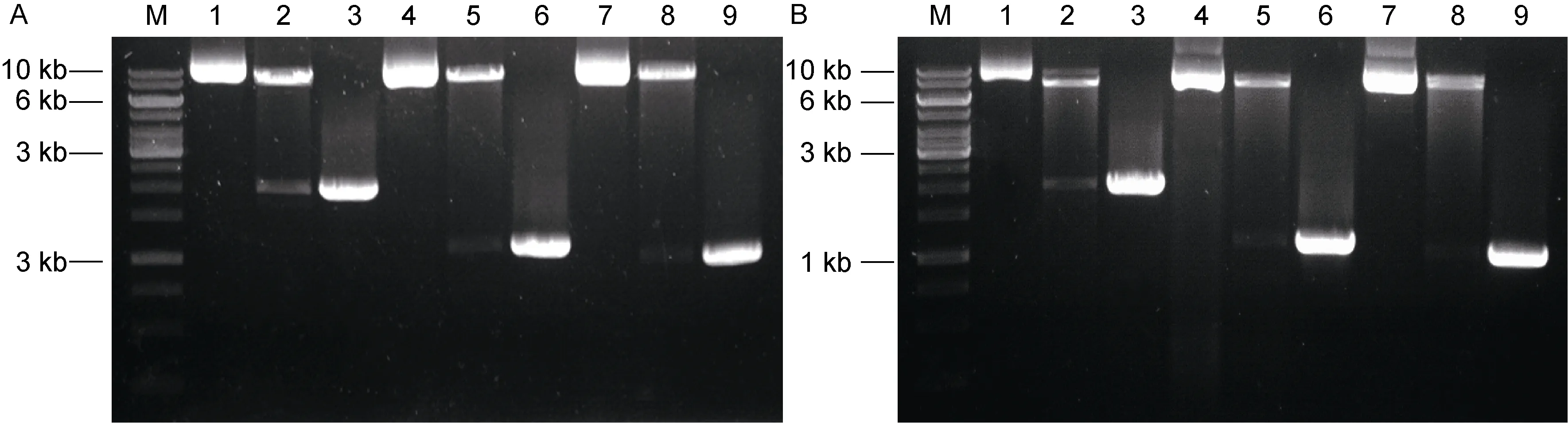
图2 重组猎物载体及诱饵载体双酶切及PCR鉴定Fig.2 Digestion and PCR identification of recombined prey vectors and bait vectors

图3 猎物蛋白和诱饵蛋白自激活作用检测Fig.3 Self-activation analysis of prey proteins and bait proteins
2.5 编码蛋白间相互作用的鉴定
共转化有pGADT7-NS1/pGBKT7-NS1、pGADT7-NS1/pGBKT7-NS2、pGADT7-NS1/pGBKT7-CP、pGADT7-NS2/pGBKT7-NS1、pGADT7-NS2/pGBKT7-NS2、pGADT7-NS2/pGBKT7-CP、pGADT7-CP/pGBKT7-NS1、pGADT7-CP/pGBKT7-NS2、pGADT7-CP/pGBKT7-CP、pGADT7-T/pGBKT7-lam、pGADT7-T/pGBKT7-53的酵母重组子AH109均能够在SD-Leu/-Trp固体培养基上生长, 挑取各酵母重组子点种至 SD/-Ade/-His/-Leu/-Trp/X-α-gal固体培养基, 只有转化有pGADT7-CP/pGBKT7-CP的酵母重组子能够在该固体培养基上上生长并显蓝, 与阳性对照(共转化有pGADT7-T/pGBKT7-53的酵母AH109)生长状况一致,而其它酵母重组子均与阴性对照(共转化有pGADT7-T/pGBKT7-lam 的酵母 AH109)一样不能在该培养基上生长(图4)。说明IHHNV编码蛋白CP-CP能够相互作用, 并激活AH109报告基因ADE2、HIS3和MEL1的表达, 而其它编码蛋白间不存在相互作用(表 2)。
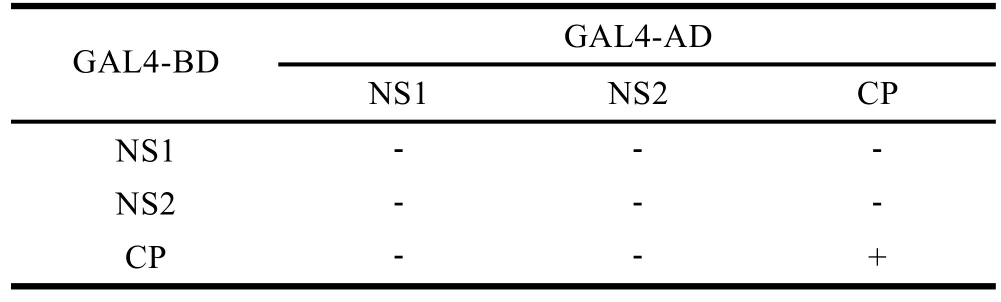
表2 酵母双杂交分析IHHNV编码蛋白间的相互作用Tab.2 Yeast two hybrid analysis of the interactions among IHHNV encoding proteins
2.6 IHHNV CP自身作用区域的鉴定
为进一步确定 CP蛋白自身互作的作用位点, 分别对CP蛋白的N端和C端进行逐段缺失, 构建新的CP缺失型诱饵载体 pGBKT7-CP151(+)、pGBKT7-CP301(+)、pGBKT7-CP451(+)、pGBKT7-CP601(+)、pGBKT7-CP387(-)、pGBKT7-CP537(-)、pGBKT7-CP687(-)、pGBKT7-CP837(-)(图 5A), 并与猎物载体 pGADT7-CP共转至酵母 AH109中, 涂布于SD/-Leu/-Trp平板, 并进一步点种于 SD/-Ade/-His/-Leu/-Trp/X-α-gal平板。结果显示构建的任何缺失体均未观察到酵母重组子生长(图5B), 说明IHHNV CP序列任何较小片段的缺失都会导致自身互作的丧失。
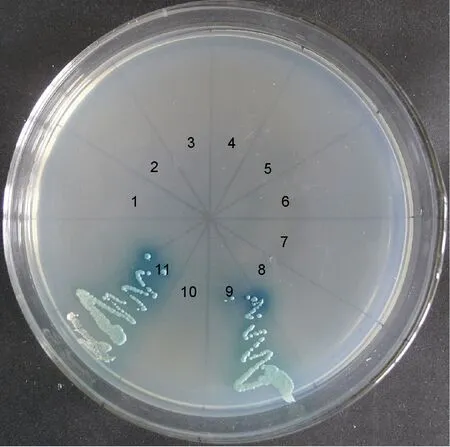
图4 IHHNV编码蛋白间相互作用鉴定Fig.4 Identification of the interactions between IHHNV encoding proteins

图5 IHHNV CP自身互作作用位点鉴定Fig.5 Identification of the functional sites of CP for self-interaction
3 讨论
酵母双杂交是一种体内鉴定和分析蛋白-蛋白相互作用的重要技术方法, 能够检测蛋白间微弱和短暂的相互作用。此外, 由于蛋白之间的相互作用是在真核酵母细胞内进行, 蛋白质可以其天然折叠状态存在, 能够增加检测的灵敏度和可信度。目前, 该技术已广泛应用于病毒蛋白相互作用的研究。例如,Tacken等(2003)利用酵母双杂交技术鉴定传染性法氏囊病毒(Infectious bursal disease virus, IBDV) VP2、VP3、VP4和VP5同型蛋白间的相互作用, 并用筛减突变法进一步确定这些蛋白自身互作的位点; Wang等(2015)利用酵母双杂交技术鉴定瓜类褪绿黄化病毒(Cucurbit chlorotic yellows virus, CCYV)各编码蛋白间的相互作用, 发现P59和P9能够自身互作, 进一步分析其自身互作的作用位点后, 发现P59的中间区域(第 173—344个氨基酸)是自身互作的必要位点, 而P9的3个不同截短位点与全长P9均无相互作用; Lian等(2014)用酵母双杂交系统发现水稻条纹病毒(Rice stripe virus, RSV)的核蛋白(nucleocapsid protein,NP)N端第 1—47个氨基酸是其自身互作所必需的,并且第 42—47个氨基酸是该作用最重要的氨基酸残基。本研究成功构建了IHHNV编码的NS1、NS2和CP蛋白酵母猎物载体pGADT7-NS1、pGADT7-NS2、pGADT7-CP和诱饵载体 pGBKT7-NS1、 pGBKT7-NS2、pGBKT7-CP, 并且这些重组载体无自激活活性,不能激活酵母AH109下游报告基因的表达。此外, 由于某些融合蛋白可能对酵母具有一定的毒性作用,本研究也测定了各酵母重组子在 SD/-Leu/Amp或SD/-Trp/Kan液体培养基中振荡培养24h后的吸光值,结果显示各重组子的 OD600均大于 0.8, 表明融合蛋白对酵母AH109无毒性作用。
病毒蛋白-蛋白间的相互作用在病毒基因组复制及病毒粒子的组装等过程中发挥重要的作用(Li et al,2013b)。如风疹病毒(Rubella virus, RV)结构蛋白CP能够通过与非结构蛋白p150相互作用促进病毒RNA的复制(Tzeng et al, 2006; Sakata et al, 2014); 水稻矮缩病毒(rice dwarf virus, RDV)非结构蛋白 Pns12与Pns11间存在相互作用, 而Pns6与Pns11间也存在相互作用, 这两组非结构蛋白间的相互作用均能影响病毒的复制(Chen et al, 2015); 轮状病毒(Rotavirus)非结构蛋白 NSP5与非结构蛋白 NSP2的相互作用,以及结构蛋白VP1与NSP5间强烈的相互作用共同促进病毒的复制及病毒粒子的形成(Eichwald et al, 2004;Arnoldi et al, 2007)。本研究对IHHNV各编码蛋白间的相互作用进行研究, 结果显示, IHHNV的结构蛋白CP与非结构蛋白间NS1、NS2或者非结构蛋白NS1与 NS2间不存在异型相互作用, 而只有病毒的结构蛋白 CP存在同型相互作用。然而, 人们在研究同属细小病毒科的脊椎动物病毒时发现, 非结构蛋白NS1能够增强病毒CP基因的表达(Shike et al, 2000), 因此, 细小病毒科的病毒非结构蛋白 NS1通过何种方式调控CP基因的表达有待进一步研究。
研究病毒衣壳蛋白间的自身互作是开展病毒自我复制和装配的基础性研究, Hallan和Gafni在研究番茄黄曲叶病毒(tomato yellow leaf curl virus,TYLCV)全长CP和诱变CP同型蛋白间的相互作用时,全长 CP能够自身互作, 截短的蛋白序列能够与全长CP互作, 而不与其自身截短的蛋白序列互作, 从而推测 CP-CP间的相互作用是源于其中一个 CP的 N端序列与另一个CP的C末端序列相互作用(Hallan et al, 2001); Kang等在研究大豆花叶病毒(soybean mosaic virus, SMV)编码蛋白间的相互作用时发现CP能够自身互作, C末端序列(第170—256 个氨基酸)的截短会导致其自身互作的丧失, 由于预测C末端具有很强的螺旋结构, 研究认为C末端参与CP的自身互作能够促进病毒的组装(Kang et al, 2004; Kang et al,2006)。然而, 本研究发现IHHNV的CP截短后不能够与全长 CP发生相互作用, 这表明 CP的自身互作是高度敏感的, 任何较少氨基酸序列的截短将导致其自身互作的丧失。Hou 等(2009)在对IHHNV的CP进行原核表达时, 发现 CP能够自我组装成与天然IHHNV病毒粒子相同大小和形状的VLPs, 因此, 我们推测 CP-CP的自身互作参与病毒颗粒的自我组装过程, 而 CP部分氨基酸序列的缺失将导致其自我组装效率的降低。
对虾养殖业是世界经济的重要组成部分, 其中南美白对虾为主要的对虾养殖种类。虽然近几年对虾养殖产量不断增长, 但因对虾疾病造成的经济损失每年约达10亿美元(Li et al, 2013a)。大部分的对虾疾病是由细菌和病毒引起, 而抗生素的使用并不能有效防治病毒引起的疾病。对虾缺乏脊椎动物所特有的获得性免疫应答, 由于缺乏抗体应答, 利用 VLPs进行药物传递或进行基因治疗将成为防治对虾病毒感染的有效方法。近些年研究认为利用 VLPs传递siRNA 或 dsRNA等干扰 RNA (interfering RNA,iRNA)将能够有效抑制对虾病毒的复制(Hou et al,2009), 本研究中, IHHNV CP的自身互作及其自身互作的高度敏感性, 将为寻找有效途径防治对虾病毒感染提供指导意义。
Arnoldi F, Campagna M, Eichwald C et al, 2007. Interaction of rotavirus polymerase VP1 with nonstructural protein NSP5 is stronger than that with NSP2. Journal of Virology, 81(5):2128— 2137
Chen Q, Chen H Y, Jia D S et al, 2015. Nonstructural protein Pns12 of rice dwarf virus is a principal regulator for viral replication and infection in its insect vector. Virus Research,210: 54—61
Dhar A K, Lakshman D K, Natarajan S et al, 2007. Functional characterization of putative promoter elements from infectious hypodermal and hematopoietic necrosis virus(IHHNV) in shrimp and in insect and fish cell lines. Virus Research, 127(1): 1—8
Eichwald C, Rodriguez J F, Burrone O R, 2004. Characterization of rotavirus NSP2/NSP5 interactions and the dynamics of viroplasm formation. Journal of General Virology, 85(3):625—634
Encinas-García T, Mendoza-Cano F, Enríquez-Espinoza T et al,2015. An improved validated SYBR green-based real-time quantitative PCR assay for the detection of the Penaeus stylirostris densovirus in penaeid shrimp. Journal of Virological Methods, 212: 53—58
Galván-Alvarez D, Mendoza-Cano F, Hernández-López J et al,2012. Experimental evidence of metabolic disturbance in the white shrimp Penaeus vannamei induced by the infectious hypodermal and hematopoietic necrosis virus (IHHNV).Journal of Invertebrate Pathology, 111(1): 60—67
Geng Y, Xiao X, Yin D S et al, 2012. The interaction between viral protein and host actin facilitates the virus infection to host. Gene, 507(2): 139—145
Guo D, Rajamäki M-L, Saarma M et al, 2001. Towards a protein interaction map of potyviruses: protein interaction matrixes of two potyviruses based on the yeast two-hybrid system.Journal of General Virology, 82(4): 935—939
Hallan V, Gafni Y, 2001. Tomato yellow leaf curl virus (TYLCV)capsid protein (CP) subunit interactions: implications for viral assembly. Archives of Virology, 146(9): 1765—1773
Hou L H, Hao W, Xu L M et al, 2009. Expression and self-assembly of virus-like particles of infectious hypodermal and hematopoietic necrosis virus in Escherichia coli. Archives of Virology, 154(4): 547—553
Hsieh C Y, Chuang P C, Chen L C et al, 2006. Infectious hypodermal and haematopoietic necrosis virus (IHHNV)infections in giant freshwater prawn, Macrobrachium rosenbergii. Aquaculture, 258(1—4): 73—79
Kang S H, Lim W S, Hwang S H et al, 2006. Importance of the C-terminal domain of soybean mosaic virus coat protein for subunit interactions. Journal of General Virology, 87(1):225—229
Kang S H, Lim W S, Kim K H, 2004. A protein interaction map of soybean mosaic virus strain G7H based on the yeast two-hybrid system. Molecules & Cells, 18(1): 122—126
Li F H, Xiang J H, 2013a. Signaling pathways regulating innate immune responses in shrimp. Fish & Shellfish Immunology,34(4): 973—980
Li J, Xue J, Zhang H M, et al, 2013b. Interactions between the P6 and P5-1 proteins of southern rice black-streaked dwarf fijivirus in yeast and plant cells. Archives of Virology,158(8): 1649—1659
Lian S, Cho W K, Jo Y et al, 2014. Interaction study of rice stripe virus proteins reveals a region of the nucleocapsid protein(NP) required for NP self-interaction and nuclear localization. Virus Research, 183: 6—14
Lightner D V, 1999. The penaeid shrimp viruses TSV, IHHNV,WSSV, and YHV. Journal of Applied Aquaculture, 9(2):27—52
Lightner D V, 2011. Virus diseases of farmed shrimp in the Western Hemisphere (the Americas): A review. Journal of Invertebrate Pathology, 106(1): 110—130
Mendoza-Cano F, Enríquez-Espinoza T, Encinas-García T et al,2014. Prevalence of the infectious hypodermal and hematopoietic necrosis virus in shrimp (Penaeus vannamei)broodstock in northwestern Mexico. Preventive Veterinary Medicine, 117(1): 301—304
Sakata M, Otsuki N, Okamoto K et al, 2014. Short self-interacting N-terminal region of rubella virus capsid protein is essential for cooperative actions of capsid and nonstructural p150 proteins. Journal of Virology, 88(19):11187—11198
Shen H X, Zhang W, Shao S H, 2015. Phylogenetic and recombination analysis of genomic sequences of IHHNV.Journal of Basic Microbiology, 55(8): 1048—1052
Shike H, Dhar A K, Burns J C et al, 2000. Infectious hypodermal and hematopoietic necrosis virus of shrimp is related to mosquito brevidensoviruses. Virology, 277(1): 167—177
Silva D C D, Nunes A R D, Teixeira D I A et al, 2014. Infectious hypodermal and hematopoietic necrosis virus from Brazil:Sequencing, comparative analysis and PCR detection. Virus Research, 189: 136—146
Tacken M G J, van den Beuken P A J, Peeters B P H et al, 2003.Homotypic interactions of the infectious bursal disease virus proteins VP3, pVP2, VP4, and VP5: mapping of the interacting domains. Virology, 312(2): 306—319
Tang K F J, Durand S V, White B L et al, 2000. Postlarvae and juveniles of a selected line of Penaeus stylirostris are resistant to infectious hypodermal and hematopoietic necrosis virus infection. Aquaculture, 190(3—4): 203—210 Tang K F J, Lightner D V, 2001. Detection and quantification of infectious hypodermal and hematopoietic necrosis virus in penaeid shrimp by real-time PCR. Diseases of Aquatic Organisms, 44(2): 79—85
Tang K F J, Poulos B T, Wang J et al, 2003. Geographic variations among infectious hypodermal and hematopoietic necrosis virus (IHHNV) isolates and characteristics of their infection. Diseases of Aquatic Organisms, 53(2): 91—99
Tzeng W P, Matthews J D, Frey T K, 2006. Analysis of rubella virus capsid protein-mediated enhancement of replicon replication and mutant rescue. Journal of Virology, 80(8):3966—3974
Vega-Heredia S, Mendoza-Cano F, Sánchez-Paz A, 2012. The infectious hypodermal and haematopoietic necrosis virus: a brief review of what we do and do not know. Transboundary and Emerging Diseases, 59(2): 95—105
Wang Z Y, Wang Y Z, Sun H et al, 2015. Two proteins of Cucurbit chlorotic yellows virus, P59 and P9, are self-interacting. Virus Genes, 51(1): 152—155

