上海某高校新校区中抗生素抗性基因污染状况分析
娄 阳,张昭寰,肖莉莉,刘海泉,2,3,潘迎捷,2,3,赵 勇,2,3*
(1.上海海洋大学 食品学院,上海 201306;2.农业部 水产品贮藏保鲜质量安全风险评估实验室,上海 201306; 3.上海水产品加工及贮藏工程技术研究中心,上海 201306)
上海某高校新校区中抗生素抗性基因污染状况分析
娄 阳1,张昭寰1,肖莉莉1,刘海泉1,2,3,潘迎捷1,2,3,赵 勇1,2,3*
(1.上海海洋大学 食品学院,上海 201306;2.农业部 水产品贮藏保鲜质量安全风险评估实验室,上海 201306; 3.上海水产品加工及贮藏工程技术研究中心,上海 201306)
探究新型环境污染物—抗生素抗性基因(ARGs)在校园环境中的分布状况。通过聚合酶链式反应(PCR)对上海某高校使用5年新校区不同区域污水检查井污泥中8种四环素类、4种磺胺类、7种β-内酰胺类、4种链霉素类和5种氯霉素类ARGs进行定性研究,并利用变性梯度凝胶电泳(DGGE)技术分析污泥中细菌群落的多样性。结果显示,校园各区域中共检出19种ARGs,有8种ARGs的检出率大于50%,其中磺胺类抗性基因sulI、sulII的检出率最高,为100%。实验区及餐饮区的ARGs检出种类最多,均为14种,其次为宿舍区(12种),教学区的ARGs检出最少(8种)。通过DGGE分析细菌群落结构,证明该地区的ARGs分布与细菌多样性无明显关系。新校区使用5年但ARGs污染严重,可能是由于人类活动(尤其是科研活动)对ARGs的产生及扩散存在促进作用。此外,细菌群落多样性与ARGs种类的关系表明ARGs在环境中的迁移可能受到除细菌种类之外其他环境因素的影响。
抗生素抗性基因(ARGs);聚合酶链式反应(PCR);变性梯度凝胶电泳(DGGE);污泥
抗生素具有预防、治疗疾病和促进机体生长的功效[1],长期以来作为医药和饲料添加物在人类和动物中大量使用。多数抗生素在人畜体内未能被充分吸收和代谢,而随排泄物进入到污水、废水和土壤等环境介质中[2]。环境中大量残留的抗生素,引起微生物耐药性选择压力增强,从而诱导细菌抗生素抗性基因(Antibiotic Resistance Genes, ARGs)的产生。研究表明,ARGs可以在环境中持续性残留,并且可通过基因水平转移(Horizontal Gene Transfer, HGT)机制与质粒、转座子、整合子等可移动基因元件结合,在种间甚至属间进行迁移[3]。因此,ARGs在环境中的迁移、转化和传播,比抗生素残留本身对生态环境的危害更大[4-5]。自2006年Pruden等[3]将ARGs作为一种新型的环境污染物提出后,国内外有关其在各种介质中传播和污染的研究与报道日益增多,以探究ARGs对环境和人类健康存在的潜在生态风险。然而这些研究主要集中于抗生素用量较大的养殖场[6-7],易造成ARGs富集的污水处理厂[8],或者能够导致ARGs迁移的河流[9]等环境介质中,而对其他环境中ARGs的分布情况关注度较小。高校校园具有人口密度较高、环境复杂等特点,而且一些科研活动不可避免地涉及到抗生素的使用,这些因素都对ARGs的产生与传播扩散具有促进作用,因此应将校园作为一个重要环境介质进行ARGs污染的普查,目前国内外还未见对校园环境中ARGs分布状况的相关报道。本研究以上海某高校使用5年的新校区为研究对象,通过聚合酶链式反应(Polymerase Chain Reaction,PCR)定性分析校园环境中5大类抗生素抗性基因(四环素类、磺胺类、β-内酰胺类、链霉素类和氯霉素类)的分布情况,并利用变性梯度凝胶电泳(Denaturing Gradient Gel Electrophoresis, DGGE)技术研究样品中细菌的多样性,旨在揭示校园这一特殊环境中ARGs的污染状况。
1 材料与方法
1.1 材料
样品的采集与处理:所选地点为上海某高校的新校区,使用5年,此前该地区为上海郊区,人类活动较少,且土质多为盐碱沙土[10],不利于大多数细菌的生长与繁殖。采样地点为该校区的实验区(A)、宿舍区(B)、餐饮区(C)及教学区(D)周边的所有污水检查井(共96个),如图1所示。所采集的7个样本为井中水体液面下5~10 cm处的污泥,样品采集于2013年10月。实验楼A1、A2、A3分别从事水产类、食品类和生命科学类研究。其中,实验区的实验楼A1共采集5个污水检查井,混匀作为样本A1,实验楼A2、A3分别采集4个污水检查井,混匀作为A2、A3。男生宿舍区采集32个污水检查井,混匀作为样本B1,女生宿舍区采集25个污水检查井,混匀作为样本B2。餐饮区采集15个污水检查井,混匀作为样本C。教学区采集21个污水检查井,混匀作为样本D,混匀后每个样品约1 kg。同时,记录采样日期、天气及温度,于-80 ℃下保存。
1.2 方法
1.2.1 DNA的提取 采用试剂盒FastDNA Spin kit for soil(MP Biomedicals),按照说明书的方法提取污泥中细菌基因组DNA,提取的DNA用酶标仪(BioTek, Synergy 2)检测含量以及纯度(A260/A280值在1.8~2.0之间),表明用该试剂盒提取的DNA纯度较高。
1.2.2 样品中ARGs的检测 选取5类(28种)常见的ARGs: 8种四环素类ARGs(tet(A)、tet(B)、tet(K)、tet(M)、tet(O)、tet(Q)、tet(S)、tet(W))、4种磺胺类ARGs(sulI、sulII、sulIII、sulA)、7种β-内酰胺类ARGs(CARB、TEM、SHV、SHV-5、ampC、ermB、mecA)、5种氯霉素类ARGs(catI、catII、catIII、catIV、floR)和4种链霉素类ARGs(strA、strB、aadA、aadE)作为目的基因,用PCR扩增技术对这些目的基因进行扩增。引物序列、片段长度及退火温度见表1。

图1 该校园采样点示意及其周围污水检查井分布Fig.1 Map showing the sampling sites and the number of sewage inspection wells in the campus

引物目标基因引物序列(5′➝3′)产物长度/bp退火温度/℃文献四环素类ARGstet(A)⁃FWtet(A)⁃RVtet(A)GCGCTNTATGCGTTGATGCAACAGCCCGTCAGGAAATT3876211tet(B)⁃FWtet(B)⁃RVtet(B)TACGTGAATTTATTGCTTCGGATACAGCATCCAAAGCGCAC2066011tet(M)⁃FWtet(M)⁃RVtet(M)ACAGAAAGCTTATTATATAACTGGCGTGTCTATGATGTTCAC1716011tet(O)⁃FWtet(O)⁃RVtet(O)ACGGARAGTTTATTGTATACCTGGCGTATCTATAATGTTGAC1716011tet(Q)⁃FWtet(Q)⁃RVtet(Q)AGAATCTGCTGTTTGCCAGTGCGGAGTGTCAATGATATTGCA1696311tet(S)⁃FWtet(S)⁃RVtet(S)GAAAGCTTACTATACAGTAGCAGGAGTATCTACAATATTTAC1695011tet(W)⁃FWtet(W)⁃RVtet(W)GAGAGCCTGCTATATGCCAGCGGGCGTATCCACAATGTTAAC1686411tet(K)⁃FWtet(K)⁃RVtet(K)TCGATAGGAACAGCAGTACAGCAGATCCTACTCCTT1696111

续表1
PCR反应程序如下:94 ℃ 6 min,94 ℃ 30 s,退火30 s,72 ℃ 60 s,25~30个循环;最后72 ℃下延伸6 min,4 ℃保存。根据目标片段的大小(128~822 bp),PCR产物用2%的琼脂糖电泳检测。
1.2.3 PCR-DGGE分析污泥中细菌种群多样性 将上述提取的基因组DNA作为模板,用PCR技术扩增其16S rDNA的V3区,引物选择细菌的16S rDNA的V3高变区通用引物[14],引物序列 338F-GC:5′-CGCGCCGCCCGCGCGCCGGGCGCGGCGGGGGCACGGGGGGCCTAGGGGAGGCAGCAG-3′,518R:5′-ATTACCGCGGCTGCTGG-3′。PCR反应条件为94 ℃ 3 min,然后94 ℃ 1 min,55 ℃ 1 min,72 ℃ 1 min,30个循环;最后72 ℃延伸5 min,4 ℃保存。用BIO-RAD Dcode系统对PCR产物进行DGGE分析,聚丙烯酰胺胶浓度为8%(质量分数),变性梯度为40%~60%。在1×TAE电泳缓冲液中,100 V、60 ℃下电泳10 h。采用SYBR Green I进行染色,用Quantity One软件对凝胶图像进行分析。
2 结果与分析
2.1 抗性基因的污染状况分析

表2 该校园中抗性基因的检出情况
样本中28种抗性基因检出率统计结果见表2。结果表明,在所选的28种ARGs中,除tet(M)、catI、catII、catIII、catIV、SHV-5、ampC、strB、sulA外,其余19种抗性基因(7种四环素类、3种磺胺类、5种β-内酰胺类、3种链霉素类和1种氯霉素类ARGs)都有不同程度的检出。总体上看,该校园污泥中普遍存在四环素类、磺胺类、β-内酰胺类、链霉素类、氯霉素类抗性基因污染。
在所检测的8种四环素类ARGs中,7种四环素类ARGs(tet(A)、tet(B)、tet(K)、tet(O)、tet(Q)、tet(S)、tet(W))被检出,大部分ARGs的检出率大于42.9%,其中tet(W)的检出率可高达85.7%。其中tet(O)和tet(S)的检出率比其在九龙江河口和厦门污水处理设施中的检出率略高[15],tet(A)、tet(W)的检出率甚至大于珠江口典型水产养殖区中这两种基因的检出率[16]。造成这种现象的原因可能是四环素类抗生素以其广谱低毒的特点,现已成为水产养殖中使用的最广泛的抗生素,该校为水产类院校,四环素类抗生素使用量较大,其大量使用增加了该校园环境中四环素类抗性基因的检出率。磺胺类ARGs定性检测结果表明,除sulA未被检出外,其余3种有不同程度的检出情况,其中sulI和sulII的检出率高达100%。本研究共选择4种磺胺类ARGs作为普查对象,其中sulI和sulII在环境中较常见[17-19],而sulIII和sulA在自然环境下被检出的报道比较少[11]。在校园环境中sulI和sulII抗性基因100%的检出率以及sulIII的检出,这一现象反映出该校园中磺胺类ARGs污染较为严重。此外,在选取的7种β-内酰胺类ARGs中,除SHV-5和ampC的检出率为零外,其余5种抗性基因(CARB、TEM、SHV、ermB、mecA)在该校园污泥中均存在。而Jiang[11]在研究黄浦江及饮用水源头中的ARGs时,仅发现TEM,而本研究检出5种β-内酰胺类ARGs,说明该校园环境中存在多种β-内酰胺类ARGs。对链霉素类ARGs的检测时发现,除strB未被检出外,其余3种链霉素类ARGs均被检出,其中strA和aadA的检出率高达至87.5%。氯霉素类ARGs中仅floR被检出,检出率为28.6%。综上所述,ARGs在该校园内的检出种类多且检出率较高,因此说明该校园中普遍存在ARGs污染,污染较严重。
2.2 各区域抗性基因的分布结果分析
该校园污泥中ARGs的分布特征如图2所示,按检出ARGs种类排序:实验区(14种)、餐饮区(14种)>宿舍区(12种)>教学区(8种);按检出ARGs类别排序:实验区(5类)、宿舍区(5类) >餐饮区(4类)、教学区(4类)。可见该校园各区域均检出多种ARGs,其中实验区和餐饮区污染较为严重,宿舍区次之,教学区污染最轻。

图2 各采样点ARGs分布情况Fig.2 Distribution condition of ARGs in each site
实验区ARGs的类别和种类均多于其他几个区域,导致这一现象的原因可能是科研活动对ARGs的影响大于其余的人类活动,这一重要结论与Chemical and Engineering news中的一篇报道基本一致[20],该研究认为实验室合成的抗生素抗性基因可能通过污水排放,从而造成河流的污染。此外,表3显示:在实验区域,实验楼A1检出ARGs的种类(14种),远大于实验楼A2(7种)
及实验楼A3(5种),A1楼为生命科学类学院的实验楼,内设多个实验室大多涉及抗生素类药物的研究或使用,这可能是造成这一现象的主要原因。在餐饮区域,ARGs的检出种类处于第二位,其原因可能是食物中携带大量的ARGs[21-22]通过基因水平转移进入污泥,从而在该区域的污泥中造成污染。而宿舍区和教学区也不同程度的检出了ARGs,表明人类日常活动可能对周边环境造成ARGs污染。女宿舍ARGs的检出数(10种)略高于男宿舍(8种),可能是多数女生日常食用药物较男生多有关。
2.3 细菌群落多样性与抗性基因种类的相关性分析
由DGGE图谱(图3)分析可知,不同区域样品中细菌群落在条带数目、带型以及亮度方面均存在一定差异,说明不同区域细菌的种类和生物量有所不同;不同区域样品对应的泳道存在许多位置相同但亮度不同的条带,表明各区域均存在这些相同条带所对应的细菌种群,但丰富度有一定差异。从不同区域样品污泥中细菌DGGE指纹图谱的条带数(表3)可以看出,餐饮区的条带最多,表明其污泥中细菌种群多样性最高;A1楼、A3楼、男女宿舍、教学区的条带数目接近,A2楼条带数最少,说明A2楼污泥细菌的多样性明显低于其他样品污泥群落。细菌群落多样性与ARGs的种类相关性分析如图4,表明各样品中细菌群落多样性与该样品中ARGs的种类不存在相关性。

图3 不同样品中细菌DGGE图谱及泳道对比Fig.3 DGGE fingerprint and lane comparison of bacteria from different soils

泳道样品污泥PCR⁃DGGE条带数ARGs种类1A1楼18142A2楼1273A3楼1854男宿舍2185女宿舍19106餐饮区38147教学区198
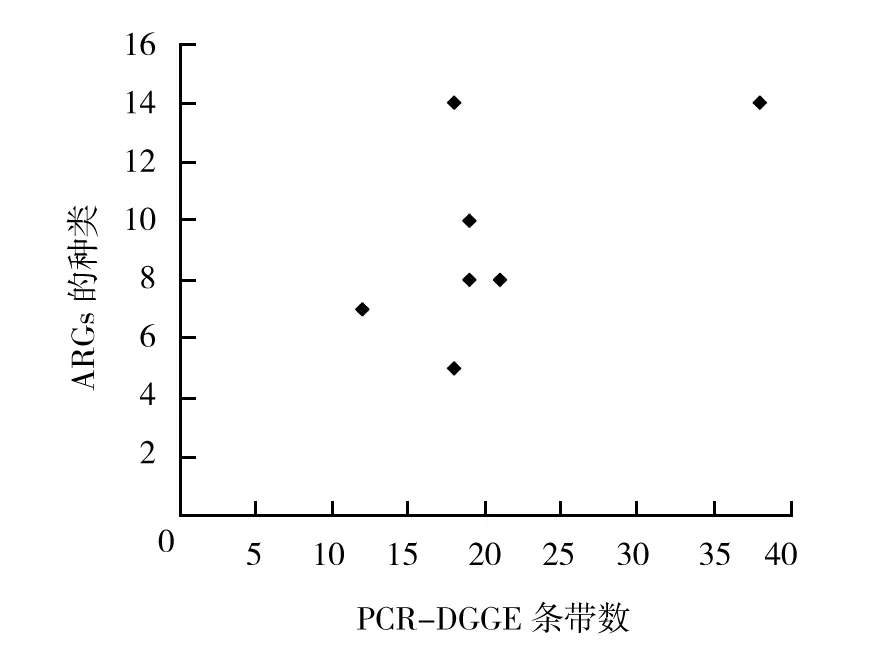
图4 ARGs数量与细菌群落多样性的相关性分析Fig.4 Correlation analysis for the number of ARGs and diversity of bacterial community
3 讨 论
目前,环境中ARGs的大量存在给人类健康和流行病学防治造成了巨大的潜在威胁[23],有关于其在养殖场、河流和污水处理厂等环境中的分布、转移等问题的研究已逐步开展[7-9],但校园这一特殊环境中ARGs污染状况的探究仍处于匮乏。因此,本研究选取上海某高校使用5年的新校区作为研究对象,通过普通PCR对该高校不同区域污水检查井污泥中5大类28种,包括8种四环素类、4种磺胺类、7种β-内酰胺类、4种链霉素类和5种氯霉素类的ARGs进行定性研究,并利用DGGE技术分析污泥中细菌群落的多样性。
定性PCR 结果表明,校园各区域中共检出19种ARGs,有8种ARGs的检出率大于50%,其中sulI、sulII的检出率最高,为100%。从ARGs在该校园的分布特征可以看出,实验区及餐饮区的ARGs检出种类最多,均为14种,其次为宿舍区(12种),教学区的ARGs检出最少(8种),结果表明该校园中ARGs污染较严重,已成为ARGs的一个重要储存库。该校区使用5年,之前该地区人类活动较少,且土质多为盐碱沙土[10],不利于大多数细菌的生长与繁殖。Takahiro等[24]研究表明,环境中细菌的浓度与ARGs的数量成正比例关系。由于该区域土壤条件不适宜多数细菌生长,因此在学校刚建成时ARGs的数量应相对较少,而本研究结果表明使用5年后的该校园环境中ARGs污染状况严重,造成这一现象的原因可能是人类活动对ARGs的产生及扩散存在一定的促进作用。许多相关研究也得出类似的推论,David等[25]研究发现随着人口密度和制药厂的增加,河流底泥中抗性基因的丰度增长了3个数量级。Pei等[26]在2006年证实了人类活动与河流中抗性基因的出现存在显著的正相关性。Pruden等[3]对美国科罗拉多州北部的河流、农田灌溉渠、湖泊等中的ARGs浓度进行检测,发现ARGs浓度在受城市和农业影响的区域显著高于那些未受影响或影响较小的区域。此外,通过DGGE分析细菌群落结构,对该校园ARGs与其微生物多样性的关系进行探究,发现各样品中ARGs的种类与该样品中细菌群落多样性不存在相关性,表明ARGs在环境中的存在可能受到除细菌种类之外的其他环境因素影响。
[1] Sapkota A, Sapkota AR, Kucharski M, et al. Aquaculture practices and potential human health risks: current knowledge future priorities[J]. Environ Int, 2008, 34(8): 1215-1226.
[2] Chee-Sanford JC, Aminov RI, Krapac IJ, et al. Occurrence and diversity of tetracycline resistance genes in lagoons and groundwater underlying two swine production facilities[J]. Appl Environ Microb, 2001, 67(4): 1494-1502.
[3] Pruden A, Pei R, Storeboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in Northern Colorado[J]. Environ Sci Technol, 2006, 40: 7445-7450.
[4] Schmitt H, Stoob K, Hamscher G, et al. Tetracyclines and tetracycline resistance in agricultural soils: Micrososm and field studies[J]. Microbial Ecol, 2006, 51(3): 267-276.
[5] Dantas G, Sommer MO, Oluwasegun RD, et al. Bacteria subsisting on antibiotics[J]. Science, 2008, 320(5872): 100-103.
[6] Cheng W, Chen H, Su C,et al. Abundance and persistence of antibiotic resistance genes in livestock farms: A comprehensive investigation in eastern China[J]. Environ Int, 2013, 61: 1-7.
[7] Marti R, Scott A, Tien YC, et al. Impact of manure fertilization on the abundance of antibiotic resistance bacteria and frequency of detection of antibiotic resistance genes in soil and on vegetables at harvest[J]. Appl Environl Microb, 2013, 79(18): 5701-5709.
[8] Gao P, Mariya M, Xagoraraki I. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J]. Sci Total Environ, 2012: 421-422: 173-183.
[9] 郭业彬,舒为群,常晓松,等. 温度与pH值对长江水系产超广谱β-内酰胺酶大肠埃希菌耐药基因转移影响分析[J]. 微生物学杂志, 2010, 30(5): 29-35.
[10]郭龙云. 上海临港新城绿化陷盐碱之困[N].中国花卉报, 2007:12-06.
[11]Jiang L, Hu XL, Xu T, et al. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Sci Total Environ, 2013, 458-460: 267-272.
[12]Ouoba LI, Lei V, Jensen LB, et al. Resistance of potential probiotic lactic acid bacteria and bifidobacteria of African and European origin to antimicrobials: Determination and transferability of the resistance genes to other bacteria[J]. Int J Food Microbiol, 2008, 121(2): 217-224.
[13]Maynard C, Fairbrother JM, Bekal S, et al. Antimicrobial resistance genes in enterotoxigenic Escherichia coli O149:K91 isolates obtained over a 23-year period from pigs[J]. Antimicrob Agents Ch, 2003, 47(10): 3214-3221.
[14]Muyzer G, Dewaal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel eletrophoresis analysis of polymerise chain reaction-amplified genes coding for 16S rRNA[J]. Appl Environ Microb, 1993, 59 (3): 695-700.
[15]何基兵, 胡安谊, 陈猛, 等. 九龙江河口及厦门污水处理设施抗生素抗性基因污染分析[J]. 微生物学通报, 2012, 39(5): 683-695.
[16]梁惜梅, 聂湘平, 施震. 珠江口典型水产养殖区抗生素抗性基因污染的初步研究[J]. 环境科学, 2013, 34(10): 4073-4080.
[17]Chad WM, Keith AL, Michael TM, et al. Tet and sul antibiotic resistance Genes in livestock lagoons of various operation type, configuration, and antibiotic occurrence[J]. Environ Sci Technol, 2010, 44(16): 6102-6109.
[18]Luo Y, Xu L, Rysz M, et al. Occurrence and transport of tetracycline, sulfonamide, quinolone, and macrolide antibiotics in the Haihe River Basin, China[J]. Environl Sci Technol, 2011, 45(5): 1827-1833.
[19]Gao PP, Mao DQ, Luo Y, et al. Occurrence of sulfonamide and tetracycline-resistant bacteria and resistance genes in aquaculture environment[J]. Water Res, 2012, 46(7): 2355-2364.
[20]邓彦阁. 实验室合成抗生素抗性基因可能造成河流污染[J]. 中国环境科学, 2013, 1: 160.
[21]Kim M, Kwon TH, Jung SM, et al. Antibiotic resistance of bacteria isolated from the internal organs of edible snow crabs[J]. Plos One, 2013, 8(8): e70887.
[22]Yutaka Y, Kaoru H, Masataka S, et al. Prevalence and antimicrobial susceptibility of vibrio species related to food safety isolated from shrimp cultured at inland ponds in Thailand[J]. Food Control, 2014, 38: 30-36.
[23]苏建强, 黄福义, 朱永官. 环境抗生素抗性基因研究进展[J]. 生物多样性, 2013, 21(4): 481-487.
[24]Takahiro S, Nozomu T, Andres B, et al. Distribution of antibiotic resistance genes in glacier environments[J]. Env Microbiol Rep, 2013, 5(1): 127-134.
[25]David WG, Susana OR, Charles WK, et al. Antibiotic resistance gene abundances associated with waste discharges to the Almendares River near Havana, Cuba [J]. Environl Sci Technol, 2010, 45(2): 418-424.
[26]Pei R, Kim SC, Carlson KH, et al. Effect of River Landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Res, 2006, 40(12): 2427-2435.
Analysis of the Occurrence of Antibiotic Resistance Genes in a New University Campus in Shanghai
LOU Yang1, ZHANG Zhao-huan1, XIAO Li-li1, LIU Hai-quan1, 2, 3,PAN Ying-jie1, 2, 3, ZHAO Yong1, 2, 3
(1.Coll.ofFoodSci.,ShanghaiOceanUni.;2.Lab.ofQual. &SafetyRiskAssessm'tforAquaticProd.onStor. &Preserv.Shanghai,Minist.ofAgric.;3.ShanghaiEngin.Res.Ctr.ofAquat-Prod.Process. &Preserv.,Shanghai201306)
The purpose of this research was to investigate the distribution of antibiotic resistance genes (ARGs) a new environmental pollutant in a university campus circumstances. Qualitative studies through PCR assays were conducted for ARGs encoding 8 categories of tetracycline, 4 sulfonamide, 5 chloramphenicol, 4 streptomycin, and 7 β-lactam from sludge collected in different wastewater checking wells in the only-5-year new university campus. Denaturing gradient gel electrophoresis (DGGE) was used to analyze the diversity of bacterial community in the sludge. The results showed that 19 ARGs were detected in samples from every area in the campus, and the detection rates of more than 50% were as high as 8 categories. Among them the sulphonamide resistance genessulI andsulII were the genes of the highest detection rate (100%). In experimental and dining areas, the largest number of ARGs (14) was detected, followed by 12 in dormitory area, and 8 ARGs were the least detected in teaching areas. DGGE analyses was done for the bacterial community structure and showed that there was no significant relationship between the distribution of ARGs and the diversity of bacterial community. The new campus was set up only 5 years, however, the ARGs pollution was considerably serious. The appearance of ARGs may have been proliferated and promoted by human activities; especially scientific research activities. Moreover, the relationship between bacterial diversity and ARGs categories suggested that some other environmental factors beyond the bacterial categories may have affected the migration of ARGs in environment.
antibiotic resistance genes (ARGs); PCR; DGGE; sludge
国家自然科学基金面上项目(31271870);上海市科委计划项目(14DZ1205100,14320502100);上海市科技兴农
娄阳 女,硕士研究生。研究方向为食品安全与生态风险。E-mail: louyangsunny@foxmail.com
* 通讯作者。男,博士,教授。研究方向为食品安全学。Tel: 021-61900503, E-mail: yzhao@shou.edu.cn
2014-08-25;
2014-09-29
Q938.1+1;X17
A
1005-7021(2015)04-0046-08
10.3969/j.issn.1005-7021.2015.04.009
重点攻关项目(沪农科攻字2014第3-5号、2015第4-8号);上海水产品加工及贮藏工程技术研究中心平台项目(11DZ2280100);
上海市教委人才培养项目(B5201120040)

