烟草青枯菌FQY_4基因组中原噬菌体生物信息学分析
蔡刘体,刘艳霞,孟琳,罗正友,石俊雄
贵州省烟草科学研究院,贵州 贵阳 550081
烟草青枯菌FQY_4基因组中原噬菌体生物信息学分析
蔡刘体,刘艳霞,孟琳,罗正友,石俊雄
贵州省烟草科学研究院,贵州 贵阳 550081
为了解青枯菌(ralstonia solanacearum)基因组中原噬菌体和噬菌体元件的信息,在烟草青枯菌FQY_4全基因组序列的基础上,分析了FQY_4染色体上的原噬菌体,结果显示FQY_4染色体上有1个具有完整噬菌体特征的原噬菌体,3个噬菌体元件。比较青枯菌GMI1000和FQY_4中的原噬菌体的序列信息,结果显示它们具有一定的相似性,其中青枯菌FQY_4的原噬菌体-2与噬菌体ϕRSA1的相似度为82%,它们有40个编码基因高度同源,相似度为99%以上。这些信息可为青枯菌原噬菌体的多样性分析,噬菌体和青枯菌的协同进化研究奠定基础。
烟草;青枯病;青枯菌;基因组;原噬菌体
噬菌体(bacteriophage,phage)是感染细菌、真菌、放线菌或螺旋体等微生物的病毒的总称,噬菌体无处不在,只要有细菌存在的地方就可能有噬菌体的身影,在土壤中可找到大量细菌的噬菌体[1-2]。几乎所有的细菌都可能被噬菌体侵蚀[3],包括引起农作物细菌性病害的土传性病原菌[4-5],已报道的能侵蚀青枯菌的噬菌体有短尾噬菌体科、肌尾噬菌体科和丝状噬菌体[9]。由于协同进化和竞争关系,事实上噬菌体与细菌性病原菌的基因组有密切关系[6-8],噬菌体侵入细菌细胞后,在细菌细胞中有两种循环方式,裂解性噬菌体通过裂解细菌细胞有效地自我复制,而温和性噬菌体侵入细菌细胞后不裂解宿主,而是整合到宿主基因组中,形成原噬菌体(prophage)[10]。原噬菌体一般情况下不引起病原菌细胞裂解,而是随着病原菌染色体的复制而复制,繁殖和传递噬菌体本身遗传信息。虽然原噬菌体没有裂解宿主的特性,但是需要表达一些调节基因和附属基因来维持原噬菌体的温和性状态[11-12]。这些调节基因和附属基因也可能赋予宿主细胞新的表型特性,比如原噬菌体中的遗传单元可能对宿主的致病性有贡献[13-14],另一方面,原噬菌体在宿主中也会发生突变和衰退,在宿主不断的繁衍传代过程中,丢失一些关键基因,最终导致原噬菌体的丧失[10]。
随着基因组测序技术的不断发展,自青枯菌GMI1000基因组测序完成之后,越来越多的青枯菌株系的全基因组测序工作已经完成[15-16],包括烟草青枯菌株系Y45和FQY_4[17-18]。青枯菌基因组中的原噬菌体序列也有报道,Askora等[19]从复杂种青枯菌株系中发现两株噬菌体RSM3和RSM4,这两株噬菌体的相关性非常近,但与丝状体噬菌体不同,青枯菌株系MAFF30139的基因组中有RSM3噬菌体的前体,能通过PCR和转化的方式使它反转成有侵蚀能力的噬菌体。通过序列比较分析显示,除了编码未知蛋白的读码框(ORF2)和编码决定宿主范围的吸附蛋白外,噬菌体RSM3和RSM1的核酸序列具有高度的保守性[20],被噬菌体RSM1和RSM3侵染后的青枯菌细胞,其群体聚合力增强,且青枯菌细菌对其宿主番茄的致病能力降低。但烟草青枯菌基因组中原噬菌体的研究未见报道,本文在烟草青枯菌FQY_4全基因组测序的基础上,分析FQY_4基因组中原噬菌体的序列和噬菌体元件,以期为从青枯菌基因组中诱导和从植烟土壤环境中筛选对烟草青枯菌具有侵蚀能力的噬菌体的研究提供信息。
1 材料与方法
材料:青枯菌GMI1000基因组信息从GenBank数据库中获取[15]。烟草青枯菌FQY_4,来自贵州省烟草青枯病病圃,其全基因组序列通过Hiseq 2000高通量测序完成,FQY_4基因组在GenBank的登录号分别是 CP004012 和 CP004013[18]。
方法:采用Prohinder(网址:http://aclame.ulb.ac.be/Tools/Prophinder/)、PHAST(PHAge Search Tool,噬菌体搜索工具)(网址http://phast.wishartlab.com/)和ACLAME数据库在线分析烟草青枯菌FQY_4基因组中的原噬菌体。在Prohinder和PHAST网站上提交FQY_4基因组的GenBank登录号,获取分析后得分在50以上的原噬菌体信息,然后在ACLAME数据库中噬菌体比较分析。原噬菌体的读编框(Open Reading Frame,ORF)分析通过NCBI网站的ORFs完成。
2 结果与分析
2.1 烟草青枯菌FQY_4基因组中的原噬菌体
采用Prohinder软件对青枯菌FQY_4基因组染色体和巨大质粒中的原噬菌体进行预测分析,结果显示FQY_4染色体上有4个原噬菌体及原噬菌体元件(图1),其中1个是原噬菌体(Prophage_2),具有典型噬菌体基因组的完整的特性,另外3个是不完整的原噬菌体,即噬菌体元件。FQY_4的巨大质粒上没有发现原噬菌体或噬菌体元件的序列。FQY_4染色体基因组上的4个原噬菌体和噬菌体元件的起始点和终止点位置信息如表1所示。
图1显示,原噬菌体-2(Prophage_2)具有完整噬菌体的编码功能,它包含了53个编码区域,具有典型的噬菌体特征,如外壳蛋白、附着点、尾部因子、tRNA等编码序列(图2)。原噬菌体-2的整个序列长度为40285 bp,GC含量为65.64%,与FQY_4整个基因组中的GC含量类似[18],其大小与青枯菌一般的噬菌体基因组类似[20-22],但是比青枯菌噬菌体ΦRSL1基因组小[23]。

图1 青枯菌FQY_4染色体中原噬菌体位置分布Fig.1 Position of prophages in the chromosome of Ralstonia Solanacearum strain FQY-4
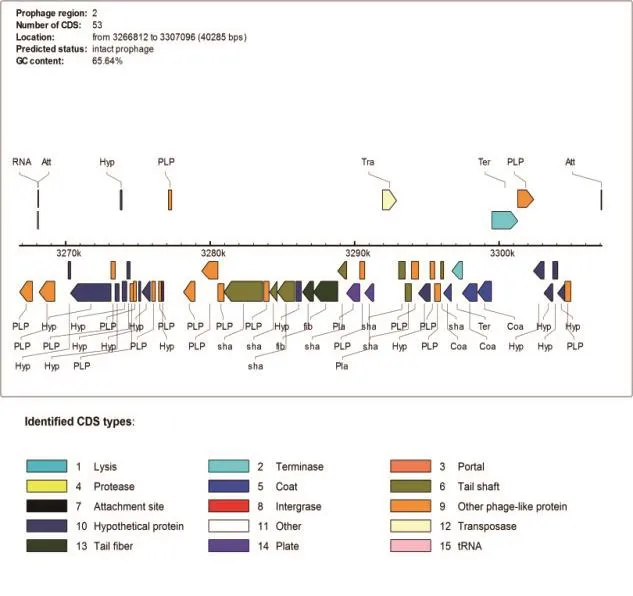
图2 青枯菌FQY_4染色体的Prophage_2编码基因示意图Fig.2 Coding genes of prophage-2 in the chromosome of Ralstonia Solanacearum strain FQY-4
2.2 青枯菌FQY_4的原噬菌体与青枯菌GMI1000的原噬菌体、噬菌体ϕRSA1的比较
采用Prohinder预测青枯菌GMI1000和FQY_4的原噬菌体信息如表1所示,青枯菌FQY_4和GMI1000染色体上的原噬菌体或噬菌体元件的数目和大小相似,且各包含有一个完整的原噬菌体,原噬菌体和噬菌体元件编码的总基因数目相近,但是它们在各自染色体上的分布位置差别比较大。Salanoubat等[15]报道青枯菌株系GMI1000染色体上至少有4个原噬菌体或噬菌体元件,它们与GMI1000染色体一个接合转座子Tn4371结合在一起,推断噬菌体元件是通过基因水平转移方式获得。GMI1000染色体上具有完整噬菌体特征的原噬菌体ϕRSΧ与能侵蚀裂解多株青枯菌的噬菌体ϕRSA1很相似[15]。Blast比较分析显示,青枯菌FQY_4的原噬菌体-2与ϕRSA1具有很高的同源性,尤其是编码的末端酶、外壳蛋白、尾丝、附着点蛋白的相似性(表2),说明青枯菌FQY_4中原噬菌体及噬菌体元件可能在它们协同进化过程中获得后整合到染色体上。
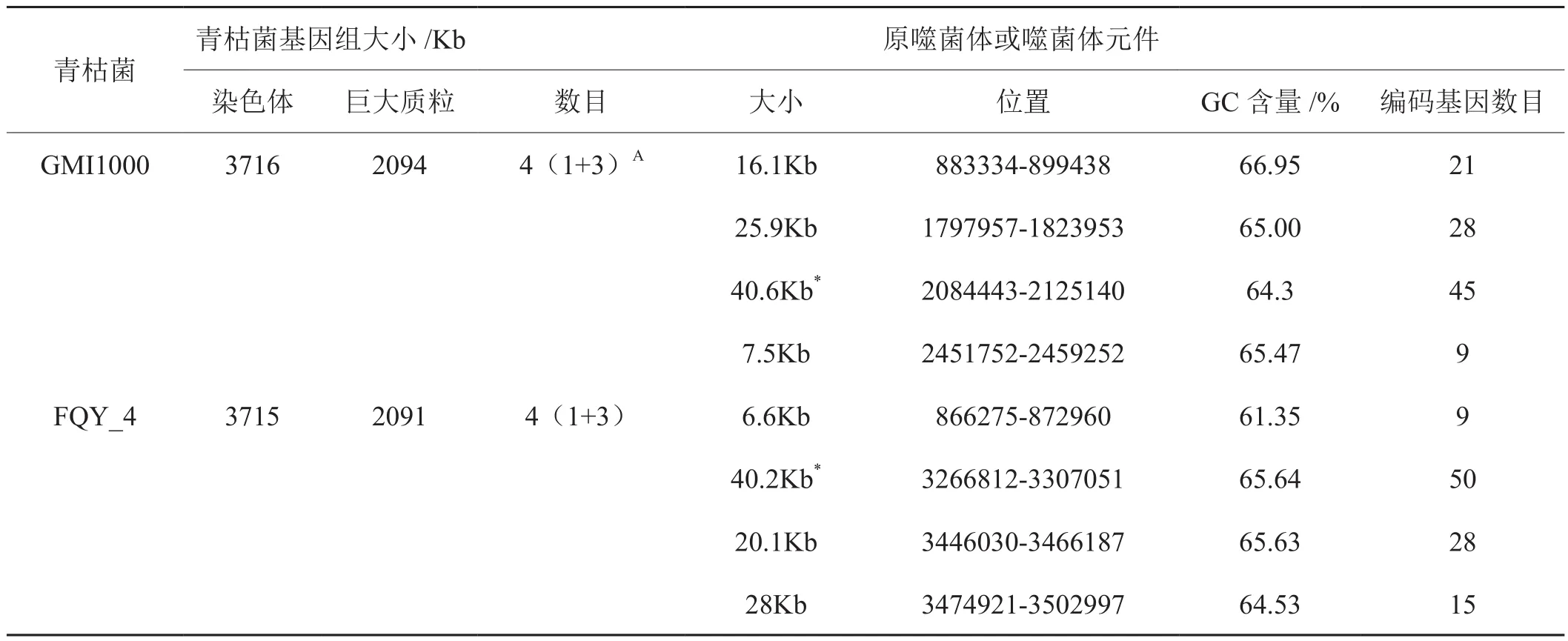
表1 青枯菌FQY_4和GMI1000染色体上原噬菌体及噬菌体元件Tab.1 Prophages and its elements in chromosome of Ralstonia Solanacearum strain FQY-4 and GMI1000

表2 青枯菌FQY_4原噬菌体-2与噬菌体ϕRSA1的相似性比较Tab.2 Homology comparison between prophage-2 in Ralstonia Solanacearum strain FQY-4 and phage ϕRSA1

续表2
3 讨论
噬菌体是细菌进化的主要驱动力之一,通常认为噬菌体驱动细菌的进化,在噬菌体的压力下,迫使在细菌群体中产生具有适应性的细菌,细菌与裂解性噬菌体的竞争性协同进化会加速协同进化物种之间的分子进化速率[24]。在测序中的青枯菌株系基因组中,许多的基因岛与预测的原噬菌体或噬菌体元件相关[25]。有趣的是原噬菌体-3的位置与染色体上一个基因岛(chr_GI10)位置有部分重叠区域,暗示FQY_4基因组中原噬菌体与基因岛上基因富集区具有一定的联系,但是其关系还需要进一步的分析。
噬菌体在土壤中不仅含量丰富,而且也是影响土壤中微生物群落进化的关键因素[26-27],同时,噬菌体还是基因水平转移(HGT)的有效载体[28],它能使病原菌快速的获得适应性功能,在病原菌多样性的出现中起重要作用。原噬菌体与病原菌的致病力也有一定的关系[29-30],青枯菌原噬菌体基因簇的序列分析显示它们中分布着好几个涉及植物-细菌相互作用的基因,如III型效应因子(T3Es)的基因(popP1,popP2,ripT等),Addy等[31-32]研究发现被丝状噬菌体ϕRSS1侵染后的青枯菌在西红柿植株内的致病力增加,而被噬菌体ϕRSM侵染后的青枯菌丧失了致病力。
三个青枯菌株系(GMI1000,Po82和CMR15)含有popP2效应因子基因,该基因与一个分布在基因组不同位置上的原噬菌体相关,而且研究显示青枯菌菌株的原噬菌体显然具有菌株特异性[21,33-34],表明它们在青枯菌菌株中分布具有多样性,谭锦等[35]研究表明柑橘黄龙病病原菌株系在原噬菌体区域具有较丰富的多样性。
细菌中的原噬菌体在一定条件下可以被诱导,形成有侵染能力的噬菌体,阮萃才等[36]利用原噬菌体诱导法检测环境中的诱变剂,张子千等[37]报道利用丝裂霉素C从嗜盐古生菌Natinema sp.J7-1中诱导分离得到嗜盐古生菌溶原噬菌体SNJ1,它能侵染Natrinema sp.J7-2并在双层平板上形成清晰的噬菌斑。烟草青枯菌FQY_4基因组中原噬菌体-2具有典型噬菌体的特征,并与对青枯菌具有侵蚀裂解能力的噬菌体ϕRSA1具较高同源性,研究有效的诱导方法把原噬菌体-2诱导成对烟草青枯菌具有侵蚀性的噬菌体,利用原噬菌体-2和噬菌体ϕRSA1的序列信息特征从植烟土壤或其它环境中筛选对烟草青枯菌具有侵蚀性的噬菌体,对于利用烟草青枯菌噬菌体建立烟草青枯病的生物防控策略具有重要意义。
[1]Michiel Vos,Birkett PJ,Birch E,et al.Local adaptation of bacteriophages to their bacterial hosts in soil [J].Science,2009,325(5942):833.
[2]徐凤宇,苏胜兵,马红霞,等.不同类型土壤中分枝杆菌噬菌体分离率的比较[J].生态学报,2013,33(15):4616-4622.
[3]Wommack KE,Colwell RR.Virioplankton:viruses in aquatic ecosystems.Microbiol[J].Mol Biol Rev,2000,64(1):69-114.
[4]Kidambi SP,Ripp S,Miller RV.Evidence for phagemediated gene transfer amongPseudomonas aeruginosastrains on the phylloplane[J].Appl.Environ.Microbiol.,1994,60(2):496-500.
[5]Killiny N,Almeida RPP.Host structural carbohydrate induces vector transmission of a bacterial plant pathogen[J].Proc.Natl.Acad.Sci.USA.,2009,106(52):22416-22420.
[6]蔡刘体,汪汉成,袁赛飞,等.青枯菌特异性噬菌体的研究进展与应用[J].生物技术通讯,2012,11:145-148.
[7]Kawasaki T.,Shimizu M.,Satsuma H,et al.Genomic characterization ofRalstonia solanacearumphage ФRSB1,a T7-like wide-host-range phage[J].J.Bacteriol.,2009,191(1):422-427.
[8]Yamada T,Satoh S.,Ishikawa H,et al.A jumbo phage infecting the phytopathogenRalstonia solanacearumde fi nes a new lineage of the Myoviridae family[J].Virology,2010,398(1):135-147.
[9]Kawasaki T.,Nagata S.,Fujiwara A.,et al.Genomic characterization of the filamentous integrative bacteriophage ФRSS1 and ФRSM1,which infectRalstonia solanacearum[J].J.Bacteriol.,2007,189(16):5792-5802 .
[10]Brussow H,Canchaya C,Hardt WD.Phages and the evolution of bacterial pathogens:from genomic rearrangements to lysogenic conversion[J].Microbiol.Mol.Biol.Rev.,2004,68(3):560-602.
[11]Koide T,Zaini PA,Moreira LM,et al.DNA microarraybased genome comparison of a pathogenic and a nonpathogenic strain ofXylella fastidiosadelineates genes important for bacterial virulence[J].J.Bacteriol.,2004,186(16):5442-5449.
[12]Nunes LR,Rosato YB,Muto NH,et al.Microarray analyses ofXylella fastidiosaprovide evidence of coordinated transcription control of laterally transferred elements[J].Genome Res.,2003,13(4):570-578.
[13]Canchaya C,Fournous G,Brussow H.The impact of prophages on bacterial chromosomes[J].Mol.Microbiol.,2004,53(1):9-18.
[14]Casjens S.Prophages and bacterial genomics:What have we learned so far?[J].Mol.Microbiol.,2003,49(2):277-300.
[15]Salanoubat M,Genin S,Artiguenave F,et al.Genome sequence of the plant pathogenRalstonia solanacearum[J].Nature,2002,415:497-502.
[16]Genin and Denny.Pathogenomics of theRalstonia solanacearumspecies complex[J].Annu.Rev.Phytopathol.,2012,50(4):1-23.
[17]Li Zefeng,Wu Sanling,Bai Χuefei,et al.Genome Sequence of the Tobacco Bacterial Wilt PathogenRalstonia solanacearum[J].J Bacteriol.,2011,139(21):6088-6089.
[18]Cao Yi,Tian Baoyu,Liu Yanxia,et al.Genome Sequencing ofRalstonia solanacearumFQY_4,Isolated from a Bacterial Wilt Nursery Used for Breeding Crop Resistance[J].Genome announcements,2013,(1)3:1-2.
[19]Askora A.,Kawasaki T.,Usami S.,et al.Host recognition and integration of filamentous phage RSM in the phytopathogenRolstonia Solanacearum[J].Virology,2009,(384):69-76.
[20]Askora A,Kawasaki T,Fujie,et al.Resolvase-like serine recombinase mediates integration/excision in the bacteriophage φRSM[J].J Biosci Bioeng,2011,111(2):109-116.
[21]Fujiwara A.,Kawasaki T.Usami S.,et al.Genomic characterization ofRalstonia solanacearumphage ФRSA1 and its related prophage(ФRSΧ) in strain GMI1000[J].J.Bacteriol.,2008,190(1):143-150.
[22]Kawasaki T.,Nagata S.,Fujiwara A.et al.Genomic characterization of the filamentous integrative bacteriophage ФRSS1 and ФRSM1,which infectRalstonia solanacearum[J].J.Bacteriol.,2007,189(16):5792-5802 .
[23]Effantin G,Hamasaki R,Kawasaki T,et al.Cryo-Electron Microscopy Three-Dimensional Structure of the Jumbo Phage ΦRSL1 Infecting the PhytopathogenRalstonia solanacearum[J].Structure,2013,21(2):298-305.
[24]Clokie MR,Millard AD,Letarov AV,et al.Phages in nature[J].Bacteriophage,2011; 1:31-45.
[25]Remenant B,de Cambiaire JC,Cellier G,et al.Ralstonia syzygii,the blood disease bacterium and some asianR.solanacearumstrains form a single genomic species despite divergent lifestyles[J].PLoS ONE,2011.6:e24356.
[26]Gomez P and Buckling A.Bacteria-phage antagonistic coevolution in soil[J].Science,2011,(332):106-109.
[27]Ronning CM,Losada L,Brinkac L,et al.Genetic and phenotypic diversity inBurkholderia:contributions by prophage and phage-like elements[J].BMC Microbiol,2010(10):202.
[28]Kenzaka T,Tani K,Nasu M.High-frequency phagemediated gene transfer in freshwater environments determined at single-cell level[J].ISME J,2010(4):648-659.
[29]Varani AM,Monteiro-Vitorello CB,Nakaya H I,et al.The Role of Prophage in Plant-Pathogenic Bacteria[J].Annual review of phytopathology,2013(51):429-451.
[30]Tyler JS,Beeri K,Reynolds J L,et al.Prophage Induction Is Enhanced and Required for Renal Disease and Lethality in an EHEC Mouse Model[J].PLoS pathogens,2013,9(3):e1003236.
[31]Addy H S,Askora A,Kawasaki T,et al.Loss of virulence of the phytopathogenRalstonia solanacearumthrough infection by ϕRSM fi lamentous phages[J].Phytopathology,2012,102(5):469-477.
[32]Addy H S,Askora A,Kawasaki T,et al.The filamentous phage φRSS1 enhances virulence of phytopathogenicRalstonia solanacearumon tomato[J].Phytopathology,2012,102(3):244-251.
[33]Gabriel DW,Allen C,Schell M,et al.Identification of open reading frames unique to a select agent:Ralstonia solanacearumrace 3 biovar 2[J].Mol.Plant-Microbe Interact,2006(19):69-79.
[34]Guidot A,Elbaz M,Carrere S,et al.Specific genes from the potato brown rot strains ofRalstonia solanacearumand their potential use for strain detection[J].Phytopathology,2009( 99):1105-1112.
[35]谭锦,王雪峰,苏华楠,等.中国柑橘黄龙病病原菌两个原噬菌体超变异基因遗传多样性[J].中国农业科学,2013,46(18):3784-3792.
[36]阮萃才,梁远,刘锦玲,等.原噬菌体诱导法在环境诱变剂研究中的应用[J].环境科学,1990,11(3):41-43.
[37]张子千,刘莹,陈向东.Natrinema属极端嗜盐古生菌溶原性噬菌体 SNJ1 的相关研究[C]//2012年湖北生物产业发展高端论坛暨湖北省生物工程学会2012年度学术交流会论文汇编,2012.
Bioinformatic analysis of prophage in genome of tobacco pathogenRalstonia solanacearumFQY_4
CAI Liuti,LIU Yanxia,MENG Lin,LUO Zhengyou,SHI Junxiong
Guizhou Tobacco Research Institute,Guiyang,550081,China
Prophages in chromosome of FQY-4 was analyzed on the basis of its complete genomic sequence.Results showed that there was one prophage which contained typical characteristics of phage,and three incomplete prophage elements.Compared with theRalstonia Solanacearumstrain GMI1000,FQY_4 shared similarity in prophage,and homology between the prophage-2 in FQY_4 and prophage ϕRSA1 was 82%.There were 40 coding sequences with highly signi fi cant homology level of more than 99%,which laid solid foundation for diversity analysis of prophage ofRalstonia Solanacearum,and for co-evolution ofRalstonia solanacearumand phages.
tobacco; bacterial wilt;Ralstonia solanacearum; genome; prophage
蔡刘体,刘艳霞,孟琳,等.烟草青枯菌FQY_4基因组中原噬菌体生物信息学分析[J].中国烟草学报,2015,21(1)
中国烟草总公司贵州省公司科技项目“烟草青枯菌致病基因家族分析”(合同号:黔烟科合2012-03);农业部公益性行业(农业)科研专项项目“利用有机(类)肥料调控我国土壤微生物区系关键技术研究”(合同号:201103004)
蔡刘体(1974— ),博士,副研究员,从事土肥植保工作,Email:cailiuti01 @163.com
石俊雄(1966— ),研究员,主要从事烟草营养与施肥,植烟土壤微生物生态修复工作,Email:sjx2196@163.com
2013-12-13
:CAI Liuti,LIU Yanxia,Meng Lin,et al.Bioinformatic analysis of prophage in genome of tobacco pathogenRalstonia solanacearumFQY_4 [J].Acta Tabacaria Sinica,2015,21(1)

