DNA指纹图谱技术在奶酪发酵菌群中的应用研究
王 娟,张铭霞,曹雁平,杨贞耐,王 蓓,,*
(1.北京工商大学,北京市食品添加剂工程技术研究中心,北京100048;2.北京工商大学,北京市食品风味化学重点实验室,北京100048;3.北京工商大学,食品质量与安全北京实验室,北京100048)
DNA指纹图谱技术在奶酪发酵菌群中的应用研究
王 娟1,张铭霞2,曹雁平1,杨贞耐3,王 蓓1,2,*
(1.北京工商大学,北京市食品添加剂工程技术研究中心,北京100048;2.北京工商大学,北京市食品风味化学重点实验室,北京100048;3.北京工商大学,食品质量与安全北京实验室,北京100048)
DNA指纹图谱技术是近年来较为常用的分子生物学技术,由于发酵菌群的DNA指纹图谱直接反映DNA水平上的差异,因而作为微生物指纹图谱的一个组成部分,DNA指纹图谱这一现代分子生物学技术在微生物研究中有其独特的作用,这种技术在发酵菌群的鉴定、遗传育种及多样性等研究领域有广阔的应用前景。常用于发酵菌群研究的DNA指纹图谱技术有RFLP、T-RFLP、SSCP、RAPD、DGGE、AFLP等。本论文在综合介绍DNA指纹图谱的一般常用技术的基础上,结合近年来国内外奶酪发酵菌群中DNA指纹图谱技术研究现状,在探讨其在奶酪发酵菌群相关领域应用的基础上,逐一介绍了各种技术的原理、优缺点及研究现状,对我国传统奶酪发酵菌群的研究具有重要的实际意义与理论价值。
DNA指纹图谱,传统奶酪,发酵菌群,分子生物学
DNA指纹图谱技术是通过物质在DNA水平上的差异,以现代分子生物学方法来标记,同时构建DNA指纹图的一种技术。由于同种微生物的核苷酸序列具有相对稳定性,而不同种类微生物核苷酸序列具有一定差异性,因而可以通过现代分子生物学技术构建体系微生物DNA指纹图谱的方法,达到对微生物菌群多样性的研究。DNA指纹图谱技术已成功地应用于许多领域中,如发酵食品中的微生物群落分析研究[1],样品原料或中间产物中微生物群落的多样性分析等[2]。
传统奶酪中发酵菌群丰富又复杂,对奶酪的风味和流变学性质有重要的影响。因而近年来奶酪发酵过程中微生物多样性及其对奶酪感官品质的影响的相关研究越来越为人们所关注。人们采用不同的DNA指纹图谱技术对奶酪中的发酵菌群进行系统研究,常用的DNA指纹图谱技术有限制性片段长度多态性、末端限制性片段长度多态性、单链构象多态性、变性梯度凝胶电泳、随机扩增多态DNA技术、扩增片段长度多态性、分子杂交技术等,本文拟从这些方法的基本原理及其在奶酪微生物多样性相关分析应用等方面进行阐述。
1 限制性片断长度多态性
限制性片断长度多态性(restriction fragment length polymorphism,RFLP),又称为核糖体DNA扩增片段限制性内切酶分析(amp lified ribosomal DNA restriction analysis,ARDRA),该技术是通过DNA的多态性来研究微生物群落多样性的方法。RFLP技术主要是基于PCR技术扩增目的基因序列,然后选择特异限制性内切酶将扩增的目的基因序列切割成大小不同的DNA片断,通过聚丙烯酰胺凝胶电泳来分析这些片段,从而获得待测样品中微生物群个体差异信息,以达到对样品中菌群分析并鉴定的目的。RFLP方法适用于比较分析不同样品或是样品在不同时期微生物菌落结构的变化,尤其适用于大批量样品的分类、鉴定研究[3],由于该方法操作简单,因而在奶酪微生物菌群研究中仍被广泛使用。Dariush等对伊朗游牧部落Motal奶酪的益生菌群落结构进行了PCR-RFLP技术分析,并在此技术基础上确定得到Motal奶酪中益生菌的主要类型是嗜酸乳杆菌和植物乳杆菌[4]。
RFLP技术虽然简单,但由于基因组信息较大,酶切图谱条带过于复杂,因而存在评价结果准确度相对较低的问题。因此近年来在RFLP技术的基础上,又发展出了两种新技术,分别是末端限制性片断长度多态性(term inal restriction fragment length polymorphism,T-RFLP)技术和tRNAAla-23S rDNA限制性片断长度多态性(tRNAAla-23S ribosomal DNA Restriction Fragment Length Polymorphism,tRNAAla-23S rDNA-RFLP)技术。
T-RFLP与RFLP技术主要的区别是在T-RFLP中,一个引物的5'末端用荧光物质来标记,因而检测得到的为带有荧光标记的末端片断[5],该步骤极大地简化了图谱带型,使得T-RFLP技术更易于进行复杂菌落结构分析并获得发酵菌群多样性的更多信息,同时该技术也可通过图谱条带来评价微生物的丰度、均度,以及样品间的相似性,因而得到的信息更加准确[6]。Rossetti等[7]在对奶酪微生物种群(乳酸菌,柠檬串珠菌株等)研究过程中,同时对比了以逆转录酶(reverse transcriptase,RT)-PCR为基础的T-RFLP技术进行的半定量分析结果和利用菌株表型鉴定以及生理生化实验进行的传统培养所得到的分析结果,对比结果表明T-RFLP技术可以对奶酪发酵过程中菌群的细微变化进行检测,并且重复性较好,而传统的菌落计数法灵敏度较低,对菌群的细微变化无法检测。此外,虽然和传统培养技术相比T-RFLP技术具有快捷、灵敏度高的特点,但T-RFLP技术的使用范围仍具有一定限制,尤其对复杂环境内的样品而言,由于在该环境下不同的微生物在酶切后可能会产生相同的切割片断,因而易对样品中微生物的种类与含量作出错误估计,导致结果的不确定性[3]。
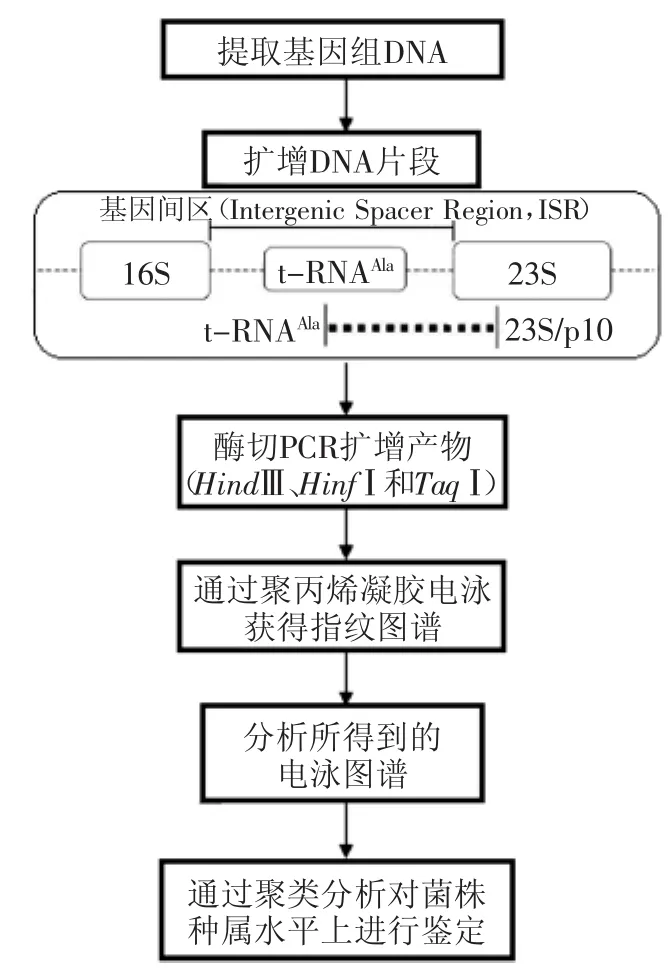
图1 tRNAAla-23S rDNA-RFLP技术的具体流程Fig.1 tRNAAla-23S rDNA-RFLPworkflow
tRNAAla-23S rDNA-RFLP技术是将16S-23S rDNA基因间区(Intergenic Spacer Region,ISR)分析技术与RFLP技术联合的一种新方法。该方法是利用转录核糖体30S小亚基的16S rDNA和50S大亚基的23S rDNA的保守区段与基因间区(ISR)来分析样品中发酵菌群系统发育情况,并对菌群进行分类和鉴定的重要方法。其中由于16S-23S rDNA的序列具有长度和序列的可变性,变异的程度比16S rDNA和23S rDNA要大,因而能够得到更加准确的结果。该方法的操作流程如图1所示:同时采用两个特异引物:tRNAAla(由酒类酒球菌的tDNAAla的一个保守的序列进行设计)与23S/p10(位于大肠杆菌中23S rRNA基因的456~474),利用PCR技术,对基因区间序列进行扩增,扩增产物再用限制性内切酶(HindⅢ、HinfⅠ和TaqⅠ)进行酶切,最后酶切产物用聚丙烯酞胺凝胶电泳,观察酶切片段的多态性,统计分析电泳图谱,即可得到待测样品中不同菌株的遗传相似水平,还可以用于鉴定样品中发酵菌株之间的系统发育关系。Mancini等[8]同时运用tRNAAla-23S rDNA-RFLP技术与其他两种DNA指纹图谱技术,即随机扩增多态性DNA(random amplified polymorphism DNA,RAPD)和重复基因外回文序列分析技术(repetitive extragenic palindrom ic-PCR,rep-PCR),对Grana Padano奶酪成熟过程中分离得到的典型乳酸菌种间和种内的多样性进行研究,分析结果表明,tRNAAla-23S rDNA-RFLP技术的重现性要高于RAPD和rep-PCR技术,并且靶tRNAAla-23S rDNA序列在细菌鉴定应用中可获得更加准确及有价值的信息,从而能够更好地对奶酪中乳酸菌在种属水平进行进一步的分析与鉴定。
2 逆转录变性梯度凝胶电泳
变性梯度凝胶电泳技术(denaturing gradient gel electrophoresis,DGGE)是一种快速、经济、可靠、重复性好的分子生物学技术,能够同时实现多个样品的分析。该技术能较为客观与直观地鉴定及分析微生物,因而被广泛用于微生物分子生态学研究的各个领域。目前已经发展成为研究微生物群落结构的主要分子生物学方法之一[9-10],也常用于检测奶酪发酵过程中微生物菌落变化[11]。该技术的主要原理是:在变性梯度聚丙酰胺凝胶电泳中,长度相同但序列不同的DNA分子会在变性剂(尿素、甲酰胺)的作用下产生不同的解链行为,在外加电场的作用下会在凝胶中形成不同的迁移位置,从而使DNA片断得到分离。电泳结束后,经过染色即可观察图谱中分开的不同带型。虽然DGGE技术已广泛用于多种食品发酵过程中菌群研究,然而同其他基于PCR的分子生物技术一样,DGGE技术除了会受到PCR扩增时产生偏差的影响外,也具有一定的自身局限性。例如,DGGE适于分离200~500bp的目的基因片断,超出此范围的片断则难以分析,因此所得序列仅能提供较为有限的系统发育信息。其次,研究表明DGGE仅能检测到发酵乳制品中微生物含量达到1%~2%以上的优势菌种,而弱势菌种一般不易被检测到[12],因而会造成对样品中某些发酵菌群多样性的低估或者高估。
由于受到传统加工技术与地域的影响,奶酪中的微生物具有独特性,并且不同微生物群体在发酵过程的不同阶段活跃程度不同,因而为了更好地揭示手工奶酪的微生物代谢活性,一些研究者利用RT-RNA技术对奶酪中发酵菌株的多样性进行进一步分析。通过结合RT-PCR-DGGE(基于RNA)和PCRDGGE(基于DNA),从奶酪总体微生物(DNA衍生的)中区分代谢活性较强(RNA衍生的)的微生物,从而更好地研究奶酪发酵过程中微生物菌群变化。Randazzo等[13]对手工Sicilian奶酪成熟过程中发酵菌群的DNA衍生的DGGE图谱与RNA衍生的DGGE图谱进行比较,表明了该奶酪在不同发酵阶段微生物群体具有不同的代谢活性。已有研究结果表明RTPCR-DGGE方法对发酵时间较长的奶酪中菌群变化的相关研究非常有效[14],并且由于RNA的稳定性相对于DNA而言较差,RNA会在已死亡的微生物中迅速地降解,因此基于RNA的测定比基于DNA的测定法更为敏感。为了获得Fontina PDO奶酪在发酵过程中细菌群落的变化以及该奶酪中的特征微生物菌群更加详细的信息,Dolci等[15]将PCR-DGGE和RTPCR-DGGE技术同时应用于Fontina PDO奶酪的表面,亚表层以及奶酪内部。研究结果表明:RT-PCRDGGE分析方法得到的电泳条带更为丰富,因而基于RNA的RT-PCR-DGGE技术能够给出奶酪在发酵过程中微生物多样性的更详细信息。
3 单链构象多态性
单链构象多态性(single strand conformation polymorphism,SSCP)技术是近年来随着对人类基因组进一步深入研究而发展起来的一种与PCR技术相结合,利用电泳方法分离构象不同的DNA序列,从而检测和分析碱基突变的一种分子生物学技术。由于单链DNA自身核苷酸序列以及物理化学环境折叠成的三级结构不同导致其在非变性凝胶中的电泳迁移率存在差异,因而利用此点可以对DNA单链中基因突变进行检测和分析。
这项技术是由Orita等[16]建立的,最初用来检测已知或未知的DNA多态性或基因的点突变,目前已成为继PCR-DGGE后,研究手工奶酪中复杂的微生物发酵菌群最常用的分析方法。当前大量研究表明SSCP方法操作简单快捷、灵敏度高,但对实验条件(电泳条件)要求相对较严格。Callon等[17]结合SSCP技术与PCR技术对3种已注册产地标记(Registered Designation of Origin,RDO)的传统Salers奶酪中的酵母菌群落结构进行分析,通过与传统培养方法的对比表明SSCP技术分析得到的菌群结果准确度高,即该技术能够快速分析奶酪在制作和成熟阶段中的酵母菌群落的演替过程。
Retureau等[18]将培养基平板计数与SSCP技术相结合确定奶酪发酵过程中微生物多样性,并以此为指标对Saint-Nectaire鲜乳奶酪的微生物安全性进行检测,检测结果表明命名为TR15的天然混合菌体与分离于Saint-Nectaire鲜乳奶酪表面的菌株在奶酪发酵过程中微生物多样性变化有所不同,并且TR15对李斯特菌的抗性较强,能够显著地抑制该菌在奶酪表面生长。平板培养结果和SSCP分析结果均表明由TR15混合菌株制得的奶酪中含有较高水平的乳杆菌和明串珠菌,同时SSCP分析结果还显示TR15奶酪中同时含有疏松肉杆菌(Carnobacterium mobile),阿氏节杆菌(Arthrobacter arilaitensis)等菌株,这些菌株也能够有效的对李斯特菌进行抑制,因此该研究表明SSCP技术可以更好地对抑制食品中致病菌株生长的微生物的交互作用进行研究,同时对于阐明奶酪中微生物相互作用关系具有重要意义。
4 随机扩增多态DNA技术
随机扩增多态性DNA(random amp lified polymorphism DNA,RAPD)标记技术是1990年由W illiams[19]和Welsh[20]同时发展起来的一种分类鉴定技术。RAPD的基本原理是根据不同的基因组中与随意引物匹配的碱基序列的位点和数目可能不同,用一组人为设计的核苷酸作为引物,通过PCR随机扩增得到该生物种特异性的DNA图谱。该技术可用于细菌种间、亚种间的亲缘关系分析,还可以用于未知菌株的快速测定等。当前研究结果表明RAPD技术的优点是简单便捷,在不了解基因组DNA的任何序列信息的情况下就可以进行鉴定,但缺点是重复性差,鉴定结果不稳定,很难在种以及亚种的水平上准确鉴定。
杨吉霞等[21]探讨了RAPD技术对不同地区牦牛奶酪乳酸菌分类鉴定中的应用,结果表明该技术能够简单快速的实现对发酵奶酪中的乳酸菌种间和种内的区分,并且该研究结果还表明鉴定得到的39株菌株的RAPD图谱的聚类表现出一定的地域性,即西藏与云南的地理位置相邻,菌株较多聚在一起,而新疆的地理位置较远,菌株很少与其余两地的菌株聚为一组,该结论很好地证明了RAPD技术可用于不同地域菌株间的遗传亲缘关系研究。Ruiz等[22]使用三种分子技术,即随机扩增多态性-聚合酶链反应(RAPDPCR)、肠杆菌基因间重复一致序列(enterobacterial repetitive intergenic consensus,ERIC-PCR)和聚三核苷酸(polytrinucleotide(GTG)5-PCR)等,对分离于山羊奶酪和Manchego奶酪中的乳酸菌和参考菌株进行分析,从而比较这些技术对细菌的鉴定能力。结果表明,RAPD-PCR方法得到的电泳结果中的条带数量最多,因而其能够对复杂微生物体系中同种属的菌株进行更好的归类,而ERIC-PCR和(GTG)5-PCR方法则对某些不同种属的菌株鉴定的结果显示没有差异性,因而RAPD-PCR方法具有较好的分辨能力,可用于对奶酪中不同种属的乳酸菌菌株种属分型鉴定。
5 扩增片段长度多态性分析技术
扩增片段长度多态性分析(amp lified fragment length polymorphism,AFLP)是在RFLP技术与PCR相结合的基础上衍生出来的一种新技术,其基本原理是先通过限制性内切酶切割基因组DNA产生不同大小的限制性片段,再使双链人工接头与酶切片段相连接,作为扩增反应的模板DNA,然后以人工接头的互补链为引物进行预扩增,最后在接头互补链的基础上添加1~3个选择性核苷酸作引物,对模板DNA基因再进行选择性扩增,最后通过聚丙烯酰胺凝胶电泳分离检测获得DNA扩增片段,并根据扩增片段长度的不同检测出多态性。
AFLP与其他DNA指纹技术有相似之处,也有其独特的优点[23]:可用于各种大小不同基因组的指纹分析;具有一定灵活性,可通过特异性PCR引物设计和内切酶组合的选择,来调整AFLP指纹图谱中限制性片段的适宜数目;采用严格的PCR条件和高分辨率的聚丙烯酰胺凝胶电泳,重复性好,分辨率高;操作简单,可作为连接遗传图谱与物理图谱间的桥梁,用于微生物基因组的分析研究。
AFLP技术建立初期用于植物育种的研究,后来发展成为可以分析任何来源DNA指纹图谱的一项通用技术,近些年来被广泛应用于奶酪相关发酵菌群的分类鉴定。嗜热链球菌是牛奶发酵过程中常用的发酵剂,Lazzi等[24]分别利用AFLP和RAPD对嗜热链球菌的遗传多样性进行比较分析,研究数据表明和RAPD方法相比,AFLP为嗜热链球菌的多样性研究提供了更为精密的检测结果,因而该技术对不同种的微生物识别效果更好。此外,近几年来AFLP技术在奶酪发酵过程中菌体代谢通量相关研究领域也取得较大进展。鼠李糖乳杆菌(Lactobacillus rhamnosus)是属于非发酵剂乳酸菌,其对大多数奶酪成熟过程中风味的形成有重要作用。Lazzi等[25]根据互补DNA的扩增片段长度多态性(cDNA-AFLP)和实时定量逆转录PCR(qPCR)方法对鼠李糖乳杆菌PR1019在CB(cheese-like medium,CB)培养基中生长代谢的相关分子机制进行进一步研究,并取得了较好结果。
6 分子杂交技术
核酸分子杂交技术(nucleic acid hybridization,NAH)是细菌分子生态学中利用特异探针来进行定性和定量分析微生物的一个重要工具,可以通过萃取样品中得到的DNA或RNA,或者是原位杂交来进行[26]。一般来说,核酸杂交技术都是根据已知序列来设计的特异的寡核苷酸探针,然后用荧光或放射性元素对该探针进行标记,并通过碱基互补的原则与目的基因进行杂交,最后利用检测得到的杂交信号对发酵微生物群落结构进行分析的方法。分子杂交法自身也存在一定的局限性,它一般对目的片断(如优势菌种)检测灵敏,但是由于其原理是根据已知基因设计探针,因而较难发现新的基因类型。
当前在发酵菌群多样性分析中应用较多的分子杂交技术是数量斑点印迹杂交和原位杂交,它们的特点是不需要对目的片断进行PCR扩增,因而可避免在PCR扩增中造成的误差。此方法简便快速,主要应用于快速检测。Ercolini等[27]对Stilton奶酪进行16S rRNA基因荧光原位杂交,并对其中的细菌进行观察检测,观察到了大量的菌落细胞均匀分布在奶酪基质中,因而该技术在研究食品中微生物群体空间分布,尤其是那些质地较脆弱不易进行检测操作的食品基质中具有良好的应用前景。Babot等[28]针对Gruyère奶酪中丙酸杆菌(propionibacteria)的16S rRNA基因来设计寡核苷酸探针,并优化荧光原位杂交对该类细菌数目进行检测,结果表明和平板计数法相比,该方法能够快速有效地检测奶酪样品中的丙酸杆菌含量,再次验证分子杂交技术可以对奶酪中的一些细菌达到快速检测的效果。
7 展望
奶酪中含有丰富的发酵菌群,其对奶酪的风味及流变学性质具有重要影响,因此奶酪发酵菌群的多样性研究是传统奶酪工业化生产的基础。相对于繁琐的传统培养方法而言,应用DNA指纹图谱技术分析,可以快速、准确、灵敏、简便地对发酵菌群进行分类、鉴定和分析,在近年来的研究中已经展现出了巨大的优越性,已成为研究奶酪以及其他食品中微生物群落结构的有效工具。不过每种方法都有其自身的优势与不足,虽然利用不同分子生物学技术可以获得更加真实的微生物群落多样性,弥补了传统培养方法的不足,但要得到奶酪发酵过程中的主要发酵菌群则仍需要通过传统分离培养的方法。因此,用分子技术指导传统培养的方向,用传统培养的结果对分子方法进行验证,二者互为补充,已经成为研究奶酪以及其他发酵食品中微生物多样性的新方向。
[1]ErcoliniD,MoschettiG,Blaiotta G,et al.Behavior of variable V3 region from 16S rDNA of lactic acid bacteria in denaturing gradient gel electrophoresis[J].Current Microbiology,2001,42(3):199-202.
[2]Ercolini D,Coppola S.Cheese|Use of Microbial DNA Fingerprinting[J].Encyclopedia of Dairy Sciences(Second Edition),2011:632-638.
[3]Liu W T,Marsh T L,Cheng H,et al.Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16S rRNA[J].Applied and Environment Microbiology,1997,63(11):4516-4522.
[4]Dariush S,Alireza D,Ainaz A,et al.Motal cheese of Iranian nomadic tribes as an untouched source of potentially probiotic Lactobacilli[J].African Journal of Microbiology Research,2013,7(22):2751-2756.
[5]Liu W J,Sun ZH,Zhang Y B,etal.A survey of the bacterial composition of kurut from Tibet using a culture-independent approach[J].Journal of Dairy Science,2012,95(3):1064-1072.
[6]Dijkshoorn L,Ursing BM,Ursing JB,et al.Strain,clone and species:comments on three basic concepts of bacteriology[J]. Journal of Medical Microbiology,2000,49(5):397-401.
[7]Sánchez J I,Rossetti L,Martínez B,et al.Application of reverse transcriptase PCR-based T-RFLP to perform semiquantitative analysis of metabolically active bacteria in dairy fermentations[J].Journal of Microbiological Methods,2006,65(2):268-277.
[8]Mancini A,Lazzi C,Bernini V,et al.Identification of dairy lactic acid bacteria by tRNAAla-23S rDNA-RFLP[J].Journal of Microbiological Methods,2012,91(3):380-390.
[9]Muyzer G.DGGE/TGGE amethod for identifying genes from natural ecosystems[J].Current Opinion in Microbiology,1999,2(3):317-322.
[10]Muvzer G,Smalls K.Application of denaturing gradient gel electrophoresis(DGGE) and temperature gradient gel electrophoresis(TGGE)in microbial ecology[J].Antonie van Leeuwenhoek,1998,73(1):127-141.
[11]徐玲玲,刘亚洁,李江,等.变形梯度凝胶电泳在微生物多样性分析中的应用及其技术发展[J].东华理工学院学报,2007,30(3):279-282.
[12]Dar S A,Kuenen JG,Muyzer G.Nested PCR-denaturing gradient gel electrophoresis approach to determine the diversity of sulfate-reducing bacteria in complex microbial communities[J].Applied and Environmental Microbiology,2005,71(5):2325-2330.
[13]Randazzo C L,TorrianiS,Akkermans A D L,etal.Diversity, dynamics and activity of bacterial communities during production ofan artisanal Sicillian cheese as evaluated by 16S rRNA analysis[J].Appl Environ Microbiol,2002,68:1882-1892.
[14]Pogacˇic' T,Kelava N,Zamberlin š,et al. Methods for cultureindependentidentification of lactic acid bacteria in dairyproducts[J]. Food Technology and Biotechnology. 2010,48(1):3-10.
[15]Dolci P,Zenato S,Pramotton R,et al.Cheese surface microbiota complexity:RT-PCR-DGGE,a tool for a detailed picture?[J].International Journal of Food Microbiology,2013,162(1):8-12.
[16]Orita M,Suzuki Y,Sekiya T,et al.A rapid and sensitive detection of point mutations and genetic polymorphisms using polymerase chain reaction[J].Genomics,1989,5(4):874-879.
[17]Callon C,Delbès C,DuthoitF,et al.Application of SSCPPCR fingerprinting to profile the yeast community in raw milk Salers cheeses[J].Systematic and Applied Microbiology,2006,29(2):172-180.
[18]RetureauÉ,Callon C,Didienne R,etal.Ismicrobial diversity an asset for inhibiting Listeria monocytogenes in rawmilk cheeses?[J].Dairy Science and Technology,2010,90(1):375-398.
[19]Williams J,Kubelik A,Livak K,et al.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Research,1990,18(22):6531-6535.
[20]Welsh J,Mcclelland M.Fingerprinting genomes using PCR with arbitrary primers[J].Nucleic Acids Research,1990,18(24):7213-7218.
[21]杨吉霞,陈芝兰,杨海燕,等.牦牛奶酪中乳酸菌的随机扩增多态性研究[J].食品科学,2013,34(7):206-211.
[22]Ruiz P,Seseña S,Palop M L.A comparative study of different PCR-based DNA fingerprinting techniques for typing of lactic acid bacteria[J].European Food Research and Technology,2014,239(1):87-98.
[23]雷正瑜.16S rDNA序列分析技术在微生物分类鉴定中的应用[J].湖北生态工程职业术学院学报,2006,4(1):4-7.
[24]Lazzi C,Bove C G,Sgarbi E,et al.Application of AFLP fingerprint analysis for studying the biodiversity of Streptococcus thermophilus[J].Journal of Microbiological Methods,2009,79(1):48-54.
[25]Lazzi C,Turroni S,Mancini A,et al.Transcriptomic clues to understand the growth of Lactobacillus rhamnosus in cheese[J]. BMCMicrobiology,2014,14:1-14.
[26]张春林.内蒙古传统发酵酸粥中微生物多样性分析[D].呼和浩特:内蒙古农业大学,2010:3-6.
[27]Ercolini D,Hill P J,Dodd C E R.Development of a fluorescence in situ hybridization method for cheese using a 16S rRNA probe[J].Journal ofMicrobiologicalMethods,2003,52(2):267-271.
[28]Babot J D,Hidalgo M,Argañaraz-Martínez E,et al. Fluorescence in situ hybridization for detection of classical propionibacteria with specific 16S rRNA-targeted probes and its application to enumeration in Gruyère cheese[J].International Journal of Food Microbiology,2011,145(1):221-228.
The application research of DNA fingerprinting technology in the bacterial fermentation microflora of cheese
WANG Juan1,ZHANG M ing-xia2,CAO Yan-ping1,YANG Zhen-nai3,WANG Bei1,2,*
(1.Beijing Engineering and Technology Research Centre of Food Additives,Beijing Technology&Business University(BTBU),Beijing 100048,China;2.Beijing Key Laboratory of Flavor Chemistry,Beijing Technology&Business University(BTBU),Beijing 100048,China;3.Beijing Laboratory for Food Quality and Safety,Beijing Technology Beijing Technology&Business University(BTBU),Beijing 100048,China)
In recent years DNA fingerprinting technique was more and more popular in molecular biologyresearching. The DNA fingerprinting of bacterial fermentation microflora could directly reflect their differenceson the DNA level. As a necessary part of the microbial fingerprinting,DNA fingerprinting of the modernmolecular biology techniques had played an important role on the microbiological studies,and as well as in thefield of identification,and genetic diversity researching of fermentation microflora. Usually DNA fingerprintingtechniques such as RFLP,T-RFLP,SSCP,RAPD,DGGE,AFLP and so on have been applied in most fermentationmicroflora studies. Based on introduction of the common techniques of DNA fingerprinting technique anddiscussion of the application of DNA fingerprinting on the fermentation micoflora of cheese,the paper hadcombined the current situation of DNA fingerprinting on cheese,and introduced the principle,advantages anddisadvantages,current researching situation of DNA fingerprinting techniques. And all of these discussions hadimportant the practical significance and theoretical value to study the fermentation microflora of our traditionalcheese.
DNA fingerprinting;traditional cheese;fermentation microflora;molecular biology
TS252.1
A
1002-0306(2015)08-0395-05
10.13386/j.issn1002-0306.2015.08.074
2014-12-05
王娟(1989-),女,硕士研究生,研究方向:乳制品风味。
*通讯作者:王蓓(1981-),女,博士,副教授,研究方向:乳制品呈味肽分析。
国家自然基金青年基金资助项目(31201392);国家“863”计划项目(2011AA100903);北京市教委科研计划资助项目(KM201310011004);北京市属高等学校创新团队建设与教师职业发展计划项目(IDHT20130506)。

