智利大麦染色体的荧光原位杂交分析
刘成等

摘要:为建立智利大麦染色体新的细胞遗传学标记,用多聚核苷酸探针对中国春-智利大麦双二倍体、1Hch和1HchS杂合附加系、4Hch、5Hch、6Hch和7Hch附加系进行原位杂交(FISH)分析。以Oligo-pTa535.1和Oligo-pSc119.2进行的双色原位杂交结果发现,前者能在1Hch~7Hch染色体上产生不同信号,而后者能在除3Hch外的6对染色体末端产生杂交信号;以(GAA)8进行的单色原位杂交在智利大麦1Hch~7Hch染色体上的杂交信号也各不相同。以上3个探针在智利大麦染色体上的杂交信号和小麦染色体信号不同,并且在染色体转移过程中未发现智利大麦多聚核苷酸序列明显删除现象。因此,可以用这3个探针进行原位杂交对小麦背景中的智利大麦染色体进行有效识别,并辅助利用智利大麦进行小麦染色体工程育种工作。
关键词:智利大麦;附加系;染色体;荧光;原位杂交;细胞遗传标记
中图分类号:S512.303.53文献标识号:A文章编号:1001-4942(2015)05-0010-05
Fluorescence In Situ Hybridization Analysis
of Hordeum chilense Chromosomes
Liu Cheng, Gong Wenying, Li Haosheng, Liu Aifeng, Song Jianmin, Song Guoqi, Li Genying, Liu Jianjun*
(Crop Research Institute, Shandong Academy of Agricultural Sciences/Key Laboratory of Wheat Biology and Genetic Improvement in the
North Yellow-Huai River Valley, Ministry of Agriculture/National Engineering Laboratory for Wheat and Maize, Jinan 250100,China)
AbstractIn order to develop new cytogenetic markers specific for Hordeum chilense chromosomes, the Chinese Spring-H. chilense amphipliod, heterozygous 1Hch﹠1HchS additon, 4Hch, 5Hch, 6Hch and 7Hch additions were analyzed by fluorescence in situ hybridization (FISH) using Oligo-pTa535.1, Oligo-pSc119.2 and (GAA)8 as probes. The results of double-color FISH using Oligo-pTa535.1 and Oligo-pSc119.2 as probes showed that the individual 1Hch~7Hch chromosomes were painted by different signals except 3Hch chromosomes with Oligo-pSc119.2 probe; the FISH using (GAA)8 as probe suggested that most of subtelomeric and centromere regions of 1Hch~7Hch chromosomes were labeled by hybridization signals. The FISH signals of H. chilense chromosomes using these three probes were different from those of wheat chromosomes, and no oligonucleotide sequence elimination occurred during chromosome transformation. Therefore, FISH with these three probes could be used to identify H. chilense chromosomes in wheat background and be assistant to wheat breeding by chromosome engineering using the agronomical useful genes of H. chilense.
Key words Hordeum chilense; Addition line; Chromosome;FISH; Cytogenetic marker
智利大麦(Hordeum chilense),2n=2x=14,基因组HchHch,是一个主要分布于智利和阿根廷的二倍体物种。西班牙、新西兰和英国科学家们对该物种的抗病性进行了综述,认为该物种高抗叶锈病、白粉病、全蚀病、颖枯病、腥黑穗病、麦二叉蚜、麦双尾蚜和禾谷孢囊线虫;此外,该物种对盐胁迫和干旱胁迫也具有一定的抗性,是小麦抗病抗逆育种的三级基因源。
智利大麦不仅易与大麦属物种杂交,与小麦属的山羊草、簇毛麦、偃麦草、黑麦和小麦等物种也很容易杂交成功。英国John Innes Centre的Miller教授课题组将智利大麦与小麦进行杂交后对其进行染色体加倍获得了可育的小麦-智利大麦双二倍体,进而,以该双二倍体为桥梁,将其与小麦进行杂交回交,获得了小麦-智利大麦染色体附加系与代换系,为定位相应优异农艺性状基因提供了材料。以这些材料为基础,研究者分别将小麦叶枯病抗性基因定位在4Hch染色体上,将腥黑穗病抗病基因和控制类胡萝卜素高含量的基因定位在7Hch染色体上,将抗盐基因定位在1Hch、4Hch和5Hch染色体上,将禾谷孢囊线虫抗性基因定位在1Hch染色体上,将细胞质雄性不育基因的育性恢复基因定位在6Hch染色体上。endprint
分子标记和细胞学标记从不同研究层面对智利大麦染色体上的优异基因向小麦转育提供有力指导。智利大麦1Hch~7Hch染色体分子标记已经建立起来,部分细胞学结果也已经发表。越多的分子和细胞标记被建立起来,就越容易对该物种进行染色体工程诱导改造并将其优异基因用于小麦育种进行后续指导。研究证明,利用多聚核苷酸Oligo-pTa535.1和Oligo-pSc119.2为探针可以对小麦42条染色体进行有效区分,(GAA)8可以对大部分小麦染色体进行鉴别,然而,是否可以利用前两个探针或结合(GAA)8同时鉴定小麦背景中的小麦和智利大麦染色体尚不可知。鉴于此,我们开展了如下试验。
1材料与方法
1.1试验材料
试验所用植物材料列于表1,由英国约翰英纳斯中心(John Innes Centre,JIC)的Reader 教授和美国堪萨斯州立大学小麦遗传与基因资源中心(Wheat Genetic and Genomic Resources Center,WGGRC)的Gill 教授提供。
1.2染色体制片及FISH分析方法
试验材料的根尖处理和染色体制片方法参见文献[16];以(GAA)8为探针进行的原位杂交所用探针标记方法、探针浓度、染色体制片洗脱步骤等参见文献[15];以Oligo-pTa535.1和 Oligo-pSc119.2为探针进行的原位杂交所用探针合成、探针浓度及具体操作步骤等参见文献[14]。原位杂交染色体制片自然干燥后,滴加0.25 μg/mL碘化丙啶并盖上盖玻片,在Olympus BX51荧光显微镜下进行照相。
2结果与分析
2.1中国春-智利大麦双二倍体的荧光原位杂交(FISH)分析
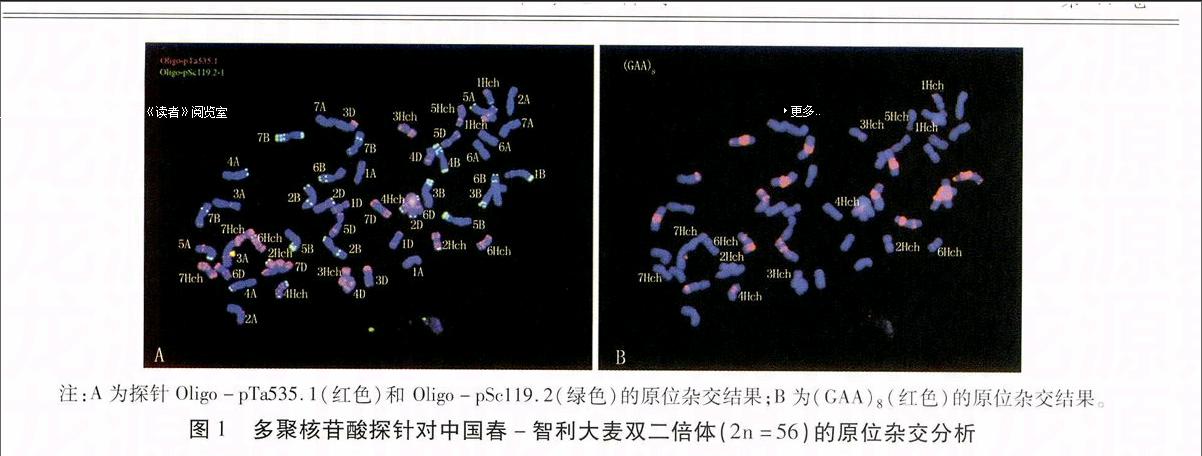
以Oligo-pTa535.1和 Oligo-pSc119.2为探针对中国春-智利大麦双二倍体进行FISH分析,发现该探针组合可以有效辨识42条小麦染色体(图1A),与文献[14]的报道吻合。而剩余14条智利大麦染色体虽有不同杂交信号,但是无法辨别其所属同源群。Martin和Cabrera发现探针(GAA)8可以用于辨别智利大麦染色体同源群,因此,本研究对上述两探针杂交过的染色体制片进行信号洗脱后,用(GAA)8对其进行二次杂交,结果显示,剩余14条染色体杂交信号(图1B)和文献[4]报道相同,可以对其进行染色体同源群认定。
染色体制片质量(主要指染色体是否被染色质覆盖)、探针量的多少及洗脱程度不同可能造成信号强度有所差异,因此,为了更好地描述Oligo-pTa535.1和 Oligo-pSc119.2在智利大麦染
注:A为探针Oligo-pTa535.1(红色)和 Oligo-pSc119.2(绿色)的原位杂交结果;B为(GAA)8(红色)的原位杂交结果。
图1多聚核苷酸探针对中国春-智利大麦双二倍体(2n=56)的原位杂交分析
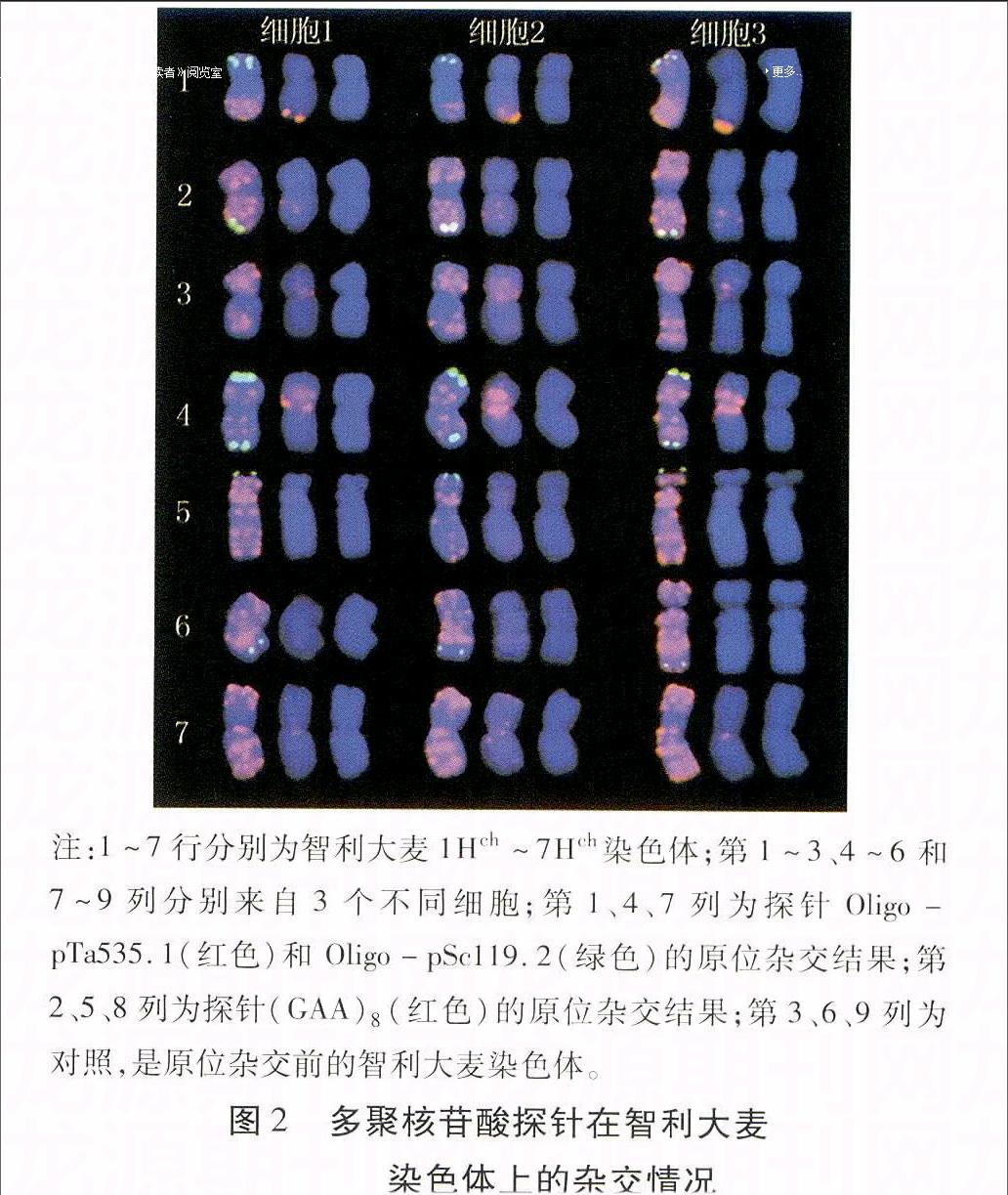
色体上的杂交情况,提取处理FISH过的智利大麦10个完整细胞,其中3个细胞中的智利大麦染色体FISH情况如图2所示。可见,5Hch和6Hch具有较大随体。Oligo-pSc119.2在1Hch、5Hch和7Hch染色体短臂末端具有杂交信号,其中,1Hch杂交信号较强,而7Hch信号较弱;在2Hch和6Hch染色体长臂末端具有杂交信号;在4Hch染色体长臂和短臂末端均具有强杂交信号;而在3Hch染色体上没有杂交信号;3个细胞间,仅7Hch杂交信号差异较明显,可能是由于信号在该染色体上较弱而被Oligo-pTa535.1的强烈红色信号覆盖的缘故。
Oligo-pTa535.1在1Hch~7Hch上均具有强烈的杂交信号,且杂交信号几乎布满整条染色体,但各染色体杂交模式各不相同。仅1Hch的杂交信号在3个细胞间有所差异,即第1和第2个细胞在染色体长臂末端及亚端部有强杂交信号,而在染色体短臂的末端有非常弱或未见杂交信号;第3个细胞在短臂末端也有杂交信号,且在长臂末端至着丝粒区域具有强杂交信号。
(GAA)8在智利大麦各染色体上的信号位置及强度各不相同,3个细胞中相同的同源群染色体杂交信号几乎完全相同,仅1Hch短臂末端弱信号有所差异(图2)。染色体1Hch长臂末端具有较强的杂交信号,而短臂末端有非常弱的信号或未见杂交信号;2Hch长臂和短臂的亚端部都具有杂交信号,长臂信号相对较强;3Hch的信号主要分布在着丝粒处、短臂亚端部和长臂近端部,并且长臂近端部信号较弱; 4Hch着丝粒区域的信号非常强,而短臂近端部信号相对较弱; 5Hch的信号分布在短臂末端和近末端(随体不包含在内);6Hch的杂交信号分布在长臂的亚端部;7Hch的信号分布在其着丝粒处、长臂和短臂近着丝粒处、短臂末端和近末端。
注:1~7行分别为智利大麦1Hch~7Hch染色体;第1~3、4~6和7~9列分别来自3个不同细胞;第1、4、7列为探针Oligo-pTa535.1(红色)和 Oligo-pSc119.2(绿色)的原位杂交结果;第2、5、8列为探针(GAA)8(红色)的原位杂交结果;第3、6、9列为对照,是原位杂交前的智利大麦染色体。
图2多聚核苷酸探针在智利大麦
染色体上的杂交情况
2.2中国春-智利大麦附加系的FISH分析
研究表明,在植物多倍体化或染色体转移过程中往往会出现序列的删除或插入现象。因此,本研究除对中国春-智利大麦双二倍体进行FISH分析外,还对中国春-智利大麦1Hch和1HchS杂合附加系及4Hch、5Hch、6Hch、7Hch附加系进行了FISH分析,结果发现,从双二倍体到附加系转移过程中,Oligo-pTa535.1和 Oligo-pSc119.2的杂交信号并未发现明显改变或丢失。中国春-智利大麦1Hch和1HchS杂合附加系的FISH图如图3所示。endprint
注:A为探针Oligo-pTa535.1(红色)和 Oligo-pSc119.2(绿色)的原位杂交结果;B为未原位杂交的染色体中期图。
图3多聚核苷酸探针对1Hch+1HchS附加系(2n=43+t,其中t=端体)的原位杂交分析
3结论与讨论
智利大麦的染色体C分带标准图已经发表,但尚没有其染色体C带序列组成的相关信息。Prieto等利用HvT01和(GAA)n对智利大麦染色体进行杂交,发现前者仅在染色体末端有杂交信号,而后者在大多数染色体着丝粒和少数染色体末端有杂交信号,与智利大麦标准C带位置明显不同,因此,这两类序列不可能是其染色体C带的组成部分。Cabrera等利用探针pAs1和18S-26S核糖体RNA对智利大麦进行分析,发现前者在所有Hch上具有杂交信号,然而,仅1Hch、6Hch和7Hch的杂交信号位置和染色体C分带信号位置部分类似,而2Hch~5Hch信号与C分带信号位置有所差异,因此,不是智利大麦染色体C分带的组成部分;后者的杂交信号仅分布在5Hch和6Hch染色体上,与智利大麦标准C带位置明显不同,因此,18S-26S核糖体RNA也不可能是其染色体C分带的组成部分。因而,我们对智利大麦染色体C带序列组成还知之甚少。但本研究发现探针Oligo-pTa535.1在智利大麦上的杂交信号位置与其染色体C分带位置几乎全部吻合,推测其是其染色体C分带的重要组成部分,该发现增加了对该物种染色体C带序列的认识。
智利大麦高抗小麦多种病害和胁迫,迄今为止,仅将少数几个抗病基因定位在其染色体上,未被定位的抗病基因仍需要进一步深入研究。因为大麦品质相对小麦差,因此,其基因组内优异基因在小麦育种上并未得到有效利用,然而,可以利用染色体工程将相应种质诱导成小片段抗病易位系进而应用于小麦抗病育种工作。本研究发现Oligo-pTa535.1和Oligo-pSc119.2、(GAA)8在智利大麦各染色体上具有不同的杂交信号,能够有效辨识智利大麦各条染色体,提供了一种检测小麦背景中智利大麦染色体的新方法,可以为后续追踪和检测小麦中的智利大麦染色体提供细胞学指导。
参考文献:
[1]Rubiales D, Niks R E, Ballesteros J, et al. Prospects for exploitation of disease resistance from Hordeum chilense in cultivated cereals [J]. Hereditas, 2001, 135(2/3):161-169.
[2]Foster B P, Philips M S, Miller T E, et al. Chromosome location of genes controlling tolerance to salt (NaCl) and vigour in Hordeum vulgare and H. chilense [J]. Heredity, 1990, 65: 99-107.
[3]Nevo E, Chen G X. Drought and salt tolerances in wild relatives for wheat and barley improvement [J]. Plant, Cell and Environment, 2010, 33:670-685.
[4]Martin A, Cabrera A. Cytogenetics of Hordeum chilense: current status and considerations with reference to breeding[J]. Cytogenet. Genome Res., 2005, 109(1-3):378-384.
[5]Miller T E, Reader S M, Chapman V. The addition of Hordeum chilense chromosomes to wheat[C]// International symposium of the section mutation and polyploidy of eucarpia: induced variability in plant breeding. Wageningen, the Netherlands, 1981: 79-81.
[6]Rubiales D, Martin A. Chromosome location in Hordeum chilense and expression of common bunt resistance in wheat addition lines [J]. Euphytica, 1999, 109: 157-159.
[7]Mattera M G, Avila C M, Atienza S G, et al. Cytological and molecular characterization of wheat-Hordeum chilense chromosome 7Hch introgression lines [J/OL]. Euphytica, 2015,DOI:10.1007/s10681-014-1292-0.
[8]Person-Dedryver F, Jahier J, Miller T E. Assessing the resistance to cereal root-knot nematode, Meloidogyne naasi in a wheat line with the added chromosome arm 1HchS of Hordeum chilense [J]. J. Genet. Breed., 1990, 44: 291-296.endprint
[9]Castillo A, Atienza S G, Martin A C. Fertility of CMS wheat is restored by two Rf loci located on a recombined acrocentric chromosome [J]. J. Exp. Bot., 2014, 65(22):6667-6677.
[10]Hagras A A, Kishii M, Tanaka H, et al. Genomic differentiation of Hordeum chilense from H. vulgare as revealed by repetitive and EST sequences [J]. Genes Genet. Syst., 2005, 80(3):147-159.
[11]Rodriguez-Suarez C, Gimenez M J, Gutierrez N, et al. Development of wild barley (Hordeum chilense)-derived DArT markers and their use into genetic and physical mapping [J]. Theor. Appl. Genet., 2012, 124(4):713-722.
[12]Cabrera A, Friebe B, Jiang J, et al. Characterization of Hordeum chilense chromosomes by C-banding and in situ hybridization using highly repeated DNA probes [J]. Genome, 1995, 38(3): 435-442.
[13]Prieto P, Martin A, Cabrera A. Chromosomal distribution of telomeric and telomeric-associated sequences in Hordeum chilense by in situ hybridization [J]. Hereditas, 2004, 141(2): 122-127.
[14]Tang Z X, Yang Z J, Fu S L. Oligonucleotides replacing the roles of repetitive sequences pAs1, pSc119.2, pTa-535, pTa71, CCS1, and pAWRC.1 for FISH analysis [J]. J. Appl. Genet., 2014, 55(3):313-318.
[15]Danilova T V, Friebe B, Gill B S. Single-copy gene fluorescence in situ hybridization and genome analysis: Acc-2 loci mark evolutionary chromosomal rearrangements in wheat [J]. Chromosoma, 2012, 121(6):597-611.
[16]任正隆,张怀琼. 一个改良的染色体C带技术 [J]. 四川农业大学学报,1995,13(1): 1-5.
[17]Oliver K R,McComb J A, Greene W K. Transposable elements: powerful contributors to angiosperm evolution and diversity [J]. Genome Biol. Evol., 2013,5(10):1886-1901.endprint

