洋葱细胞质雄性不育基因分子标记的开发
李永博+刘冰江+霍雨猛+缪军+杨建平+吴雄+杨妍妍


摘 要:
根据细香葱orfA501开发的特异引物,以洋葱细胞质雄性不育系为试材进行PCR扩增,对获得的片段进行回收测序,根据测序结果设计引物进行染色体步移,获得部分侧翼序列,开发了鉴定洋葱细胞质基因型的SCAR标记,命名为OC2175。该标记在洋葱不育系中扩增出一条2 175 bp的特异条带和1 053 bp的条带,而保持系中仅扩增出1 053 bp的条带。对17组不同遗传背景的不育系及其相应的保持系进行验证,该SCAR标记的鉴定结果与田间表型判断结果完全吻合。
关键词:洋葱;细胞质雄性不育;分子标记
中图分类号:S633.203.6 文献标识号:A 文章编号:1001-4942(2015)06-0001-04
Development of a Molecular Marker Identifying
Cytoplasmic Male Sterility Gene in Onion(Allium cepa L.)
Li Yongbo1,2,Liu Bingjiang2,Huo Yumeng2,Miao Jun2,Yang Jianping1,Wu Xiong2,Yang Yanyan2*
(1. College of Horticulture Science and Engineering of Shandong Agricultural University, Taian 271018, China;
2. Vegetable Research Institute of Shandong Academy of Agricultural Sciences,Jinan 250100,China)
Abstract The specific primers of orfA501 in chives and cytoplasmic male sterile line of onion were used to PCR amplification. The amplified fragment was extracted and sequenced. The primers were designed according to the obtained sequence for genome walking, and partial flanking sequence was obtained. Then a SCAR marker was developed and named as OC2175. With this marker, a special fragment of 2 175 bp could be obtained from the male sterile lines and a 1 053 bp fragment was amplified from both (s)-cytoplasm and (N)-cytoplasm. Seventeen pairs of male sterile lines and their corresponding maintainer lines with different genetic backgrounds were used to verify this SCAR marker. The results indicated that the SCAR marker was perfectly suitable for distinguishing the cytoplasm types S or N in onion.
Key words Onion; Cytoplasmic male sterility; Molecular marker
洋葱(Allium cepa L.),又称圆葱、葱头,百合科葱属,已有5 000多年的栽培历史。1936年美国的Jones和Emsweller 首次在“Italian Red”品种中发现雄性不育株[1],1943年Jones和Clarke又发现了雄性不育系[2]。随后洋葱成为世界上最早利用杂种优势的蔬菜作物之一。洋葱是两年生植物,其育种年限是白菜、萝卜等蔬菜的2~4倍,与传统的测交-回交、自交等方法相比,分子标记技术直接以DNA形式出现,在植物体的各个组织、各发育时期均可检测,不受季节、环境的限制,可大大缩短不育系和保持系的选育年限,减少工作量,加快育种进程。
国内外关于洋葱细胞质雄性不育的研究报道较少,Havey[3]利用RFLP分子标记手段找出了洋葱雄性保持系和不育系线粒体基因组之间的差异。Engelke等[4,5]得到了鉴定洋葱S型、N型和T型细胞质的分子标记。Sato[6]利用CMS特殊线粒体核苷酸序列的PCR扩增来鉴定洋葱细胞质基因型。李园园等[8]对洋葱细胞质雄性不育系及其保持系进行RAPD分析,成功获得了一个RAPD标记。Kim等[7]在S型和T型细胞质中发现了一个嵌合的开放阅读框orf725,并且开发了一个区分三种洋葱细胞质类型的分子标记。
由于细胞质中的DNA序列易产生缺失、插入等变异现象,再加上遗传背景、人工选择和自然选择的差异等,上述很多研究,存在可操作性差、重复性低、不稳定等缺点。本研究旨在结合TAIL-PCR[9]和SCAR标记方法开发一种鉴定洋葱细胞质育性的分子标记,以准确区分洋葱S型和N型细胞质,避免选择的盲目性,提高选择效率,加快洋葱细胞质雄性不育系及保持系的选育进程。
1 材料与方法
1.1 试验材料
试验材料为本课题组选育的17组洋葱细胞质雄性不育系及其相应的保持系,于2011年9月10日播种于山东省农业科学院蔬菜花卉研究所实验基地,11月初定植,并提取洋葱基因组总DNA,2013年6月田间检测花粉育性。endprint
1.2 DNA的提取及基因池的建立
洋葱基因组总DNA提取方法采用北京天根生化科技有限公司生产的快捷型植物基因组提取试剂盒,提取方法参照说明书。琼脂糖凝胶电泳及分光光度计检测DNA的浓度和质量。
应用分离群体分组分析法(Bulked Segregation Analysis)即BSA法,将不育系的10个单株和与之对应的保持系10个单株的DNA样品分别等量混合,组成洋葱细胞质雄性不育系及其保持系的基因池。
1.3 引物的设计与合成
根据Engelke等[5]报道的特异引物5′-ATGGCTCGCCTTGAAAGAGAGC-3′和5′-CCAAGCATTTGGCGCTGAC-3′对洋葱细胞质雄性不育系S118及其相应的保持系N218的DNA进行PCR扩增,对所得片段按照北京天根生化科技有限公司生产的琼脂糖凝胶DNA回收试剂盒使用说明进行回收纯化,纯化片段克隆至Promega公司的pGEM-T Easy 载体,转化大肠杆菌DH5α,挑取阳性克隆委托生工生物工程(上海)股份有限公司进行序列测定,根据测序结果,应用Premier 5.0设计TAIL-PCR引物进行染色体步移,获得了该片段的侧翼序列。根据获得的侧翼序列设计了一对PCR引物,OC2175-F:5′-ATGCCACTTCTCCTTCCTCATATGGT-3′;OC2175-R:5′-CCAAGGATTGCCAAGCATTTGGCACTGAC-3′。由北京博尚生物技术有限公司合成,PAGE纯化。
1.4 PCR扩增及检测
PCR扩增均在美国伯乐的TC-XP-D型基因扩增仪上进行,回收纯化片段的扩增参照Engelke和Tatlioglu的方法。所得标记OC2175采用如下体系: 10×PCR Buffer(with Mg2+)2.5 μL,dNTPs (each 2.5 mmol/L)2.0 μL,Primer1、2(0.5 μmol/L)各1.0 μL,Taq DNA polymerase 1 U,ddH2O补至25 μL。反应程序为:94℃预变性4 min;94℃变性30 s,56℃退火30 s,72℃延伸2.5 min,35个循环;72℃延伸10 min,4℃保存。PCR产物经1.0%琼脂糖凝胶电泳,溴化乙啶染色,凝胶成像系统自动成像。
1.5 单株验证
将所得标记在17组洋葱雄性不育系及相应的保持系组合中进行单株验证。从每个株系中随机选取10株提取基因组总DNA,进行PCR扩增与检测,检测结果与田间观察判断结果进行对比。
1.6 DNA序列分析
将获得的侧翼序列在NCBI数据库中进行BLAST分析。
2 结果与分析
2.1 基因组总DNA的检测分析
提取所得洋葱基因组总DNA溶液清亮,无褐化现象。0.8%琼脂糖凝胶电泳结果显示所提取的DNA在点样孔处无明显残留,表明多糖和蛋白质含量均较低; DNA带位于同一位置,清晰、无弥散、无降解现象(图1)。在紫外可见分光光度计上测定DNA样品的OD260/OD280值均在1.8~2.0之间,表明提取的洋葱基因组总DNA纯度较高,适用于PCR扩增、AFLP分子标记等生物学试验。
M:DNA Marker DL2000;S1~S10:不育系;N1~N10:保持系
图1 洋葱基因组总DNA的电泳图谱
2.2 洋葱细胞质雄性不育SCAR标记的获得及序列分析
根据Engelke等[5]报道的特异引物5′-ATGGCTCGCCTTGAAAGAGAGC-3′和5′-CCAAGCATTTGGCGCTGAC-3′对洋葱细胞质雄性不育系S118及其相应的保持系N218的DNA进行PCR扩增,结果仅在S118部分个体中扩增出长度为473 bp的片段,根据473 bp的核苷酸序列,应用TAIL-PCR方法进行染色体步移,获得了全长为2 175 bp的侧翼序列,在此基础上开发出鉴定洋葱细胞质基因型的SCAR标记,其扩增引物为OC2175-F和OC2175-R。 应用该标记对洋葱细胞质雄性不育系S118及其相应的保持系N218进行PCR扩增,结果在洋葱不育系中均扩增出一条2 175 bp的特异性条带和一条1 053 bp的条带,而相应的保持系N218只扩增出1 053 bp的条带(图2),将此标记命名为OC2175。
利用NCBI数据库对2 175 bp的序列进行BLAST搜索比对分析,发现该片段的5′端与洋葱的COXⅠ基因(GU138027.2)高度同源,同源性达到99%。
M:DNA Marker DL2000;S:S118的单株;N:N218的单株
图2 引物OC2175-F、OC2175-R对洋葱不育系及其保持系的PCR扩增结果
2.3 SCAR标记在洋葱不育系及保持系中的验证
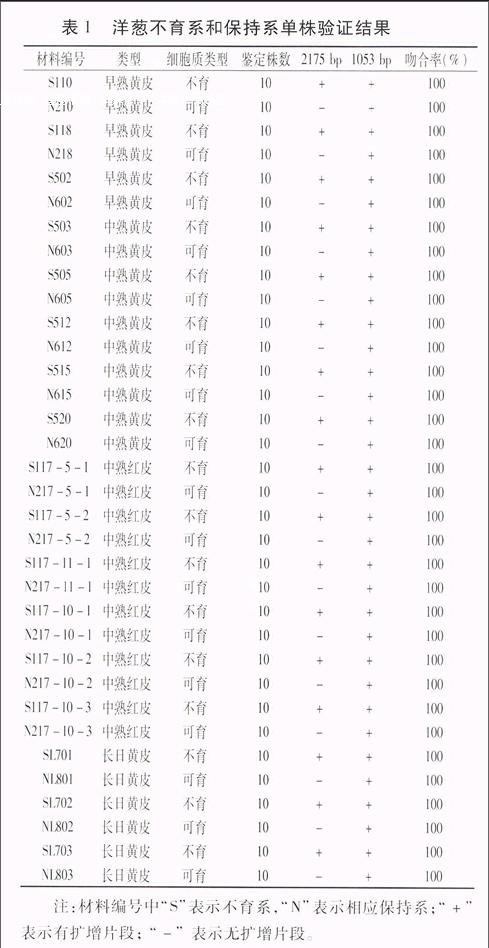
为了验证SCAR标记OC2175在分子标记辅助选择应用上的可行性,利用具有不同遗传背景的已知细胞质基因型的17个组合材料进行PCR验证(表1),结果表明,所有的不育系中均扩增出一条2 175 bp的特异性条带和一条1 053 bp的条带,而相应的保持系只扩增出1 053 bp的条带,PCR扩增结果与田间判断结果吻合。此结果表明应用该引物完全可以鉴别本课题组洋葱的细胞质基因型。
3 结论与讨论
洋葱是最早发现和利用细胞质雄性不育的蔬菜作物之一[2]。利用Engelke等[5]报道的特殊引物扩增时,有的株系中条带非常弱或无扩增,认为靶基因可能是以Sublimon的形式存在,因此无法应用到实际育种工作中。刘杰等[10]运用RAPD技术对洋葱细胞质雄性不育系和保持系的线粒体DNA进行了多态性研究,最终获得了两个多态性片段。但由于线粒体DNA的提取过程较复杂,费时费力,使得该标记在大规模田间材料的鉴定工endprint
表1 洋葱不育系和保持系单株验证结果
材料编号类型细胞质类型鉴定株数2175 bp1053 bp吻合率(%)
S110早熟黄皮不育10++100
N210早熟黄皮可育10-+100
S118早熟黄皮不育10++100
N218早熟黄皮可育10-+100
S502早熟黄皮不育10++100
N602早熟黄皮可育10-+100
S503中熟黄皮不育10++100
N603中熟黄皮可育10-+100
S505中熟黄皮不育10++100
N605中熟黄皮可育10-+100
S512中熟黄皮不育10++100
N612中熟黄皮可育10-+100
S515中熟黄皮不育10++100
N615中熟黄皮可育10-+100
S520中熟黄皮不育10++100
N620中熟黄皮可育10-+100
S117-5-1中熟红皮不育10++100
N217-5-1中熟红皮可育10-+100
S117-5-2中熟红皮不育10++100
N217-5-2中熟红皮可育10-+100
S117-11-1中熟红皮不育10++100
N217-11-1中熟红皮可育10-+100
S117-10-1中熟红皮不育10++100
N217-10-1中熟红皮可育10-+100
S117-10-2中熟红皮不育10++100
N217-10-2中熟红皮可育10-+100
S117-10-3中熟红皮不育10++100
N217-10-3中熟红皮可育10-+100
SL701长日黄皮不育10++100
NL801长日黄皮可育10-+100
SL702长日黄皮不育10++100
NL802长日黄皮可育10-+100
SL703长日黄皮不育10++100
NL803长日黄皮可育10-+100
注:材料编号中“S”表示不育系,“N”表示相应保持系;“+”表示有扩增片段; “-” 表示无扩增片段。
作中受到限制,且RAPD标记重复性和稳定性较差,其判断结果可信度不高。
本研究以遗传背景几乎完全相同的洋葱不育系与保持系为试材,利用TAIL-PCR进行染色体步移,获得侧翼序列,在此基础上设计引物OC2175-F/OC2175-R,开发了鉴定洋葱细胞质基因型的SCAR标记OC2175。对17个不同遗传背景的不育系和保持系组合进行验证,PCR检测结果和田间判断结果完全吻合,扩增条带清晰,无模糊条带。该结果表明,OC2175是洋葱不育细胞质的特异性标记,可应用于洋葱分子标记辅助育种。此外,该SCAR标记只需在苗期提取基因组总DNA,通过简单的PCR扩增即可鉴定洋葱细胞质基因型,不仅避免了常规育种方法的繁琐过程和盲目性,更有效避免了错误分类,减少了工作量和育种成本,加快了育种进程,为建立具有普遍意义的洋葱分子标记辅助育种技术体系奠定了基础。
参 考 文 献:
[1]
Jones H A, Emsweller S L. A male-sterile onion[J]. Proc. Am. Soc. Hort. Sci., 1936, 34: 582-585.
[2] Jones H A, Clarke A E. Inheritance of male sterility in the onion and the production of hybrid seed[J]. Proc. Am. Soc. Hort. Sci., 1943, 43: 189-194.
[3] Havey M J. Identification of cytoplasms using the polymerase chain reaction to aid in the extraction of maintainer lines from open-pollinated populations of onion[J]. Theor. Appl. Genet., 1995, 90: 263-268.
[4] Engelke T, Tatlioglu T A. PCR-marker for the CMS1-inducing cytoplasm in chives derived from recombination events affecting the mitochondrial gene atp9[J]. Theor. Appl. Genet., 2002, 104: 698-702.
[5] Engelke T, Terefe D, Tatlioglu T. A PCR-based marker system monitoring CMS-(S) , CMS-(T) and (N)-cytoplasm in the onion (Allium cepa L.) [J]. Theor. Appl. Genet., 2003, 107: 162-167.
[6] Sato Y. PCR amplification of CMS-specific mitochondrial nucleotide sequences to identify cytoplasmic genotypes of onion (Allium cepa L.) [J]. Theor. Appl. Genet., 1998, 96:367-370.
[7] Kim S, Lee E T, Cho D Y, et al. Identification of a novel chimeric gene, orf725, and its use in development of a molecular marker for distinguishing among three cytoplasm type in onion (Allium cepa L.) [J]. Theor. Appl. Genet., 2009, 118: 433-441.
[8] 李园园, 王忠,梁峰,等. 洋葱细胞质雄性不育系70及其保持系71基因组DNA的RAPD分析[J]. 生物技术通报, 2006, 6:100-102.
[9] 缪军, 霍雨猛,杨妍妍,等. 高效TAIL-PCR的改良及在洋葱中的应用[J]. 山东农业科学, 2009, 41(10): 1-4.
[10]刘杰, 崔成日,崔崇士,等. 洋葱细胞质雄性不育系与相应保持系线粒体DNA的RAPD 分析[J]. 东北农业大学学报, 2004, 35(3): 322-324.endprint

