大豆根瘤素蛋白家族的生物信息
刘小健,王巍杰,顾婷,郑强
(1.华北理工大学 生命科学学院,河北 唐山063000;2.山东大学 齐鲁软件学院,山东 济南250101)
大豆是食用油和植物蛋白的重要来源,在农业和经济领域越来越受到重视[1]。2008年12月8日在美国墨西哥州举行的国际豆科基因组与遗传会议上,美国能源部联合基因组研究所(DOE/JGI)公布了大豆基因组序列的初步科学分析结果:大豆基因组由10亿个碱基对组成,是人类基因组的三分之一,大豆基因组中约有66 000个基因。2010年1月14日的《Nature》杂志公布了由美国农业部、美国能源部联合基因组研究所和普渡大学等多家科研机构联合完成的豆科植物最重要的物种大豆的完整基因组序列草图[2]。
大豆基因组复制发生在距今5 900万年和1 300万年间[1],产生了一个复制率很高的基因组,其中近75%的基因以多版本存在,现代人为干预事件也使大豆基因组更加复杂[3]。拥有了大豆基因组序列,科学家们可以进行大豆后基因组进一步的研究,分析大豆相关蛋白的作用机制和功能,深入了解大豆的遗传和生理特性,对挖掘物种重要功能基因及加快分子育种奠定重要的科研基础。因此,大豆全基因组信息也会促进根瘤菌遗传特性的分析[4]。
自然界中,生物固氮约占自然固氮的90%,其中豆科植物与根瘤菌的共生固氮作用是重要的固氮方式。豆科植物与根瘤菌所形成的共生固氮体系必须要有豆科植物的根瘤素参与,根瘤素诱导根部形成的根瘤是固氮的前提条件。本研究就大豆的根瘤素蛋白家族23个蛋白进行生物信息分析。
1 材料与方法
1.1 大豆根瘤素蛋白家族数据来源
在NCBI(http://www.ncbi.nlm.nih.gov/)数据库中通过E值为1e-15的blast搜索,确定大豆根瘤素基因。23个大豆根瘤素蛋白数据来源于NCBI中的蛋白数据库(http://www.ncbi.nlm.nih.gov/protein/)。
1.2 大豆根瘤素基因分析
从GenaBank获得大豆根瘤素基因在大豆染色体上的位置信息和整个大豆基因组的长度及序列信息,利用MapInspect对大豆根瘤素基因进行染色体物理定位。
1.3 大豆根瘤素蛋白家族生物信息分析
对大豆根瘤素蛋白进行亚细胞定位分析,研究中使用了在线分析软件PSORT Prediction(http://psort.hgc.jp/form.html)。大豆根瘤素蛋白氨基酸基本理化性质、氨基酸数目、分子量、等电点、不稳定性指数和脂肪指数均采用在线分析工具 ProtParam (http://expasy.org./tools/protparam.html)[5]分析得到。二级结构的分析采用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)[6]分析预测。大豆根瘤素蛋白信号肽相关信息由 CBS(http://www.cbs.dtu.dk/index.shtml)[8]在线分析获得。
1.4 大豆根瘤素蛋白家族基因结构分析
从NCBI得到大豆根瘤素蛋白的基因序列和cDNA序列,利用Spidey(http://www.ncbi.nlm.nih.gov/IEB/Research/Ostell/Spidey/)分析内含子和外显子组成。
1.5 大豆根瘤素蛋白的保守序列查找
对大豆根瘤素蛋白序列的多重对比,使用了ClustalX2软件,参数均为默认值。
1.6 大豆根瘤素蛋白的系统进化分析
利用ClustalX2软件对大豆根瘤素蛋白序列的多重对比,将结果输出保存,参数为默认值。随后,继续使用MEGA6[7]选用最大似然法(Maximum Likelihood)构建系统进化树,并进行1 000次Bootsrat抽样。
2 实验结果与分析
2.1 大豆根瘤素基因分析
从GenBank中确定大豆根瘤素基因家族共有23个成员,从数据库中序列信息分析基因位置,进行了染色体物理定位。从染色体的物理定位看,23个大豆根瘤素基因在大豆染色体上分布不均匀,只在15条染色上有分布,1号染色体上分布2个,2号染色体上分布2个,5号染色体上分布1个,6号染色体上分布2个,7号染色体上分布2个,8号染色体上分布1个,10号染色体上分布3个,12染色体上分布1个,13号染色体上分布3个,14号染色体上分布1个,15号染色体上分布1个,16号染色体上分布1个,17号染色体上分布1个,18号染色体上分布1个,19号染色体上分布1个,结果见图1。
从表1统计数据可知,23个根瘤素蛋白的氨基酸数目差距较大,15个根瘤素蛋白序列长度都在100~400个氨基酸之间,小于100个氨基酸的蛋白1个,序列超过了400个氨基酸的蛋白7个,最大的CLV1B有987个氨基酸。根瘤素蛋白家族成员的蛋白分子量在10186.1~108908.3。等电点分析表明:14个根瘤素蛋白等电点小于6.5,为酸性蛋白;9个根瘤素蛋白等电点大于7.5,为碱性蛋白。脂融指数分析表明,共有18个蛋白的脂溶性指数小于100,另外5个蛋白的脂溶性指数大于100,说明大多数的根瘤素蛋白属于亲水性蛋白。不稳定指数分析表明:NP_001235855.1、NP_001241451.1、NP_001238376.1、ABD77418.1、NP_001237636.1、NP_001237618.1、NP_001238498.1、NP_001237453.1和 NP_001235885.1这9个蛋白的不稳定指数不小于40.00,为不稳定蛋白。
如表2所示,对大豆根瘤素蛋白家族的23个成员二级结构预测结果分析:根瘤素蛋白的二级结构有α-螺旋、β-折叠、转角、卷曲4个结构。在 NP_001235855.1、NP_001236691.1、NP_001238376.1、ABD77418.1、NP_001238498.1、NP_001237453.1、NP_001237695.1、NP_001235870.1、NP_001235885.1、XP_006572990.1、NP_001238004.1这11个蛋白中各组成成分的百分比卷曲>α-螺旋>β-折叠>转角;在NP_001241451.1、NP_001237618.1中各组成成分的百分比卷曲>β-折叠>α-螺旋>转角;在NP_001237525.1、NP_001237749.1、NP_001237653.1、NP_001237669.1、NP_001236825.1、AAA33993.1、XP_003535653.1、NP_001235599.1、NP_001237748.1共9个蛋白中各组成成分的百分比为α-螺旋>卷曲>β-折叠>转角;在NP_001237636.1中各组成成分的百分比为卷曲=α-螺旋>β-折叠>转角。
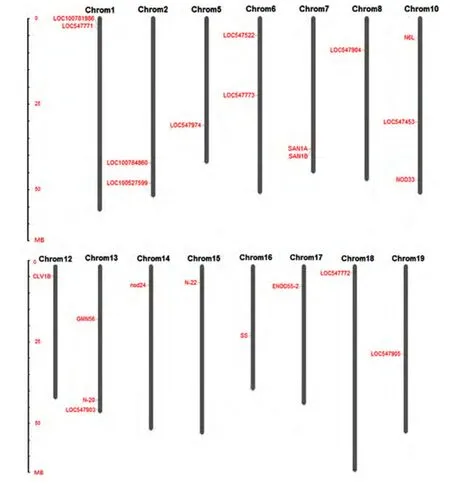
图1 大豆根瘤素基因的染色体物理定位
2.2 大豆根瘤素蛋白家族生物信息分析

表1 大豆根瘤素蛋白家族成员基本信息

表2 大豆根瘤素蛋白家族蛋白二级结构和亚细胞定位
用PSORT Prediction对大豆根瘤素蛋白家族的蛋白进行亚细胞定位,一部分蛋白属于分泌蛋白,绝大多数的蛋白都属于膜蛋白,其余的定位于细胞质细胞浆和细胞器。定位于质膜的8个蛋白:NP_001241451.1、NP_001237653.1、NP_001237669.1、NP_001236825.1、NP_001235870.1、NP_001235599.1、XP_006572990.1、NP_001238004.1。定位于胞外的7个蛋白:NP_001235855.1、NP_001237618.1、NP_001238498.1、NP_001237453.1、NP_001237695.1、AAA33993.1、NP_001235885.1。定位于微体5个蛋白:NP_001236691.1、NP_001237525.1、NP_001238376.1、ABD77418.1、NP_001237749.1。定位于细胞质细胞浆的2个蛋白:NP_001237636.1、XP_003535653.1。定位于内质网膜有1个蛋白:NP_001237748.1。

表3 大豆根瘤素蛋白家族蛋白信号肽预测
利用CBS分析软件分析大豆根瘤素蛋白家族蛋白,表3结果数据显示出:NP_001235855.1、NP_001241451.1、NP_001237618.1、NP_001238498.1、NP_001237453.1、NP_001237695.1、AAA33993.1、NP_001235885.1、NP_001235599.1、XP_006572990.1、NP_001238004.1这11个蛋白具有信号肽,其中包括亚细胞定位胞外的7个蛋白和定位到质膜上的4个蛋白。
2.3 大豆根瘤素蛋白家族基因结构分析
大豆根瘤素蛋白家族基因结构分析以及外显子的数量统计见表1。根据统计数据显示出基因上外显子数小于10个的有18个基因:NP_001235855.1、NP_001236691.1、NP_001236691.1、NP_001238376.1、ABD77418.1、NP_001237636.1、NP_001237669.1、NP_001237618.1、NP_001238498.1、NP_001237453.1、NP_001236825.1、NP_001237695.1、AAA33993.1、NP_001235870.1、NP_001235885.1、XP_003535653.1、NP_001237748.1、NP_001238004.1,其中LOC547771、LOC547974、N-22中都只有1个外显子;其余5个基因外显子的数目大于10个:NP_001237525.1、NP_001237749.1、NP_001237653.1、NP_001235599.1、XP_006572990.1,其中LOC100781986中外显子的数目最多,达到14个。所有的大豆根瘤素蛋白家族基因的结构组成如图2所示。

图2 大豆根瘤素基因外显子和内含子组成分析
2.4 大豆根瘤素蛋白的保守序列查找分析
使用MEGA6对ClustalX2软件对大豆根瘤素蛋白序列的多重对比结果选用最大似然法(Maximum Likelihood)构建系统进化树,进行系统的分析,由图2可知,存在6个相对保守的区域。Motif1在6个相对保守的区域中是最优的,Motif2、Motif3、Motif4、Motif5、Motif6这5个基序属于次级保守区。NP_001238498.1的 Motif2、Motif3、Motif4、Motif5、Motif6不完整。NP_001237636.1无 Motif3、Motif4、Motif5、Motif6。NP_001237669.1无 Motif5、Motif6基序,Motif4表现不完整。
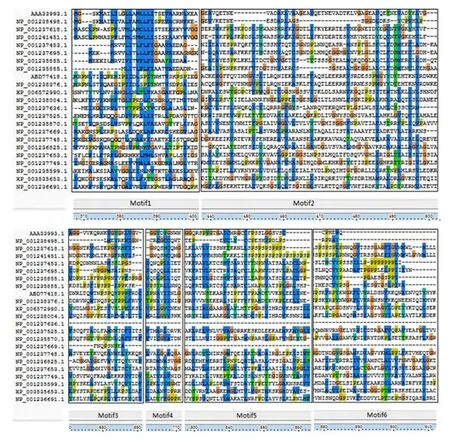
图3 大豆根瘤素家族蛋白序列的多重比较
2.5 原材料大豆根瘤素蛋白的系统进化分析
利用MEGA6选用最大似然法(Maximum Likelihood)构建系统进化树,以便分析大豆根瘤素蛋白的差异和系统进化关系。由图3可知,23个大豆根瘤素蛋白6个亚族,最大的一个亚族拥有5个成员:NP_001237749.1、NP_001237748.1、XP_006572990.1、NP_001238004.1、NP_001236691.1。最小的一个亚族拥有2个成员:NP_001237636.1、NP_001237525.1。
3 结论
在NCBI数据库中,确定了23个大豆根瘤素蛋白,并且获取对应的基因信息。基因在染色体上的物理定位结果显示23个大豆根瘤素基因在大豆染色体上分布并不均匀,并且每条染色体根瘤素基因所处位置也是变化无常的,基因表达与此相关,是导致各个基因间发生变化的因素之一,与ClustalX2比对分析结果吻合。另外,23个大豆根瘤素蛋白亚细胞定位,发现定位于胞外的蛋白有7个,这些蛋白可能参与诱导根瘤菌产生结瘤因子。
23个大豆根瘤素蛋白氨基酸数目有较大差距,大多数根瘤素蛋白序列长度都在100~400之间;蛋白分子量在10186.1(NP_001237636.1)~108908.3(CLV1B)之间变化。等电点分析结果表明:变化范围在5.0~10.13,14个根瘤素蛋白等电点小于6.5,表现出酸性,9个根瘤素蛋白等电点大于7.5,表现出碱性;脂融指数分析表明,共有18个蛋白的脂溶性指数小于100,另外5个蛋白的脂溶性指数大于100,说明大多数的根瘤素蛋白属于亲水性蛋白。不稳定指数分析表明:9个蛋白的不稳定指数不小于40,为不稳定蛋白。二级结构预测α-螺旋与卷曲是大豆根瘤素蛋白的主要构成原件;一部分蛋白被预测出来属于分泌蛋白,定位于胞外的概率最大的有7个蛋白,没有蛋白定位于细胞核中,绝大多数的蛋白都属于膜蛋白,定位于质膜的概率最大的有8个蛋白,定位于内质网膜的概率最大的有1个蛋白,其余的分贝定位于细胞质细胞浆和细胞器,定位于微体的概率最大的有5个蛋白,定位于细胞质细胞浆的概率最大的有2个蛋白;11个蛋白具有信号肽,其中包括亚细胞定位到胞外的7个蛋白,剩余的4个蛋白均是定位到质膜上的蛋白;大豆根瘤素蛋白基因上外显子数目在1~14变化。
到目前为止,大豆根瘤素蛋白的功能研究还不是很清楚,除少数根瘤素通过遗传学方法确定遗传学功能外,大多数根瘤素的功能仍是未知。本次研究对大豆根瘤素蛋白家族进行初步分析,为深入了解该家族蛋白的合成调控、结构和功能等提供了参考数据。加快了将大豆与根瘤菌这种共生固氮作用人为控制应用于实践的进程,采用基因工程技术育种,有针对性地进行固氮菌的遗传改造,构建高效的固氮菌株,以提高固氮效率,减少化肥施用,为作物提供更多的固氮。同时,研究其他禾本科植物是否具有大豆根瘤素的同源基因具有更加潜在的意义。

图4 大豆根瘤素蛋白系统进化树
[1] Gary Stacey,Lila Vodkin,Wayne A,et al.Parrott.National Science Foundation-Sponsored Workshop Report.Draft Plan for Soybean Genomics[J].Plant Physiology,2004,135(1):59-70.
[2] Jeremy Schmutz,Steven B.Cannon,Jessica Schlueter,et al.Genome sequence of the palaeopolyploid soybean[J].Nature,2010,463(7278):178-120.
[3] David L.Hyten,Qijian Song,Youlin Zhu,et al.Impacts of genetic bottlenecks on soybean genome diversity[J].PNAS,2006,103(45):16666-16671.
[4] Xiangyang Xua,Liang Zeng,Ye Tao,et al.Pinpointing genes underlying the quantitative trait loci for root-knot nematode resistance in palaeopolyploid soybean by whole genome resequencing[J].PNAS,2013,110(33):13469-13474.
[5] Wilkins MR,Gasteiger E,Bairoch A,et al.Protein Identification and Analysis Tools on the ExPASy Server[J].Methods Mol Biol.1999,112:571-607.
[6] Geourjon C,Deléage G.SOPMA:Significant improvement in protein secondary structure prediction by cprediction from alignments and joint prediction[J].CABIOS,1995,11(6):681-684.
[7] Koichiro Tamura,Glen Stecher,Daniel Peterson,et al.MEGA6:Molecular Evolutionary Genetics Analysis Version 6.0[J].Mol.Biol.2013,30(12):2725-2729.

