基因间长非编码RNA: T-UCRs转录活性的验证
王瑞雨,张 靖,朱丽媛,彭小忠,2*
(中国医学科学院 1.基础医学研究所 北京协和医学院 基础学院 医学分子生物学国家重点实验室;2.科学院神经科学中心, 北京 100005)
基因间长非编码RNA: T-UCRs转录活性的验证
王瑞雨1,张 靖1,朱丽媛1,彭小忠1,2*
(中国医学科学院 1.基础医学研究所 北京协和医学院 基础学院 医学分子生物学国家重点实验室;2.科学院神经科学中心, 北京 100005)
目的选择小鼠大脑皮层胚胎发育阶段转录的一类非编码RNA,即基因间区的T-UCRs作为研究对象,探究小鼠基因组上基因间超保守区域的转录活性。方法1)利用UCSC数据库(NCBI37/mm9)通过生物信息学预测筛选在小鼠14.5 d胎脑中可能表达的基因间UCR;2)Coding Potential Assessment Tool(CPAT)和Coding Potential Caculator(CPC)对筛选结果进行编码能力预测;3)RT-PCR对筛选结果进行验证;4)Real-time PCR分析T-uc.62在不同组织中的表达差异以及在小鼠不同发育阶段脑组织中的表达变化。结果1)通过UCSC网站上的RNA-seq数据库筛选得到16个可能在小鼠E14.5脑组织中表达的基因间UCRs;2)通过CPAT和CPC分析,16个UCRs均不具有蛋白编码能力,提示可能是非编码RNA;3)经过RT-PCR验证发现有15个是可以表达的;4)以T-uc.62为例,其主要在小鼠发育阶段的脑组织中高表达,而在小鼠的成年组织中低表达甚至不表达。结论基因间超保守区域可以转录生成长非编码RNA,其中,T-uc.62主要在小鼠发育阶段的脑组织中高表达;其可能在大脑发育过程中发挥重要功能。
UCR;小鼠大脑发育;T-uc.62
哺乳动物大脑发育一直是神经科学研究领域的重点和热点,其具有严格的时空特异性,这种时空特异性受到多种非编码RNA的调控[1- 2]。有一类长非编码RNA是由超保守区域(ultra-conserved regions,UCRs)转录生成的,称为超保守区域转录本(transcribed ultra-conserved regions, T-UCR)。目前关于T-UCR的研究仅限于肿瘤组织中[3- 4],在神经系统发育过程中的研究尚未见报道。UCRs多位于重要调控部位,参与转录调节和发育过程;基因间区的UCRs多位于转录和发育相关基因的附近[5]。因此,本研究以基因间区可能转录的UCRs为研究对象,通过生物信息学分析及RT-PCR等实验方法寻找小鼠大脑发育阶段可能表达的基因间T-UCR,并考察这类长非编码是否具有转录的时空特异性,以期为后续研究这一类非编码RNA的生物学功能提供基础数据。
1 材料与方法
1.1 材料
1.1.1 实验动物:SPF级ICR品系小鼠: 8~10周龄 (26~30 g),孕鼠与 E10.5、E12.5、E14.5、E16.5和E18.5的胎鼠以及P1、P3、P7、P14和P21的幼鼠 [北京维通利华公司,合格证号:SCXK (京) 2012-0001]于中国医学科学院基础医学研究所动物中心培养。
1.1.2 试剂: Trizol(Invitrogen公司)、RNase抑制剂、RNase-Free DNaseI和SYBR green Mix(TaKaRa公司)、反转录试剂盒(Transgen公司)、Taq DNA聚合酶(北京天根公司)。
1.2 方法
1.2.1 生物信息学分析: 利用Mestdagh等[6]2010年在Oncogene发表文献中的数据库分析整理出186个基因间UCR的序列信息,将186个序列信息逐一输入到UCSC Genome Browser on Mouse July 2007 (NCBI37/mm9) Assembly网站(http://genome.ucsc.edu/cgi-bin/hgBlat?command=start)上,经过BLAT和Browser后,获取在Whole Brain Embryonic day 14.5 RNA-seq Signal from ENCODE/LICR 通道中有表达信号的T-UCRs。
通过两种生物学软件Coding Potential Assessment Tool[7](CPAT, http://lilab.research.bcm.edu/cpat/)和Coding Potential Caculator[8](CPC, http://cpc.cbi.pku.edu.cn/programs/run_cpc.jsp)在线预测筛选到的16个可能在小鼠胚胎脑组织中表达的T-UCR的编码能力。
1.2.2 RNA的提取及real-time PCR: 取新鲜鼠脑组织,立即液氮中冻存,称取组织重量,按照20 mg/mL Trizol的比例进行消化,匀浆器匀浆后释放RNA,氯仿-异丙醇抽提,75% DEPC-乙醇漂洗后,空气干燥,加入适量DEPC处理过的水溶解。琼脂糖凝胶电泳检测RNA的质量后,用RNase Free DNase I对提取的RNA进行消化DNA处理,使用比例为RNA 10 mg/mL RNase Free DNase I,37 ℃水浴20 min,然后异丙醇沉淀,纯化RNA样品。普通PCR鉴定RNA中DNA除净后,用反转录试剂盒将2 μg的RNA使用随机引物反转录成20 μL体系的cDNA。使用小鼠E14.5全脑cDNA进行RT-PCR表达验证;各基因均使用1 μL的反转录产物为模板,使用引物及扩增长度(表1),在CFX- 96仪器(Bio-Rad公司)上进行扩增反应,条件为:95 ℃ 1 min、95 ℃ 30 s、57~58 ℃ 30 s, 循环45次,并作熔解曲线。使用Gapdh为内参,数据分析采用2-ΔΔCt法,重复样本(n=3)。
1.2.3 引物设计: 本实验中所用引物均是使用DNAMAN7.0进行设计后NCBI进行比对。
1.3 统计学分析
2 结果
2.1 UCSC RNA-seq数据库表达能力预测
筛选到16个有表达信号的UCRs,初步认为这些UCRs是可以被转录出来的(表2)。
2.2 编码能力预测
16个UCRs的转录产物均不具备编码能力(表3)。

表1 实验中所使用的引物序列及扩增长度Table 1 The sequences of RT-PCR and real-time PCR primers in this study
*F=forward, R=reverse, M=mouse, H=human

表2 小鼠胚胎期14.5天脑组织中表达的基因间T-UCRs
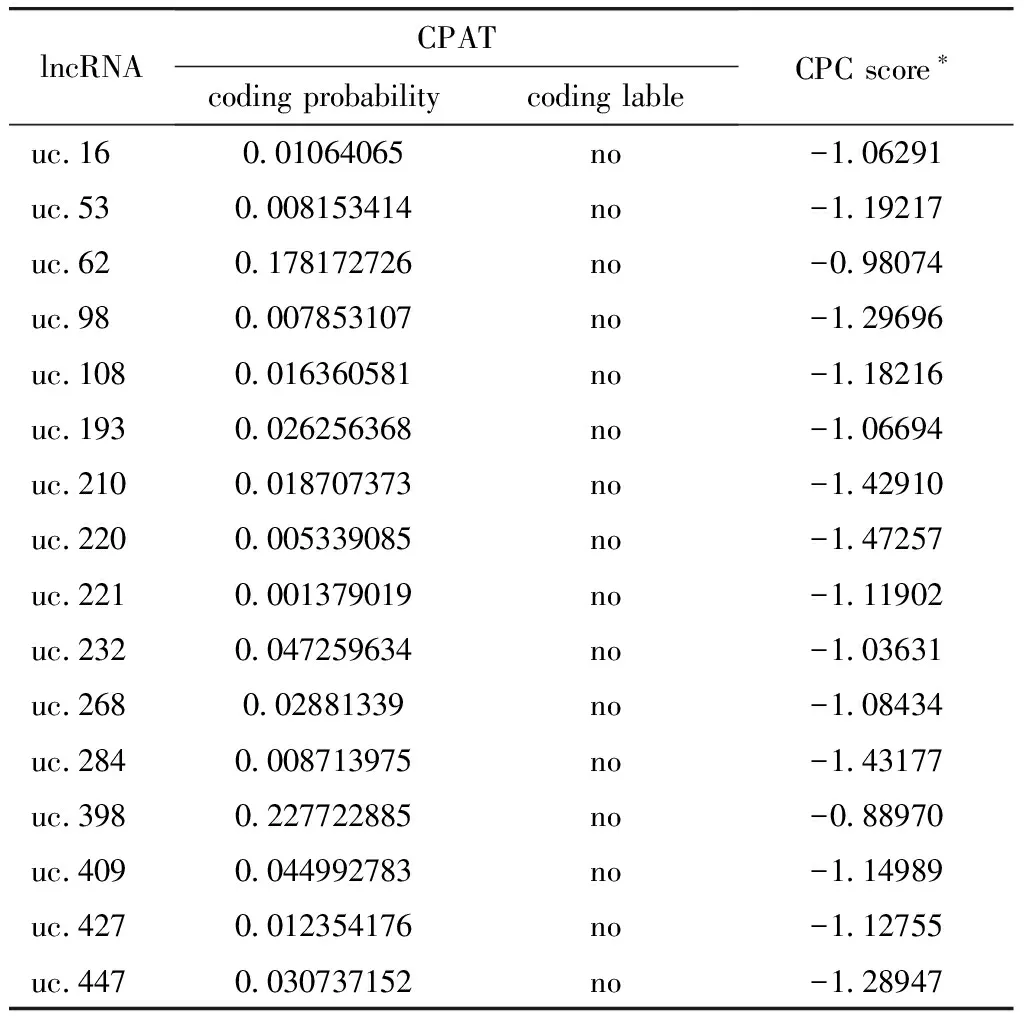
表3 16个基因间T-UCRs的编码能力预测
*CPC score:<0 noncoding RNA;0~1 weakly coding RNA;>1 protein coding RNA.
2.3 RT-PCR表达验证
上述16个备选UCRs有15个在小鼠14.5天胎脑组织中具有转录能力,uc.284除外(图1)。

图1 16个基因间UCRs在小鼠E14.5全脑的表达Fig 1 Expression of 16 intergenic UCRs in mouse E14.5 Brain
2.4 T-uc.62在小鼠不同组织中的表达情况
除脾脏外,T-uc.62在其他成年组织中的表达量相对于14.5天胎脑组织的表达量几乎为零(图2)。
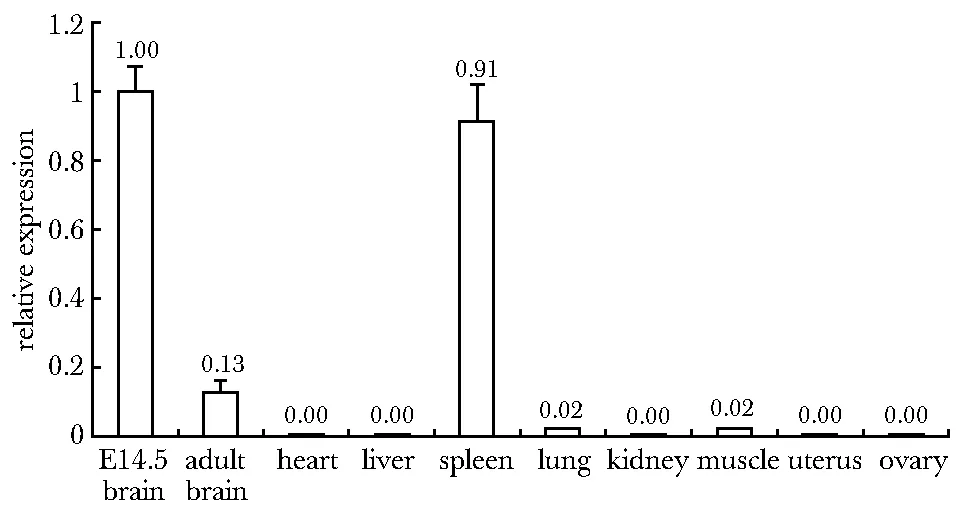
图2 T-uc.62在小鼠不同组织中的表达Fig 2 Expression of T-uc.62 in different mouse tissues
2.5T-uc.62在小鼠脑组织发育的不同阶段的表达
T-uc.62在胚胎时期及幼年早期的表达量比较高;随时间推移,其表达量逐渐降低,在成年时维持在一定的水平(图3)。

图3 T-uc.62在小鼠发育不同阶段脑组织中的表达Fig 3 Expression of T-uc.62 at different develop-mental stages of mouse brain
3 讨论
超保守区域是指在人、小鼠和大鼠的基因组上完全保守的区域,无任何碱基的缺失、插入或者突变。UCRs的长度范围为200~779 bp,共有481个[5]。根据UCRs与基因的位置关系,将其分为5类:包含外显子的(exon containing)、外显子内的(exonic)、包含部分外显子的(partly exonic)、基因间的(intergenic)和内含子区的(intronic)[6]。基因间的超保守区域在物种进化过程中被完整的保存下来,大多出现在调控转录和发育相关基因的附近,它们的存在或许不是偶然的,可能转录生成某些非编码RNA。本研究通过生物信息学预测以及实验验证发现,有15个基因间的UCRs在小鼠胚胎脑组织中可以转录生成长非编码RNA T-UCRs,说明基因间的超保守区域确实具有转录的能力。
有研究者对8个数据库中的RNA表达芯片进行了组织特异性分析,共涵盖了12 115个非编码RNA和6 856个编码基因[9],结果显示,编码基因多呈现广泛性表达,然而内含子长非编码RNA、反义长非编码RNA和基因间的长非编码RNA往往呈现组织特异性表达,尤其是基因间的长非编码RNA以脑组织特异性表达最显著。本研究中,通过对T-uc.62在小鼠不同成年组织、脑发育不同时间点中的表达谱分析显示,T-uc.62在脑组织中的表达量高于其他组织,且在小鼠胚胎期脑组织中的表达量高于成年组织,说明T-uc.62在小鼠胚胎脑组织中特异性表达,这一结果为T-uc.62在小鼠脑胚胎发育阶段可能发挥功能的探索提供了初步证据。
研究发现,T-UCRs在多种肿瘤组织中具有重要的调控作用。例如, T-uc.300+A对全反式维甲酸诱导的神经母细胞瘤SK-N-BE和SH-SY5Y的增殖和侵袭具有促进作用[10]。再比如,在结直肠癌细胞系HCT116 细胞中,uc.283+A(T-uc.283+A)在pri-miR- 195的Drosha剪切位点上可以与Drosha剪切酶竞争性结合pri-miR- 195,通过占位作用阻碍Drosha剪切酶对pri-miR- 195的剪切作用,从而使成熟miR- 195生成减少[11]。T-UCRs在肿瘤的发生发展中的作用正在逐步被揭示出来,说明T-UCRs的功能和机制研究已经起步,相信T-UCRs在生命体的发育过程中的功能与作用也将会被一一呈现。
[1] Martynoga B, Drechsel D, Guillemot F,etal. Molecular control of neurogenesis: a view from the mammalian cerebral cortex [J]. Cold Spring Harb Perspect Biol, 2012, 1- 14.
[2] Clark BS, Blackshaw S. Long non-coding RNA dependent transcriptional regulation in neuronal development and disease [J]. Front Genet, 2014, 5, 1- 19.
[3] Calin GA, Liu CG, Ferracin M,etal. Ultraconserved regions encoding ncRNAs are altered in human leukemias and carcinomas [J]. Cancer Cell, 2007, 12, 215- 229.
[4] Chiara B, Nicola V, Takayuki K,etal. Expression and functional role of a transcribed noncoding RNA with an ultraconserved element in hepatocellular carcinoma [J]. Proc Natl Acad Sci U S A, 2011, 108, 786- 791.
[5] Bejerano G, Pheasant M, Makunin I,etal. Ultraconserved elements in the human genome [J]. Science, 2004, 304, 1321- 1325.
[6] Mestdagh P, Fredlund E, Pattyn F,etal. An integrative genomics screen uncovers ncRNA T-ucR functions in neuroblastoma tumours [J]. Oncogene, 2010, 29: 3583- 3592.
[7] Wang L, Hyun JP, Surendra D,etal. CPAT: coding-potential assessment tool using an alignment-free logistic regression model [J]. Nucleic Acids Res, 2013, 41,74.
[8] Kong L, Zhang Y, Ye ZQ,etal. CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine [J]. Nucleic Acids Res, 2007, 35: 345- 349.
[9] Morten M, Disa T, Soren V,etal. Identification of expressed and conserved human noncoding RNAs [J]. RNA, 2014, 20: 236- 251.
[10] Karen M, Kenneth B, Niamh H,etal. Expressional alterations in functional ultra-conserved non-coding rnas in response to all-trans retinoic acid-induced differentiation in neuroblastoma cells [J]. BMC Cancer 2013, 13: 184- 197.
[11] Liz J, Portela A, Soler M,etal. Regulation of pri-miRNA processing by a long noncoding RNA transcribed from an ultraconserved region [J]. Mol Cell, 2014, 55: 1- 10.
新闻点击
活动与支架舒缓关节炎疼痛
据美国WebMD医学新闻网2013-11-06报道,研究指出,骨关节炎会痛,但运动能改善患者或高风险族群的生活质量。另一篇研究指出,膝关节炎患者可以用简单的支架舒缓疼痛。
骨关节炎或磨损性关节炎与老化有关,是最常见的关节炎,症状包括关节疼痛与僵硬。膝关节炎是渐进式的关节软骨受损,所谓关节软骨是指长骨尾端的缓冲物,这种伤害会导致肌肉萎缩、疼痛以及肿大。
芝加哥西北大学的研究人员Kai Sun医生表示,即使是少量运动也有帮助。运动越多,整体生活质量越好。
膝盖有穿戴支架的患者表示,6周后减少约40%的疼痛。研究人员建议,可以用软的尼奥普林支架(Neoprene-like)。另外,专家们建议,患者可做有氧运动以及重量训练,那些不曾运动的人可以从每周运动3次、每次15 min开始。
Validation of intergenic long non-coding RNA: T-UCRs for their transcriptional activities
WANG Rui-yu1, ZHANG Jing1, ZHU Li-yuan1, PENG Xiao-zhong1,2*
(1.State Key Laboratory of Medical Molecular Biology, Dept. of Molecular Biology and Biochemistry, Institute of Basic Medical Sciences, CAMS; 2.Neuroscience Center, CAMS, Beijing 100005,China)
ObjectiveExploration of the embryonic development related transcriptional activities of a specific class of lncRNA, T-UCR (transcribed ultra-conserved regions) in the brain.MethodsUCSC Genome Browser(NCBI37/mm9) RNA-seq, Coding Potential Assessment Tool (CPAT) and Coding Potential Calculator (CPC), RT-PCR and real-time PCR were employed to identify T-UCR, to predict coding potential, to display the spatio-temporal patterns in different mouse adult tissues and different mouse development stages of embryonic brain, respectively.ResultsThere are 15 intergenic T-UCRs expressed in mouse embryonic brains, which may function as long noncoding RNAs. T-uc.62 is prominently expressed in mouse embryonic brains, rather than other adult tissues.ConclusionsThe intergenic T-UCR can be transcribed to long noncoding RNAs, and T-uc.62 is expressed mainly
UCR; mouse brain development; T-uc.62
2015- 01- 23
:2015- 03- 13
国家自然科学基金(31370789)
*通信作者(correspondingauthor):pengxiaozhong@pumc.edu.cn
1001-6325(2015)05-0632-05
研究论文
Q522
:A
in mouse embryonic brains, which infers that T-uc.62 may be important in brain development.

