液相质谱联用的代谢组方法优化及其在蓝细菌分析中的应用
牛向凤,张晓清,于馨恒,苏营雪,陈磊,张卫文
液相质谱联用的代谢组方法优化及其在蓝细菌分析中的应用
牛向凤,张晓清,于馨恒,苏营雪,陈磊,张卫文
天津大学化工学院合成微生物学实验室,天津 300072

牛向凤, 张晓清, 于馨恒, 等. 液相质谱联用的代谢组方法优化及其在蓝细菌分析中的应用. 生物工程学报, 2015, 31(4): 577–590.Niu XF, Zhang XQ, Yu XH, et al. Optimization and application of targeted LC-MS metabolomic analyses in photosynthetic cyanobacteria. Chin J Biotech, 2015, 31(4): 577–590.
为了准确鉴定光合蓝细菌中的各种代谢物,需要对基于液相色谱–质谱联用仪 (LC-MS) 的代谢组学分析方法进行有针对性的优化。本研究选取了24种涉及中心碳代谢和能量代谢的代谢物作为LC-MS的检测目标,获得了每个代谢物的最适色谱分离条件和质谱参数;同时以光合蓝细菌sp. PCC6803为主要对象,针对性地优化了样品前处理条件,结果显示适当延长梯度洗脱顺序表的时间并将流速设为0.2 mL/min可以得到最佳的分离效果,同时选择80% () 甲醇 (−80 ˚C) 作为代谢物萃取剂。分析结果证明这一代谢组分析技术可以成功地应用到光合蓝细菌的研究中。
LC-MS,代谢物,代谢组学,蓝细菌
光合微生物能够利用太阳能固定大气中的CO2,这对于减缓和扭转全球变暖的趋势有着重要意义。同时因其高效的光合作用[1-3]、较强的环境适应能力[4]、简单的结构和遗传背景[5],光合微生物在生物工程中的应用近年来备受关注。首先,光合微生物自身能够产生多种化学品,例如油脂[6]、糖类[7]等;其次,利用基因工程技术改造后的光合微生物可以成为工业中生产重要化合物的非常有潜力的工程宿主菌。例如已有研究者将光合微生物投入到生物工程应用中,实现了在光合微生物中生物燃料和精细化学品的生产,如乙醇[8]、正丁醇[9]、异丁醇[10]、脂肪酸[6]、糖类[7]、氢[11]等。为了进一步提高对自然环境中光合微生物的了解,发掘光合微生物在生物工程领域的应用潜力,需要开发高效灵敏的分析技术对它们的代谢机制进行探究。
代谢组学作为一种检测细胞内小分子的多样性和丰度差异的分析方法,是对基因组学、转录组学和蛋白组学研究的重要补充[12-18]。质谱分析技术因其很高的灵敏度和广泛适用性已成为代谢组学中的主要分析技术,它和不同分离技术的耦合,如气相色谱-质谱 (GC-MS) 和液相色谱−质谱 (LC-MS),也已成为了研究细胞代谢组的理想工具[19]。其中,GC-MS方法尽管有较高的色谱分辨率、高重现性及灵敏性[20],但由于许多关键化合物因为不能被气化或离子化而无法被检测,有一定的使用局限性[12]。而与此同时,LC-MS方法因其高通量和软电离技术 (例如ESI和APCI),针对一些化学性质不稳定的代谢物,例如在中心碳代谢途径和能量代谢途径中有着重要作用的化合物 (NADP (H)) 和NAD (H) 或者一些易水解的核苷酸类化合物 (ATP、GTP、cAMP和PEP),能够得到更好的结果,可以弥补GC-MS的一些应用上的不足。通常基于GC-MS的代谢组分析都是以全扫描的方式获得生物代谢所反映的全部信息,而基于LC-MS的代谢组分析可以利用仪器的选择离子监测方式对一些特定的化合物进行定量分析,这种做法可以降低信噪比并能对代谢网络中重要的代谢物进行选择性检测,提高了检测的灵敏性[21-22]。
目前已有研究利用LC-MS的分析方法对蓝细菌中的模式生物如sp. PCC 7002中的特定代谢物进行分析[13,23]。然而由于很多目标代谢物在水溶液或细胞内易被水解,要获得准确反映生物信息的代谢物组,还需要优化LC-MS代谢组分析方法中的各个步骤,如样品前处理、代谢物的萃取、色谱分离以及质谱参数等。其中,样品前处理和萃取剂是获取完整代谢物组信息的关键因素,有研究估计样品前处理中造成的误差占整个测量误差的60%−80%[24]。和常规的异养菌相比,光合蓝细菌在面对环境条件如光照强度的变化时会发生代谢的迅速改变[25],因此对其代谢组样品的制备要求更高。目前针对光合蓝细菌的完整LC-MS代谢组分析方法不多,对方法的系统优化也还未见于文献。
本研究以光合蓝细菌模式菌sp. PCC 6803为对象,对其代谢物组样品的制备和基于LC-MS的代谢组学检测方法进行了系统优化,证明了该方法能够运用于光合蓝细菌的代谢组研究中。
1 材料与方法
1.1 材料
1.1.1 菌株
本实验使用的菌株为sp. PCC 6803。
1.1.2 试剂
HPLC重蒸水,色谱级别甲醇,三丁胺、乙酸以及24种代谢物标品 (图1) 均购自Sigma公司。
1.1.3 主要仪器
三重四级杆液质联用质谱仪 (Agilent 6410,美国)、台式高速冷冻离心机 (Eppendorf 5430R,德国)、超低温冰箱 (ThermoForma 700 series,美国)、紫外可见分光光度计 (岛津UV−1750,日本)。
1.1.4 培养基
培养sp. PCC6803所用培养基为pH 7.5的BG−11培养基,成分包括:1.5 g/L NaNO3,0.04 g/L K3HPO4·3H2O,0.075 g/L MgSO4·7H2O,0.006 g/L柠檬酸 (Citric acid (C6H8O7)),0.006 g/L柠檬酸铁铵 (Ammonium ferric citrate ((NH4)3Fe(C6H5O7)2)),0.036 g/L CaCl2·2H2O,0.001 g/L EDTA,0.02 g/L NaCO3,2.86 mg/L H3BO3,1.81 mg/L MnCl2·4H2O,0.222 mg/L ZnSO4·7H2O,0.390 mg/L NaMoO4·5H2O,0.079 mg/L CuSO4·5H2O, 0.049 4 mg/L Co(NO3)2·6H2O,并用稀盐酸将pH调至7.5。
1.1.5 培养条件
sp. PCC 6803在装有50 mL BG−11液体培养基的250 mL三角瓶中培养。将摇瓶放置在光照摇床中,光照强度约为50 μmol photons/(m2·s),转速130 r/min,温度控制在 30 ˚C[26−28]。
1.2 方法
1.2.1 样品制备方法
取蓝细菌培养样品后在常温下离心 (8 000×) 8 min,弃上清后立即加入900 μL 80% () 甲醇/水溶液 (−80 ˚C) 并在液氮中反复冻融3次。将冻融后的混合液离心 (−4 ˚C, 15 000×) 5 min,收集上清液并在沉淀物里加入500 μL 80% () 甲醇/水溶液 (−80 ˚C),重复之前步骤,最后合并两次上清并存于−80 ˚C,以备LC-MS分析。

图1 化合物的质谱参数优化结果及其在代谢途径中的分布 (用粗体表示的为本研究中所选择分析的代谢物,代谢途径参照文献[13]和[25];1最强准分子离子的碎裂电压值;2最强准分子离子的碰撞池电压值)
1.2.2 LC-MS代谢物检测方法
色谱条件为:液相色谱仪器为Agilent 1260系列,色谱柱为SYnergi Hydro−RP (C18, 150 mm×2.0 mm I.D., 4 μm 80 Å particles, Phenomenex, Torrance, CA, USA);柱温箱温度为40 ˚C;洗脱方式为梯度洗脱,流动相A为10 mmol/L三丁胺溶液 (pH 4.95),流动相B为甲醇,洗脱时间表为0 min (0% B),8 min (35% B),18 min (35% B),24 min (90% B),28 min (90% B),30 min (50% B),31 min (0% B);流速为0.2 mL/min。
质谱条件:质谱仪为Agilent 6410 Triple Quadrupole Mass Analyser,离子源为ESI;进样量为10 μL;毛细管电压为4 000 V;载气流速和压力分别为10 L/min和50 psi,温度为300 ˚C。
1.3 数据分析及处理
实验中涉及优化数据以及样品分析结果都是在负离子模式下用多反应监测 (Multiple reaction monitoring,MRM) 扫描得到的。选取了24个涉及中心碳代谢和能量代谢的代谢物 (图1[13,25]) 进行分析和方法优化,其标准品混合液的浓度为50 μmol/L。数据采集软件为Agilent Mass Hunter workstation LC/QQQ acquisition software (Version B.04.01),数据分析软件为Agilent Qualitative Analysis software (Version B.04.00)。每种代谢物的相对定量是由MRM模式下色谱峰的峰面积决定的,经过标准化处理后,将样品的代谢组数据输入软件SIMCA−P 11.5进行主成分 (PCA) 分析[29]。
2 结果与讨论
2.1 LC-MS检测代谢物方法的优化
2.1.1 色谱条件优化结果
基于LC-MS的代谢组学分析方法,限制步骤在于色谱分离而非质谱检测。全扫描的分析方式,即想要获得生物代谢的全部信息时,采用较长的分离时间确实能够检测到更多的化合物,有时分离时间可以长达1 h[12],但对于检测特定化合物的代谢组分析,在相对较短的时间内获得需要分离的代谢物信息即可。本实验以24种代谢物的化学标准品为分析对象,从流速和梯度洗脱的洗脱顺序表两方面进行了优化。其中24种重要代谢物及其在代谢途径中的分布见图1。
1) 洗脱顺序表的优化: 本研究选取24种标准化学品中的15种标准化学品为分析对象,在文献报道的总时间为18 min的顺序表[13]基础上,同时在不改变流动相比例的前提下,分别尝试了总时长为26、31、36以及51 min的洗脱时间。实验结果发现当洗脱时间为18和26 min时,分离效果不好;当洗脱时间延长到31 min以上时,分离效果明显改善;但介于31、36和51 min,分离效果差别不大。下图显示了3种总时间为18 min、31 min和51 min的不同洗脱顺序表 (分别命名为顺序表TT1、TT2和TT3) 下得到的EIC图 (选择离子流图,Elect Ion Current) (流速为0.3 mL/min)。图2A−C所示结果显示 15 min后的一些化合物在分离上呈现出不同的结果,其中TT1中15.8 min处的一个峰在TT2中分裂成两个峰,其中包含了4种化合物:ADP、NADH、NADP和PEP。而且TT1中16.5 min左右紧密相邻的两个峰包含3种化合物:ATP、NADPH和AcCOA,在TT2的EIC图中在24 min到26 min期间出峰。而在TT3中,51 min的长时间下分离效果没有比洗脱时间为31 min时的分离效果有所提高,因此我们选择了分离效果相当且时间相对较短的方法,即认为31 min为最佳的洗脱时间。
2) 流速的优化: 在以上洗脱顺序表的基础上,以24种标准化学品为研究对象,本实验比较了3个流速下的EIC图,流速分别为 0.15 mL/min、0.20 mL/min和0.3 mL/min (图3A−C)。当流速为0.15 mL/min时,化合物之间可以互相分离,但除了GLU外,其他化合物均在13 min后才出峰,并且27 min以后的7个化合物紧密相连,这就需要再延长洗脱顺序表以得到更好的分离效果 (图3A)。而提高流速到0.30 mL/min时,在总时间为31 min的洗脱中各化合物均能被检测到,但3PG被包含在15.5 min的峰中,NADH和PEP这两个化合物也紧密相连 (图3C)。在0.20 mL/min的流速下,3PG、FBP均以单峰的形式分离出来,而且NADH和PEP也分离开,NADP也以一个肩缝的形式被分离出来,而且总时间为31 min的洗脱顺序表也适用 (图3B)。因此0.2 mL/min成为了最适分离流速。

图2 三种梯度洗脱时间顺序表的比较 (TT1、TT2、TT3分别为18 min、31 min、51 min的洗脱时间。MPB为流动相B (甲醇);各化合物缩写见Abbreviations;EIC:选择离子流图)

图3 不同流速间的比较 (不同流速下获得的EIC图 (选择离子流图):(A) 0.15 mL/min、(B) 0.20 mL/min、(C) 0.30 mL/min。具体流速和各化合物的标注见图中。各化合物的缩写见Abbreviations)
2.1.2 质谱条件优化结果
本研究采用MRM的扫描方式精确鉴定了24个与中心碳代谢及能量代谢紧密相关的化合物,建立了自己的数据库。对其质谱参数的优化是利用Agilent optimizer software,在各化合物的准分子离子[M−H]−质量数以及特征碎片离子 (Product ion) 质量数已知的情况下,设置Fragmentation voltage (FV) 值和Collision voltage (CV) 值的区间[13]和步长,由Agilent optimizer software软件即可自动得到最适的FV值和CV值。各个化合物的优化结果显示于 图1中。
2.2 样品前处理条件的优化
通过细胞淬灭可以迅速终止胞内的代谢活动,获得准确反映细胞瞬时生理状态的代谢物浓度信息。萃取剂可以破坏细胞壁,使胞内代谢物释放,同时溶解化合物。对于本实验所选的24种与中心碳代谢和能量代谢相关的化合物来说,使用已有报道甲醇水的萃取剂能够获得sp. PCC 6803的代谢物组。事实上,在sp. PCC 6803的早期代谢组学研究中,有人提出过使用100%冷甲醇作为萃取剂[30-31],也有人提出过使用80% () 甲醇/水溶液作为萃取剂[13,25]。此外,在针对不同生长时期的代谢组学分析的早期研究发现,直接使用不经过离心收集的微生物培养物和萃取剂 (100%甲醇) 按1:4的比例混合离心提取代谢物能够获得较好的分析结果,这样做的优势是能够减少在样品离心收集过程中可能造成的代谢物变化[32]。本研究中我们首先对比了不经过离心收集的sp. PCC 6803培养物与萃取 (100%甲醇) 按1∶4的比例直接混合后再离心收集 (M1),以及先离心收集菌体后再向菌体细胞中添加萃取剂 (M2) 的提取代谢物方法。结果发现用M1方法难以提取到ATP、ADP这些能量型的磷酸化合物,并且最终在样品中可检测到的每个代谢物的响应值均小于用M2方法提取到的代谢物的响应值,例如用M2方法提取到的样品中检测到AcCOA的响应值可以达到M1方法提取的两倍。因此,先将培养物离心后直接将萃取剂添加到收集的细胞中能够起到更好的萃取效果。
在此基础上,采用MRM的扫描方式,比较了80% () 甲醇/水溶液 (−80 ˚C) 作为萃取剂 (M3) 和100%甲醇 (−80 ˚C) 作为萃取剂 (M2) 提取24种代谢物的结果。用80% () 甲醇/水溶液作萃取剂提取到的代谢物的种类比100%甲醇作萃取剂提取的代谢物种类多了8种,且80% () 甲醇/水溶液作萃取剂提取到的代谢物含量要比100%甲醇作萃取剂提取的代谢物含量高,例如AcCOA、NADPH、NADP、UDP-glucose、ATP和GLU等,尤其是ATP和GLU这样的化合物在细胞内能够快速被分解,甚至在离心的过程中,其含量就能发生很大的变化[33]。随后又进行了两组对比实验,结果都证明用80% () 甲醇/水溶液作萃取剂提取到的代谢物的种类比100%甲醇作萃取剂提取的代谢物种类多,且提取到的代谢物含量更高。因此,80% () 甲醇/水溶液作为萃取剂能够取得更好的结果。样品前处理条件的优化结果见表1。
2.3 方法的可靠性和重复性检测
LC-MS仪器的稳定性是获得精确数据的前提。为了检验仪器的稳定性,本研究首先对标准品混合样 (各物质浓度均为50 μmol/L) 进行两次重复进样检测,取两次数据的平均值及标准差后计算变异系数 (Coefficient of variation) 列于表2。对各化合物的保留时间进行分析,结果显示优化后的方法可以精确区分结构类似的化合物如GAP (12.14 min) 和DHAP (14.26 min)、G6P (11.04 min) 和F6P (11.63 min)、ADP (21.10 min) 和ATP (27.36 min)。此外,除了ADP和RiBP的保留时间变异系数在1%左右外,其余所有化合物的保留时间变异系数均小于1% (表2)。同时对各组分的峰面积进行分析,结果显示大部分化合物的色谱峰峰面积变异系数均小于10% (表2)。这些结果表明仪器的重复性很好。
为了检验生物学样品的重现性,实验选取了一组sp. PCC6803的3个生物重复样进行分析,并且将每个生物重复样品进样两次 (两个技术重复样)。本研究以其中一组生物重复样为例,对检测到的代谢物数据从色谱峰的峰面积和保留时间两方面进行分析,可以看出各代谢物的色谱峰的峰面积的变异系数在7.08%到46.02%之间 (表3),而各代谢物的保留时间的变异系数除FBP外,均小于1%。这说明生物重复样间代谢物的色谱峰的峰面积变化较大。考虑到生物样之间的生理代谢差异,以及代谢物提取过程中复杂的样品前处理过程和代谢物本身的不稳定性,这种偏离应该是可以接受的。检测到的代谢物中,只有3种代谢物的色谱峰的峰面积的变异系数在40%左右,它们分别是AMP、F6P和AKG (表3)。
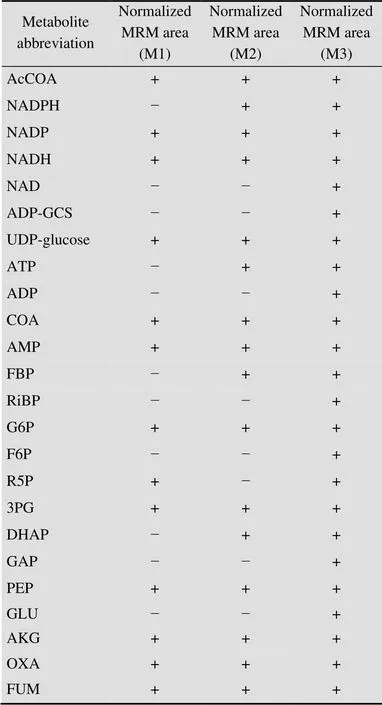
表1 样品前处理条件的优化结果
“+” represents detected metabolites; “−” represents nondetected metabolites.

表2 LC-MS代谢组分析的稳定性检验
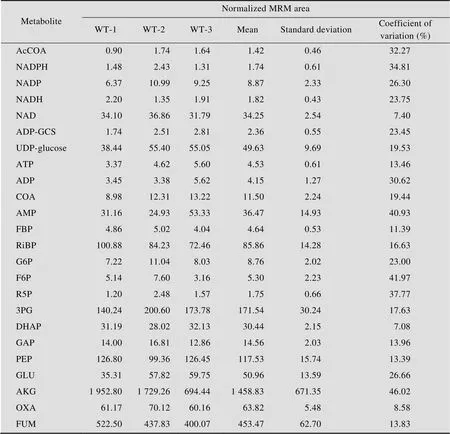
表3 生物样间的重复性检验
WT-1, WT-2 and WT-3 represent different biological replicates, respectively.
为了检验技术重复样的分析重现性,实验选取同一样品进行两次分析并对检测到的代谢物数据从色谱峰的峰面积和保留时间两方面进行比较,结果发现两个重复样WT-1-a和WT-1-b的线性拟合系数2分别为0.985 5和0.999 7 (图4A和4B),这说明技术重复样中各个化合物的色谱峰峰面积和保留时间相差较小,仪器的稳定性良好。
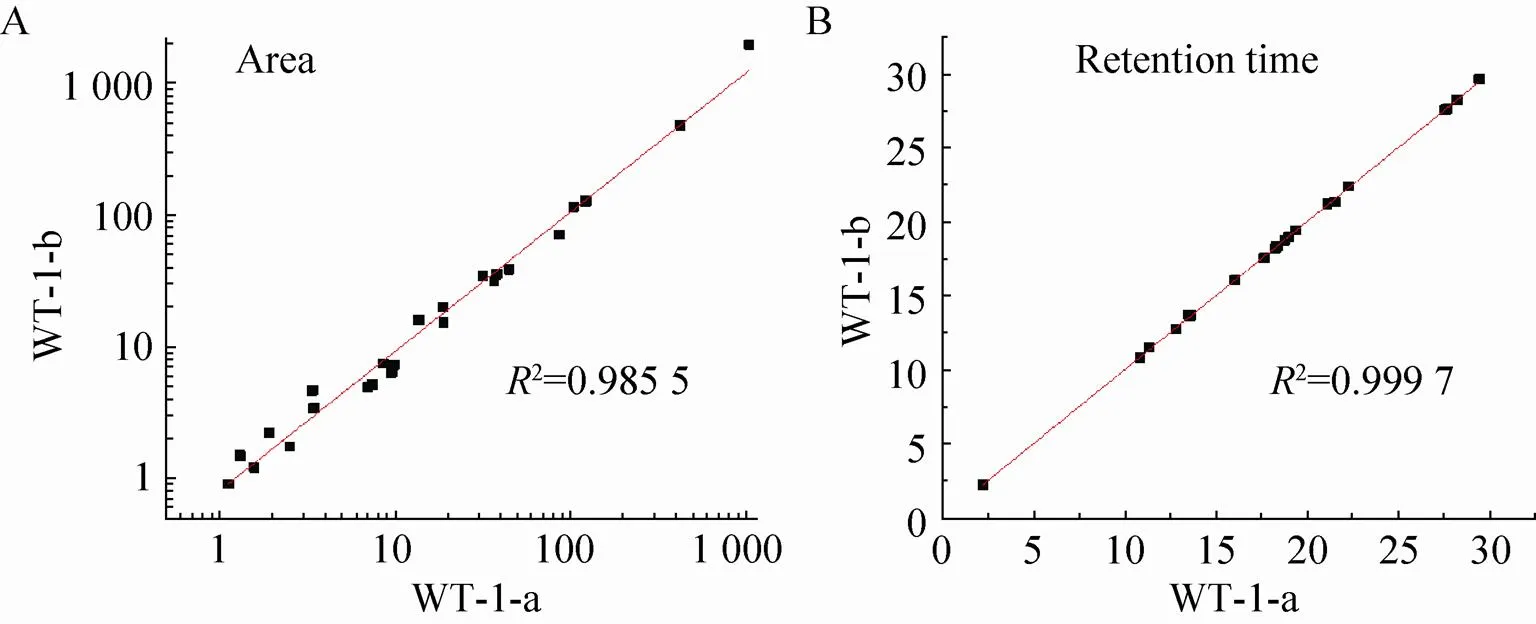
图4 代谢组分析的重复性检验 (图中每个点均代表一种代谢物,WT-1-a、WT-1-b为WT的不同技术重复样)
3 结论与展望
本文以模式光合蓝细菌sp. PCC 6803作为研究对象,建立并优化了基于LC-MS的代谢组学分析方法。该方法可以对涉及中心碳代谢和能量代谢的24种重要代谢物进行检测,包括氧化还原对 (NADPH和NADH) 或者一些易水解的核苷酸类化合物 (ATP、GTP、cAMP和PEP) 等。研究结果证明选择具有更好溶解性的萃取剂80% () 甲醇/水溶液,能够快速破解细胞,获取目标代谢物。另外,合适的流速和洗脱梯度时间表不仅能将化合物分离,还能保证整个分离时间不至于过长,节约时间和流动相,研究也优化了质谱参数。同时,对sp. PCC 6803生物重复样品进行了误差分析,结果证明了此代谢组学分析方法在光合蓝细菌中的合理应用性。在今后的研究中,我们希望将此分析方法结合其他组学分析例如蛋白组学,对蓝细菌及微藻等多种类型的光合微生物的生理机制进行研究。此外,这一基于LC-MS的代谢组学分析方法还可以和同位素标记示踪分析等技术结合[34-35],获得更为准确和全面的代谢组学分析,以实现对光合微生物生理代谢更为深入的研究。
Abbreviations
3PG:-(-)-3-Phosphoglyceric acid; AcCOA: Acetyl coenzyme A; ADP: Adenosine 5’- diphosphate; ADP-GCS: Adenosine-5’- diphosphoglucose; AKG: α-Ketoglutaric acid; AMP: Adenosine 5’-monophosphate; ATP: Adenosine 5’-triphosphate; COA: Coenzyme A hydrate; DHAP: Dihydroxyacetone phosphate; FBP:-Fructose 1,6-bisphosphate; F6P:-Fructose 6-phosphate; FUM: Sodium fumarate dibasic; G6P:-Glucose 6-phosphate; GAP:-Glyceraldehyde 3-phosphate; GLU:-Glutamic acid; NAD: α-Nicotinamide adenine dinucleotide; NADH: Reduced α-Nicotinamide adenine dinucleotide; NADP: Nicotinamide adenine dinucleotide phosphate; NADPH: Reduced nicotinamide adenine dinucleotide phosphate; OXA: Oxaloacetic acid; PEP: Phospho (enol) pyruvic acid; R5P:-Ribose 5-phosphate; RiBP:-Ribulose1,5-bisphosphate; UDP-GCS: Uridine 5’-diphosphoglucose.
REFERENCES
[1] Wang B, Wang J, Zhang W, et al. Application of synthetic biology in cyanobacteria and algae. Front Microbiol, 2012, 3: 344.
[2] Dismukes GC, Carrieri D, Bennette N, et al. Aquatic phototrophs: efficient alternatives to land-based crops for biofuels. Curr Opin Biotechnol, 2008, 19(3): 235−240.
[3] Gupta RS. Molecular signatures for the main phyla of photosynthetic bacteria and their subgroups. Photosynth Res, 2010, 104(2): 357−372.
[4] Ahsan M, Habib B, Parvin M, et al. A review on culture, production and use of spirulina as food for humans and feeds for domestic animals and fish (FAO Fisheries and Aquaculture Circular no. 1034). Rome: Food and Agriculture Organization (FAO) of the United Nations, 2008.
[5] Koksharova O, Wolk C. Genetic tools for cyanobacteria. Appl Microbiol Biotechnol, 2002, 58(2): 123−137.
[6] Liu X, Sheng J, Curtiss R. Fatty acid production in genetically modified cyanobacteria. Proc Natl Acad Sci USA, 2011, 108(17): 6899−6904.
[7] Xu Y, Guerra LT, Li Z, et al. Altered carbohydrate metabolism in glycogen synthase mutants ofsp. strain PCC 7002: cell factories for soluble sugars. Metab Eng, 2013, 16: 56−67.
[8] Gao Z, Zhao H, Li Z, et al. Photosynthetic production of ethanol from carbon dioxide in genetically engineered cyanobacteria. Energy Environ Sci, 2012, 5: 9857−9865.
[9] Lan EI, Liao JC. ATP drives direct photosynthetic production of 1-butanol in cyanobacteria. Proc Natl Acad Sci USA, 2012, 109(16): 6018−6023.
[10] Atsumi S, Higashide W, Liao JC. Direct photosynthetic recycling of carbon dioxide to isobutyraldehyde. Nat Biotechnol, 2009, 27(12): 1177−1180.
[11] Kumaraswamy GK, Guerra T, Qian X, et al. Reprogramming the glycolytic pathway for increased hydrogen production in cyanobacteria: metabolic engineering of NAD+-dependent GAPDH. Energy Environ Sci, 2013, 6: 3722−3731.
[12] Krall L, Huege J, Catchpole G, et al. Assessment of sampling strategies for gas chromatography-mass spectrometry (GC-MS) based metabolomics of cyanobacteria. J Chromatogr B, Anal Technol Biomed Life Sci, 2009, 877(27): 2952−2960.
[13] Bennette NB, Eng JF, Dismukes GC. An LC-MS-based chemical and analytical method for targeted metabolite quantification in the model cyanobacteriumsp. PCC 7002. Anal Chem, 2011, 83(10): 3808−3816.
[14] Dexter J, Fu P. Metabolic engineering of cyanobacteria for ethanol production. Energy Environ Sci, 2009, 2: 857−864.
[15] Park SJ, Lee SY, Cho J, et al. Global physiological understanding and metabolic engineering of microorganisms based on omics studies. Appl Microbiol Biotechnol, 2005, 68(5): 567−579.
[16] Wang QZ, Wu CY, Chen T, et al. Integrating metabolomics into a systems biology framework to exploit metabolic complexity: strategies and applications in microorganisms. Appl Microbiol Biotechnol, 2006, 70(2): 151−161.
[17] Trethewey RN. Metabolite profiling as an aid to metabolic engineering in plants. Curr Opin Plant Biol, 2004, 7(2): 196−201.
[18] Kell DB. Metabolomics and systems biology: making sense of the soup. Curr Opin Microbiol, 2004, 7(3): 296−307.
[19] Zhou B, Xiao JF, Tuli L, et al. LC-MS-based metabolomics. Mol Biosyst, 2012, 8(2): 470−481.
[20] Wang J, Chen L, Tian X, et al. Global metabolomic and network analysis ofresponses to exogenous biofuels. J Proteome Res, 2013, 12(11): 5302−5312.
[21] Fiehn O. Metabolomics-the link between genotypes and phenotypes. Plant Mol Biol, 2002, 48(1/2): 155−171.
[22] Issaq HJ, Van QN, Waybright TJ, et al. Analytical and statistical approaches to metabolomics research. J Sep Sci, 2009, 32(13): 2183−2199.
[23] Dempo Y, Ohta E, Nakayama Y, et al. Molar-based targeted metabolic profiling of cyanobacterial strains with potential for biological production. Metabolites, 2014, 4(2): 499−516.
[24] Yin P, Peter A, Franken H, et al. Preanalytical aspects and sample quality assessment in metabolomics studies of human blood. Clin Chem, 2013, 59(5): 833−845.
[25] Eisenhut M, Huege J, Schwarz D, et al. Metabolome phenotyping of inorganic carbon limitation in cells of the wild type and photorespiratory mutants of the cyanobacteriumsp. strain PCC 6803. Plant Physiol, 2008, 148(4): 2109−2120.
[26] Katoh A, Sonoda M, Katoh H, et al. Absence of light-induced proton extrusion in a cotA-less mutant ofsp. strain PCC6803. J Bacteriol, 1996, 178(18): 5452−5455.
[27] Stanier RY, Kunisawa R, Mandel M, et al. Purification and properties of unicellular blue-green algae (order Chroococcales). Bacteriol Rev, 1971, 35(2): 171−205.
[28] Kaneko T, Nakamura Y, Sasamoto S, et al. Structural analysis of four large plasmids harboring in a unicellular cyanobacterium,sp. PCC 6803. DNA Res, 2003, 10(5): 221−228.
[29] Roessner U, Luedemann A, Brust D, et al. Metabolic profiling allows comprehensive phenotyping of genetically or environmentally modified plant systems. Plant Cell, 2001, 13(1): 11−29.
[30] Young JD, Shastri AA, Stephanopoulos G, et al. Mapping photoautotrophic metabolism with isotopically nonstationary(13)C flux analysis. Metab Eng, 2011, 13(6): 656−665.
[31] Baran R, Bowen BP, Northen TR. Untargeted metabolic footprinting reveals a surprising breadth of metabolite uptake and release bysp. PCC 7002. Mol BioSyst, 2011, 7(12): 3200−3206.
[32] Park CH, Park CH, Lee YJ, et al. Determination of the intracellular concentrations of metabolites incollected during the exponential and stationary growth phases using liquid chromatography-mass spectrometry. Bull Korean Chem Soc, 2011, 32(2): 524−530.
[33] Lu WY, Bennett BD, Rabinowitz JD. Analytical strategies for LC-MS-based targeted metabolomics. J Chromatogr B Anal Technol Biomed Life Sci, 2008, 871(2): 236−242.
[34] You L, Berla B, He L, et al.13C-MFA delineates the photomixotrophic metabolism ofsp. PCC 6803 under light- and carbon-sufficient conditions. Biotechnol J, 2014, 9(5): 684−692.
[35] Wu Y, Li L. Development of isotope labeling liquid chromatography-mass spectrometry for metabolic profiling of bacterial cells and its application for bacterial differentiation. Anal Chem, 2013, 85(12): 5755−5763.
(本文责编 陈宏宇)
Optimization and application of targeted LC-MS metabolomic analyses in photosynthetic cyanobacteria
Xiangfeng Niu, Xiaoqing Zhang, Xinheng Yu, Yingxue Su, Lei Chen, and Weiwen Zhang
,,,300072,
To accurately analyze metabolites in industry-important photosynthetic microbes, LC-MS based metabolomics protocol needs to be optimized specifically for individual species. In this study, an LC-MS based metabolomics method was optimized for cyanobacteriumsp. PCC 6803. With the optimized extraction, liquid chromatographic and mass spectral parameters, the method was capable of detecting 24 important metabolites related to central carbohydrate and energy metabolism insp. PCC 6803. The study laid an important foundation for the metabolomics analysis of cyanobacteria.
LC-MS, metabolites, metabolomics, cyanobacterium
July 19, 2014; Accepted:December 1, 2014
Weiwen Zhang. Tel: +82-22-27406394; Fax: +86-22-27403389; E-mail: wwzhang8@tju.edu.cn
Supported by:National Basic Research Program of China (973 Program) (Nos. 2011CBA00803, 2012CB721101), National High Technology Research and Development Program of China (863 Program) (No. 2012AA02A707).
国家重点基础研究发展计划 (973计划) (Nos. 2011CBA00803, 2012CB721101),国家高技术研究发展计划 (863计划) (No. 2012AA02A707) 资助。
网络出版时间:2015-01-15
http://www.cnki.net/kcms/detail/11.1998.Q.20150115.0941.005.html

