北美鹅掌楸NAC基因的克隆与表达分析
杨 颖, 李火根
(南京林业大学南方现代林业协同创新中心, 江苏 南京 210037)
北美鹅掌楸NAC基因的克隆与表达分析
杨 颖, 李火根①
(南京林业大学南方现代林业协同创新中心, 江苏 南京 210037)
基于鹅掌楸〔Liriodendronchinense(Hemsl.) Sarg.〕转录组信息,对北美鹅掌楸(L.tulipiferaLinn.)NAC(NAM、ATAF1/2及CUC2)家族基因的结构和功能进行研究。采用RACE技术从北美鹅掌楸叶片中克隆出16个LtNAC基因的开放阅读框(ORF),编号为LtNAC01至LtNAC16。16个LtNAC基因的ORF长度为561~1 830 bp,编码187~610个氨基酸。根据理论等电点(pI),在LtNAC基因编码的16个LtNAC蛋白中,9个为碱性蛋白质,7个为酸性蛋白质。根据蛋白质疏水性(GRAVY),16个LtNAC蛋白中,13个为亲水蛋白质,3个为两性蛋白质。蛋白质二级结构预测结果显示:16个LtNAC蛋白均以无规则卷曲为主要构成元件,α螺旋、延伸链和β转角则散布于整个蛋白质中。系统进化分析结果表明:北美鹅掌楸16个LtNAC基因可能并不参与细胞壁的生物合成,其中,LtNAC02、LtNAC03和LtNAC04基因可能参与北美鹅掌楸的胁迫响应。LtNAC03、LtNAC07、LtNAC08、LtNAC09、LtNAC11、LtNAC12、LtNAC14、LtNAC15和LtNAC16 基因的相对表达量与北美鹅掌楸叶片衰老程度呈现相关性,推测该9个LtNAC基因可能参与调控叶片衰老。
北美鹅掌楸;LtNAC基因; 叶片衰老; qRT-PCR
植物NAC(NAM、ATAF1/2及CUC2)基因家族是植物特有的、最大的转录因子基因家族。该基因家族成员有一个共同的、保守的DNA结合结构域,即N端的NAC保守结构域;相反,其C端(通常包含转录激活结构域)的长度与序列组成高度变异。最早的研究发现NAC基因的功能与顶端分生组织(SAM)及子叶中的器官分离有关[1-2]。随后的研究结果表明:NAC基因家族对于植物器官边界的建成[3-5]、叶缘形态发育[3,6-8]、细胞次生壁合成[9-12]、叶片衰老[13-17]以及植物对生物和非生物胁迫的响应[18-19]等多个生物学阶段的基因表达具有重要的调控作用。
叶片衰老是叶片发育的重要阶段,其生物学意义是将营养物质从正在衰老的器官中转移到仍在生长的组织中,最终转移到发育中的种子中[20]。在营养生长阶段,叶片延长绿叶期意味着将会产生更多的营养物质,然而,在种子成熟阶段延迟衰老将导致农作物产量下降,这对营养物质的转移至关重要[21]。NAC基因家族是调控叶片衰老的一个重要的转录因子家族。在大麦(HordeumvulgareLinn.)[21]、柳枝稷(PanicumvirgatumLinn.)[22]和拟南芥〔Arabidopsisthaliana(Linn.) Heynh.〕[14]中均发现多个调控叶片衰老的NAC基因,它们对植物叶片衰老起到正向或负向的调控作用。
目前,关于植物NAC基因对林木叶片衰老的调控机制的研究还未见报道。北美鹅掌楸(LiriodendrontulipiferaLinn.)是原产北美东部的落叶大乔木,属于古老的被子植物,在植物进化研究中占据举足轻重的地位,因此,研究北美鹅掌楸NAC基因对叶片衰老的调控作用具有非常重要的意义。
本文以北美鹅掌楸为研究对象,通过挖掘北美鹅掌楸转录组信息,采用RACE技术分离克隆NAC家族基因,同时,利用qRT-PCR技术进行基因定量表达分析,以初步分析LtNAC基因的功能,以期为鹅掌楸属(LiriodendronLinn.)植物叶片衰老调控及候选基因筛选提供参考。
1 材料和方法
1.1 材料
供试材料均采自南京林业大学下蜀实习林场鹅掌楸种源试验林。于2014年5月采集成年北美鹅掌楸刚刚开始黄化的叶片(ES)作为基因克隆材料;为研究叶片不同衰老阶段的基因表达,同年9月,采集成年北美鹅掌楸相同冠层、相同方位不同生长阶段的叶片,包括嫩叶(LY)、完全展开未衰老叶片(LEN)、衰老早期叶片(LE)以及衰老后期叶片(LL)4种类型的叶片,其中嫩叶为完全张开且中脉长度5~6 cm的叶片;完全展开未衰老叶片为中脉长度9~10 cm的绿色叶片;衰老早期叶片为叶表黄化面积小于25%且中脉长度9~10 cm的叶片;衰老后期叶片为叶表黄化面积大于50%且中脉长度9~10 cm的叶片。每种类型叶片各取3枚,液氮速冻后,带回实验室并于-80 ℃保存。
1.2 基因克隆
1.2.1 引物设计 作者所在实验室前期已利用Illumina测序平台对鹅掌楸〔L.chinense(Hemsl.) Sarg.〕的花瓣和叶片进行转录组测序,通过拼接共获得87 841 unigene,并进行功能注释[23]。从Pfam的注释结果中搜索包含NAC结构域的序列。Liang等[24]利用454测序平台开发了北美鹅掌楸转录组数据库(http:∥ancangio.uga.edu/content/liriodendron-tulipifera),其unigene的序列平均长度为478 bp。为获得北美鹅掌楸NAC家族基因信息,将已经搜索到的鹅掌楸NAC结构域序列与北美鹅掌楸转录组数据库进行BLAST比对,获得目的基因片段序列信息,根据目的基因片段序列设计RACE引物。
1.2.2 RNA提取及克隆 采用RNA Prep Pure Tissue Kit试剂盒〔天根生化科技(北京)有限公司〕克隆全长目的基因。5′RACE反转录使用TaKaRa 5′-Full RACE Kit with ATP试剂盒〔宝生物工程(大连)有限公司〕,3′RACE反转录使用TaKaRa 3′-Full RACE Core Set Ver.2.0试剂盒〔宝生物工程(大连)有限公司〕。用Thermo Scientific Revert Aid First Strand cDNA Synthesis Kit〔赛默飞世尔科技(中国)有限公司〕合成第1链cDNA并用于全长开放阅读框(ORF)的扩增。具体操作步骤参照各试剂盒说明书。
1.3LtNAC基因的生物信息学分析
采用ExPASy提供的在线工具Protparam(http:∥web.expasy.org/protparam/)对预测的北美鹅掌楸NAC基因编码的蛋白质进行一级结构分析。利用在线工具SOPMA对北美鹅掌楸NAC基因编码的蛋白质进行二级结构分析(https:∥npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)[25],并预测其α螺旋、延伸链、β转角和无规则卷曲等在整体结构中所占的比例。采用在线分析软件TMHMM Server v. 2.0(http:∥www.cbs.dtu.dk/services/TMHMM/)进行蛋白质跨膜区预测。
从拟南芥蛋白质数据库(TAIR,http:∥www.arabidopsis.org/)中下载拟南芥所有105个NAC家族成员(ANAC001至ANAC105)的蛋白质序列。同时,从GenBank下载23个其他物种中已验证功能的NAC蛋白序列,包括水稻(OryzasativaLinn.)的OsNAP(NP_912423)、OsNAC4(BAA89798)、OsNAC5(BAA89799)、OsNAC6(BAA89800)和OsNAC9,大麦的NAM-B1(ACL31422.1),BambusaemeiensisL. C. Chia et H. L. Fung的BeNAC1(ADP69102.1),Ipomoeanil(Linn.) Roth的InNAP(BAO57486.1),MedicagotruncatulaGaertn.的MtNAM(AFI56799.1),Petunia×hybridaE. Vilm.的NAM(CAA63102),SolanumlycopersicumLinn.的GOBLET(HM210879.1)以及PopulustrichocarpaTorr. et A.Gray ex Hook.的PtrWND1A(NAC068,XM_002317023)、PtrWND1B(NAC063, XM_002300464)、 PtrWND2A (NAC065, XM_002320861)、PtrWND2B(NAC061, XM_002302636)、PtrWND3A(NAC050,XM_002322362)、PtrWND3B(NAC037,XM_002318252)、PtrWND4A(NAC038, XM_002329829)、 PtrWND4B (NAC046, XM_002304392)、PtrWND5A(NAC025,XM_002310261)、PtrWND5B(NAC039,XM_002327730)、PtrWND6A(NAC055,XM_002327206)和PtrWND6B(NAC060,XM_002325955)12个NAC蛋白质序列。将北美鹅掌楸LtNAC基因编码的蛋白质保守结构域序列和上述NAC蛋白质保守结构域序列一起进行系统进化分析。利用Clustal X(version 1.83)软件进行蛋白质序列的多序列比对。利用MEGA 6.0软件、采用neighbor-joining(NJ)法构建无根的系统进化树,bootstrap test值设为1 000。
1.4LtNAC基因的qRT-PCR分析
采用Primer 5.0软件设计qRT-PCR的LtNAC基因和内参基因的引物,引物序列见表1。
表1 北美鹅掌楸LtNAC基因和内参基因的引物序列
Table 1 Primer sequence ofLtNACgene and internal control gene fromLiriodendrontulipiferaLinn.
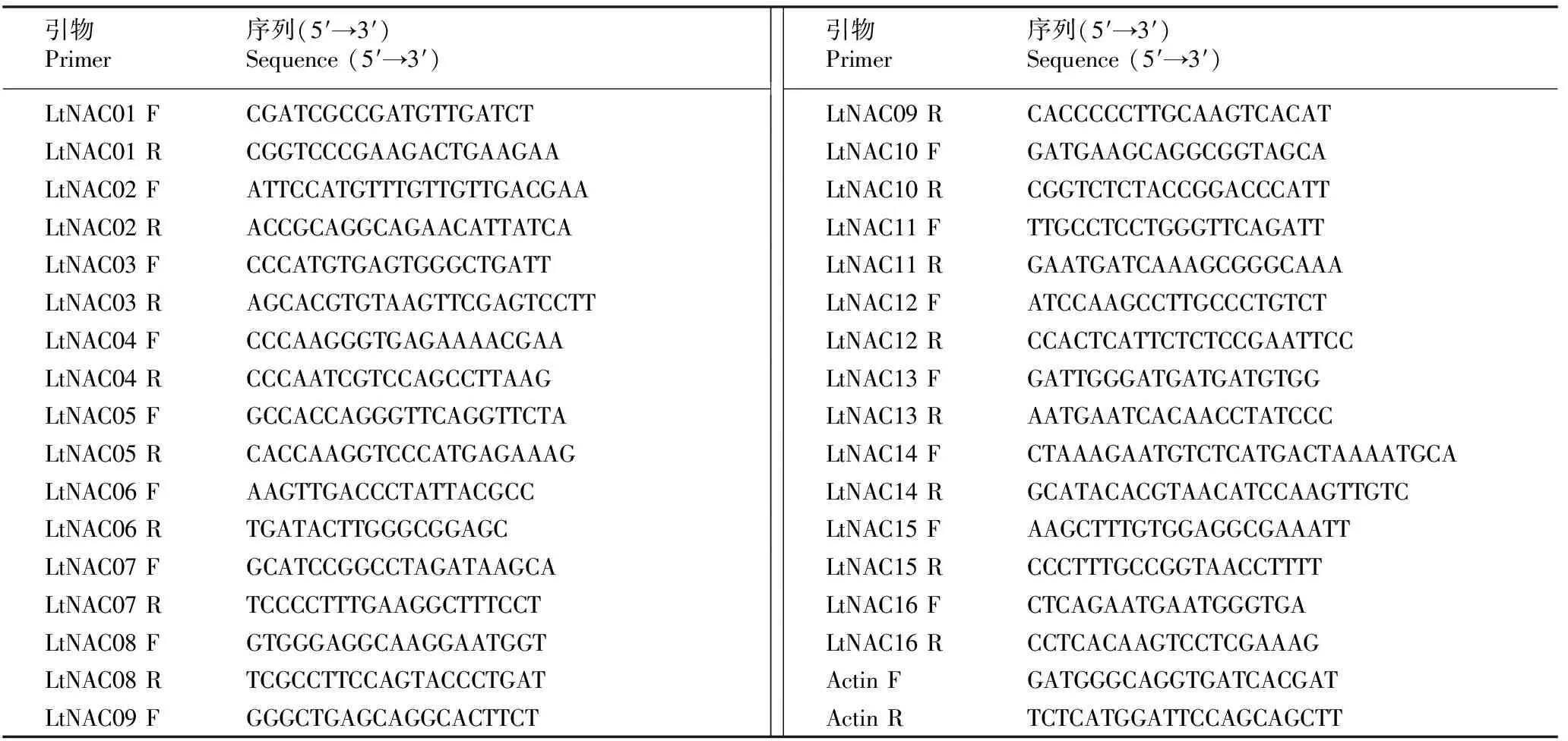
引物 Primer序列(5′→3′)Sequence(5′→3′) 引物 Primer序列(5′→3′)Sequence(5′→3′) LtNAC01FCGATCGCCGATGTTGATCT LtNAC09RCACCCCCTTGCAAGTCACAT LtNAC01RCGGTCCCGAAGACTGAAGAA LtNAC10FGATGAAGCAGGCGGTAGCA LtNAC02FATTCCATGTTTGTTGTTGACGAA LtNAC10RCGGTCTCTACCGGACCCATT LtNAC02RACCGCAGGCAGAACATTATCA LtNAC11FTTGCCTCCTGGGTTCAGATT LtNAC03FCCCATGTGAGTGGGCTGATT LtNAC11RGAATGATCAAAGCGGGCAAA LtNAC03RAGCACGTGTAAGTTCGAGTCCTT LtNAC12FATCCAAGCCTTGCCCTGTCT LtNAC04FCCCAAGGGTGAGAAAACGAA LtNAC12RCCACTCATTCTCTCCGAATTCC LtNAC04RCCCAATCGTCCAGCCTTAAG LtNAC13FGATTGGGATGATGATGTGG LtNAC05FGCCACCAGGGTTCAGGTTCTA LtNAC13RAATGAATCACAACCTATCCC LtNAC05RCACCAAGGTCCCATGAGAAAG LtNAC14FCTAAAGAATGTCTCATGACTAAAATGCA LtNAC06FAAGTTGACCCTATTACGCC LtNAC14RGCATACACGTAACATCCAAGTTGTC LtNAC06RTGATACTTGGGCGGAGC LtNAC15FAAGCTTTGTGGAGGCGAAATT LtNAC07FGCATCCGGCCTAGATAAGCA LtNAC15RCCCTTTGCCGGTAACCTTTT LtNAC07RTCCCCTTTGAAGGCTTTCCT LtNAC16FCTCAGAATGAATGGGTGA LtNAC08FGTGGGAGGCAAGGAATGGT LtNAC16RCCTCACAAGTCCTCGAAAG LtNAC08RTCGCCTTCCAGTACCCTGAT ActinFGATGGGCAGGTGATCACGAT LtNAC09FGGGCTGAGCAGGCACTTCT ActinRTCTCATGGATTCCAGCAGCTT
对于各RNA样本,cDNA第1条链的合成采用第1链cDNA合成试剂盒(美国Invitrogen公司),实验步骤参照说明书。使用1 μg总RNA进行反转录,得到20 μL产物cDNA,作为qRT-PCR扩增反应的模板。qRT-PCR使用光学96孔平板在7500型实时荧光定量PCR仪(美国ABI公司)中进行。采用单一PCR扩增法:样本和内对照分别在独立的反应孔中进行扩增。使用actin作为内对照,每个样本和内对照使用3个重复反应孔进行扩增。反应体系参照试剂盒说明书进行qRT-PCR扩增和检测。20.0 μL的反应体系包括10.0 μL SYBRMIX、10 mmol·L-1正向引物和反向引物各0.5 μL、0.8 μL第1链cDNA模板(40 ng)及8.2 μL ddH2O。反应条件如下:50 ℃, 2 min; 95 ℃,10 min;95 ℃,20 s;60 ℃,60 s;共40个循环。反应结束后通过熔解曲线鉴定产物的特异性。采用2-ΔΔCt计算各基因的相对表达量。
2 结果和分析
2.1LtNAC基因的克隆与序列分析
利用RACE技术从北美鹅掌楸叶片中共克隆出16个包含完整ORF的NAC基因,分别命名为LtNAC01至LtNAC16,ORF长度为561~1 830 bp,编码187~610个氨基酸。
北美鹅掌楸LtNAC家族基因编码的16个LtNAC蛋白的理化性质见表2。由表2可以看出:其中9个LtNAC蛋白的理论等电点(pI)大于7,属于碱性蛋白质;另外7个LtNAC蛋白的pI值小于7,属于酸性蛋白质。LtNAC16和LtNAC08蛋白的氨基酸数目较少,分别为187和190,二者的理论相对分子质量也较小,分别为21 811.9和21 667.3。LtNAC02蛋白的氨基酸数目最多(610),其理论相对分子质量也最大,为68 011.5。
表2 北美鹅掌楸LtNAC家族基因编码的LtNAC蛋白的理化性质1)
Table 2 Physical-chemical property of LtNAC protein encoded byLtNACfamily gene fromLiriodendrontulipiferaLinn.1)
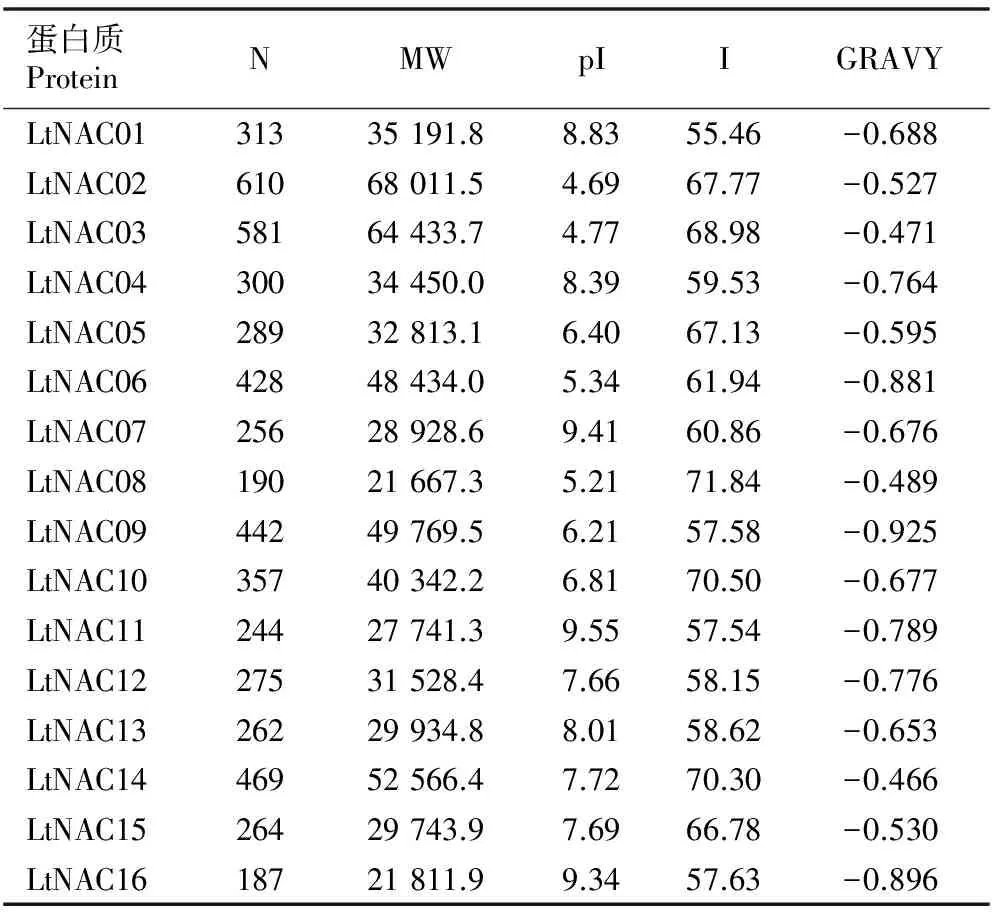
蛋白质ProteinNMWpIIGRAVYLtNAC0131335191.88.8355.46-0.688LtNAC0261068011.54.6967.77-0.527LtNAC0358164433.74.7768.98-0.471LtNAC0430034450.08.3959.53-0.764LtNAC0528932813.16.4067.13-0.595LtNAC0642848434.05.3461.94-0.881LtNAC0725628928.69.4160.86-0.676LtNAC0819021667.35.2171.84-0.489LtNAC0944249769.56.2157.58-0.925LtNAC1035740342.26.8170.50-0.677LtNAC1124427741.39.5557.54-0.789LtNAC1227531528.47.6658.15-0.776LtNAC1326229934.88.0158.62-0.653LtNAC1446952566.47.7270.30-0.466LtNAC1526429743.97.6966.78-0.530LtNAC1618721811.99.3457.63-0.896
1)N: 氨基酸数目 Number of amino acids; MW: 理论相对分子质量 Theoretical relative molecular weight; pI: 理论等电点 Theoretical isoelectric point; I: 脂肪族氨基酸指数 Aliphatic amino acid index; GRAVY: 蛋白质疏水性 Grand average of hydropathicity.
蛋白质疏水性(GRAVY)的分析结果显示:16个LtNAC蛋白的GRAVY值均为负值,其中13个LtNAC蛋白的GRAVY值小于-0.5,为亲水蛋白质;只有LtNAC03、LtNAC08和LtNAC14蛋白的GRAVY值大于-0.5,分别为-0.471、-0.489和-0.466,为两性蛋白质。
2.2LtNAC基因编码的蛋白质二级结构预测
利用SOPMA程序对16个北美鹅掌楸LtNAC家族基因编码的LtNAC蛋白的二级结构进行预测,结果见表3。由表3可以看出:16个LtNAC蛋白均以无规则卷曲为主要构成元件,其中LtNAC03蛋白的二级结构中无规则卷曲所占比例最少,仅39.59%;LtNAC04蛋白的二级结构中无规则卷曲所占比例最高,达到61.00%;α螺旋、延伸链和β转角则散布于整个LtNAC蛋白中。
蛋白质跨膜区预测研究结果显示:16个LtNAC蛋白中,LtNAC02和LtNAC03蛋白包含跨膜基序(TM,transmembrane motif),且TM都存在于C端。
表3 北美鹅掌楸LtNAC家族基因编码的LtNAC蛋白二级结构的预测结果
Table 3 Predicting result of secondary structure of LtNAC protein encoded byLtNACfamily gene fromLiriodendrontulipiferaLinn.
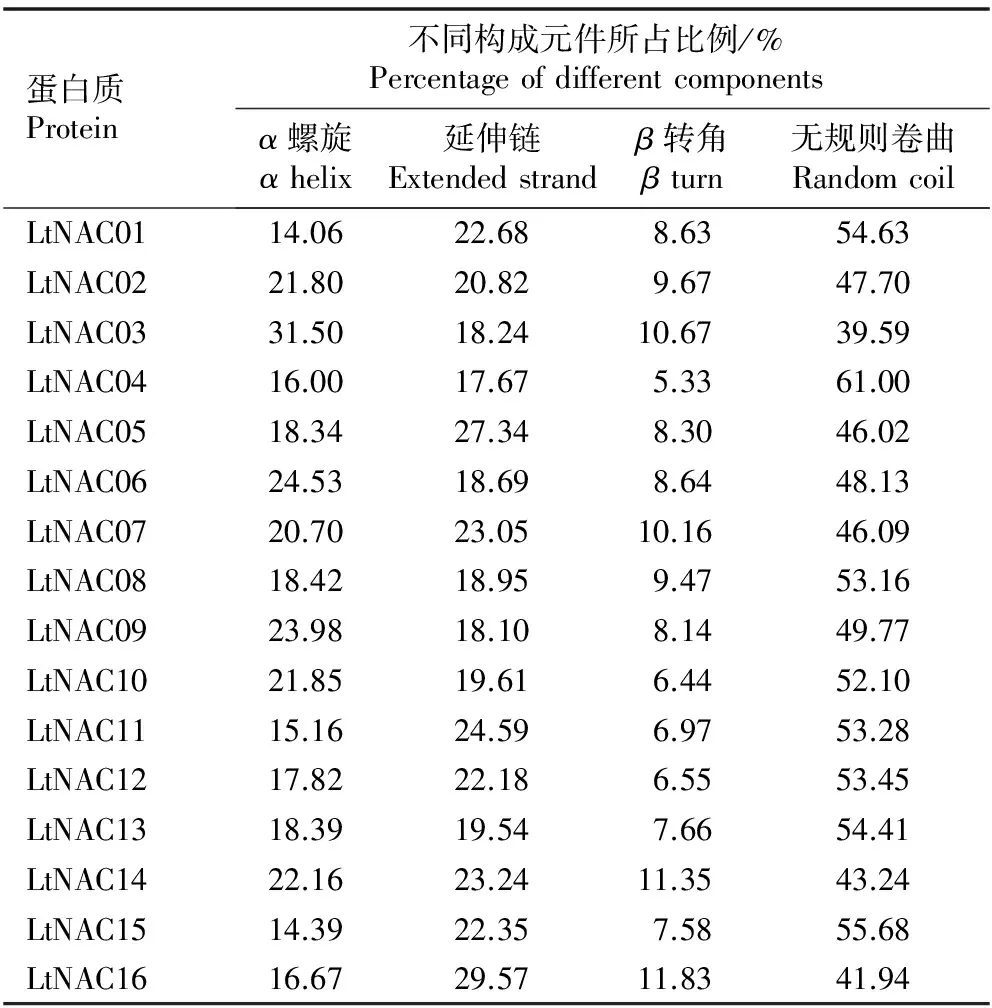
蛋白质Protein不同构成元件所占比例/%Percentageofdifferentcomponentsα螺旋αhelix延伸链Extendedstrandβ转角βturn无规则卷曲RandomcoilLtNAC0114.0622.688.6354.63LtNAC0221.8020.829.6747.70LtNAC0331.5018.2410.6739.59LtNAC0416.0017.675.3361.00LtNAC0518.3427.348.3046.02LtNAC0624.5318.698.6448.13LtNAC0720.7023.0510.1646.09LtNAC0818.4218.959.4753.16LtNAC0923.9818.108.1449.77LtNAC1021.8519.616.4452.10LtNAC1115.1624.596.9753.28LtNAC1217.8222.186.5553.45LtNAC1318.3919.547.6654.41LtNAC1422.1623.2411.3543.24LtNAC1514.3922.357.5855.68LtNAC1616.6729.5711.8341.94
2.3LtNAC基因编码的蛋白质的聚类分析
对北美鹅掌楸16个LtNAC基因编码的LtNAC蛋白与拟南芥及23个其他物种中功能已知的NAC蛋白构建系统进化树,选用保守的N端的NAC保守结构域对NAC蛋白进行多序列比对及系统进化分析,结果见图1。由图1可以看出:144个NAC蛋白被划分为NAC-Ⅰ、NAC-Ⅱ、NAC-Ⅲ、NAC-Ⅳ、NAC-Ⅴ、NAC-Ⅵ、NAC-Ⅶ和NAC-Ⅷ 8类,8类中分别包含0、2、0、1、5、4、0和4个LtNAC蛋白。其中,NAC-Ⅶ类被认为与细胞壁生物合成的转录调控有关,而北美鹅掌楸16个LtNAC基因编码的LtNAC蛋白均未出现在NAC-Ⅶ类中,说明北美鹅掌楸LtNAC基因可能未参与细胞壁的生物合成。
2.4LtNAC基因在叶片不同生长阶段的表达分析
北美鹅掌楸16个LtNAC基因在叶片不同生长阶段的相对表达量见图2。由图2可以看出:北美鹅掌楸LtNAC03、LtNAC07、LtNAC08、LtNAC09、LtNAC11、LtNAC12、LtNAC15和LtNAC16 基因在嫩叶、完全展开未衰老叶片、衰老早期叶片和衰老后期叶片中的相对表达量呈逐渐升高的趋势,推测这些基因可能正向调控植物叶片的衰老。而LtNAC14基因在完全展开未衰老叶片、衰老早期叶片和衰老后期叶片中的相对表达量呈逐渐下降的趋势,推测该基因可能是叶片衰老的负向调控因子。
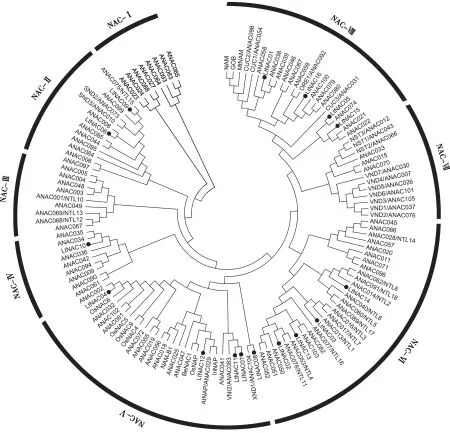
图中涉及16个北美鹅掌楸NAC蛋白(LtNAC01至LtNAC16)、105个拟南芥NAC蛋白(ANAC001至ANAC105)和23个其他物种已知的NAC蛋白,其中,16个LtNAC蛋白用“●”标出 Proteins in this figure include 16 NAC proteins fromL.tulipifera(from LtNAC01 to LtNAC16), 105 NAC proteins fromA.thaliana(from ANAC001 to ANAC105) and 23 known NAC proteins from other plants, in which, 16 LtNAC proteins are marked with “●”.
图1 北美鹅掌楸LtNAC蛋白与拟南芥NAC蛋白及23个其他物种已知NAC蛋白的系统进化分析
Fig. 1 Phylogenetic analysis on LtNAC protein fromLiriodendrontulipiferaLinn., NAC protein from
Arabidopsisthaliana(Linn.) Heynh. and 23 known NAC proteins from other plants
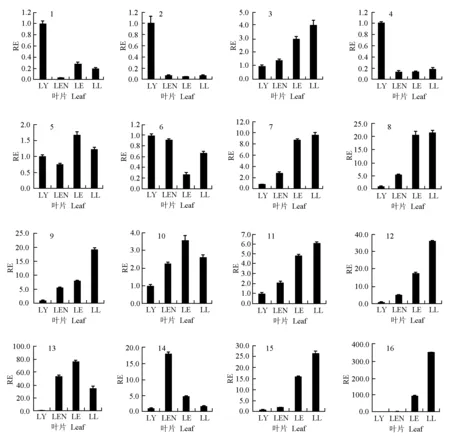
RE: 相对表达量Relative expression. LY: 嫩叶Young leaf; LEN: 完全展开未衰老叶片Fully expanded and non-senescent leaf; LE: 衰老早期叶片Leaf at the early stage of senescence; LL: 衰老后期叶片Leaf at the late stage of senescence.
1:LtNAC01基因LtNAC01 gene; 2:LtNAC02基因LtNAC02 gene; 3:LtNAC03基因LtNAC03 gene; 4:LtNAC04基因LtNAC04 gene; 5:LtNAC05基因LtNAC05 gene; 6:LtNAC06基因LtNAC06 gene; 7:LtNAC07基因LtNAC07 gene; 8:LtNAC08基因LtNAC08 gene; 9:LtNAC09基因LtNAC09 gene; 10:LtNAC10基因LtNAC10 gene; 11:LtNAC11基因LtNAC11 gene; 12:LtNAC12基因LtNAC12 gene; 13:LtNAC13基因LtNAC13 gene; 14:LtNAC14基因LtNAC14 gene; 15:LtNAC15基因LtNAC15 gene; 16:LtNAC16基因LtNAC16 gene.
图2 北美鹅掌楸16个LtNAC基因在叶片不同生长阶段的相对表达量
Fig. 2 Relative expression of sixteenLtNACgenes at different growth stages of leaf ofLiriodendrontulipiferaLinn.
3 讨 论
对北美鹅掌楸NAC预测蛋白质理化特征的分析结果显示:16个LtNAC蛋白的GRAVY值均为负值,其中81.25%LtNAC蛋白的GRAVY值小于-0.5,与王洋等[26]对大豆〔Glycinemax(Linn.) Merr.〕的研究结果一致,152条大豆NAC蛋白的GRAVY平均值均为负值,且大多数小于-0.5。北美鹅掌楸全基因组范围内NAC家族理化性质是否也具有相同特征尚待进一步研究。
Ooka等[27]将拟南芥以及水稻全部NAC蛋白分为2个大类,分别包含14和4个亚类。Shen等[28]将11种植物的1 232个NAC蛋白分成8类。本研究中144个NAC蛋白序列共聚成8类,目前在不同植物NAC蛋白的系统进化研究中,NAC蛋白被聚成不同数目的类别,它们均受到所分析的蛋白数目、采用的算法和软件以及选用的是保守结构域还是全长氨基酸序列进行比对等因素的影响。本研究采用的是使用较普遍的NAC保守结构域及NJ法进行系统进化分析,结果显示具有相同功能的基因编码的蛋白趋向于划分在同一类中。根据系统进化分析结果,胁迫应答NAC基因(SNAC),包括水稻OsNAC4、OsNAC5、OsNAC6和OsNAC9(SNAC1)以及拟南芥ANAC019、ANAC055和ANAC072 基因编码的蛋白被归到NAC-Ⅴ类中。Ricachenevsky等[29]认为该类基因参与胁迫应答的功能在不同物种中可能是保守的。在本研究中,LtNAC04基因与SNAC基因编码的蛋白聚为一类,因而,从系统进化关系上预测,LtNAC04 基因有可能与胁迫应答有关。LtNAC蛋白跨膜区预测研究表明:LtNAC02和LtNAC03蛋白包含TM螺旋。植物NAC家族一些成员C端含有1个至多个α螺旋的跨膜基序,这些C端含有跨膜基序的NAC基因被称为NTL(NTM1-like)。全基因组研究结果表明:拟南芥中有至少13个NTL(NTLs)、水稻中有5个NTL(OsNTLs)、马铃薯(SolanumtuberosumLinn.)中有14个NTL[18]、大豆中有11个NTL[30],这些NTL被发现参与植物的胁迫响应[18]。根据系统进化分析结果,大多数拟南芥NTL聚集在NAC-Ⅵ类中;少数聚集在NAC-Ⅲ和NAC-Ⅳ类中;北美鹅掌楸LtNAC02和LtNAC03蛋白也聚集在NAC-Ⅵ类中,据此预测LtNAC02和LtNAC03 基因也可能参与北美鹅掌楸的胁迫响应。
植物组织自然衰老过程的主要作用是在种子日渐饱满的过程中完成营养物质的转移。植物NAC基因对叶片衰老的调控作用包括正向调控作用和负向调控作用,正向调控基因如拟南芥的AtNAP[14]、ATAF1[31]、ORE1和ORS1(ORE1SISTER1)[32],小麦(TriticumaestivumLinn.)的NAM-B1[17],水稻的OsNAC5[33]及Bambusaemeiensis‘Viridiflavus’ 的BeNAC1[15]等;负向调控基因如JUB1[34]和VNI2[35]。本研究中北美鹅掌楸LtNAC03、LtNAC07、LtNAC08、LtNAC09、LtNAC11、LtNAC12、LtNAC15和LtNAC16 基因的相对表达量与叶片衰老程度呈正相关,为叶片衰老的正向调控候选因子;LtNAC14 基因的相对表达量与叶片衰老程度呈负相关,为叶片衰老的负向调控候选因子。在拟南芥的109个NAC基因家族成员中,其中约20%的基因在衰老叶片中表达[13]。在本研究中,北美鹅掌楸的16个LtNAC基因中有9个基因被认为是与叶片衰老有关的候选基因,所占比例远高于拟南芥。这是由于本实验克隆获得的北美鹅掌楸LtNAC基因均基于鹅掌楸叶片和花瓣转录组数据库,而在这2个组织中不表达的LtNAC基因无法克隆获得,因此推测实际上与叶片衰老相关的LtNAC基因所占比例应低于50%。
已有的研究结果显示:不同植物的NAC基因可能作用于不同的信号传导途径中,处于同一信号传导途径中的NAC基因也可能处于上、下游的不同位置,这些都可能是由于聚类上的分化导致的。如,在拟南芥Ethylene-insensitive2(EIN2)介导的叶片衰老代谢途径中,信号传递分子EIN3直接结合ORE1和AtNAP的启动子并激活表达,且ORE1和AtNAP作用于2条不同但交叉的信号传导途径[36]。Garapati等[31]认为,ATAF1 基因是ABA和H2O2介导的衰老代谢途径的上游调控因子,通过激活衰老调控NAC转录因子ORE1和抑制叶绿素维持关键因子GLK1 的表达使叶片向衰老转变。Hollmann等[20]认为,在冬季大麦叶片中HvNAC026是衰老相关的N元素再利用代谢途径的重要调控基因,然而,同样是衰老相关的基因,HvNAC001、HvNAC005和HvNAC013则可能并不参与N元素的回收再利用。北美鹅掌楸9个与叶片衰老相关的LtNAC基因被聚为多个类别。在9个LtNAC基因中,LtNAC12基因与许多已知的叶片衰老调控基因(如AtNAP、InNAP、BeNAC1、OsNAP及NAM-B1)聚在一起,它们都是经过功能验证并在植物叶片衰老过程中起重要调控作用的基因。所以,LtNAC12 基因也可能在北美鹅掌楸叶片衰老的过程中起到重要的调控作用。
[1] SOUERE, VanHOUWELINGENA, KLOOSD, et al. Thenoapicalmeristemgene ofPetuniais required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell, 1996, 85: 159-170.
[2] AIDA M, ISHIDA T, TASAKA M. Shoot apical meristem and coty-ledon formation duringArabidopsisembryogenesis: interaction among theCUP-SHAPEDCOTYLEDONandSHOOTMERISTEMLESSgenes[J]. Development, 1999, 126: 1563-1570.
[3] CHENGX,PENGJ,MAJ, et al.NOAPICALMERISTEM(MtNAM) regulates floral organ identity and lateral organ separation inMedicagotruncatula[J]. New Phytologist, 2012, 195: 71-84.
[4] AIDAM, TASAKAM. Geneticcontrol of shootorganboundaries[J]. Current Opinion in Plant Biology, 2006, 9: 72-77.
[5] RAST M I, SIMON R. The meristem-to-organ boundary: more than an extremity of anything[J]. Current Opinion in Genetics and Development, 2008, 18: 287-294.
[6] NIKOVICSK,BLEINT,PEAUCELLEA, etal. The balance between theMIR164AandCUC2 genes controls leaf margin serration inArabidopsis[J]. The Plant Cell, 2006, 18: 2929-2945.
[7] KAWAMURAE,HORIGUCHIG,TSUKAYAH.Mechanisms of leaf tooth formation inArabidopsis[J]. The Plant Journal, 2010, 62: 429-441.
[8] BEN-GERAH,SHWARTZI,SHAO M R, et al. ENTIRE and GOBLET promote leaflet development in tomato by modulating auxin response[J]. The Plant Journal, 2012, 70: 903-915.
[9] ZHOU J, ZHONG R, YE Z H.ArabidopsisNAC domain proteins, VND1 to VND5, are transcriptional regulators of secondary wall biosynthesis in vessels[J]. PLoS One, 2014, 9: e105726.
[10] MITSUDAN, SEKIM, SHINOZAKIK,et al. The NAC trans-cription factors NST1 and NST2 ofArabidopsisregulate secondary wall thickenings and are required for anther dehiscence[J]. The Plant Cell, 2005, 17: 2993-3006.
[11] MITSUDAN,IWASEA,YAMAMOTOH,et al. NAC trans-cription factors, NST1 and NST3, are key regulators of the formation of secondary walls in woody tissues ofArabidopsis[J]. The Plant Cell, 2007, 19: 270-280.
[12] ZHONGR,YEZH.Complexity of the transcriptional network controlling secondary wall biosynthesis[J]. Plant Science, 2014, 229: 193-207.
[13] GUO Y, CAI Z, GAN S. Transcriptome ofArabidopsisleaf senes-cence[J]. Plant, Cell and Environment, 2004, 27: 521-549.
[14] GUO Y, GAN S. AtNAP, a NAC family transcription factor, has an important role in leaf senescence[J]. The Plant Journal, 2006, 46: 601-612.
[15] CHEN Y, QIU K, KUAI B, et al. Identification of an NAP-like transcription factor BeNAC1 regulating leaf senescence in bamboo (Bambusaemeiensis‘Viridiflavus’)[J]. Physiologia Plantarum, 2011, 142: 361-371.
[16] LIANG C, WANG Y, ZHU Y, et al. OsNAP connects abscisic acid and leaf senescence by fine-tuning abscisic acid biosynthesis and directly targeting senescence-associated genes in rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111: 10013-10018.
[17] UAUYC, DISTELFELDA, FAHIMAT, et al.ANACgene regulating senescence improves grain protein, zinc, and iron content in wheat[J]. Science, 2006, 314: 1298-1301.
[18] SINGHAK, SHARMAV, PALAK, etal.Genome-wide organization and expression profiling of the NAC transcription factor family in potato (SolanumtuberosumL.)[J]. DNA Research, 2013, 20: 403-423.
[19] FANG Y, YOU J, XIE K, et al. Systematic sequence analysis and identification of tissue-specific or stress-responsive genes of NAC transcription factor family in rice[J]. Molecular Genetics and Genomics, 2008, 280: 547-563.
[20] HOLLMANN J, GREGERSEN P L, KRUPINSKA K. Identification of predominant genes involved in regulation and execution of senescence-associated nitrogen remobilization in flag leaves of field grownbarley[J]. JournalofExperimental Botany, 2014, 65: 3963-3973.
[21] CHRISTIANSEN M W, GREGERSEN P L. Members of the barley NAC transcription factor gene family show differential co-regulation with senescence-associated genes during senescence of flag leaves[J]. Journal of Experimental Botany, 2014, 65: 4009-4022.
[22] PALMERNA,DONZE-REINERT,HORVATHD,etal. Switchgrass (PanicumvirgatumL.) flag leaf transcriptomes reveal molecular signatures of leaf development, senescence, and mineral dynamics[J]. Functional and Integrative Genomics, 2015, 15: 1-16.
[23] YANG Y, XU M, LUO Q, et al. De novo transcriptome analysis ofLiriodendronchinensepetals and leaves by Illumina sequencing[J]. Gene, 2014, 534: 155-162.
[24] LIANG H, AYYAMPALAYAM S, WICKETT N, et al. Generation of a large-scale genomic resource for functional and comparative genomics inLiriodendrontulipiferaL.[J]. Tree Genetics and Genomes, 2011, 7: 941-954.
[25] GEOURJON C, DELÉAGE G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Computer Applications in the Biosciences: CABIOS, 1995, 11: 681-684.
[26] 王 洋, 柏 锡. 大豆NAC基因家族生物信息学分析[J]. 大豆科学, 2014, 33(3): 325-333.
[27] OOKA H, SATOH K, DOI K, et al. Comprehensive analysis ofNACfamily genes inOryzasativaandArabidopsisthaliana[J]. DNA Research, 2003, 10: 239-247.
[28] SHEN H, YIN Y, CHEN F, et al. A bioinformatic analysis ofNACgenes for plant cell wall development in relation to lignocellulosic bioenergy production[J]. BioEnergy Research, 2009, 2: 217-232.
[29] RICACHENEVSKYFK, MENGUERPK,SPEROTTO R A. kNACking on heaven’s door: how important are NAC transcription factors for leaf senescence and Fe/Zn remobilization to seeds?[J]. Frontiers in Plant Science, 2013, 4: 226.
[30] LE DT, NISHIYAMAR, WATANABE Y, et al. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J]. DNA Research, 2011, 18: 263-276.
[31] GARAPATI P, XUE G P, MUNNÉ-BOSCH S, et al. Transcription factorATAF1 inArabidopsispromotes senescence by direct regula-tion of key chloroplast maintenance and senescence transcriptional cascades[J]. Plant Physiology, 2015, 168: 1122-1139.
[32] BALAZADEH S, KWASNIEWSKI M, CALDANA C, et al. ORS1, an H2O2-responsive NAC transcription factor, controls senescence inArabidopsisthaliana[J]. Molecular Plant, 2011, 4: 346-360.
[33] SPEROTTO R A, BOFF T, DUARTE G L, et al. Identification of putative target genes to manipulate Fe and Zn concentrations in rice grains[J]. Journal of Plant Physiology, 2010, 167: 1500-1506.
[34] WU A, ALLU A D, GARAPATI P, et al.JUNGBRUNNEN1, a reactive oxygen species-responsive NAC transcription factor, regulates longevity inArabidopsis[J]. The Plant Cell, 2012, 24: 482-506.
[35] YANGS, SEOPJ, YOONH, etal.TheArabidopsisNAC transcription factor VNI2 integrates abscisic acid signals into leaf senescence via theCOR/RDgenes[J]. The Plant Cell, 2011, 23: 2155-2168.
[36] KIM H J, HONG S H, KIM Y W, et al. Gene regulatory cascade of senescence-associated NAC transcription factors activated by ETHYLENE-INSENSITIVE2-mediated leaf senescence signalling inArabidopsis[J]. Journal of Experimental Botany, 2014, 65: 4023-4036.
(责任编辑: 张明霞)
欢迎订阅2016年《植物资源与环境学报》
全国中文核心期刊 中国科技核心期刊
中国科学引文数据库核心期刊 RCCSE中国核心学术期刊(A)
“中国期刊方阵”双效期刊 “江苏期刊方阵”优秀期刊
季刊,单价20元,邮发代号28-213,国内统一连续出版物号CN 32-1339/S
《植物资源与环境学报》系江苏省·中国科学院植物研究所、江苏省植物学会等单位联合主办的学术期刊,国内外公开发行。本刊为BA、CA、CAB、Elsevier’s、中国生物学文摘、中国环境科学文摘、中国科学引文数据库、万方数据——数字化期刊群、中国学术期刊(光盘版)和中文科技期刊数据库等国内外著名刊库收摘,并被Ulrich’s期刊指南等所收录。本刊在2013年荣获首届“江苏省新闻出版政府奖——报刊奖”及江苏省科学技术协会“精品科技期刊”称号,在2015年荣获“第六届江苏省科技期刊金马奖——精品期刊奖”。
本刊围绕植物资源与环境两个中心命题,报道我国植物资源的考察、开发利用和植物物种多样性保护,自然保护区与植物园的建设和管理,植物在保护和美化环境中的作用,环境对植物的影响以及与植物资源和植物环境有关学科领域的原始研究论文、研究简报和综述等。凡从事植物学、生态学、自然地理学以及农、林、园艺、医药、食品、轻化工和环境保护等领域的科研、教学、技术人员及决策者均可以从本刊获得相关学科领域的研究进展和信息。
本刊为季刊,大16开本。每期定价20元,全年80元,全国各地邮局均可订阅。若错过征订时间或需补齐1992年至2015年各期者,请直接与编辑部联系邮购。1992年至1994年每年8元,1994年至2000年每年16元,2001年至2005年每年24元,2006年至2008年每年40元,2009年至2011年每年60元,2012年至2015年每年80元 (均含邮资),如需挂号另付邮挂费。
编辑部地址:江苏省南京市中山门外 江苏省中国科学院植物研究所内(邮编210014);电话: 025-84347014; E-mail: zwzy@cnbg.net; QQ:2219161478。投稿网址:http:∥www.cnbg.net/Tg/Contribute/Login.aspx。
欢迎订阅! 欢迎投稿!
Cloning ofNACgene fromLiriodendrontulipiferaand its expression analysis
YANG Ying, LI Huogen①
(Co-Innovation Center for Sustainable Forestry in Southern China, Nanjing Forestry University, Nanjing 210037, China),J.PlantResour. &Environ., 2015, 24(3): 1-9
Based on the transcriptome data ofLiriodendronchinense(Hemsl.) Sarg., structure and function ofNAC(NAM,ATAF1/2 andCUC2) family genes fromL.tulipiferaLinn. were researched. Open reading frame (ORF) of 16LtNACgenes was cloned from leaf ofL.tulipiferaby RACE technology with Nos. fromLtNAC01 toLtNAC16. Length of ORF of 16LtNACgenes is 561-1 830 bp, and 187-610 amino acids are encoded. According to theoretical isoelectric point (pI), there are 9 basic proteins and 7 acidic proteins in 16 LtNAC proteins encoded byLtNACgenes. According to grand average of hydropathicity (GRAVY), there are 13 hydrophilic proteins and 3 hydrophonic proteins in 16 LtNAC proteins. The predicting result of secondary structure of LtNAC protein shows that random coil is main component in all of 16 LtNAC proteins, andαhelix, extended strand andβturn distribute widely in the whole protein. The result of phylogenetic analysis shows that 16LtNACgenes ofL.tulipiferamay not participate in cell wall biosynthesis, in which,LtNAC02,LtNAC03 andLtNAC04 genes may participate in stress response ofL.tulipifera. Relative expression ofLtNAC03,LtNAC07,LtNAC08,LtNAC09,LtNAC11,LtNAC12,LtNAC14,LtNAC15 andLtNAC16 genes is related to leaf senescence progression ofL.tulipifera, and it is predicted that the nineLtNACgenes may participate in regulating leaf senescence progression.
LiriodendrontulipiferaLinn.;LtNACgene; leaf senescence; qRT-PCR
2015-05-22
国家自然科学基金资助项目(31470660; 31170621); 江苏高校优势学科建设工程资助项目(PAPD)
杨 颖(1988—),女,江苏南京人,博士研究生,主要从事森林遗传学等方面的研究。
①通信作者 E-mail: hgli@njfu.edu.cn
Q943.2; S792.21
A
1674-7895(2015)03-0001-09
10.3969/j.issn.1674-7895.2015.03.01

