利用Gibson Assembly技术构建乙型肝炎病毒1.1倍体复制子
方琳琳,宋浏伟,杨 林,吴 勇,袁 权,夏宁邵
(厦门大学公共卫生学院,国家传染病诊断试剂与疫苗工程技术研究中心,福建厦门361101)
利用Gibson Assembly技术构建乙型肝炎病毒1.1倍体复制子
方琳琳,宋浏伟,杨 林,吴 勇,袁 权*,夏宁邵
(厦门大学公共卫生学院,国家传染病诊断试剂与疫苗工程技术研究中心,福建厦门361101)
乙型肝炎病毒(HBV)基因组各开放阅读框高度重叠,因此构建可在体外细胞模型中复制的HBV DNA质粒至少要包含1.1倍以上的HBV基因组.构建HBV1.1倍体复制子的传统克隆方法涉及多步DNA酶切/连接,操作复杂,耗时较长.本研究发展了基于新型克隆技术Gibson Assembly构建HBV1.1倍体复制子质粒的新方法,在获得HBV基因组DNA后仅需1步体外装配即可完成,利用这一方法,本研究成功从4份HBV感染者标本中获得HBV1.1倍体复制子,并通过瞬时转染Huh7细胞,证明其能够支持HBV的体外复制.本研究为从临床标本中快速获得HBV复制子克隆以研究病毒表型功能提供了更为高效的新方法.
乙型肝炎病毒;Gibson Assembly;1.1倍体
乙型肝炎病毒(hepatitis B virus,HBV)感染是世界范围内重要的公共卫生问题之一[1].它通过血液、体液等传播,HBV感染可导致急性或者慢性感染,急性感染有可能引起暴发性肝病,导致死亡,而对于慢性感染人群,则患肝硬化或肝癌的概率大大上升,全球慢性乙肝患者已经超过2亿4千万人,每年大约有60万人死于HBV感染相关疾病[2].中国是HBV高流行区,有近1亿HBV携带者,其中每年新发HBV感染达10万以上,慢性乙型肝炎患者高达3 000万人[3],多种肝脏疾病与HBV的持续感染相关,例如,原发性肝细胞癌(hepatocellular carcinoma,HCC),其发病率和死亡率分别位于全球恶性肿瘤的第五位和第三位,在我国癌症死亡率中排名第二[4-5].因此,对HBV的预防和治疗研究任重而道远.
HBV为正嗜肝DNA病毒属[6],其基因组大小约3.2 kb,为部分双链的松弛环状DNA,共包含4个开放阅读框(open reading frames,ORF),即S-ORF、PORF、C-ORF、X-ORF,4种ORF分别有各自的启动子.其中S-ORF分别编码3个包膜蛋庄:L-HBsAg (编码序列:nt2854-nt3221/1-nt832)、M-HBs Ag(编码序列:nt3211-nt3221/1-nt832)和S-HBsAg(编码序列:nt155-nt832);C-ORF编码Pre-Core蛋庄(HBe Ag,编码序列:nt1814-nt2456)和Core蛋庄(HBc Ag,编码序列:nt1901-nt2456),HBe Ag是一种分泌型蛋庄,在慢性感染中起到耐受宿主免疫应答的作用,Pre-Core组成病毒Capsid,并包裹病毒基因组.由于HBV基因组结构紧密,高度压缩,重复利用,不同编码基因相互重叠,启动子和增强子等调控序列又位于编码基因之内,ORF分布于全长DNA,某个部位的基因突变将影响多个基因的表达,因此需要含大于3.2 kb的HBV基因序列的载体才能在转染细胞内建立HBV的复制状态[7].通常需要构建HBV1.1或者以上倍体复制子用于病毒功能的研究.
本实验利用新型克隆技术“Gibson Assembly”[8],从临床标本中构建了HBV1.1倍体复制子,并转染Huh7细胞验证其功能;避免了传统方法中多步酶切等繁复的实验步骤,大大提高了克隆构建的效率,为HBV临床研究和病毒功能研究提供了更快速高效的方法.

1 材料与方法
1.1 材 料
1.1.1 HBV阳性血清
4份HBs Ag阳性标本来自西藏林芝医院(血清样本的处理及病毒DNA提取均在P2级病毒室操作).标本的基本信息如表1所示.
1.1.2 质粒、菌株及主要试剂
真核表达载体p TT22E由实验室自己构建; PrimeSTAR®GXL DNA Polymerase为大连宝生物(TaKaRa)公司产品;QIAamp DNA Blood MiniKit试剂盒为QIAGEN公司产品;感受态细胞DH5α、质粒小提试剂盒(离心柱型)、琼脂糖凝胶DNA纯化回收试剂盒(离心柱型)均为天根生化科技(北京)有限公司产品(TIANGEN);Gibson Assembly®Master Mix为纽英伦(NEW ENGLAND Bio Labs)生物技术有限公司产品;Turbofect Transfection Reagent为MBIFermentas公司的产品.
1.1.3 细胞内颗粒中DNA提取所需试剂
除了QIAamp DNA Blood Mini Kit试剂盒为QIAGEN公司产品以外,还需以下试剂:PBS溶液:20 mmol/L NaCl,2.68 mmol/L KCl,10 mmol/L Na2HPO4,1.76 mmol/L KH2PO4;细胞裂解液:10 mmol/L TB8.0,1 mmol/L乙二胺四乙酸(EDTA), 2%(质量分数)蔗糖,1%(质量分数)非离子变性剂乙基苯基聚乙二醇(NP40).
1.2 方 法
1.2.1 引物设计
根据GenBank中HBV全基因序列设计PCR所需引物.另外,根据Gibson Assembly的引物设计原则,为了保证引物特异性,特异性结合区设为18~25个碱基,在引物的5′端加入交叠序列.交叠序列与临近序列的末端同源,交叠序列的长度≥20 bp.
引物Ec1816F/n224R用于扩增A片段,引物n199F/Hd2049R用于扩增B片段.p TT1369F、p TT1342R分别为扩增载体的上下游引物,S3、S4分别为鉴定上下游引物,所有引物均由上海英骏生物技术有限公司合成.引物序列如表2(下划线部分为相应酶切位点).
1.2.2 标本的处理及血清HBV DNA提取
标本于¯20℃保存,检测前取出自然化冻,去血清标本200μL,用QIAamp DNA Blood Mini Kit试剂盒提取,按照核酸提取试剂盒说明书中的步骤进行操作,最后得到100μL血清DNA提取物溶液.
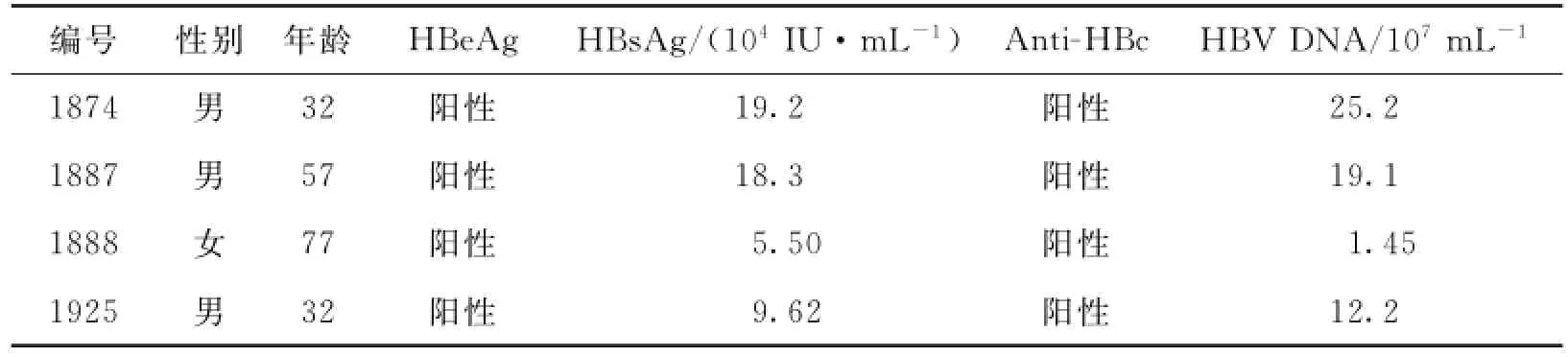
表1 临床样本具体信息Tab.1 Sample demographic and clinical characteristics

表2 PCR分段扩增HBV基因组所用引物(I)、扩增载体所用引物(II)以及鉴定引物(III)Tab.2 Primers used for HBV DNA amplification(I),primers used for vector amplification(II)and primers for identification(III)
1.2.3 A、B片段的扩增
以血清中提取的HBV DNA为模板,以Ec1816F、n224R为引物扩增A片段,以n199F、Hd2049R为引物扩增B片段.采用高保真性聚合酶PrimeSTAR GXL DNA Polymerase进行PCR扩增, PCR反应体系为50μL,反应体系包括5×Prime-STAR GXL Buffer 10μL,d NTP混合液(2.5 mmol/ L)4μL(终浓度0.2 mmol/L),引物各1μL(终浓度0.4μmol/L),PrimeSTAR®GXL酶1μL,HBV DNA 5μL,补加超纯水至50μL;扩增条件:98℃预变性10 min;98℃变性30 s,58℃退火30 s,68℃延伸2 min,30个循环;最后72℃延伸10 min.用1.5% (质量分数,下同)的琼脂糖凝胶于TAE缓冲液中电泳回收PCR产物,采用TIANGEN公司生产的DNA回收试剂盒,按其说明书使用回收.
1.2.4 载体的制备
用PCR扩增出线性载体再用Dpn1处理的方法制备.以p TT22E质粒作为模板,以p TT1369F、p TT1342R为引物,采用高保真性聚合酶PrimeSTAR GXL DNA Polymerase扩增载体,扩增条件:98℃预变性10 min;98℃变性30 s,58℃退火30 s,68℃延伸6 min,30个循环;最后72℃延伸10 min.之后,用Dpn1处理,1μL Dpn1+1μL Dpn1 Buffer/50μL的比例,37℃反应60 min,80℃5 min使Dpn1失活.用1%的琼脂糖凝胶于TAE缓冲液中电泳回收PCR产物,采用TIANGEN公司生产的DNA回收试剂盒,按其说明书使用回收.
1.2.5 pTT22E-1.1HBV克隆的构建
Gibson Assembly是Dr.Daniel Gibson发展出的体外高效快速合成DNA的新方法,现被广泛应用于分子克隆的构建,尤其是大片段及多片段的分子克隆构建.其原理如图1(a)所示,Gibson Assembly的体系主要由T5 exonuclease、Phusion DNA polymerase、Taq DNA ligase 3种核心酶组成.T5 exonuclease具有5′-3′DNA外切酶活性,制造载体与片段或片段与片段之间的互补末端;Phusion DNA polymerase具有3′-5′DNA聚合酶活性,互补单链退火后将缺口补齐; Taq DNA ligase具有连接酶活性,将两条单链间切口连接.利用这3种酶和缓冲液按一定比例配成预混液,即可实现50℃一步反应的目的[8].同时,Gibson Assembly合成的DNA没有缺口或切口,不依赖大肠杆菌(Escherichia coli)的修复,因此效率高,可在转化前跑胶鉴定组装是否成功.
根据图1(b)中所示的流程图,在获得片段和载体之后,将片段1、2与载体进行Gibson Assembly,体系为:2×Gibson mix为4μL,片段A、B分别为1.5μL,载体为1μL,50℃反应1 h.连接产物直接做转化,用TIANGEN公司的DH5α感受态.挑取单菌落,接种于5 m L LB/AMP+液体培养基中,180 r/min,37℃过夜培养.用引物S3、S4进行菌液PCR鉴定,挑选菌液PCR阳性的提质粒,电泳,通过质粒大小鉴定是否插入了外源片段.
1.2.6 核苷酸测序鉴定
将PCR鉴定阳性的克隆送上海Invitrogen公司进行核酸序列测定.在美国Applied Biosystem公司生产的3730自动测序仪上分段进行测序.
1.2.7 转染Huh7细胞
人肝癌细胞株Huh7细胞来自于本实验室,转染前,将细胞培养于含10%(体积分数)胎牛血清的基础培养基(DMEM),于转染前1 d转至十二孔板培养,按照Turbofect的转染方法说明,将上述HBV体外重组质粒与GFP质粒(m(HBV质粒)∶m(GFP质粒)=9∶1)共转转染人肝癌细胞株Huh7.4 d后收集细胞上清,利用酶联免疫吸附法(ELISA)检测细胞培养上清液的HBs Ag与HBe Ag水平.
1.2.8 细胞内病毒颗粒中DNA的定量检测
根据之前的报道,用PEG8000沉淀HBV core,提取core DNA[9],并用实时荧光定量PCR检测HBV DNA[10].
2 实验结果
用Gibson Assembly的方法构建1.1倍体时,一些重要步骤所对应的结果如图2所示,先用PCR的方法获取载体,然后用Dpn1消化后,采用柱法回收.处理完的载体,与回收好的2个片段,一起进行50℃Gibson Assembly后,转化、涂板、挑菌并鉴定即可获得目的克隆.
2.1 两片段PCR扩增结果
按前述PCR反应体系及反应条件,获得PCR产物,将其在1.5%和1%的琼脂糖凝胶中电泳,蓝光灯下观察到约1.6和1.9 kb的条带(片段1和2),以及6 kb的条带(载体).结果与预期片段大小相符(图2(a)和(b)).
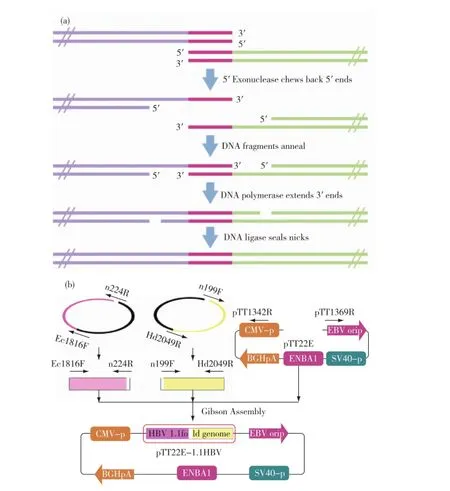
图1 Gibson Assembly原理示意图(a)和Gibson Assembly构建1.1倍HBV感染性克隆流程图(b)Fig.1 Overview of the Gibson Assembly method(a)and diagram for process of the cloning(b)
2.2 Gibson Assembly质粒鉴定
2.2.1 PCR鉴定结果
以p TT22E-1.1HBV为模板,PCR扩增S片段,扩增产物经1.5%琼脂糖凝胶电泳,可得到500 bp的片段,与预计片段大小相符,说明从临床样本中PCR得到的1.1倍的HBV DNA已和载体连接,亦即HBV1.1倍体复制子已经构建成功.如图2(c)所示。
2.2.2 测序分析
测序结果经核对后完全正确,也进一步证实插入的基因片段方向及核昔酸序列正确.
测序结果已提交GenBank后获得的收录号分别为:KP276253、KP276254、KP276255、KP276256(分别来自表1中的编号1874、1887、1888、1925).
2.3 构建质粒转染Huh7
根据测序结果,选择没有突变的质粒,将此HBV体外重组质粒与GFP共转到人肝癌细胞株Huh7中,转染2 d后荧光显微镜观察,结果如图3(a)所示,另单转GFP质粒用以质控转染效率,如图3(b)所示.转染4 d后收集细胞上清,检测其HBs Ag与HBe Ag,结果发现均呈阳性,如图4(a)和(b)所示;并裂解细胞,提取胞内的HBV病毒颗粒中的DNA,进行HBV DNA的定量.
再经过细胞数量及转染效率的标化后获得如图4 (c)所示的每个细胞中的病毒拷贝量的结果.结果显示,除编号1874对应的样本相对较低外,其余3株1.1倍克隆均能在细胞中有效复制.
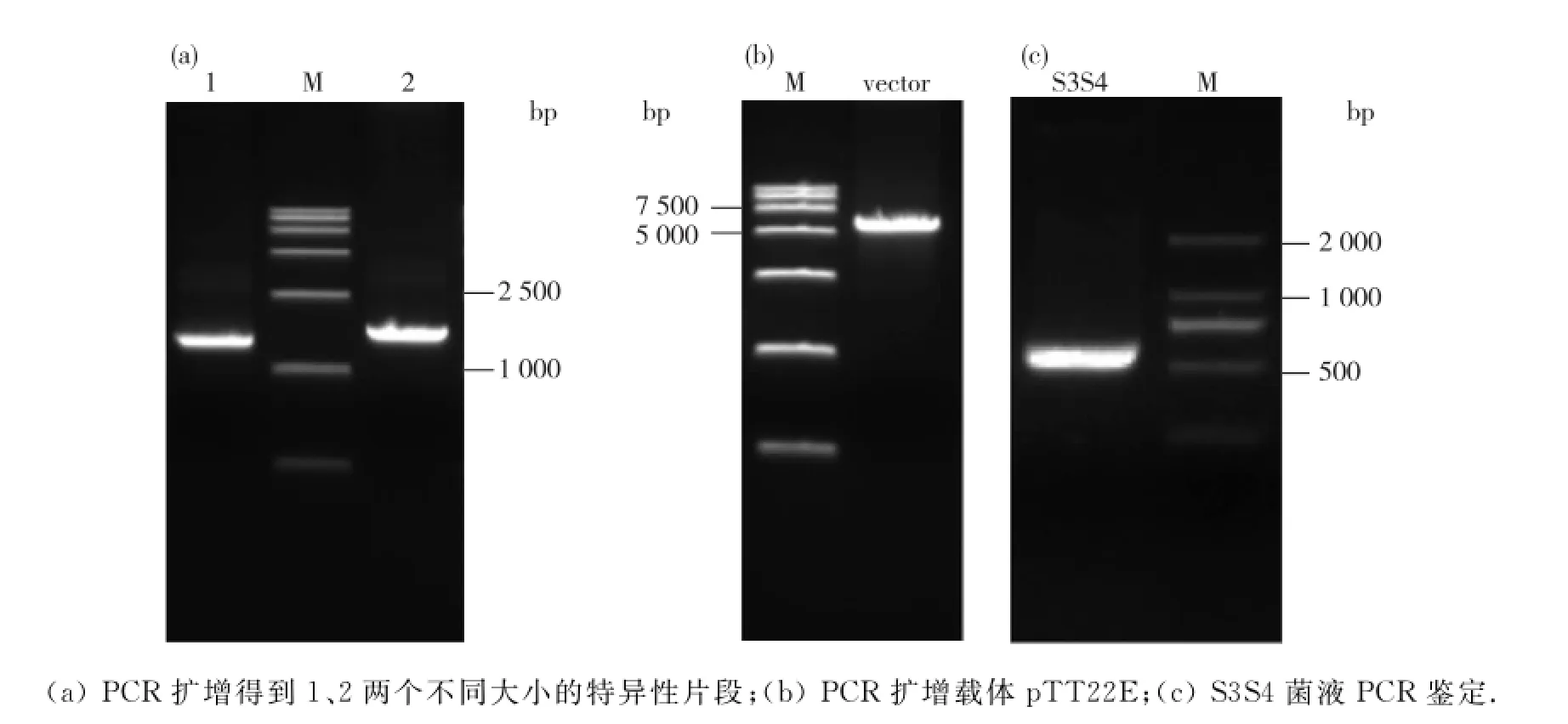
图2 用Gibson Assembly方法构建HBV 1.1倍体复制子Fig.2 Construction of 1.1-fold-overlength genome plasmids by Gibson Assembly
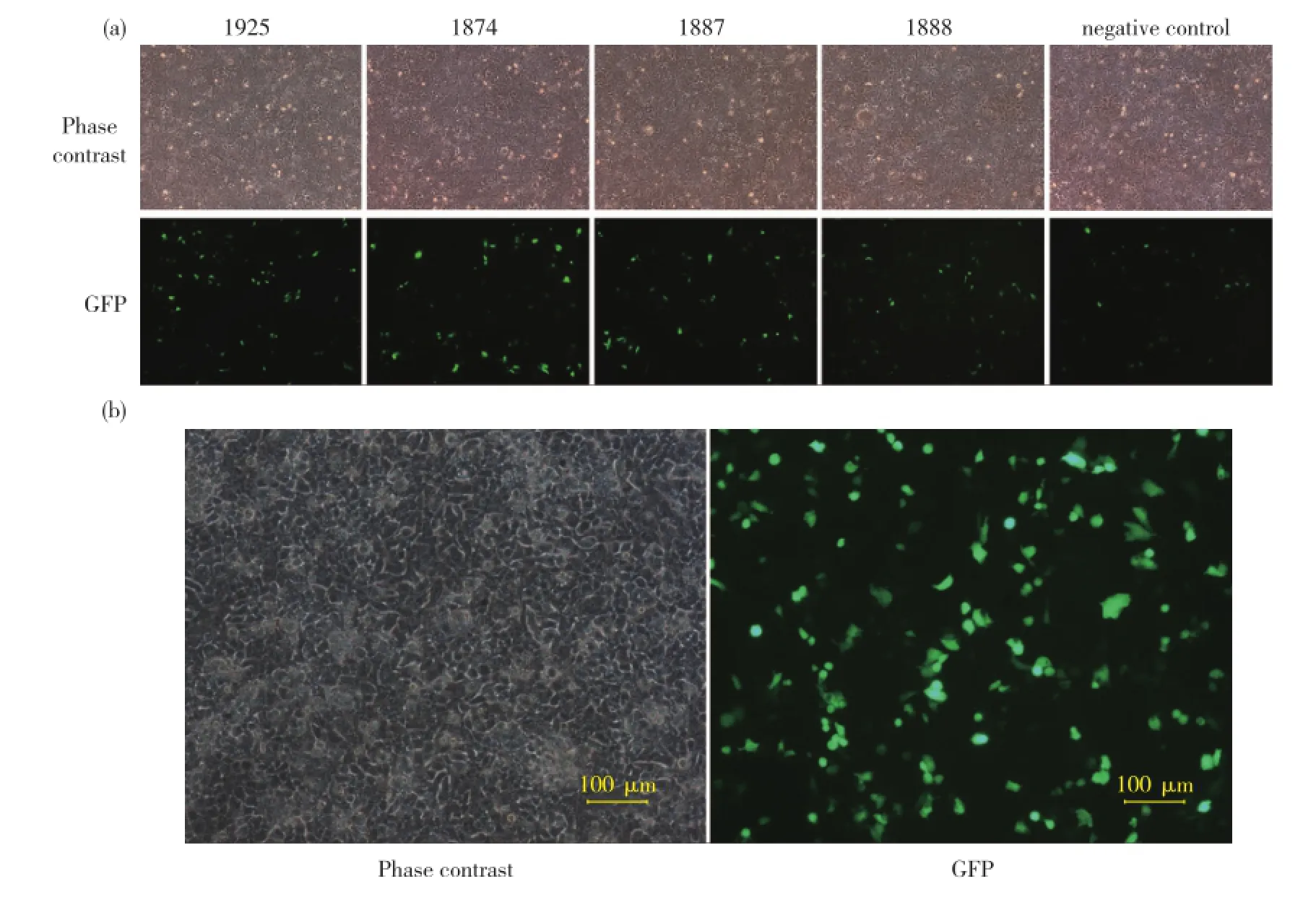
图3 1.1倍HBV重组质粒与GFP共转Huh7 2 d后得到的常光拍摄图和荧光图(a);单转GFP指征每一孔细胞的转染效率(b)Fig.3 GFP channel and phase contrast channel were recorded at 48 h after co-transfection(a)and GFP transfection only(b)
3 讨 论
从感染机制上来看,HBV感染宿主细胞后,基因组DNA进入细胞核,经DNA聚合酶修复缺失部分,形成共价闭合环状双链DNA(covalently closed circular DNA,cccDNA)[11].HBV基因组DNA在复制过程中至少转录5种主要的mRNA,其中pregenomic (pg)RNA,长约3.5 kb,由Core Promoter启动,包含HBV基因组的全部序列,是HBV用作病毒复制的RT模板,并可以翻译Core和Pol蛋庄,长度约为HBV全基因组的1.1倍.形成正确的pgRNA是HBV DNA重组质粒支持体外HBV复制的必要条件[12-13],所以在体外可复制的HBV DNA重组体至少要包含1.1倍以上的HBV基因组,而由于HBV基因组的特殊结构,构建体外可复制重组体比较困难.

图4 转染Huh7细胞后检测上清的HBsAg水平(a)和HBeAg水平(b)和细胞内的颗粒中DNA水平(c)Fig.4 Detection of HBs Ag(a)and HBeAg(b)in supematants 96 h after transfection and the quantity of HBV DNA in cells 96 h after transfection(c)
有效HBV体外感染模型的缺乏,极大地限制了HBV的研究.据文献[14]报道,多数与HBV相关的体外研究都是通过HBV DNA重组质粒转染到肝癌细胞系(如HepG2和Huh7细胞)来进行.而现有的HBV体外重组体基本是通过多步复杂的酶切连接过程对标准病毒株进行片段置换而形成,这些方法会受制于HBV高度异质性[15-16],因此通用性较差,而且,酶切方法具有序列依赖性,对酶切位点有严格的要求,也会造成通用性差,因此难以用于多样本的临床试验研究.
此外,为了防止PCR过程中HBV序列发生变异,影响实验结果,我们应用了高保真性聚合酶PrimeSTAR GXL DNA Polymerase来保证实验结果的准确可靠.相比于r Taq DNA聚合酶,具有高保真性、适合于“热启动”PCR、可用于高GC含量模板等优点,它还配有最佳的缓冲液,对多种的目标物都可进行长链PCR.但是此高保真性聚合酶也有缺点,即其PCR产物为平末端,需要另外加A尾才能进行T克隆的连接,但是本文用的Gibson Assembly的方法避免了T克隆连接这一步,所以用此高保直性聚合酶非常合适.
从本实验的结果也可以看出4份HBs Ag阳性的标本,都能有HBeAg的表达,但是HBsAg与HBeAg没有呈现出明显的相关性.在临床中,不同的病人中HBs Ag和HBe Ag是有差异的,因为不同病人体内感染的HBV的复制表达能力并不相同,同时一些常见突变,例如HBs Ag MHR区突变,前C区/C区基本核心启动子突变等可以显著影响HBs Ag和HBe Ag的分泌.血清中HBsAg与HBeAg的水平有一定的相关性,在临床病例中,一般HBe Ag阳性的病人HBs Ag定量水平相对HBe Ag阴性的病人普遍较高,但HBeAg的定量水平目前由于缺乏临床意义而极少研究,另外由于HBs Ag和HBe Ag由HBV基因组不同的ORF分别编码,所以二者并不是绝对的相关.
4 结 论
本研究从HBV感染者血清中直接提取DNA作为模板,应用分子生物学新技术Gibson Assembly的方法快速构建HBV全基因组1.1倍体,能够尽量避免复杂的酶切和连接过程,并且得到的HBV DNA序列完全来自临床样本病毒株基因组,能够支持HBV体外复制.仅需3个步骤就可以将临床血中的基因组构建为1.1倍基因组克隆,周期仅仅只有2 d.我们采取的这种新型的基于Gibson Assembly的1.1倍基因组克隆构建方法,获得的产物效率高、操作简便、实验周期短,可以作为一种简单快速的方法验证分析临床调取的HBV表型,例如复制能力和病毒抗原表达水平.
[1] Dienstag J L.Hepatitis B virus infection[J].The New England Journal of Medicine,2008,359:1486-1500.
[2] Organization WHO.Hepatitis B[EB/OL].[2014-07-26] http:∥who.int/mediacentre/factsheets/fs204/en/.
[3] 中华人民共和国国家卫生和计划生育委员会.我国控制乙肝成效显著[EB/OL].(2013-07-26)[2014-07-26]http:∥www.nhfpc.gov.cn/jkj/s3582/201307/518216575e544 109b2caca07fca3b430.shtml.
[4] 钦伦秀,孙惠川,汤钊猷.原发性肝癌研究进展——2006沪港国际肝病大会纪要[J].中华外科杂志,2006,44 (15):1070-1074.
[5] 肖开银,彭民浩.原发性肝癌流行病学研究进展[J].中国普外基础与临床杂志,2000,7(4):272-274.
[6] Nassal M,Schaller H.Hepatitis B virus replication[J]. Trends in Microbiology,1993,1:221-228.
[7] Durantel D,Carrouee-Durantel S,Werle-Lapostolle B,et al.A new strategy for studying in vitro the drug susceptibility of clinical isolates of human hepatitis B virus[J]. Hepatology,2004,40(4):855-864.
[8] Gibson D G,Young L,Chuang R Y,et al.Enzymatic assembly of DNA molecules up to several hundred kilobases [J].Nature Methods,2009,6(5):343-345.
[9] Guo H,Jiang D,Zhou T,et al.Characterization of the intracellular deproteinized relaxed circular DNA of hepatitis B virus:an intermediate of covalently closed circular DNA formation[J].Journal of Virology,2007,81:12472-12484.
[10] Yuan Q,Ou S H,Chen C R,et al.Molecular characteristics of occult hepatitis B virus from blood donors in southeast China[J].Journal of Clinical Microbiology, 2010,48:357-364.
[11] Tang H,Mc Lachlan A.A pregenomic RNA sequence adjacent to DR1 and complementary to epsilon influences hepatitis B virus replication efficiency[J].Virology, 2002,303:199-210.
[12] Pollack J R,Ganem D.An RNA stem-loop structure directs hepatitis B virus genomic RNA encapsidation[J]. Journal of Virology,1993,67:3254-3264.
[13] Wang G H,Zoulim F,Leber E H,et al.Role of RNA in enzymatic activity of the reverse transcriptase of hepatitis B viruses[J].Journal of Virology,1994,68: 8437-8442.
[14] Durantel D,Brunelle M N,Gros E,et al.Resistance of human hepatitis B virus to reverse transcriptase inhibitors:from genotypic to phenotypic testing[J].Journal of Clinical Virology:the Official Publication of the Pan A-merican Society for Clinical Virology,2005,34(Sup 1): S34-S43.
[15] Medley G F,Lindop N A,Edmunds W J,et al.Hepatitis-B virus endemicity:heterogeneity,catastrophic dynamics and control[J].Nature Medicine,2001,7:619-624.
[16] de Maddalena C,Giambelli C,Tanzi E,et al.High level of genetic heterogeneity in S and P genes of genotype D hepatitis B virus[J].Virology,2007,365:113-124.
Using Gibson Assembly to Construct Replication-competent 1.1 Copies of Hepatitis B Virus Genome
FANG Lin-lin,SONG Liu-wei,YANG Lin,WU Yong,YUAN Quan*,XIA Ning-shao
(National Institute of Diagnostics and Vaccine Development in Infectious Diseases, School of Public Health,Xiamen University,Xiamen 361101,China)
:Hepatitis B virus(HBV)has a highly compact genetic organization,that is why the replication-competent genome which can fulfill the entire HBV lifecycle in vitro is arranged as an over 100%head to tail DNA genome,at least 1.1 copies of HBV genome.There always concludes several times of restriction enzyme digestion and connection in the traditional construction methods, leading to the steps of previous methods are cumbersome and time consuming.In this report,we develop a novel strategy based on Gibson Assembly and result in a fast and convenient method which just needs one step to complete the construction after HBV DNA obtained.In this method,we successfully constructed 4 1.1-fold-overlength genome plasmids directly extracted from serum specimens and confirmed the plasmid is replication-competent by transfecting in Huh7 cells.This new strategy simplifies the construction procedures and results in a fast and high-efficiency method that is especially suitable to be applied in clinical samples.
hepatitis B virus;Gibson Assembly;1.1 copies
R 373.2+1,Q 785
A
0438-0479(2015)03-0324-07
10.6043/j.issn.0438-0479.2015.03.005
2014-07-26 录用日期:2015-01-04
*通信作者:yuanquan@xmu.edu.cn
方琳琳,宋浏伟,杨林,等.利用Gibson Assembly技术构建乙型肝炎病毒1.1倍体复制子[J].厦门大学学报:自然科学版,2015,54(3):324-330.
:Fang Linlin,Song Liuwei,Yang Lin,et al.Using Gibson Assembly to construct replication-competent 1.1 copies of hepatitis B virus genome[J].Journal of Xiamen University:Natural Science,2015,54(3):324-330.(in Chinese)

