蓝藻遗传多样性的分子生物学研究进展
曹焜,荆瑞勇,王光华,郭永霞
(1.中国科学院东北地理与农业生态研究所黑土区农业生态重点实验室,黑龙江哈尔滨150081; 2.黑龙江八一农垦大学,黑龙江大庆163319)
蓝藻遗传多样性的分子生物学研究进展
曹焜1,2,荆瑞勇1,2,王光华1,郭永霞2
(1.中国科学院东北地理与农业生态研究所黑土区农业生态重点实验室,黑龙江哈尔滨150081; 2.黑龙江八一农垦大学,黑龙江大庆163319)
蓝藻是一类能光合产氧的原核微生物,广泛存在于海洋、湖泊、稻田、沙漠甚至各种极端环境中。研究蓝藻的遗传多样性对了解物种进化和物种资源开发与利用等方面具有重要意义。文章综述了海洋、湖泊和稻田中蓝藻遗传多样性分子生物学研究进展,阐述了不同自然环境中都存在着独特的蓝藻类群及其蓝藻的分布规律,讨论了环境因子对蓝藻遗传多样性的影响,并提出了环境中蓝藻多样性研究中的存在问题及未来研究趋势。图1,表1,参52。
蓝藻多样性;海洋;湖泊;稻田;分子生物学技术
0 引言
蓝藻也称蓝细菌,因其含藻青蛋白而呈现蓝绿色,也被称为蓝绿藻[1]。蓝藻属革兰氏阴性、光合产氧原核微生物,蓝藻细胞比一般细菌大,作为生物圈的初级生产者,大约27亿年前出现在地球上[2-3]。全球蓝藻生物量高达10亿t[4],是生态系统的重要组成部分[5]。蓝藻在地球从无氧到有氧环境的转变过程中起到重要作用[1]。
蓝藻在进化过程中逐步分化成具有固氮作用的异形胞和抗逆性强的原垣孢子[2],使其能够在水生和陆生环境中广泛存在,如海洋[6]、湖泊[7]、稻田[8]、沙漠[9]和极地[10]等。研究发现,不同生态系统中蓝藻多样性存在一定的差异,在同一生态系统中蓝藻多样性也会发生一定程度的时空变化[6-10]。研究蓝藻多样性常采用两种方法,分别为形态特征的显微观察法和分子生物学方法。显微观察法对蓝藻多样性的研究相对粗略,有时,一些蓝藻的鉴定仍需要结合分子生物学方法开展[11]。蓝藻核糖体DNA以及一些功能基因已被广泛用于不同环境中蓝藻多样性的研究,用于核糖体DNA以及一些功能基因PCR扩增的引物有很多,这些引物的扩增范围和目的片段大小存在一定差异,见表1。近年来,采用分子生物学技术解析环境中蓝藻遗传多样性已取得很多成果。为此,文章综述了海洋、湖泊和稻田等环境中蓝藻多样性的研究进展,分析不同分子生物学方法的利弊,为促进蓝藻分子生态学研究提供一些参考信息。
1 海洋环境中蓝藻多样性
海洋中的蓝藻主要由原绿球藻(Prochlorococcus)和聚球藻(Synechococcus)组成。20世纪70和80年代末先后发现海洋中存在大量的聚球藻[12]和原绿球藻[13],这两类蓝藻是海洋生态系统中重要的初级生产者[6],其中原绿球藻被认为是地球上最多的光合产氧微生物[14]。两类蓝藻的生态功能、多样性及时空分布等相关研究一直倍受科学界关注。
Moore等[15-16]和Partensky等[17]根据原绿球藻中叶绿素b与叶绿素a2的比值高低,将原绿球藻分成高含量型原绿球藻(High-B/A)和低含量型原绿球藻(Low-B/A),在较强光照条件下,低含量型原绿球藻能生长,而高含量型原绿球藻则受到抑制[16]。West等[18]根据原绿球藻的16S rDNA序列和对光适应能力,粗略的将原绿球藻分成弱光适应型(Low-Light Adapted,LLA)和强光适应型((High-Light Adapted,HLA)。LLA型与High-B/A型是同一类[19],LLA型主要分布在深层海域,HLA分布在浅层海域,这一规律受到Huang等[20]的研究所证实。到目前为止,已发现海洋中原绿球藻HLA型和LLA型各有6个进化分枝,且HLA型和LLA型间可能存在过渡型,见图1。原绿球藻的分布主要受到水体深度和纬度的影响,从全球范围看主要分布在南纬48°和北纬45°之间[6]。
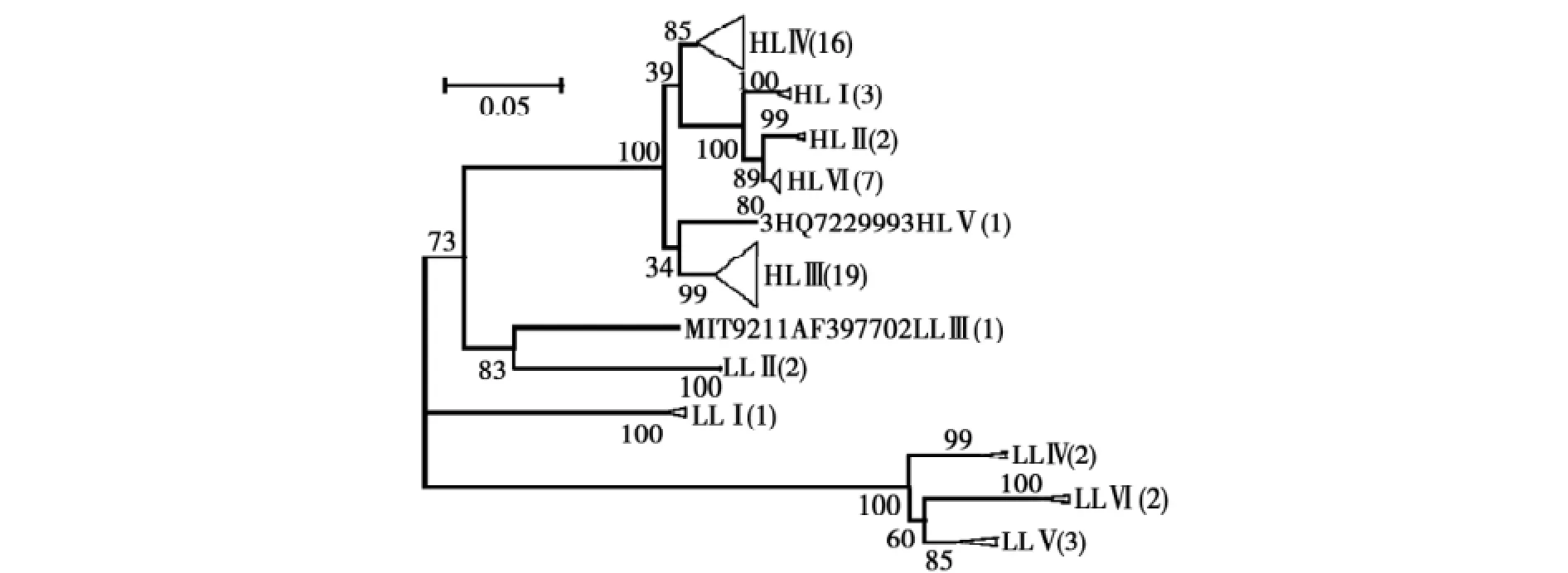
图1 海洋中原绿球藻ITS区基因序列构建的系统发育树Fig.1Phylogenetic tree constructed with ITS sequences from Prochlorococcus in marine
海洋聚球藻遗传多样性较为复杂,Waterbury等[23]基于聚球藻的生境及G+C含量,将聚球藻分成6个分枝,随后Fuller等[22]基于16S rDNA序列将海洋中的聚球藻分成10个进化簇(Clusters I-X),其中,Cluster V的聚球藻遗传多样性很丰富[15],且位于该簇内的聚球藻表型较复杂。Rocap等[24]基于ITS区将分离纯化且位于Cluster V中的25株聚球藻分成6个进化枝(Clades I-VI),其中的三个进化枝与其表型相关,即Clade III与运动型聚球藻相关[25],Clade I和III与颜色适应型聚球藻相关[21],Clade VI与缺乏藻尿胆素型聚球藻相关[24]。由于研究对象的范围狭窄,致使对聚球藻的分析较片面。为了更好的揭示聚球藻的基因多样性与其表型间的关系,学术界对Cluster V的内部分类进行了深入的研究,将其重新界定为2个进化亚簇(subclusters 5.1和5.2)[26]。Huang等[20]于2012年在河口和沿海地区发现一些独特的聚球藻菌群,它们主要分布在Cluster V中,进一步丰富了Cluster V中的亚簇分型,即3个进化亚簇(subclusters 5.1、5.2和5.3),并将Cluster V中的聚球藻分为27个进化分枝(Clades I~XVI,CB1~CB5,5.3 -I~5.3-VI)。聚球藻分布范围比原绿球藻广,主要分布在60°S~50°N之间[27]。在环境影响因素中,温度和光照对蓝藻丰度的影响较大,赤道附近温度高、光照强度大,抑制蓝藻生长,降低蓝藻丰度[27-28],导致其分布呈向两极移动的趋势。
Flombaum等[27]利用海洋聚球藻和原绿球藻的分布情况建立了生态位模型。基于16S rDNA和ITS区分析海洋中蓝藻的遗传多样性及蓝藻进化地位,发现海洋蓝藻可分成不同的进化分枝,这些进化分枝可以代表其对应的生态单元。利用这些生态单元间蓝藻分布与丰度等方面的差异来分析海洋蓝藻与环境间的互作机制,为完善生态位模型奠定基础。
海洋蓝藻多样性的研究不仅局限于原绿球藻和聚球藻,还有其它丝状蓝藻。Thacker等[29]采用引物CYA106F/CYA781Ra/b[30]发现热带海洋中存在林氏藻属(Lyngbya)、束藻属(Symploca)和一些未知的丝状蓝藻。Laamanen等[31]用引物16CITS/23CITS扩增部分间隔区序列(ITS1-S),分析波罗的海中节球藻的多样性,将其划分为两簇,即产节球藻素型和不产节球藻素型,见表1。以上研究表明在海洋生态系统中,除单细胞球状蓝藻外,还存在着人们关注度较少的丝状蓝藻。
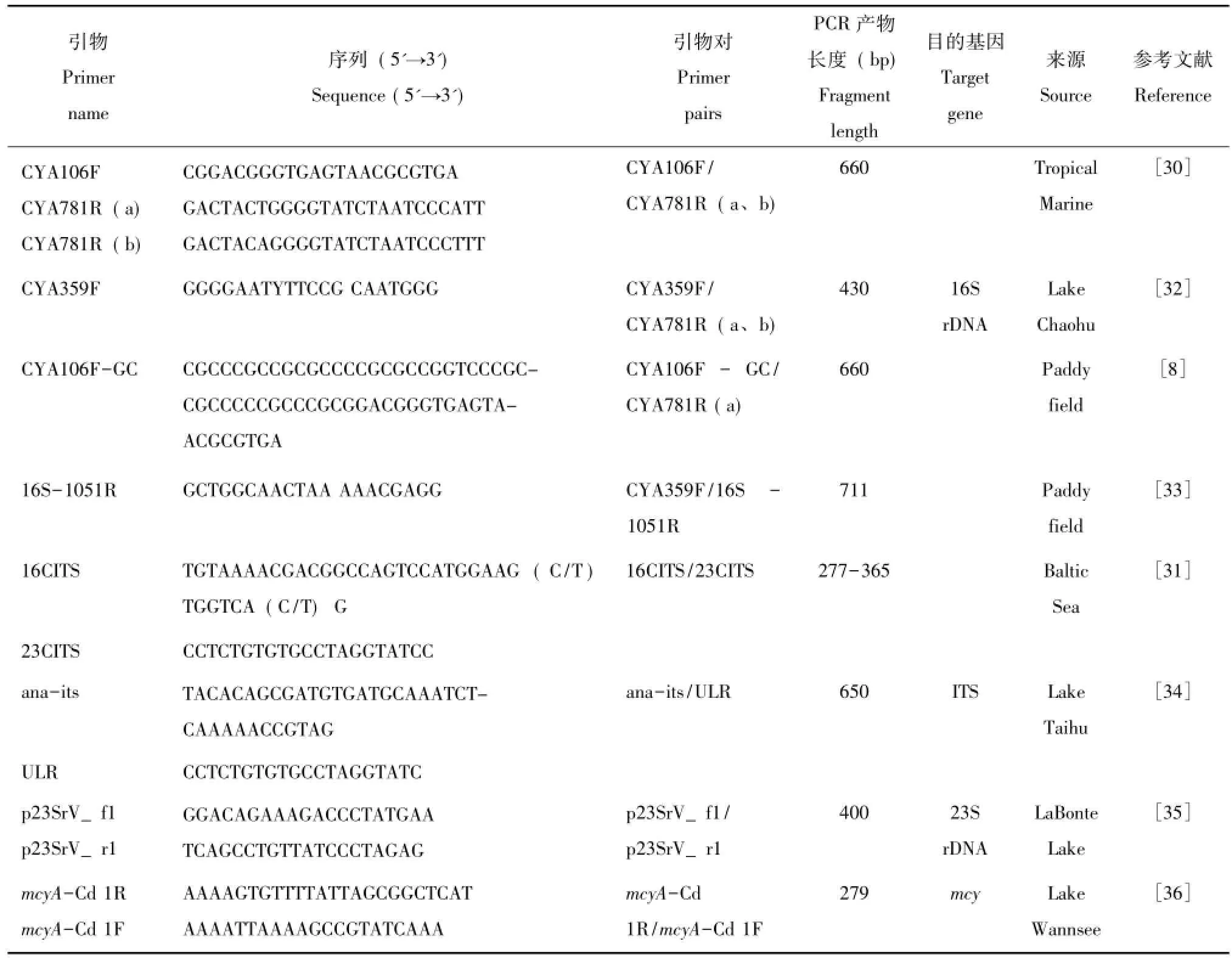
表1 研究蓝藻多样性的PCR引物Tab.1Primer sets for study of cyanobacterial diversity
2 湖泊中蓝藻多样性
湖泊是陆地表面洼地积水形成的比较宽广的水域。近年来,湖泊中藻类大量繁殖的一种自然生态现象(即“水华”)频发,引起人们的重视。蓝藻是引起“水华”的元凶之一,在不同湖泊中蓝藻组成差异较大。如瑞士的苏黎世湖(Lake Zurich)引起“水华”的主要蓝藻有微囊藻(Microcystis)、鱼腥藻(Ana-baena)和浮丝藻(Planktothrix)等[37];加拿大的安大略湖(Lake Ontario)引起“水华”的主要蓝藻有束丝藻(Aphanizomenon)、聚球藻(Synechococcus)和鱼腥藻(Anabaena)等[38];希腊的卡斯托里亚湖(Lakes Kastoria)引起“水华”的主要蓝藻有微囊藻(Microcystis)、鱼腥藻(Anabaena)、束丝藻(Aphanizomenon)和柱胞藻(Cylindrospermopsis)等[39]。可见引起“水华”现象中常见的丝状蓝藻是鱼腥藻,球状蓝藻是微囊藻[37-39],且它们呈现出时间上的规律变化[32,34]。Cai等[32]用引物CYA359F/CYA781Ra/b对巢湖“水华”中蓝藻DNA进行PCR扩增,克隆测序发现鱼腥藻和微囊藻是优势菌群,且随温度变化发生菌群的演替,7月-8月时,微囊藻是优势菌群;而3月、4月、5月和12月时,鱼腥藻是优势菌群。Liu等[34]用引物ana-its/ULR对太湖浮游性鱼腥藻(Dolichospermum)DNA进行PCR扩增并进行了序列测定,发现浮游鱼腥藻的L型和W型是其优势基因型,W型浮游性鱼腥藻在4月-9月丰度较大,且随温度发生从W型到L型的演替。微囊藻素合成酶基因(mcy)是功能基因,可用于分析产毒蓝藻(微囊藻、浮丝藻和鱼腥藻)的多样性,该基因分为10个基因型(mcyA-mcyJ),其中mcyA、mcyD、mcyG和mcyJ是关键基因型[40]。Rinta-Kanto等[36]用引物mcyA-Cd 1R/mcyA-Cd 1F对伊利湖(Lake Erie)中蓝藻DNA进行PCR扩增,克隆测序发现微囊藻和浮丝藻具有空间上的分布差异,并发现一些新的产毒蓝藻。
湖泊中蓝藻除鱼腥藻和微囊藻外,还有一些具有地域特色的蓝藻类群。Taton等[10]用变性梯度凝胶电泳(DGGE)和克隆测序技术解析了南极的弗里克塞尔湖(Lake Fryxell)中蓝藻遗传多样性,发现念珠藻(Nostoc)、节球藻(Nodularia)、颤藻(Oscillatoria)和席藻(Phormidium)是优势蓝藻菌群,其中顶突颤藻(Oscillatoria cf.subproboscidea)是该地区特有的蓝藻;Dorador等[41]用PCR-DGGE和克隆测序技术研究智利高原上的盐湖(Salar de Huasco)中蓝藻遗传多样性,发现颤藻目(Oscillatoriales)、念珠藻目(Nostocales)和色球藻目(Chroococcales)是优势菌群;Rastogi等[42]用直接测序法研究印度王舍城(Rajgir)的温泉中蓝藻遗传多样性,发现蓝杆藻(Cyanothece)、念珠藻(Nostoc)、伪枝藻(Scytonema)和胶须藻(Rivularia)是优势菌群。以上研究表明,在不同湖泊环境中都存在特有的蓝藻类群,但总体来说湖泊中蓝藻的优势菌群为丝状蓝藻,球状蓝藻相对较少[43]。
3 稻田中蓝藻多样性
蓝藻是稻田生态系统中一类重要的微生物类群[33],主要以丝状多细胞态蓝藻为主[8],如念珠藻属(Nostoc)和鱼腥藻属(Anabaena)约占稻田蓝藻菌群的80%[44]。1939年De[45]报道了固氮蓝藻在印度水稻田中繁殖并提供氮肥,在不施肥的情况下,能保证水稻连年保持较高的产量,稻谷增产10%~30%[46],稻田蓝藻的固氮作用受到人们的广泛重视。研究发现念珠藻属、筒孢藻属(Cylindrospermum)等[47]蓝藻具有固氮作用,是潜在的生物氮肥生产者。但蓝藻过量繁殖,尤其是在泡田后30天之内,会影响稻苗的生长,在一定程度上也会引起减产[48]。
在稻田生态系统中,蓝藻种类受到很多因素的影响,如土壤理化性质、水分、光照强度、化肥施用状况及农药等。当土壤pH为中性至弱碱性[48]、稻田处于渍水条件下及光照强度弱时[33],蓝藻多样性较丰富;反之蓝藻多样性会降低。肥料对蓝藻的影响较复杂,在施肥与否的条件下,念珠藻和鱼腥藻都是优势菌群;当施用肥料时,降低具有固氮作用的异形胞生长速率[49]。Kumari等[50]按照10 kg·hm-2~30 kg·hm-2量施用农药丁草胺时,发现低浓度时提高了蓝藻多样性,而高浓度处理降低了蓝藻多样性。
对稻田中蓝藻多样性的研究主要分两部分,即稻田水体和稻田土壤。针对稻田水体中细菌群落结构研究报道很多,但对蓝藻及其多样性进行调查还鲜有报道[51]。稻田土壤中蓝藻多样性的研究主要集中在中国福建省、日本及印度等水稻主产区。2005年Song等[33]用CYA359F/16S-1051R、CYA359F/CYA781R两对引物对中国福建省稻田土壤中蓝藻DNA进行半巢式PCR扩增,用DGGE技术检测,发现在9月时蓝藻多样性达到峰值,且水稻生长季时土壤中蓝藻多样性高于水稻收获后的时期。2008年Asari等[8]用引物CYA106F-GC/CYA781R(a)对日本稻田秸秆上蓝藻菌群进行研究,发现测得的DGGE条带序列归分为5个进化分枝,丝状蓝藻主要分布在III和IV进化枝上。由于CYA781R(a)是丝状蓝藻的特异性引物,该研究未发现秸秆上存在球状蓝藻,进而可能降低了对蓝藻多样性的评估。最近,Wang等[52]对吉林大安碱地稻田单细胞蓝藻16S-23S间ITS基因克隆测序分析发现,有50%左右的克隆与已知序列相似度介于50%~76%之间,并总结认为稻田中存在7个独特单细胞蓝藻类群,表明稻田中生存着一些迄今为止未知的单细胞蓝藻微生物。
4 展望
利用蓝藻的核糖体DNA以及一些功能基因等序列进行蓝藻遗传多样性研究并取得了一定的进展。通过对比发现:16S rDNA是一段耐受环境胁迫的保守序列,一般可鉴定到属以上的分类单位;ITS区虽然存在保守性,与16S rDNA相比受环境胁迫影响相对较大,它能积累比16S rDNA更多的突变位点,可用于亚属或种水平的鉴定;现有的23S rDNA数据库较小,仅能鉴定到门或科[35],只能用于传统方法的补充说明;功能基因适用范围较广,不适合单独用于蓝藻多样性的研究,可作为16S rDNA的补充信息或对特定蓝藻类群的分析,为精确的进行种属鉴定作补充说明。
大量研究发现,不同生态系统中蓝藻多样性存在较大差异,各自都有特有的类群,这表明蓝藻的分布与其所处的环境有一定的关系。上述研究均是基于从环境DNA(eDNA)中扩增出不同蓝藻的基因来进行解析,但到目前为止,采用一些已报道的蓝藻引物,都会扩增出一些非蓝藻序列,所以完全依赖于分子生物学技术研究环境中的蓝藻可能会高估其多样性。故此,应将传统的分离纯化、形态观察和分子生物学的方法进行有效结合,才能更有效地解析环境中蓝藻群落结构组成。
海洋是一个复杂的生态系统,面积大、分布广、受多种因素影响,其中蓝藻的分布问题更为复杂。尽管Flombaum等[27]已经收集了近年来的一些聚球藻和原绿球藻的分布数据及相关的环境变量,建立了蓝藻的生态位模型。可是近几十年来,全球气候变化日趋复杂,海洋中微生物的分布规律日渐模糊。因此,需要在不同地点进行长期定位试验,综合各点试验数据,才能建立出海洋中微生物的生态位模型,为日后进一步预测海洋环境变化和研究生物地球化学循环奠定基础。对湖泊生态系统中蓝藻多样性的研究较为深入,认为“水华”是蓝藻及其相关微生物共同作用的结果。在湖泊中一些新的蓝藻种属不断被发现,蓝藻及其伴生细菌间相互作用关系仍未明确。因此,需将研究重点放在蓝藻菌株分离纯化及其生理特性等方面的研究上;在全球变暖的背景下,调查蓝藻多样性对各种温室气体叠加效应的响应机理。在稻田生态系统中一些有固氮作用的蓝藻菌株不断被发现,但是它的生理特性及其与周围环境间的相互作用机理尚不明晰。因此,需将研究重点放在固氮蓝藻的分离纯化及其生理特性等方面的研究上,为将来开发生物氮肥奠定基础。
[1]Mann N H,Clokie M R.Cyanophage.In:Whitton B A(ed)Ecology of cyanobacteria II:Their diversity in space and time[M].New York: Springer,2012.
[2]Tomitani A,Knoll A H,Cavanaugh C M,et al.The evolutionary diversification of cyanobacteria:molecular–phylogenetic and paleontological perspectives[J].Proceedings of the National Academy of the Sciences of the United States of America,2006,103(14):5442-5447.
[3]Shi T,Falkowski P G.Genome evolution in cyanobacteria:The stable core and the variable shell[J].Proceedings of the National Academy of the Sciences of the United States of America,2008,105(7):2510-2515.
[4]Ferran G P,Jayne B,Susanne N,et al.Estimates of global cyanobacterial biomass and its distribution[J].Algological Studies,2003,109: 213–227.
[5]Wood S A,Rueckert A,Cowan D A,et al.Sources of edaphic cyanobacterial diversity in the dry valleys of eastern Antarctica[J].International Society for Microbial Ecology,2008,2(3):308-320.
[6]Zwirglmaier K,Jardillier L,Ostrowsk M,et al.Global phylogeography of marine Synechococcus and Prochlorococcus reveals a distinct partitioning of lineages among oceanic biomes[J].Environmental Microbiology,2008,10(1):147-161.
[7]Zwart G,Kamst-van Agterveld M P,van der Werff-Staverman I,et al.Molecular characterization of cyanobacterial diversity in a shallow eutrophic lake[J].Environmental Microbiology,2005,7(3):365-377.
[8]Asari N,Ishihara R,Nakajima Y,et al.Cyanobacterial communities of rice straw left on the soil surface of a paddy field[J].Biology and Fertility of Soils,2008,44:605-612.
[9]Garcia-Pichel F,López-Cortés A,Nübel U.Phylogenetic and morphological diversity of cyanobacteria in soil desert crusts from the ColoradoPlateau[J].Applied and Environmental Microbiology,2001,67(4):1902-1910.
[10]Taton A,Grubisic S,Brambilla E,et al.Cyanobacterial diversity in natural and artificial microbial mats of lake fryxell(McMurdo Dry Valleys,Antarctica):a morphological and molecular approach[J].Applied and Environmental Microbiology,2003,69(9):5157-5169.
[11]Wu Z X,Shi J Q,Xiao P,et al.Phylogenetic analysis of two cyanobacterial genera Cylindrospermopsis and Raphidiopsis based on multi-gene sequences[J].Harmful Algae,2011,10(5):419-425.
[12]Waterbury J B,Watson S W,Guillard R R L,et al.Widespread occurrence of a unicellular,marine,planktonic,cyanobacterium[J].Nature,1979,277(5694):293-294.
[13]Chisholm S W,Olson R J,Zettler E R,et al.A novel free-living prochlorophyte abundant in the oceanic euphotic zone[J].Nature,1988,334:340-343.
[14]Scanlan D J,West N J.Molecular ecology of the marine cyanobacterial genera Prochlorococcus and Synechococcus[J].FEMS Microbiology E-cology,2002,40(1):1-12.
[15]Moore L R,Rocap G,Chisholm S W.Physiology and molecular phylogeny of coexisting Prochlorococcus ecotypes[J].Nature,1998,393 (6684):464-467.
[16]Moore L R,Chisholm S W.Photophysiology of the marine cyanobacterium Prochlorococcus:ecotypic differences among cultured isolates[J].Limnology and Oceanography,1999,44(3):628-638.
[17]Partensky F,Hess W R,Vaulot D.Prochlorococcus,a marine photosynthetic prokaryote of global significance[J].Microbiology and Molecular Biology Reviews,1999,63(1):106-127.
[18]West N J,Schonhuber W A,Fuller N J,et al.Closely related Prochlorococcus genotypes show remarkably different depth distributions in two oceanic regions as revealed by in situ hybridization using 16S rRNA-targeted oligonucleotides[J].Microbiology-Sgm,2001,147(7):1731 -1744.
[19]Moore L R,Post A F,Rocap G,et al.Utilization of different nitrogen sources by the marine cyanobacteria Prochlorococcus and Synechococcus[J].Limnology and Oceanography,2002,47(7):989-996.
[20]Huang S J,Wilhelm S W,Harvey H R,et al.Novel lineages of Prochlorococcus and Synechococcus in the global oceans[J].The ISME Journal,2012,6(2):285-297.
[21]Palenik B.Chromatic adaptation in marine Synechococcus strains[J].Applied and Environmental Microbiology,2001,67(2):991-994.
[22]Fuller N J,Marie D,Partensky F,et al.Clade-specific 16S ribosomal DNA oligonucleotides reveal the predominance of a single marine Synechococcus clade throughout a stratified water column in the Red Sea[J].Applied and Environmental Microbiology,2003,69(5):2430-2443.
[23]Waterbury J B,Rippka R.Subsection I.Order Chroococcales Wettstein 1924,emend[M].In:Staley J T,Bryant M P,Pfennig N,eds.Bergey's manual of systematic bacteriology.Baltimore:Williams and Wilkins,1989.
[24]Rocap G,Distel D L,Waterbury J B,et al.Resolution of Prochlorococcus and Synechococcus ecotypes by using 16S-23S ribosomal DNA internal transcribed spacer sequences[J].Applied and Environmental Microbiology,2002,68(3):1180-1191.
[25]Waterbury J B,Watson S W,Valois F W,et al.Biological and ecological characterization of the marine unicellular cyanobacterium Synechococcus[J].Can Bull Fish Aquat Sci,1986,214(71):120.
[26]Herdman M,Castenholz R W,Iteman I,et al.Subsection I(Formerly Chroococcales Wettstein 1924,emend.Rippka,Deruelles,Waterbury,Herdman and Stanier 1979).In:Boone D R,Castenholz R W,Garrity G M,eds.Bergey's Manual of Systematic Bacteriology,2nd ed.,vol.1.The archaea and the deeply branching and phototrophic bacteria[M].New York:Springer Publishers,2001.
[27]Flombaum P,Gallegos J L,Gordillo R A,et al.Present and future global distributions of the marine cyanobacteria Prochlorococcus and Synechococcus[J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(24):9824-9829.
[28]Llabres M,Agusti S.Picophytoplankton cell death induced by UV radiation:Evidence for oceanic Atlantic communities[J].Limnology and Oceanography,2006,51(1):21-29.
[29]Thacker R W,Paul V J.Morphological,chemical,and genetic diversity of tropical marine cyanobacteria Lyngbya spp.and Symploca spp.(Oscillatoriales)[J].Applied and Environmental Microbiology,2004,70(6):3305-3312.
[30]Nübel U,Garcia-Pichel F,Muyzer G.PCR primers to amplify 16S rRNA genes from cyanobacteria[J].Applied and Environmental Microbiology,1997,63(8):3327-3332.
[31]Laamanen M J,Gugger M F,Lehtimaki J M,et al.Diversity of toxic and nontoxic nodularia isolates(cyanobacteria)and filaments from the Baltic Sea[J].Applied and Environmental Microbiology,2001,67(10):4638-4647.
[32]Cai Y F,Kong F X.Diversity and dynamics of picocyanobacteria and bloom-forming cyanobacteria in a large shallow eutrophic lake(lake Chaohu,China)[J].Journal of Limnology,2013,72(3):473-484.
[33]Song T,Martensson L,Eriksson T,et al.Biodiversity and seasonal variation of the cyanobacterial assemblage in rice paddy field in Fujian,China[J].FEMS Microbiology Ecology,2005,54(1):131-140.
[34]Liu Y,Xu Y,Xiao P,et al.Genetic analysis on Dolichospermum(Cyanobacteria;sensu Anabaena)populations based on the culture-independent clone libraries revealed the dominant genotypes existing in Lake Taihu,China[J].Harmful Algae,2014,31:76-81.
[35]Steven B,McCann S,Ward N L.Pyrosequencing of plastid 23S rRNA genes reveals diverse and dynamic cyanobacterial and algal populationsin two eutrophic lakes[J].FEMS Microbiology Ecology,2012,83(2):607-615.
[36]Rinta-Kanto J M,Wilhelm S W.Diversity of microcystin-producing cyanobacteria in spatially isolated regions of Lake Erie[J].Applied and Environmental Microbiology,2006,72(7):5083-5085.
[37]Deng L,Hayes P K.Evidence for cyanophages active against bloom-forming freshwater cyanobacteria[J].Freshwater Biology,2008,53 (6):1240-1252.
[38]Molot L A,Li G Y,Findlay D L,et al.Iron-mediated suppression of bloom-forming cyanobacteria by oxine in a eutrophic lake[J].Freshwater Biology,2010,55(5):1102-1117.
[39]Kormas K A,Gkelis S,Vardaka E,et al.Morphological and molecular analysis of bloom-forming cyanobacteria in two eutrophic,shallow Mediterranean lakes[J].Limnologica-Ecology and Management of Inland Waters,2011,41(3):167-173.
[40]Tanabe Y,Kaya K,Watanabe M M.Evidence for recombination in the microcystin synthetase(mcy)genes of toxic cyanobacteria Microcystis spp[J].Journal of Molecular Evolution,2004,58(6):633-641.
[41]Dorador C,Vila I,Imhoff J F,et al.Cyanobacterial diversity in Salar de Huasco,a high altitude saline wetland in northern Chile:an example of geographical dispersion?[J].FEMS Microbiology Ecology,2008,64(3):419-432.
[42]Rastogi R P,Kumari S,Richa,et al.Molecular characterization of hot spring cyanobacteria and evaluation of their photoprotective compounds[J].Canadian Journal of Microbiology,2012,58(6):719-727.
[43]Scheffer M,Rinaldi S,Gragnani A,et al.On the dominance of filamentous cyanobacteria in shallow,turbid lakes[J].Ecology,1997,78 (1):272-282.
[44]Prasanna R,Jaiswal P,Nayak S,et al.Cyanobacterial diversity in the rhizosphere of rice and its ecological significance[J].Indian Journal of Microbiology,2009,49(1):89-97.
[45]De P K.The role of blue-green algae in nitrogen fixation in rice-field[J].Proceedings of the Royal Society of London Series B,1939,127: 121-139.
[46]沈银武,黎尚豪.固氮蓝藻培养和应用的结果与展望[J].水生生物学报,1993,17(4):357-364.
[47]Kumari N,Narayan O P,Rai L C.Understanding butachlor toxicity in Aulosira fertilissima using physiological,biochemical and proteomic approaches[J].Chemosphere,2009,77(11):1501-1507.
[48]Nayak S,Prasanna R.Soil pH and its role in cyanobacterial abundance and diversity in rice field soils[J].Applied Ecology and Environmental Research,2007,5(2):103-113.
[49]Choudhary K K,Bimal R.Distribution of nitrogen-fixing cyanobacteria(Nostocaceae)during rice cultivation in fertilized and unfertilized paddy fields[J].Nordic Journal of Botany,2010,28(1):100-103.
[50]Kumari N,Narayan O P,Rai L C.Cyanobacterial diversity shifts induced by butachlor in selected indian rice fields in Eastern Uttar Pradesh and Western Bihar analyzed with PCR and DGGE[J].Journal of Microbiology and Biotechnology,2012,22(1):1-12.
[51]Shibagaki-Shimizu T,Nakayama N,Nakajima Y,et al.Phylogenetic study on a bacterial community in the floodwater of a Japanese paddy field estimated by sequencing 16S rDNA fragments after denaturing gradient gel electrophoresis[J].Biology and Fertility of Soils,2006,42 (4):362-365.
[52]Wang G H,Liu J J,Yu Z H,et al.Unique distribution of cyanobacterial podoviruses and their potential hosts in a paddy field of Northeast China[J].FEMS Microbiology Ecology,2014,90(1):331-334.
Molecular Biological Research Progress in Genetic Diversity of Cyanobacteria
CAO Kun1,2,JING Rui-yong1,2,WANG Guang-hua1,GUO Yong-xia2
(1.Key Laboratory of Mollisols Agroecology,Northeast Institute of Geography and Agroecology,CAS,Harbin 150081,China; 2.Heilongjiang BaYi Agricultural University,Daqing 163319,China)
Cyanobacteria are a kind of photosynthetic and oxygen-producing prokaryotic microorganisms which are widely spread in ocean,lake,paddy field,desert and even in some extremely adverse environments.Studying the genetic diversity of cyanobacteria is very important in understanding species evolution,exploitation and utilization of resources.In this paper,we reviewed the research progress on genetic diversity of cyanobacteria in marine,lake and paddy field based on molecular biological techniques.The distribution patterns of cyanobacteria and several unique cyanobacterial groups exist in different environments were summarized.Meanwhile,the effect of environmental factors on cyanobacterial diversity was discussed and several research problems and future research tendencies of cyanobacterial diversity study were proposed.
cyanobacterial diversity;marine;lake;paddy field;molecular biological technique
S182;Q-9
A
10.11689/j.issn.2095-2961.2015.04.007
2095-2961(2015)04-0183-07
2015-04-02;
2015-05-17.
国家自然科学基金(41271262,31300425);中国科学院"百人计划"项目.
曹焜(1989-),男,黑龙江鹤岗人,在读硕士,研究方向为农药环境毒理.
王光华(1966-),男,黑龙江海林人,博士,研究员,主要从事微生物生态研究.
郭永霞(1970-),女,山东胶南人,博士,教授,主要从事植物保护教学及科研.

