十种臂尾轮虫12S rDNA基因序列的PCR扩增及分析
程双怀, 金思宇, 陈款民, 孙道琴, 魏世祥, 项贤领
(安徽师范大学 生命科学学院、安徽省高校生物环境与生态安全省级重点实验室、重要生物资源保护和利用研究安徽省重点实验室,安徽 芜湖 241000)
十种臂尾轮虫12S rDNA基因序列的PCR扩增及分析
程双怀, 金思宇, 陈款民, 孙道琴, 魏世祥, 项贤领
(安徽师范大学 生命科学学院、安徽省高校生物环境与生态安全省级重点实验室、重要生物资源保护和利用研究安徽省重点实验室,安徽 芜湖 241000)
通过对角突臂尾轮虫、尾突臂尾轮虫、裂足臂尾轮虫、剪形臂尾轮虫、方形臂尾轮虫、壶状臂尾轮虫、红臂尾轮虫、镰形臂尾轮虫、萼花臂尾轮虫和十指臂尾轮虫等十种臂尾轮虫和矩形龟甲轮虫的12S rDNA基因进行扩增和序列测序,结合Genbank中两种褶皱臂尾轮虫的125rDNA序列,使用MEGA软件构建这13种轮虫系统发生树(NJ树),探讨了12种臂尾轮虫之间的系统关系.结果表明:本研究所涉及的轮虫12S rDNA序列差异百分比均值为34.41%,可作为分子标记应用于轮虫属内种间系统关系研究;系统树支持将十指臂尾轮虫、裂足臂尾轮虫归属于臂尾轮属;壶状臂尾轮虫和红臂尾轮虫是两个独立的种.
轮虫;臂尾轮属;12S rDNA;系统发生
轮虫隶属轮虫动物门(Rotifera),约2000多种.早期的轮虫分类及系统关系研究主要依据轮虫的形态学特征.随着分子生物学技术的发展和DNA测序费用的降低,分子分析手段被逐渐应用到轮虫的分类和系统关系研究中.12S rDNA为线粒体核糖体小亚基rRNA的编码序列.12S rDNA序列比较保守,但也具有变异区域,可以用于属内种间或种内的系统发生关系研究,也可以用于种或地理品系的鉴定.作为分子标记,12S rDNA已经被广泛用于一些物种的系统发生关系研究中[1-3].
臂尾轮属隶属于臂尾轮科,属中许多种类广泛分布于各类水体,大多数为优势种.本属目前已发现34种,我国已记载的有10多种,但常见的不到10种[4].属中壶状臂尾轮虫(B.urceolaris)、红臂尾轮虫(B.rubens)、十指臂尾轮虫(B.patulus)、裂足臂尾轮虫(B.diversicornis)和褶皱臂尾轮虫(B.plicatilis)等在种类划分和系统发生上一直存有争议[5-10].本研究扩增并测定臂尾轮属十个常见种类的12SrDNA部分序列,通过建立系统树探讨臂尾轮虫的系统发生关系,试图解决一些传统分类学上的争议和物种的分类地位问题;也为轮虫大规模培养时,物种的分子鉴定提供参考.
1 材料与方法
1.1 实验材料
研究用角突臂尾轮虫(B.angularis)、尾突臂尾轮虫(B.caudatus)、裂足臂尾轮虫、剪形臂尾轮虫(B.forficula)、方形臂尾轮虫(B.quadridentatus)、壶状臂尾轮虫、红臂尾轮虫、镰形臂尾轮虫(B.falcatus)、萼花臂尾轮虫(B.calyciflorus)、十指臂尾轮虫和矩形龟甲轮虫(Keratellaquadrata)分别采自芜湖市镜湖、汀棠湖、凤鸣湖和莲塘等水体(表1).除裂足臂尾轮虫以外的9种臂尾轮虫和晶囊轮虫采集后于实验室内在25±1℃、自然光照(光照强度约130lx,L∶D=14∶10)条件下进行“克隆”培养.轮虫培养液采用[11]配方(pH=7.3),臂尾轮虫以HB-4培养基[4]培养的、处于指数增长期的斜生栅藻(Scenedesmusobliquus)为饵料,晶囊轮虫用角突臂尾轮虫为饵料,当各“克隆”轮虫的个体数达100只以上时用轮虫培养液过滤冲洗,饥饿72h后用灭菌双蒸水冲洗,Eppendorf管收集,-20℃保存备用.裂足臂尾轮虫难以培养,直接从野外水体中采集、挑取并纯化,经上述方法处理后-20℃保存备用.
1.2 总DNA提取
用玻璃粉法提取基因组DNA[12].具体方法为:离心获得轮虫样品,ddH2O冲洗2次,置1.5ml Eppendorf管中,加500μl DNA提取缓冲液(0.5%SDS,25mM EDTA,25mmol/L NaCl,100mmol/L Tris-HCl,pH8.0)和20μl 蛋白酶K(20μg/ml),置60℃水浴2h;加500μl 8M预热后的硫氰酸胍和40μl(1∶1)的洁净玻璃粉乳液,混匀后置37℃水浴1h,不时摇动;取出,4000rpm离心2min,弃去上清液;沉淀用70%冰乙醇洗2次,再用丙酮洗1次;置真空干燥机中干燥.加TE(pH8.0)40μl,置56℃水浴30min,取出,8000rpm离心2min,吸上清液,置-20℃保存备用.
1.3 PCR扩增及序列测定

表1 13个样本的核苷酸序列来源Table 1 The origins of nucleotide sequences of 13 samples
扩增反应在MJ Research 公司生产的PTC-100TM扩增仪上进行.参照已经报道的褶皱臂尾轮虫线粒体全序列设计合成PCR引物:Br12S F:CACAAAAGACAAGGATTAG; Br12S P:GTAACAGAGGTGACGGGATTTGTAC,于上海Invitrogen公司合成.其他试剂均购于上海生工生物工程技术服务有限公司.PCR反应体系的总体积为25μl,包括1×Buffer,0.2mmol/L dNTP,1.5μmol/L引物,2mmol/L Mg2+,DNA模板2μl,1U的Taq酶,超纯水补至25μl.扩增反应程序如下:94℃预变性5min;94℃变性1min,53℃退火1.5min,72℃延伸1min,共35个循环;最后72℃延伸10min,4℃保存10h,终止反应.扩增完成后,PCR产物用乙醇沉淀或DPS系统纯化,沉淀凉干后加适量TE溶解,取2μl 于1%的琼脂糖凝胶电泳上检查,其余-20℃保存备用.将纯化产物与质粒载体连接,质粒载体为pMD 18-T vector,按《分子克隆实验指南(第2版)》[13]进行操作.用Qiagen公司的质粒提取试剂盒提取所要的克隆子质粒DNA,ABI PRISM 377型自动测序仪进行序列测定.
1.3 系统发生分析
对测序结果进行处理并分析,序列编辑使用CLUSTAL X(1.81)软件[14]进行DNA序列比对;用DNASTAR软件计算两两序列的序列差异百分比;再用DnaSP 3.5软件[15]分析变异位点;用MEGA(version 3.1)软件计算不同序列间转换/颠换数;采用DAMBE软件进行基因替换的饱和性分析.用邻接法(neighbor-joining method,NJ)重建系统发生树.NJ树构建使用MEGA软件,采用P-distance距离模型;系统树各结点的支持率以序列数据集100次重复抽样检验的自引导值(bootstrap value)表示.
2 结果
对本文测序的13个轮虫样品12S rDNA核苷酸序列分析显示,已测定的各种轮虫12S rDNA序列长度并不一致,最长的为矩形龟甲轮虫(475bp),最短的是壶状臂尾轮虫(332bp).12S rDNA基因序列中,A、C、G、T碱基的平均含量分别为 31.0%、 13.1%、 17.2%和38.7%,其中A+T含量(69.7%)较丰富,明显大于C+G含量(30.3%).经比对,339个位点中共有86个保守位点,占25.4%;251个变异位点(表2),占74.0%;单一位点114个,简约信息位点135个.用MEGA(version3.1)软件分析碱基的转换/颠换数,转换与颠换之比平均为0.928.用DNASTAR软件分析发现序列差异百分比最大值为79.8,最小值为2.5,平均值为34.41(表3).从遗传距离及转换、颠换数散点饱和图(图1)中可以看出,颠换数高于转换数,随着遗传距离的增加,转换和颠换数均伴有线性增加,未出现饱和现象.
2.3 系统发生树
根据所测序列用MEGA分析软件,基于P-distance距离模型,以矩形龟甲轮虫为外群,采用邻接法(NJ法)构建了13个样品的系统发生树(图2),树节点支持率较高.

表2 13个样本12S rDNA基因的部分核苷酸序列变异位点Table 2 Variable sites of partical 12S rDNA nucleotide sequences in thirteen samples
续表2

物种名称序 列222222222222222222222222222222222222233333333333333333333333335555556666666666777777788888999999999000001111111112222223333345678901234567890123469135890123567890123601234678901246835678B.angularisTTAGTTTACTACTTAATTCGGTGAGATATTTTGTTTATATAGTTCCTCTCTTTAGAA⁃CCGTB.calyciflorus.C.A.A.TAG⁃....................C..AA.......C.TCT.TC......CGTACB.caudatusCC.AAA.TAGG.................⁃..CA.AA.....A..TA.T.TC......⁃....B.diversicornis..TA....AA.T..T....C.AT.AT.T....TAA.T..⁃.T.....T.T...TATC⁃..C.B.falcatusA..T.AC.AA.......CT..CA...CT...A..AC...AG...TGCT.TC......CGTACB.forficulaA..T.AC.AA.......CT..CA...CT...A..AC...AG...TGCT.TC......G....B.koreanus...A.ACGGA⁃TC.....T...A....G⁃......AG..C...CA.ATCT.C.....G....B.patulus...A.A..AG⁃TC.....T....T.......A..A.G...G....TCTCT.......⁃....B.plicatilis...AAA..GG⁃.......T........G...G..AAGC.C..C.TTA....C.....G....B.quadridentatus.....A.GAG⁃T......T............G..AA.......CT.CT.TC......G....B.rubens...A..C.AG⁃.C.....T.......C.C.....AA..G.G..GATCT.T.......G....B.urceolaris...A.AA.AA⁃TC.....T............G..AAG...T..CTACT.TC......⁃....K.quadrata...AAAAT.A⁃GCC.TCAA.AC....C⁃.CC.....C.GGG..CATC..T.CCT...⁃....
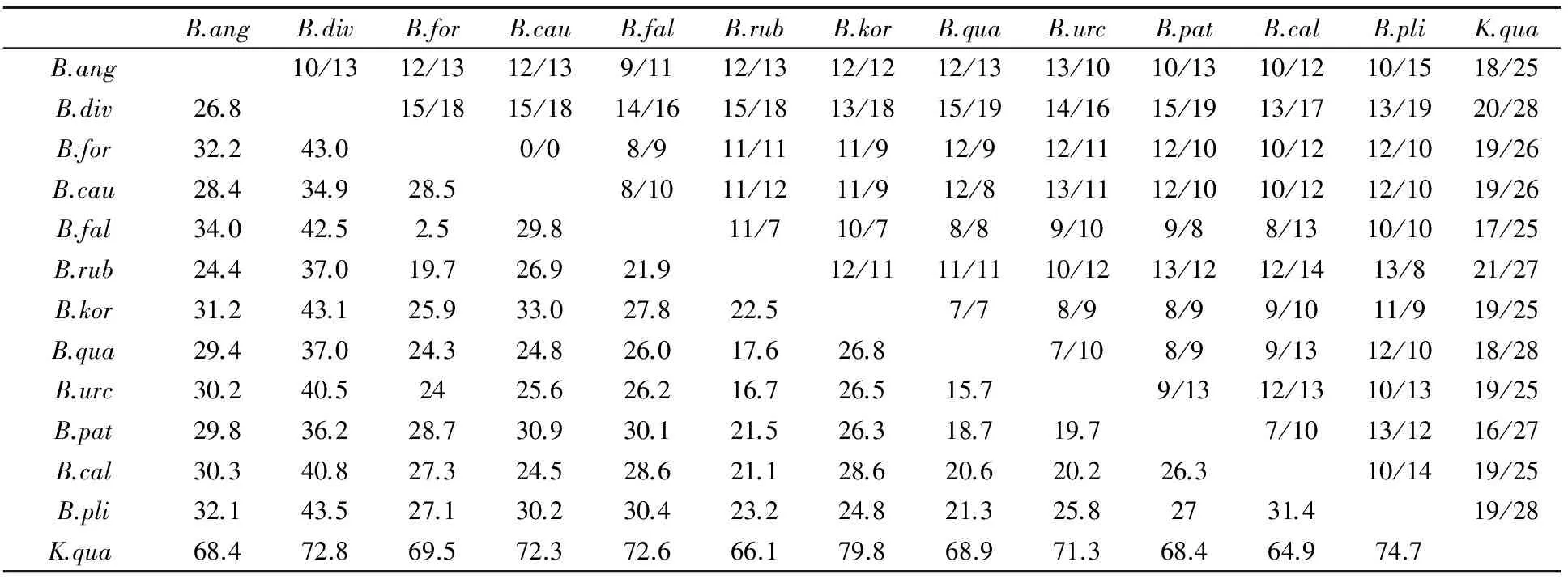
表3 13个样品12S rDNA基因序列转换/颠换数(对角线上)及差异百分比(对角线下)Table 3 Numbers of transitions/transvertions (above diagonal) and percentage of divergence (below diagonal) for 12S rDNA gene sequences in 13 samples
3 讨论
本研究中,轮虫13个样品的12S rDNA基因序列之间差异百分比最大值为79.8,最小值为2.5,平均值为34.41,因此12S rDNA基因序列可以作为分子标记应用于臂尾轮虫的分子分类和分子系统进化研究.基于12S rDNA基因序列构建的系统树能够反映臂尾轮虫种间的亲缘关系.方形臂尾轮虫和壶状臂尾轮虫都是附着型生活方式,在构建的NJ系统进化树中它们构成单系群.镰形臂尾轮虫和剪形臂尾轮虫都具有“八字形”后棘刺,NJ系统进化树中它们也构成单系群,说明两个物种的亲缘关系较近.在臂尾轮虫之间的12S rDNA序列差异百分比中,镰形臂尾轮虫和剪形臂尾轮虫为最小值为2.5,同样支持上述结论.此结果与Xiang等[16]利用ITS序列、程双怀等[17,18]利用COⅠ序列、28S rDNA所得出的研究结果基本一致.
Turner[7]和Segers等[8]通过分析比较平甲轮属(Platyias)、臂尾轮属和十指臂尾轮虫咀嚼器的超微结构,将十指臂尾轮虫立为一个新属——扁甲轮属(Plationus),而传统的分类学方法将它置于臂尾轮属中[19-21].本研究构建的NJ系统进化树中十指臂尾轮虫和方形臂尾轮虫与壶状臂尾轮虫聚在一起,而且结点自展检验值较高,为82,由此推断十指臂尾轮虫应隶属于臂尾轮属,且与方形臂尾轮虫和壶状臂尾轮虫的亲缘关系最近,此结果与Xiang[16]等利用ITS序列、程双怀[17,18]等利用线粒体基因COⅠ序列、核基因28SrDNA得出的研究结果不同,而与程双怀[22]等利用16SrDNA以及Wulfert和Koste应用传统的分类学方法所得到的研究结果一致,不支持Turner[7]和Segers等[8]将它另立新属的观点.因此十指臂尾轮虫的分类地位在分子水平的分析结果也不一致,需要更多的数据进一步研究.
壶状臂尾轮虫和红臂尾轮虫具有相似的形态,早期的研究曾将这二者归为一种,称为B.urceus,也译为壶状臂尾轮虫[5];后来Koste[20]、章宗涉和黄祥飞[4]根据虫体前端棘刺的两侧是否对称和眼点的形状不同把B.urceus分为B.urceolaris和B.rubens两种.在本研究中,壶状臂尾轮虫和红臂尾轮虫12SrDNA基因序列差异百分比高达16.7%,远远大于镰形臂尾轮虫和剪形臂尾轮虫之间序列差异百分比(2.5%),因此可以判断两者应分别为独立的、不同的种.此结论支持程双怀等利用COⅠ序列[17]、28SrDNA[18]和16SrDNA[22]研究的结果.
裂足臂尾轮虫具有臂尾轮属的一些显著特征,所以有的轮虫学家将它归属于臂尾轮属[20,4].但是它的形态又具有特殊性,如它的被甲比较长,即使不包括两端的棘刺在内,长度也大于宽度.王家楫[5]依据它的足的结构与其他臂尾轮虫不同,将它另立一属——裂足轮属(Schizocerca).本研究中系统树支持裂足臂尾轮虫隶属于臂尾轮属.此结果支持鲍蕾等[6]和程双怀等利用COⅠ[20]、利用28SrDNA[18]和16SrDNA[22]研究的结果.
在构建的系统进化树中,海产的褶皱臂尾轮虫位于淡水臂尾轮虫之间,支持海产轮虫来源于淡水轮虫,但结点自展检验值较低,可能是由于所用序列较短.因此,关于海产轮虫的起源与演化需要进一步研究.
[1] 罗育发,颜亨梅.由12SrDNA序列探讨中国狼蛛科主要类群的分子系统发育关系. 四川动物[J].2006,25(3):445-450.
[2] 张旭,潘景芝,刘福杰,等.香蒲拟发网菌12S rDNA的PCR扩增及序列分析.东北林业大学学报[J].2011,39(7):96-98.
[3] 陈冬,柏凡,周明亮,张翔宇,吴登俊.基于线粒体12S rRNA基因鉴别混合牛肉及制品的牛种来源,遗传[J].2008,30(8):1008-1014.
[4] 章宗涉,黄祥飞.淡水浮游生物研究方法[M].北京:科学出版社,1991:269.
[5] 王家楫.中国淡水轮虫志[M].北京:科学出版社,1961:69-77.
[6] 鲍蕾,牛翠娟,马蕊,李庆芬.裂足轮虫还是裂足臂尾轮虫[J].北京师范大学学报:自然科学版,2003,39(3):397-401.
[7] TURNER P. The rotifer genusplatyiasharring(1913)intheneotropica[J]. Acta Limnol Brasil, 1990,3:741-756.
[8] SEGERS H, MURUGAN G, DUMONT H J. On the taxonomy of the Brachionidae: description ofPlationusn.gen. (Rotifera,Monogononta)[J]. Hydrobiologia, 1993,268:1-8.
[9] HAGIWARA A, KOTANI T, SNELL T W. et al. Morphology, reproduction, genetics, and mating behavior of small, tropical marineBrachionusstrains(Rotifera).J.Exp.Mar.Biol.Ecol[J], 1995,194:25-37.
[10] Ciros-Perez, J. Gomez, A. and Serra, M. On the taxonomy of three sympatric species of theBrachionusplicatilis(Rotifera)complexfromSpain,withthedescriptionofB.ibericusn.sp.,J.PlanktonRes[J], 2001,23:1311-1328.
[11] GILBERT J J. Mictic female production in rotiferBrachionuscalyciflorus.JExpZool[J], 1963,153:113-124.
[12] HAO J-S, YANG Q, LI C-X, ZHANG K-Y, SUN X-Y. Molecular phylogeny of Lophotrochozoa based on 18S rRNA gene sequences-with comment on the phylogenetic position of Bryozoa. J. Genetics and Molecular Biology[J], 2003,14(2):64-72.
[13] 分子克隆实验指南[M].第2版.金冬雁,黎孟枫,译.北京:科学出版社,1993:627.
[14] THOMPSON J D, HIGGINS D G, GIBSON T J. CLUSTAL X Multiple Sequence Alignment Program, Version,1999:1.8.
[15] ROZAS J, SANCHEZ-DELBARRIO J C, MESSEGUER X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods. Bioinformatics[J], 2003,19:2496-2497.
[16] XIANG X-L, XI Y-L, HU H-Y. Phylogenetic relationships ofBrachionusrotifersbasedonrDNAITS1genesequences.ActaZoologicaSinica[J], 2006,52(6):1067-1074.
[17] 程双怀,席贻龙,项贤领,胡好远. 从线粒体COⅠ基因序列探讨臂尾轮属的系统发生关系[J]. 动物分类学报,2007,32(2):328-334.
[18] 程双怀,席贻龙,项贤领,胡好远.基于28S rDNA基因序列研究十种臂尾轮虫的系统关系和分类地位[J].动物分类学报,2007,32(3):599-605.
[19] WULFERT K. Revision der Rotatorien—Gattung Platyias Harring 1913[J]. Limnologica, 1965,3:41-46.
[20] KOSTE W. Rotatoria[M]. Die Radertiere Mitteleuropas. Borntraeger, Berlin,1978,75-79.
[21] KOSTE W, SHIEL R J. Rotifera from Australian inland waters. II. Epiphanidae and Brachionidae (Rotifera: Monogononta)[J]. Invertebr Taxon. 1987,7:959-1021.
[22] 程双怀,席贻龙.基于16S rDNA序列探讨十种臂尾轮虫的系统关系和分类地位[J].动物分类学报,2009,34(4):934-940.
PCR Amplication and Sequence Analysis of Partial 12S rDNA Gene From TenBrachionusSpecies (Rotifera)
CHENG Shuang-huai, JIN Si-yu, CHEN Kuan-min, SUN Dao-qin, WEI Shi-xiang, XIANG Xian-ling
(College of Lofe Sciences, Provincial Key Laboratory of Biotic Environment and Ecological Safety in Anhui, Anhui Provincial Key Lab of the Conservation and Exploitation of Biological Resources, Anhui Normal University, Wuhu 241000, China)
Partial sequences of 12S rDNA gene fromBrachionusangularis,B.caudatus,B.diversicornis,B.forficula,B.quadridentatus,B.urceolaris,B.rubens,B.falcatus,B.calyciflorus,B.patulusandKeratellaquadratawereamplifiedandsequenced.Combinedwith12srDNAofB.plicatilisandB.koreanusfromGenbank,phylogenetictreewerebuiltbasedonneighbor-joining(NJ)methodwithMEGAsoftware,aimingtorevealtheirphylogeneticrelationshipsandresolvesometaxonomicissues.Theresultsshowedthatthepercentageofdivergencefor12SrDNAgenesequencesin13sampleswasaveraged34.41%.Thus,the12SrDNAgenewassuitableforexploringthephylogenyandevolutioninrotifera.TheNJtreesupportedthatB.patulusandB.diversicornisbelongtothegenusBrachionus,whileB.urceolarisandB.rubensaretwopotentialspecies.
rotifera;Brachionus; 12SrDNA;phylogeny
10.14182/J.cnki.1001-2443.2015.05.011
2015-05-25
国家自然科学基金(31200324);安徽省自然科学基金(1208085QC47);国家级大学生校外实践教育基地大学生创新训练计划项目.
程双怀(1973-),男,安徽怀宁人,副教授,博士,主要研究方向:轮虫分子生物学.
程双怀,金思宇,陈款民,等.十种臂尾轮虫125rDNA基因序列的PCR扩增及分析[J].安徽师范大学学报:自然科学版,2015,38(5):460-465.
Q951.3
A
1001-2443(2015)05-0460-06

