两栖类壶菌病3种PCR鉴定方法与比较
李斌 黄艳 王宇 邵晨
摘要 壶菌病是一种新型的由蛙壶菌感染所引起的两栖动物传染疾病,其快速的传染性和广泛的暴发性导致两栖类物种的大面积灭绝,对全球两栖动物的生存产生了巨大影响。介绍了3种鉴定蛙壶菌的PCR方法:常规PCR、巢式PCR和实时定量PCR,并对这3种方法进行分析与比较。
关键词 壶菌病;蛙壶菌;两栖动物;PCR
中图分类号 S188 文献标识码A 文章编号 0517-6611(2015)11-032-03
蛙壶菌(Batrachochytrium dendrobatidis)是壶菌门(Chytridiomycota)、壶菌纲( Chytridiomycetes)、壶菌目(Chytridiales)的一个新属(Batrachochytrium),且蛙壶菌是该属的唯一物种[1]。蛙壶菌可在4~25 ℃的温度下生长,最适温度17~25 ℃,它们可以在寄主身上过冬。但蛙壶菌对热较敏感,在25 ℃以上的环境中生长较差,若高于28 ℃,生长就会停止,且在37 ℃加热4 h或47 ℃加热30 min或60 ℃加热5 min 致死率将达到100%[2]。蛙壶菌具有快速的传染性和广泛的暴发性,但同时其传染性和暴发性具有明显的季节性和地域性。壶菌病在冬季暴发率较高,且多发生在高海拔地区和某些热带雨林山区[3]。蛙壶菌主要寄生于无尾两栖类成体皮肤和蝌蚪口器中,通过寄主生活的水体进行传播,患病的蛙和蝌蚪携带的蛙壶菌游动孢子会随表皮角质层脱落而被释放到水中,健康蛙和蝌蚪则会因接触含蛙壶菌游动孢子的水而感染壶菌病[4]。蛙壶菌主要通过影响蛙体皮肤的功能来影响其机体的生理机能,皮肤对两栖动物保持物质转运和电解质平衡具有重要作用,个体感染蛙壶菌后,依赖皮肤的电解质转运被抑制率达50%以上,血浆钠、钾浓度分别减少了20%和50%,严重影响了心肌功能,从而导致心跳骤停而死亡[5]。
壶菌病广泛性暴发是导致全球两栖类种群数量急剧下降的主要原因之一。研究发现此病自1978年已经遍布整个澳洲,目前非洲、南美洲、北美洲、欧洲和亚洲均发现两栖动物感染蛙壶菌[1,6-16]。自2010年国内首次报道在云南的滇蛙(Rana pleuraden)、牛蛙(Rana catesbeiana)、云南臭蛙(Odorrana andersonii)、昭觉林蛙(Rana chaochiaoensis)中发现蛙壶菌感染以来,四川、湖北、贵州、湖南、浙江、北京、天津、河北的石家庄以及河南的郑州在野外均发现蛙壶菌的分布[15-16]。同时,研究表明在20世纪八九十年代的国内馆藏标本中也检测到蛙壶菌的DNA分子[17-18]。说明我国两栖类蛙壶菌的感染可能要追溯到20世纪八九十年代,甚至更早。目前,壶菌病检测主要依赖组织学、免疫组织化学以及PCR等方法,但组织学和免疫组织化学法对壶菌病感染早期不敏感,且对蛙体的正常生存具有一定影响。用常规PCR或改进的巢式PCR和实时定量PCR技术,灵敏性和特异性相对较高,有助于在饲养和野生的两栖动物种群中及早发现蛙壶菌[19-20]。笔者对常规PCR、巢式PCR和实时定量PCR在蛙壶菌检测中的应用分别进行概述,并对这3种PCR方法的优缺点进行分析与比较,为两栖类壶菌病检测方法的选择提供参考。
1 蛙壶菌的PCR检测方法
1.1 常规PCRPCR(polymerase chain reaction,PCR)即聚合酶链式反应,它是一种体外快速扩增特定DNA片段的方法。1985年,美国Mullis等[21]学者首创了PCR 技术,并由美国PE-Cetus 公司开发研制。 PCR反应中,利用碱基互补配对的原理,通过双链DNA的高温变性(denaturation)、低温退火(annealing)和适温延伸(extension)3步实现一个循环,在不断循环中,每一循环所合成的新DNA链,又作为下一循环中的模板,扩增产物的量以指数级方式增加,一般单一拷贝的DNA片段扩增循环25~30 次,可扩增l00 万~200 万倍[22]。自PCR 技术问世以来,已在微生物检测、临床诊断、水产养殖等领域得到广泛应用,如姜莉莉等[23]选择外毒素A基因(PEA)作为 PCR 检测的目标基因,建立了水貂绿脓杆菌 PCR 快速检测方法;张立军等[24]通过PCR技术鉴定拟南芥T-DNA 插入突变体atsuc3等。
Annis等[19]在利用常规PCR对蛙壶菌进行检测时,根据蛙壶菌位于5.8 S rDNA的特异性DNA片段设计引物,通过设计的引物Bd1a(5′-CAGTGTGCCATATGTCACG-3′)和Bd2a(5′-CATGGTTCATATCTGTCCAG-3′)进行PCR扩增得到目的片段[19],回收和纯化目的片段,再将得到的目的片段进行转化、克隆、测序,最后将得到的序列与GenBank中已发表的蛙壶菌基因序列进行序列分析。
1.2 巢式PCR巢式PCR(Nested PCR)是在常规PCR基础上发展起来的DNA扩增技术,具有较高的特异性和灵敏度,巢式PCR甚至可以在污染或已经降解的样本中进行DNA扩增[25]。其高特异性和灵敏度使得它在医学和生物学研究方面都有广泛应用[26-28]。巢式PCR通过两轮PCR反应,使用两套引物扩增特异性的DNA片断。 第一轮扩增中,外引物用以扩增得到特异性的DNA片段,作为第二轮扩增的模板,第二对引物特异性的扩增位于首轮PCR产物内的一段DNA片断,得到扩增的目的片段[29]。两轮PCR反应增加了目的基因的数量,从而提高了检测灵敏度[30]。
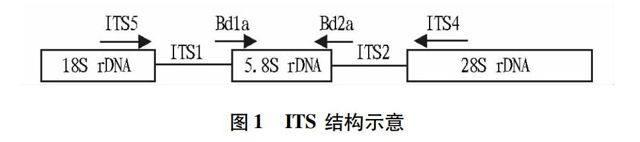
2005年Garner等[9]采用巢式PCR对欧洲壶菌病进行研究,结果表明蛙壶菌在欧洲具有广泛而不规则的分布。2009年Gaertner等[31]利用巢式PCR对美国德州5种地方性蝾螈的壶菌病调查中发现5种蝾螈都存在一定程度的蛙壶菌感染,同年,Goka等[32]同样使用巢式PCR对日本壶菌病进行调查,发现在部分两栖动物中也存在壶菌病,另外,2012年Bai等[16]使用巢式PCR对我国壶菌病的调查中发现云南、贵州、浙江等10省中均有蛙壶菌分布。在使用巢式PCR检测蛙壶菌中,蛙壶菌的一段特异性DNA片段是位于ITS4和ITS5(ITS:转录间隔区)之间的一段DNA序列(图1),试验中可以使用真菌通用引物ITS4(5′-TCCTCCGCTTATTGATATGC-3′)和ITS5(5′-GGAAGTAAAAGTCGTAACAAGG-3′)进行PCR扩增,得到一段特异性的DNA片段[33]。以得到的DNA片段作为模板,使用蛙壶菌特异性引物Bd1a(5′-CAGTGTGCCATATGTCACG-3′)和Bd2a(5′-CATGGTTCATATCTGTCCAG-3′)进行PCR扩增[19],后续步骤与常规PCR类似,将得到的目的片段依次进行回收、纯化、转化、克隆和测序,最后将得到的序列与GenBank中已发表的蛙壶菌基因序列进行序列分析。
1.3 实时定量PCR实时定量PCR(real-time TaqMan PCR)是由美国Applied Biosystems公司于1996年研发的一种新型实时定量检测特定核酸技术,该技术将核酸探针技术、PCR技术、荧光共振能量传递技术和光谱技术有机结合起来,在常规PCR基础上加入荧光标记探针来实现其实时定量功能[34-35]。
2006年Garner等[36]对北美洲的加拿大、美国等8个国家的牛蛙(Rana catesbeiana)壶菌病研究中发现7个国家的牛蛙中都检测到蛙壶菌感染。同年,Kriger等[37]对野外101只巨型斑蟾(Mixophyes iteratus)使用实时定量PCR和组织学方法对蛙壶菌进行检测,结果表明实时定量PCR的灵敏度明显高于组织学检测方法。2010年Bai等[15]在我国云南5种两栖动物中的4个物种中检测到壶菌病。Bataille等[13]对韩国壶菌病分布情况研究中发现壶菌病已在韩国广泛传播。以上几项调查研究中均使用实时定量PCR技术,在使用实时定量PCR检测蛙壶菌时,引物和探针一般采用通用壶菌检测引物和探针:
引物:ITS1-3 Chytr:5′-CCTTGATATAATACAGTGTGCCATATGTC-3′;
5.8S Chytr:5′-AGCCAAGAGATCCGTTGTCAAA-3′;
探针:ChytrMGB: 5′-6FAM CGAGTCGAACAAAAT MGBNFQ-3′。
在反应条件上实时定量PCR与常规PCR和巢式PCR有所不同:50 ℃ 2 min,95 ℃ 15 min, 95 ℃ 15 s,60 ℃ 1 min,50个循环[20]。将得到的目的片段依次进行回收、纯化、转化、克隆和测序,最后将得到的序列与GenBank中已发表的蛙壶菌基因序列进行序列分析。
2 3种PCR鉴定方法的分析与比较
2.1 巢式PCR与常规PCR比较 常规PCR中使用Bd1a和Bd2a作为引物扩增出来的DNA条带约为300 bp[19]。在巢式PCR中,第一步以ITS4和ITS5为引物扩增出的条带约600bp,第二步PCR以Bd1a和Bd2a为引物扩增出目的片段[19]。由于模板和引物的改变,降低了非特异性反应连续放大的可能性,保证了反应的特异性;同时,第一步PCR的产物作为第二步PCR的模板,克服常规PCR“平台期效应”的限制,使扩增倍数提高,从而极大地提高了PCR的灵敏度;另外,内侧引物扩增的模板是外侧引物扩增的产物,第二步PCR反应能否顺利进行,是对第一阶段反应正确性的鉴定,以增加了整个反应的准确性及可行性[29]。但经过2次PCR反应,在增加反应的特异性、灵敏度、准确性和可行性的同时也增加了两步反应之间试验污染的概率。
2.2 实时定量PCR与常规PCR比较实时定量PCR中通过引物(ITS1-3 Chytr和5.8S Chytr)和探针(ChytrMGB)与模板的特异性杂交来鉴别模板,相对于常规PCR而言,具有较高的准确性、特异性和较低的假阳性等优点[37];在实时定量PCR中将计算机和探针技术相结合,对试验中每个循环都可以实时监测,同时实现了PCR从定性到定量检测的飞跃,提高了试验的精确性,自动化程度较高,易于进行电脑化数据管理[38]。
2.3 巢式PCR与实时定量PCR比较巢式PCR和实时定量PCR都有各自的利弊,它们都具有特异性强、灵敏度高等优点。进一步比较两者,巢式PCR经历两步PCR反应会增加试验中污染的概率,而实时定量PCR则是在一个封闭的环境中完成PCR过程,减少试验中污染的可能性;而且,实时定量PCR还具有快速、自动化程度高、重复性好等特点[38]。另一方面,巢式PCR也有其优越性:首先,巢式PCR在蛙壶菌检测中具有比实时定量PCR更高的灵敏度;其次,若蛙壶菌DNA中具有相对保守性的ITS区中出现替换或缺失等突变时,实时定量PCR中的探针可能会出现与模板杂交失败的情况,影响PCR的效果,巢式PCR则可以避免此类问题的出现[32]。Goka等[32]使用3种PCR方法对26种四大类不同基因型的蛙壶菌进行检测,结果表明灵敏度由高到低依次为巢式PCR、实时定量PCR、常规PCR,另外,当蛙壶菌在 ITS1-5.8S rDNA-ITS2 区域有替换或缺失时,只有巢式PCR的结果为阳性。
3 展望
两栖动物种群全球性衰退是21世纪最紧迫的环境问题之一,全球2/3的两栖动物种群面临生存危机[39]。高致死性真菌病原体蛙壶菌是引起两栖类种群数量急剧下降的主要原因之一,其惊人的传播速度及广泛暴发对世界范围内的两栖动物种群数量构成重大威胁。蛙壶菌己在世界各地发现并流行,我国具有丰富的两栖动物资源和适合蛙壶菌生存的广阔的地理环境,由于蛙类贸易的发展,壶菌病已在我国传播蔓延。为了阻止壶菌病在我国进一步扩散蔓延与恶化,必须加强立法,在蛙类贸易进出口建立有效可靠的检疫防御措施。因此,得到快速、灵敏、简单、准确的蛙壶菌检测方法迫在眉睫。
另一方面,生物芯片(Biochip,又称DNA芯片、基因芯片)技术日新月异,利用生物芯片技术在对核酸、蛋白质、生物组织碎片或完整的活细胞进行检测时具有大规模高通量、灵敏准确、快速简便等优点[40-42]。该技术被广泛应用于基因表达分析[43-45]、DNA序列测定[46-47]、新基因发现[48-49]、医学诊疗[50-51]等方面。若能将生物芯片技术运用到蛙壶菌的检测上,不仅可以快速方便准确地检测蛙壶菌的存在,而且可以同时检测到不同种类的蛙壶菌,甚至明确不同蛙壶菌的基因型及菌株系。
参考文献
[1]BERGER L,SPEARE R,DASZAK P,et al.Chytridiomycosis causes amphibian mortality associated with population declines in the rain forests of Australia and Central America[J].Proceedings of the National Academy of Sciences,1998,95(15):9031-9036.
[2] JOHNSON M L,BERGER L,PHILLIPS L,et al.Fungicidal effects of chemical disinfectants,UV light,desiccation and heat on the amphibian chytrid,Batrachochytrium dendrobatidis[J].Diseases of Aquatic Organisms,2003,57:255-260.
[3] DASZAK P,BERGER L,CUNNINGHAM A A,et al.Emerging infectious diseases and amphibian population declines[J].Emerg Infect Dis,1999,5(6):735-748.
[4] RACHOWICZ L J,VREDENBURG V T.Transmission of Batrachochytrium dendrobatidis within and between amphibian life stages[J].Diseases of Aquatic Organisms,2004,61:75-83.
[5] VOYLES J,YOUNG S,BERGER L,et al.Pathogenesis of chytridiomycosis,a cause of catastrophic amphibian declines[J].Science,2009,326(5952):582-585.
[6] KRIGER K M,PEREOGLOU F,HERO J.Latitudinal variation in the prevalence and intensity of chytrid (Batrachochytrium dendrobatidis) infection in eastern Australia[J].Conservation Biology,2007,21(5):1280-1290.
[7] BERGER L,SPEARE S,HYATT A.Chytrid fungi and amphibian declines:overview,implications and future directions[M]//Declines and disappearances of Australian frogs.Canberra:Environment Australia,1999:23-33.
[8] WELDON C.Chytridiomycosis survey in south africa[J].Froglog,2002,51:1-2.
[9] GARNER T W,WALKER S,BOSCH J,et al.Chytrid fungus in Europe[J].Emerg Infect Dis,2005,11(10):1639-1641.
[10] BOVERO S,SOTGIU G,ANGELINI C,et al.Detection of chytridiomycosis caused by Batrachochytrium dendrobatidis in the endangered Sardinian newt (Euproctus platycephalus) in southern Sardinia,Italy[J].Journal of Wildlife Diseases,2008,44(3):712-715.
[11] UNE Y,KADEKARU S,TAMUKAI K,et al.First report of spontaneous chytridiomycosis in frogs in Asia[J].Diseases of Aquatic Organisms,2008,82(2):157-160.
[12] MCLEOD D S,SHERIDAN J A,JIRAUNGKOORSKUL W,et al.A survey for chytrid fungus in Thai amphibians[J].The Raffles Bulletin of Zoology,2008,56(1):199-204.
[13] BATAILLE A,FONG J J,CHA M,et al.Genetic evidence for a high diversity and wide distribution of endemic strains of the pathogenic chytrid fungus Batrachochytrium dendrobatidis in wild Asian amphibians[J].Molecular Ecology,2013,22:4196-4209.
[14] YANG H,BAEK H,SPEARE R,et al.First detection of the amphibian chytrid fungus Batrachochytrium dendrobatidis in free-ranging populations of amphibians on mainland Asia:survey in South Korea[J].Diseases of Aquatic Organisms,2009,86:9-13.
[15] BAI C,GARNER T W,LI Y.First evidence of Batrachochytrium dendrobatidis in China:discovery of chytridiomycosis in introduced American bullfrogs and native amphibians in the Yunnan Province,China[J].EcoHealth,2010,7(1):127-134.
[16] BAI C,LIU X,FISHER M C,et al.Global and endemic Asian lineages of the emerging pathogenic fungus Batrachochytrium dendrobatidis widely infect amphibians in China[J].Diversity and Distributions,2012,18(3):307-318.
[17] 曾朝辉,白世卓,朱蕴绮,等.蟾蜍壶菌病病原遗传分化研究[J].经济动物学报,2011,15(3):160-163.
[18] 曾朝辉,白世卓,朱蕴绮,等.馆藏泽蛙标本壶菌病原实时 PCR 检测与系统发育分析[J].经济动物学报,2012,16(3):168-171.
[19] ANNIS S L,DASTOOR F P,ZIEL H,et al.A DNA-based assay identifies Batrachochytrium dendrobatidis in amphibians[J].Journal of Wildlife Diseases,2004,40(3):420-428.
[20] BOYLE D,BOYLE D,OLSEN V,et al.Rapid quantitative detection of chytridiomycosis (Batrachochytrium dendrobatidis) in amphibian samples using real-time Taqman PCR assay[J].Diseases of Aquatic Organisms,2004,60:141-148.
[21] MULLIS K B,ERLICH H A,GELFAND D H,et al. Reacting nucleic acid with oligonucleotide primer in presence of catalytic enzyme dna polymerase; polymerase chain reaction patent: US,4965188[P].1990-10-23.
[22] 金宇良.PCR 技术的研究进展[J].现代农业科技,2012(10):47-48.
[23] 姜莉莉,樊兆斌,高文玉.水貂绿脓杆菌环介导等温扩增快速检测方法的建立[J].中国农学通报,2012,28(29):79-82.
[24] 张立军,杨双,阮燕晔,等.拟南芥T-DNA 插入突变体atsuc3 的PCR 鉴定[J].植物生理学通讯,2006,42(1):91-94.
[25] MA Z,LUO Y,MICHAILIDES T.Nested PCR assays for detection of Monilinia fructicola in stone fruit orchards and Botryosphaeria dothidea from pistachios in California[J].Journal of Phytopathology,2003,151(6):312-322.
[26] MAYER C L,PALMER C J.Evaluation of PCR,nested PCR,and fluorescent antibodies for detection of Giardia and Cryptosporidium species in wastewater[J].Applied and Environmental Microbiology,1996,62(6):2081-2085.
[27] ANDREA T.Two dimensional optimization of a semi-nested PCR for detecting Listeria monocytogenes[J].Eppendorf Bio News Application Notes,2000,6:3-4.
[28] KHAN J,SRIVASTAVA P,SINGH S.Efficacy of nested-PCR for the detection of phytoplasma causing spike disease of sandal[J].Current Science,2004,86(11):1530-1538.
[29] BECHER P,ORLICH M,KOSMIDOU A,et al.Genetic diversity of pestiviruses:identification of novel groups and implications for classification[J].Virology,1999,262(1):64-71.
[30] 兰平,李文凤,朱水芳,等.甘薯丛枝病植原体的 PCR 检测[J].植物学通报,2001,18(2):210-215.
[31] GAERTNER J P,FORSTNER M R,ODONNELL L,et al.Detection of Batrachochytrium dendrobatidis in endemic salamander species from Central Texas[J].Eco Health,2009,6(1):20-26.
[32] GOKA K,YOKOYAMA J,UNE Y,et al.Amphibian chytridiomycosis in Japan:distribution,haplotypes and possible route of entry into Japan[J].Molecular Ecology,2009,18(23):4757-4774.
[33] WHITE T J,BRUNS T,LEE S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M].San Diego,California:Academic Press,1990:315-322.
[34] HEID C A,STEVENS J,LIVAK K J,et al.Real time quantitative PCR[J].Genome Research,1996,6(10):986-994.
[35]GINZINGER D G.Gene quantification using real-time quantitative PCR:an emerging technology hits the mainstream[J].Experimental Hematology,2002,30(6):503-512.
[36] GARNER T W,PERKINS M W,GOVINDARAJULU P,et al.The emerging amphibian pathogen Batrachochytrium dendrobatidis globally infects introduced populations of the North American bullfrog,Rana catesbeiana[J].Biology Letters,2006,2(3):455-459.
[37] KRIGER K M,HINES H B,HYATT A D,et al.Techniques for detecting chytridiomycosis in wild frogs:comparing histology with real-time Taqman PCR[J].Diseases of Aquatic Organisms,2006,71(2):141-148.
[38] 钟江华,张光萍,柳小英.实时荧光定量 PCR 技术的研究进展与应用[J].氨基酸和生物资源,2011,33(2):68-72.
[39] STUART S N,CHANSON J S,COX N A,et al.Status and trends of amphibian declines and extinctions worldwide[J].Science,2004,306(5702):1783-1786.
[40] SHOEMAKER D,SCHADT E,ARMOUR C,et al.Experimental annotation of the human genome using microarray technology[J].Nature,2001,409(6822):922-927.
[41] WU J,SMITH L T,PLASS C,et al.ChIP-chip comes of age for genome-wide functional analysis[J].Cancer Research,2006,66(14):6899-6902.
[42] ROBERTS S S,MORI M,PATTEE P,et al.GABAergic system gene expression predicts clinical outcome in patients with neuroblastoma[J].Journal of Clinical Oncology,2004,22(20):4127-4134.
[43] 贺建华,刘丽莉,谢红兵,等.基因芯片筛选畜禽热应激差异表达基因的研究进展[J].动物营养学报,2012,24(12):2287-2294.
[44] 陈国宏,栾德琴,常国斌,等.利用基因芯片技术研究隐性白鸡不同时期肌肉组织相关差异表达基因[J].中国兽医学报,2012,32(1):83-88.
[45] HUANG J,SHENG H H,SHEN T,et al.Correlation between genomic DNA copy number alterations and transcriptional expression in hepatitis B virus-associated hepatocellular carcinoma[J].FEBS Letters,2006,580(15):3571-3581.
[46] 肖华胜,滕晓坤.基因芯片与高通量DNA 测序技术前景分析[J].生命科学(C辑),2008,38(10):891-899.
[47] HARRIS T D,BUZBY P R,BABCOCK H,et al.Single-molecule DNA sequencing of a viral genome[J].Science,2008,320(5872):106-109.
[48] SCHENA M,SHALON D,DAVIS R W,et al.Quantitative monitoring of gene expression patterns with a complementary DNA microarray[J].Science,1995,270(5235):467-470.
[49] SCHENA M,SHALON D,HELLER R,et al.Parallel human genome analysis:microarray-based expression monitoring of 1000 genes[J].Proceedings of the National Academy of Sciences,1996,93(20):10614-10619.
[50] LU Y,LEMON W,LIU P Y,et al.A gene expression signature predicts survival of patients with stage I non-small cell lung cancer[J].PLoS Medicine,2006,3(12):2229-22431.
[51] CLARKE P A,POELE R,WORKMAN P.Gene expression microarray technologies in the development of new therapeutic agents[J].European Journal of Cancer,2004,40(17):2560-2591.
责任编辑 宋平 责任校对 况玲玲

