基于Web的生物通路图可视化与编辑工具的设计
竺涌楠,方景龙
(杭州电子科技大学 计算机学院,浙江 杭州310012)
生物通路图是一系列化合物和反应所组成的复杂网络的一种可视化表示形式[1-2]。因此,一条生物通路可表示成节点和边的集合,节点可用来表示参与化学反应的反应物和产物,例如:蛋白质,小分子化合物,DNA,RNA等;边可表示各种相互作用关系,例如:反应和规则[3]。生物通路图以直观网络图谱的方式来显示相关的生物学信息,便于生物学家认清各种生物反应的网络图谱。了解生物通路中参与反应的生物分子之间的关系,各个生物分子的功能,以及各自的反应方式,以便于生物学家对生物实验数据进行观察、记录和分析。
随着对生物信息研究的继续,产生了许多生物通路数据库。例如,KEGG[4],Reactome[5-6],BioCyc[7]。然而这些数据库中的生物通路图均经过专业训练的生物学专家来手工绘制,数据库中展示的生物通路图也均是静态、不可编辑的[8-9]。目前,许多生物学研究者[8-9]也揭示了这种方式的不利之处,例如:费时且效率低,缺少数据分析的交互的方式,编辑困难等。特别是当一条通路中发现新的信息时,更新生物通路图很费时间。
针对这些问题,本文设计并实现了一个基于HTML5的生物通路图交互式可视化与编辑网站平台,用户只需登录即可编辑、浏览和改进生物通路图,用户并不需要安装相关插件。
1 HTML5 Canvas绘图技术
HTML5 Canvas元素是为了利用客户端绘制图形而设计的。它允许脚本语言(JavaScript)动态的渲染位图像,可用于2D和3D的图形绘制。JavaScript是一门寄宿式的脚本语言,主要的宿主环境是浏览器。JavaScript能够操作这些API得益于浏览器厂家提供的一套绘图API。开发人员只需调用这套API,就能设计开发出所需图形。目前的主流浏览器:IE9+、Chrome、Safari、FireFox、Opera都对它有着很好的支持,IE8以及之前的版本则不支持Canvas[10]。
Canvas的优点是不需要将所绘制图像中的每个图元当作对象存储,性能良好,而且它提供的API比其他编程语言简单易懂、易于上手。基于Canvas开发出来的应用无需安装任何应用插件。基于这些特点的分析,HTML5 Canvas具备实现基于Web的生物通路图可视化的潜力。
2 交互式可视化系统设计
2.1 生物通路图的图形对象可视化设计
生物通路图是一种以图的可视化形式来表示的生物通路,生物分子以及相关的反应可以被映射到网络图中。考虑到图的易读性和美观程度,以及经过与生物学家的讨论,本文对图的基本元素设计如下。
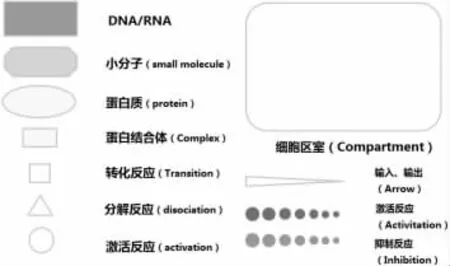
图1 生物通路可视化图的基本元素
节点:在生物通路图中,每个节点代表一种生物分子。图中的节点有许多相关的属性可供选择设置,例如:形状、背景颜色、大小、边框颜色、边框层次和边框的粗细等等。本文采用形状、颜色和大小来区分不同的生物分子。例如,用填充黄色背景的椭圆形来表示蛋白质(Protein),用填充黄色背景的矩形来表示蛋白质结合体。
边:在生物通路图中,用边来表示各种相互作用关系、反映类型和生物学功能等等。边可分为有向边和无向边。边也可以采用不同的形状,附加采用不同的颜色和粗细。在本文的设计中采用有向边来强调每一个反应的输入和输出关系。对于复杂的网络,传统的箭头很难区分方向,所以本文采用锥形箭头(Tapered Arrow)[2]来表示方向。由于有时两个结点之间有可能有多种关系,所以本文将其中的生物学功能关系(箭头)表示成由渐变圆点组成的虚线,这样当多个箭头重叠起来时,能看到底下被覆盖箭头的类型。
2.2 基于JavaScript面向对象的设计
JavaScript支持面向对象(OOP)编程。对象可以在代码执行过程中创建和增强,因此具有动态性。每一个对象都有Prototype属性,该属性所指向的对象是原型对象,因此可以指定那些应该共享的属性和方法。在生物通路图的设计中,本文需要把各种生物分子转化成各种图形对象,并找出这些对象共有的属性和方法。例如,每个节点对象Node都包含二维的位置坐标(x,y)、长(h)、宽(w)、Id编号、填充的颜色、边的颜色和text标签。每条边对象(Line)都包含起始节点对象、结束节点对象和边的类型(Arrow、Activation和Inhibition)。由于所有节点和边都有共有属性和方法,本文采用面向对象的设计思想产生出抽象对象Node和Line;其他具体生物分子的图元对象都继承自该Node对象,并添加各自特有的属性和方法;其他各种箭头表示都继承自该Line对象。所有的图形对象都被存放在一个Manage对象中。所有的事件操作Click、Mousedown、Mousemove和Mousemove也都 由Manage对象统一管理。

图2 图元对象设计
3 交互式可视化系统实现
3.1 整体界面的设计实现
使用面向对象的设计思想,将顶点和边都定义成对象,每个对象都包含特定的属性和方法,所有的对象和交互事件都由Manage类统一管理,能方便地实现图形的编辑。图3展示了生物通路图编辑软件的界面,在窗口中心显示当前的生物通路图。左上角显示边交叉数量、节点重叠数量和箭头往上的数量等统计数据,这是用来提醒用户当前图中存在的一些问题。界面右上角是功能菜单,包括用户名、本地数据导入(Load)、生物通路图保存到本地(Download)和生物通路图保存到服务器(Send)。
图3 生物通路图的在线浏览编辑界面
3.2 交互的设计实现
对于生物通路图的编辑和浏览,主要通过鼠标单击来选择移动。对于生物通路图的选择,采用两种方式:第一种是当鼠标靠近屏幕左边时,会自动弹出一列包含多种生物通路的树型结构,用户可以点击选取相应的生物通路,从而浏览特定的生物通路图;第二种是用户可以导入自己保存的生物通路图。当用户找到感兴趣的生物通路图时可以通过点击Download按钮下载该生物通路图。如果用户要保存当前修改过的生物通路图到服务器,可以点击Send发送到服务器。对于节点图元的编辑,用户只要单击选中图元,那么其边框就会高亮,用户只需移动即可。
3.3 在线编辑实例介绍
选择一条名为Abacavir transport and metabolism的生物通路,如图4所示。这条生物通路看起来有点杂乱,因为有许多边与边之间的交叉,箭头的方向也杂乱无章,这导致在该生物通路中查找特定生物分子相关的路径会变得很困难。所以,需要通过该平台对它进行编辑和改进。在线编辑平台的左上角显示着边交叉数量、节点重叠数量以及箭头向上的数量。只需要把想要排列的节点拖动到合适的位置,就可以编辑生物通路图。排列后的结果,如图5所示。看起来箭头的流向基本都是从上到下,也不存在边的交叉。完整的设计网站,请访问:http://www.csee.umbc.edu/~yongnan/drawPathway/。

图4 Abacavir transport and metabolism生物通路

图5 排列后的Abacavir transport and metabolism生物通路
4 专家评估
系统的使用者是特拉华大学的两位生物学家:Carl Schmidt教授和Liang Sun博士,对该软件平台进行了详细的测试和评估,主要针对生物通路图编辑浏览功能的方便性和实用性。
方便性:生物通路图的在线编辑和查找功能的设计,有利于创建特定数据相关的生物通路图。在线对各种生物通路数据进行编辑和保存,无需安装任何插件,具有较高的方便性。
实用性:该平台支持对生物通路图的动态操作,特别是本地生物通路数据的导入。只需简单的拖放就可以对生物通路图可视化的结果进行重排。这方便生物学家改进现有的生物通路图,同时也方便了生物学家生物通路数据的共享,以及随时随地地进行查看和浏览。
5 结束语
本文设计并实现了一个在线浏览和编辑生物通路图的工具,描述了可视化生物通路图的图形对象和交互式可视化系统的设计与实现。HTML5 Canvas的绘图功能在生物通路图可视化应用是一次创新,方便了生物研究者访问、浏览和编辑生物通路。该平台可以扩展成一个基于生物通路图的编辑和分析平台,并提供导入其他的生物实验数据进行分析的功能,这有利于提高生物学家对编辑后的生物通路图的利用率和便于在线分析实验数据。
[1]Becker M Y,Rojas I.A graph layout algorithm for drawing metabolic pathways[J].Bioinformatics,2001,17(5):461-467.
[2]Kojima K,Nagasaki M,Jeong E,et al.An efficient grid layout algorithm for biological networks utilizing various biological attributes[J].BMC Bioinformatics,2007,8(1):76-92.
[3]Schreiber F.High quality visualization of biochemical pathways in BioPath[J].Silico Biology,2002,2(2):59-73.
[4]Kanehisa,Minoru,Susumu Goto.KEGG:kyoto encyclopedia of genes and genomes[J].Nucleic Acids Research,2000,28(1):27-30.
[5]Croft D,O'Kelly G,Wu G,et al.Reactome:a database of reactions,pathways and biological processes[J].Nucleic Acids Research,2011(39):D691-D697.
[6]Joshi-Tope G,Gillespie M,Vastrik I,et al.Reactome:a knowledgebase of biological pathways[J].Nucleic Acids Research,2005(33):D428-D432.
[7]Caspi R,Foerster H,Fulcher C A,et al.The MetaCyc database of metabolic pathways and enzymes and the BioCyc collection of Pathway/Genome databases[J].Nucleic Acids Research,2008(36):D623-D631.
[9]Kojima K,Nagasaki M,Jeong E,et al.An efficient grid layout algorithm for biological networks utilizing various biological attributes[J].BMC Bioinformatics,2007(8):31-76.
[10]张泽娜.JavaScript实战[M].北京:清华大学出版社,2014.

