引入猪种与合作藏猪mtDNA D-loop的遗传多样性
赵生国, 刘丽霞, 郑王山, 权金强, 汪文强, 郭永博, 李永青, 焦 婷, 蔡 原
(1.甘肃农业大学动物科学技术学院,甘肃 兰州 730070;2.西北民族大学生命科学与工程学院,甘肃 兰州 730030;3.甘肃农业大学草业学院,甘肃 兰州 730070)
引入猪种与合作藏猪mtDNA D-loop的遗传多样性
赵生国1, 刘丽霞2, 郑王山1, 权金强1, 汪文强1, 郭永博1, 李永青1, 焦 婷3, 蔡 原1
(1.甘肃农业大学动物科学技术学院,甘肃 兰州 730070;2.西北民族大学生命科学与工程学院,甘肃 兰州 730030;3.甘肃农业大学草业学院,甘肃 兰州 730070)
测定并分析了杜洛克猪、长白猪和大白猪3个引入猪种及合作藏猪共178个个体的线粒体DNA D环(mtDNA D-loop)高变区核苷酸序列,研究引入猪种对合作藏猪遗传资源的影响.结果表明:合作藏猪核苷酸多样度和平均核苷酸差异系数均低于杜洛克猪和大白猪,仅高于长白猪,而其单倍型多样度高于3个引入猪种.单倍型分析结果表明:合作藏猪仅分布在亚洲单倍类,杜洛克猪和大白猪同时具有亚洲和欧洲两个单倍类,欧洲起源猪种没有入侵到合作藏猪母系.
合作藏猪; 线粒体DNA D环; 单倍型; 遗传多样性
合作藏猪与其他猪种相比具有独特的种质资源特征.细胞遗传学研究表明,藏猪染色体带型与其他猪种相比存在特异染色体带型[5],而且在藏猪的β-珠蛋白[6]、MHC-DQB1[7]和Myogenin[8]的基因中发现了丰富的遗传多样性.这可能是由于藏猪属于高原放牧型品种,人工选择对其影响较小,使其能够长期保持原始血统,同时在自然条件下产生的突变类型能够保留并积累在群体中.但也有研究表明,中国个别地方品种和一些国外品种猪的遗传多样性呈下降趋势[9-11].对迪庆、甘孜、阿坝、合作和西藏藏猪群体线粒体D环(mitochondrial DNA D-loop, mtDNA D-loop)序列分析发现,除合作藏猪群体外,其他几个群体的遗传变异相对比较低[12].这说明可能由于人工选择导致中国家猪遗传多样性下降[13],特别是高强度选择猪种对地方猪种杂交产生的后果更为严重.
本试验以mtDNA为分子标记,开展杜洛克猪、长白猪、大白猪和合作藏猪的遗传多样性研究,比较不同来源猪种遗传资源的差异,旨在为我国地方猪种的资源保护提供参考依据.
1 材料与方法
1.1 材料
采集合作藏猪(59头)、杜洛克猪(36头)、长白猪(42头)和大白猪(41头)共178头的猪耳(尾)组织样品.采用传统的酚氯仿抽提法[14]进行基因组DNA的提取.
1.2 引物设计
根据发表在GenBank中的沂蒙黑猪mtDNA全序列(AF486868.1)[15],采用引物设计软件Primer Premier 5.0 (http://www.premierbiosoft.com)对猪mtDNA D-loop高变区扩增引物进行设计,设计如下引物序列.上游引物P-L16337:5′-TAGAACACCCATTCATCATC-3′;下游引物P-H1356:5′-CGTTATGAGCTACCGTTATA-3′.引物由上海生工生物技术有限公司合成.
1.3 PCR反应
PCR扩增条件为:94 ℃预变性2 min;94 ℃变性20 s、55.6 ℃退火30 s、72 ℃延伸40 s,30个循环;循环结束后于72 ℃延伸10 min.扩增产物采用2%琼脂糖凝胶电泳进行检测,检测的目的产物由上海生工生物技术有限公司完成测序.
值得注意的是,在调查统计中,居住支出包括房租、水、电、燃料、物业管理等方面的支出,也包括自有住房折算租金,但没有包括购买住房的支出,因此实际居住的支出是被严重低估的。
1.4 数据处理
采用Chromas 2.3软件(http://www.technelysium.com.au/chromas.html)对原始序列进行编辑,并通过MEGA 5.0软件(http://www.megasoftware.net)[16]建立数据库后用Clustal X软件(http://www.igbmc.ustrasbg.fr/pub/ClustalX)进行同源序列比对分析,采用软件包Dnasp 4.10(http://www.ub.es/dnasp)进行遗传多样性分析.
2 结果与分析
2.1 mtDNA D-loop高变区的核苷酸多样度
通过测序获得合作藏猪、杜洛克猪、长白猪和大白猪共178个个体的mtDNA D-loop高变区长度为438 bp的核苷酸序列.采用Dnasp 4.10软件进行单核苷酸多态性分析发现,4个猪群存在19个变异位点(2个是单一变异位点,17个是简约信息位点),并由此界定了17个单倍型.遗传多样性分析结果如表1所示.
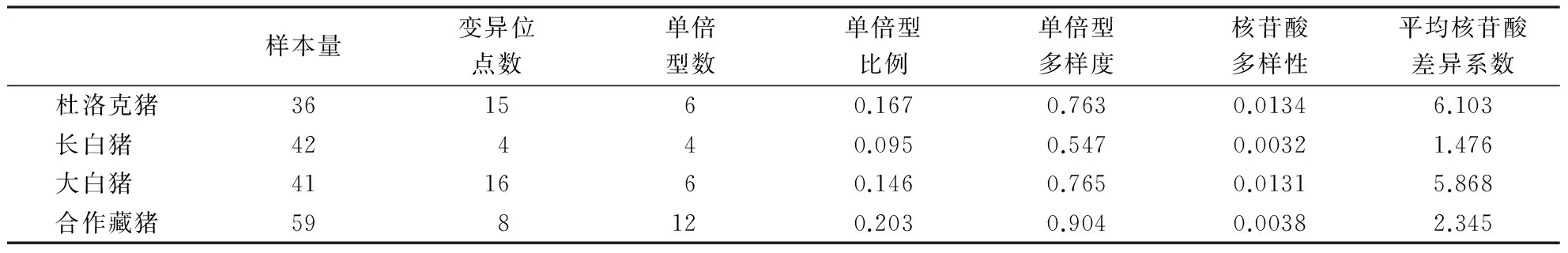
表1 4个猪群的遗传多样性
从表1可以看出,合作藏猪的单核苷酸变异位点数(8个)介于大白猪(16个)、杜洛克猪(15个)与长白猪(4个)之间,说明选育猪种杜洛克猪和大白猪的核苷酸变异比较丰富,而合作藏猪显得比较低.反映遗传多样性的核苷酸多样度和平均核苷酸差异系数与单核苷酸变异位点数的分析结果类似,这些结果表明合作藏猪核苷酸变异水平低于杜洛克猪和大白猪,仅高于长白猪.
2.2 mtDNA D-loop高变区的单倍型
采用Dnasp 4.10软件进行单倍型分析及其频率计算的结果(表2)表明,4个猪群的17个单倍型中存在5个共享单倍型,其中有两个(H1和H3)是4个猪群的共享单倍型.在12个特有单倍型中,合作藏猪的单倍型最多,达到8个,而杜洛克猪和大白猪分别只有3和1个;长白猪没有发现其特有的单倍型.显然,外来选育猪种的特有单倍型数远远低于地方猪种,这可能是由于在高强度的选育过程中,繁殖母猪的单一母系来源造成此结果.
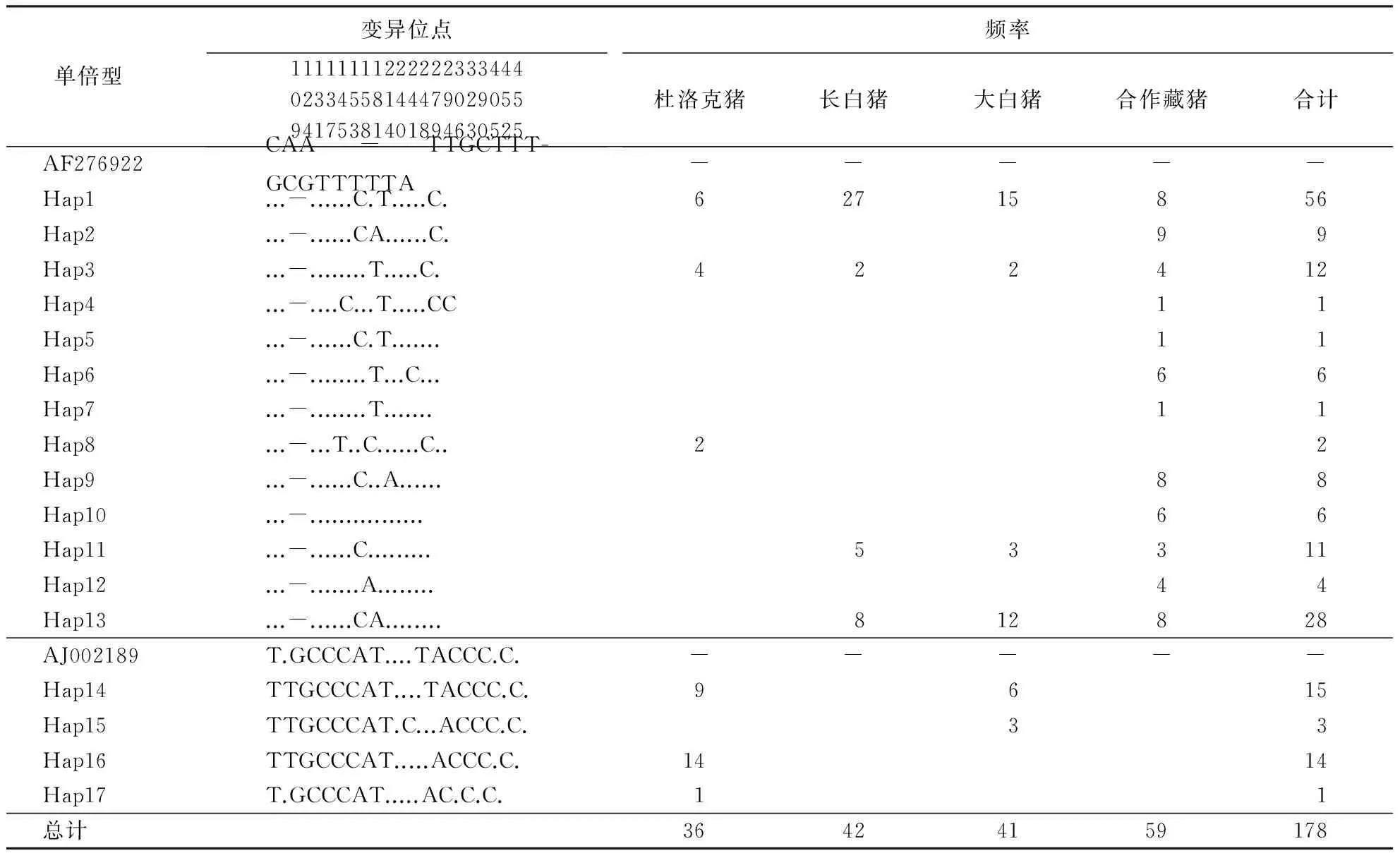
表2 4个猪群的单倍型分布及频率1)
1)-表示碱基缺失.
2.3 遗传背景
分别对亚洲和欧洲起源猪种(中国地方猪种二花脸和瑞士选育猪)[17-18]的mtDNA D-loop序列(AF276922[18]和AJ002189[19])进行遗传背景研究,经核苷酸比对,根据亚洲和欧洲来源家猪mtDNA D-loop核苷酸序列存在的特征性碱基(表2)将17个单倍型分为两个单倍类,即亚洲单倍类(Hap1-13)和欧洲单倍类(Hap14-17).中介网络关系分析结果(图1)表明,合作藏猪和长白猪的单倍型仅分布在亚洲单倍类,杜洛克猪和大白猪的单倍型同时出现在亚洲和欧洲两个单倍类中.
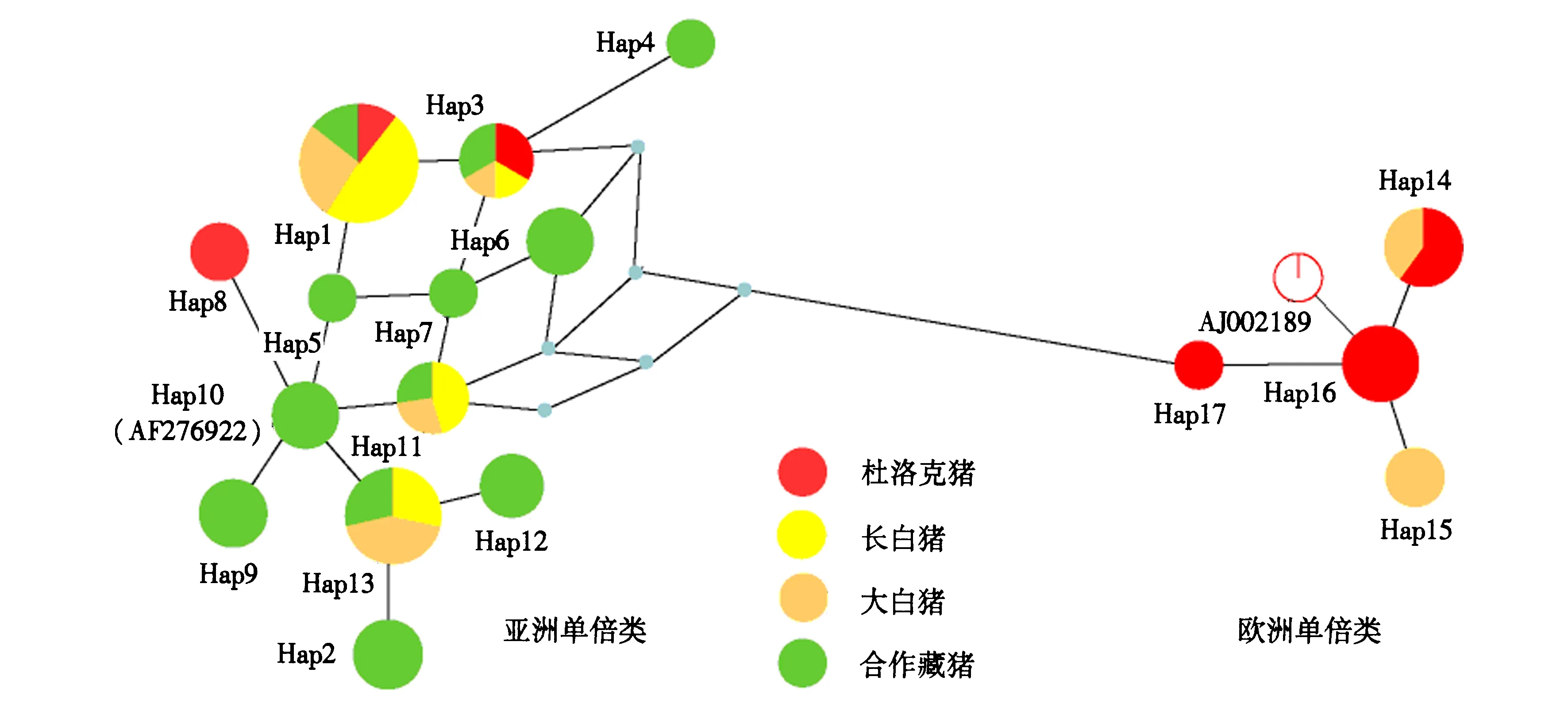
图1 4个猪群单倍型的中介网络关系
3 讨论
3.1 遗传多样性的变化趋势
Muir et al[20]研究表明:地方鸡种群数量下降意味着其遗传多样性减少,并比较了地方鸡品种和商业选育品种数千个单核苷酸多样度的遗传标记,结果发现,随着时间的推移,商业选育鸡品种失去了超过半数的单核苷酸多态性标记位点.单纯追求生产性能而开展的高强度选育,使核苷酸变异、等位基因或单倍型数量等降低的现象在猪的种质遗传资源方面也同样发生.Li et al[21]采用微卫星对中国4个地方猪种和一个澳大利亚选育猪种遗传多样性的研究中发现,澳大利亚选育猪种的等位基因数最少.本试验结果表明,合作藏猪和长白猪的核苷酸变异系数、核苷酸多样度和平均核苷酸变异系数等遗传多样性指标低于杜洛克和大白两个选育猪种.尽管杜洛克猪和大白猪经历了高强度选育,然而在选育过程中,由于这两个猪种分别来自亚洲和欧洲两个母系,其遗传背景较合作藏猪和长白猪复杂,表现出了相对较高的核苷酸多样度及其变异系数.由于外来猪种的大量饲养,合作藏猪当前的分布区域、饲养量逐渐萎缩,且由于其繁殖周期长、产仔数低(5-6头)[22],导致核苷酸的变异速率相对较慢,最终表现为遗传资源呈现下降趋势.与此相反,应用现代育种技术选育的猪种,其繁殖周期短且产仔数高(10头以上)[22],在较高的繁殖率条件下,核苷酸发生变异的速率加快.
尽管选育猪种的高繁殖力与核苷酸变异速率的加快有关,然而对单倍型的分析发现,合作藏猪的单倍型数(12个)、单倍型比例(0.203)和单倍型多样度(0.904)均高于3个选育猪种,这从分子遗传标记上印证了工业化猪生产中,选育猪种以极少的种用母猪生产大量仔猪的现象,其母系逐渐趋向于单一化,而处于自然繁殖状态下的合作藏猪保持了相对丰富的母系遗传多样性.
3.2 合作藏猪的遗传资源及其母系背景
地方畜禽在长期进化过程中形成了丰富的遗传资源宝库,在环境适应性和抗病性等方面具有独特的优势,但由于不能满足当前人们对经济利益的单纯追求,受到具有较高经济效益的选育品种的排挤(被外来品种取代或与外来品种杂交),导致地方畜禽遗传资源面临濒危,甚至灭绝.由此来看,选育猪种和地方猪种因高强度选育、种群数量下降及生态环境改变,导致了其遗传多样性下降,表现出与家鸡类似的结果[20],即抗逆性下降,使人类在应对大气变化和恶性动物疾病等突发事件方面难以应对.本试验研究合作藏猪及引入猪种的遗传多样性现状及分布规律,旨在为资源保护提供参考依据.
单倍型多样度和核苷酸多样度是衡量一个群体mtDNA 变异程度的两个重要指标[23],但各有侧重.单倍型多样度可以反映群体母系遗传背景的丰富程度,而核苷酸多样度则反映了群体内个体间的变异.本试验研究的合作藏猪和3个引入猪种中,单倍型多样度以合作藏猪最高.中介网络关系分析表明,合作藏猪单倍型均分布在亚洲来源的单倍型中,各单倍型形成的星状结构表明其已形成稳定的种群.合作藏猪以放养为主的饲养方式,繁育受人为干预较少,表现出自然交配后的种群分化结果,即在长期的自然驯化过程中形成了种群扩张.欧洲单倍类型中没有发现合作藏猪,说明欧洲起源猪种没有入侵到其母系群体中.杜洛克猪和大白猪同时具备欧洲和亚洲家猪单倍型,说明其在人工选育的过程中参入了亚洲和欧洲家猪的母系血统,高频率(58)的单倍型Hap1正是人为干预下单一母系高效率繁殖的结果.因此,从遗传资源保护的角度来看,应尽可能保持地方猪种的自然繁殖方式,降低人工选育强度.
[1] 李金元,罗章.西藏高原藏猪生活习性的调查研究[J].家畜生态学报,1993,14(1):18-21.
[2] 张牧,戴伯章.胎次和妊娠期对母猪产仔数和仔猪增重的影响[J].江苏农学院学报,1996,17(2):33-34.
[3] 周志强,金敏球,肖新良,等.长白母猪断奶至配种间隔时间与下一产的产仔数之关系[J].养猪,2002(2):16.
[4] 呼红梅,郭建凤,朱荣生,等.胎次对杜洛克和大约克母猪繁殖性能影响的研究[J].黑龙江畜牧兽医,2008(3):31-33.
[5] 吕学斌,邹方东,赵森林,等.藏猪种质资源细胞遗传学研究[J].四川动物,2009,28(6):831-834.
[6] 杨会强.藏猪β-珠蛋白基因与其低氧遗传适应的关系[D].北京:中国农业科学院,2010.
[7] 杨涛.藏猪MHC-DQB1基因多态性的研究[D].拉萨:西藏大学,2010.
[8] 朱砺.猪骨骼肌形成过程中的几种决定因子(MDF)的遗传效应及其遗传多态性和分子进化研究[D].雅安:四川农业大学,2004.
[9] ZHANG S Q, XU J C, ZHANG Y P, et al. Genetic diversity of Zhejiang native pig breeds inferred from mitochondrial DNA polymorphism [J]. Zool Res, 1998,19(2):125-130.
[10] LIU Z L, WEI H, ZENG Y Z, et al. Analysis of the porcine mtDNA D-loop PCR RFLP [J]. Shanghai Lab Anim Sci, 2000,20(1):7-10.
[11] GROSSI S F, LUI J F, GARCIA J E, et al. Genetic diversity in wild (Susscrofascrofa) and domestic (Susscrofadomestica) pigs and their hybrids based on polymorphism of a fragment of the D-loop region in the mitochondrial DNA [J]. Gene Mol Res, 2006,5(4):564-568.
[12] JIAO T, ZHAO S G, WANG C, et al. Mitochondrial DNA D-loop diversity of Tibetan Pig populations [J]. Philippine Agriculture Scientist, 2009,92(4):362-369.
[13] LI J, YANG H, LI J R, et al. Artificial selection of the melanocortin receptor 1 gene in Chinese domestic pigs during domestication [J]. Heredity, 2010,105(3):274-281.
[14] SAMBROOK J, RUSSELL D. Molecular cloning: a laboratory manual[M]. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 2002:484-485.
[15] YANG J, WANG J, KIJAS J, et al. Genetic diversity present within the near-complete mtDNA genome of 17 breeds of indigenous Chinese pigs [J]. Journal of Heredity, 2003,94(5):381-385.
[16] TAMURA K, DUDLEY J, NEI M. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0 [J]. Mol Biol Evol, 2007,24:1596-1599.
[17] GONGORA J, FLEMING P, SPENCER P B, et al. Phylogenetic relationships of Australian and New Zealand feral pigs assessed by mitochondrial control region sequence and nuclear GPIP genotype [J]. Mol Phylogenet Evol, 2004,33(2):339-348.
[18] KIM K I, LEE J H, LI K, et al. Phylogenetic relationships of Asian and European pig breeds determined by mitochondrial DNA D-loop sequence polymorphism [J]. Animal Genetics, 2002,33:19-25.
[19] URSING B M, ARNASON U. The complete Mitochondrial DNA sequence of the pig (Susscrofa) [J]. J Mol Evol, 1998,47:302-306.
[20] MUIR W M, WONG G K, ZHANG Y, et al. Genome-wide assessment of worldwide chicken SNP genetic diversity indicates significant absence of rare alleles in commercial breeds. [J]. PNAs, 2008,105(45):17312-17313.
[21] LI K, CHEN Y, MORAN C, et al. Analysis of diversity and genetic relationships between four Chinese indigenous pig breeds and one Australian commercial pig breed [J]. Animal Genetics, 2000,31:322-325.
[22] 杨公社.猪生产学[M].北京:中国农业出版社,2012.
[23] SBISA E, TANZARIELLO F, REYES A, et al. Mammalian mitochondrial D-loop region structural analysis: identification of new conserved sequences and their functional and evolutionary implications [J]. Gene, 1997,205(12):125-140.
(责任编辑:施晓棠)
Genetic diversity of commercial pigs and Hezuo Tibetan pig
ZHAO Sheng-guo1, LIU Li-xia2, ZHEN Wang-shan1, QUAN Jin-qiang1, WANG Wen-qiang1,GUO Yong-bo1, LI Yong-qing1, JIAO Ting3, CAI Yuan1
(1.College of Animal Science and Technology, Gansu Agriculture University, Lanzhou, Gansu 730070, China; 2.College of Life Science and Engineering, Northwest University for Nationalities, Lanzhou, Gansu 730030, China; 3.College of Grassland Sciences, Gansu Agriculture University, Lanzhou, Gansu 730070, China)
The mitochondrial DNA D-loop (mtDNA D-loop) sequences of 178 individuals obtained from Duroc pig, Large White pig, Landrace pig and Hezuo Tibetan pigs were sequenced and analyzed to assess the effect of imported breed on genetic resource of Hezuo Tibetan pigs. The nucleotide diversity and average number of nucleotide differences of Hezuo Tibetan pigs were lower than that of Duroc pigs and Large White pigs, and higher than that of Landrace pigs. The haplotype diversity of Hezuo Tibetan pigs was higher than that of the three imported pig breeds. The analysis of haplotypes indicated that Hezuo Tibetan pigs were droped in the clade of Asia, both of Asia and Europe clades had been found in Duroc pigs and Large White pig presented, which implied maternal of Hezuo Tibeatan pigs had not eroded by commercial pigs originated from Europe.
Hezuo Tibetan pig; mtDNA D-loop; haplotype; genetic diversity
2014-07-03
2014-09-13
甘肃农业大学盛彤笙科技创新项目(GSAU-STS-1236).
赵生国(1976-),男,副教授.研究方向:动物遗传学.Email:zhaosg@gsau.edu.cn.通讯作者蔡原(1974-),女,副教授.研究方向:动物遗传学.Email:caiyuan@gsau.edu.cn.
S813.1
A
1671-5470(2015)04-0391-05
10.13323/j.cnki.j.fafu(nat.sci.).2015.04.010

