普通烟草SBP转录因子家族的全基因组鉴定及其进化和表达分析
刘 成,李晓旭,苏玉龙,郭永峰
(1.中国农业科学院烟草研究所,青岛 266101;2.中国农业科学院研究生院,北京 100081)
普通烟草SBP转录因子家族的全基因组鉴定及其进化和表达分析
刘 成1,2,李晓旭1,2,苏玉龙1,2,郭永峰1*
(1.中国农业科学院烟草研究所,青岛 266101;2.中国农业科学院研究生院,北京 100081)
SBP转录因子家族作为植物特异性的转录因子具有重要的生物学功能,广泛参与花和果实发育等诸多生命过程。利用普通烟草(Nicotiana tabacum)TN90基因组数据,通过隐马尔科夫模型检索,SMART和Pfam分析,鉴定了普通烟草SBP转录因子家族成员;通过多序列比对鉴定并分析了普通烟草SBP结构域的结构特征;通过邻接法、极大似然法进行了系统发育分析;分别利用GSDS和MEME对SBP转录因子家族成员的基因结构和蛋白保守结构域进行了分析;利用普通烟草TN90转录组数据分析了SBP转录因子家族成员在不同组织和时期的表达情况,并对鉴定出的SBP转录因子家族成员进行了GO注释分析。结果表明,普通烟草基因组编码32个SBP转录因子家族成员,氨基酸序列长度差异较大;系统发育分析表明,所有的SBP转录因子家族成员可分为8个亚家族。转录组数据显示,SBP基因家族成员在不同组织和时期中表达迥异,但在花器官中均有较高的表达量。GO注释结果表明,该家族成员作为转录因子,在植物的生长发育、逆境响应等过程中发挥作用。本研究为烟草及其他植物中SBP转录因子家族基因的鉴定和功能研究提供了基础。
生物信息学分析;SBP转录因子家族;普通烟草
转录因子是参与转录调控的一类蛋白,通过与特定序列的DNA结合,能够激活或抑制目标基因的转录,从而调控基因的表达[1]。SQUAMOSA Promoter Binding Proteins(SBPs)转录因子家族是一类植物特有的重要转录因子,由多个成员组成,最早发现于金鱼草中,广泛参与植物生长、发育以及多种生理生化过程[2]。SPL(SBP-like)基因主要调控花和果实发育,有报道称拟南芥中SPL3,SPL8,SPL14在开花、孢子形成、赤霉素信号传导、抵抗毒素侵袭等生命过程中发挥着重要作用[3-6]。作为植物特异的转录因子,所有已知的SBP转录因子家族成员蛋白中均含有一段由79个氨基酸残基组成的高度保守的DNA结合域。拟南芥SBP转录因子的DNA结合域由两个分离的锌指结构组成,一个是C3H或C4,另一个是C2HC[7]。根据文献报道,SBP转录因子家族很多成员都受到miR156的调控。目前在拟南芥和水稻中都各发现有11个SPL基因含有miR156的识别位点[8],拟南芥营养发育过程中SPL3的时空性表达受到miR156的调控[9],水稻中的miR156和某些OsSPL基因也会发生组织特异性互作[10]。随着越来越多的植物基因组测序的完成,SBP转录因子家族在全基因组范围内已经被广泛地鉴定。
烟草是重要的经济作物,同时也是一种重要的模式植物,经常作为分子生物学的研究对象。在普通烟草基因组注释信息公布前,人们往往采取同源克隆结合RACE技术等手段研究烟草中重要的功能基因,如陆莹等[11]利用马铃薯的同源基因在烟草中克隆到了光周期的关键基因NtCO1并进行了初步分析;刘好宝等[12]在烟草中同源克隆到4个钾离子通道基因:NKT3、NKT4、NKT5和NKT6。随着生物信息学的发展和烟草基因组测序、注释的完成,烟草中如钙依赖蛋白激酶基因家族(CDPK Gene Family)[13]等基因家族都已成为研究的热点。但烟草中针对SBP转录因子家族的研究还未见报道。本研究在普通烟草的全基因组注释信息的基础上,利用生物信息学方法鉴定出普通烟草32个SBP转录因子家族成员,分析了各成员的进化关系、基因结构、蛋白保守结构域、表达情况,并进行GO注释分析,为进一步研究普通烟草SBP转录因子家族成员的功能奠定基础。
1 材料与方法
1.1 普通烟草SBP转录因子家族成员的鉴定与序列分析
根据文献报道,在TAIR数据库(http://www. arabidopsis.org/)下载了拟南芥SBP转录因子家族成员的蛋白全长序列,在PlantTFDB(http:/ /planttfdb.cbi.pku.edu.cn/)数据库[14]下载了番茄、马铃薯的SBP转录因子家族成员的蛋白全长序列。对于拟南芥、番茄、马铃薯的SBP蛋白全长序列,利用MAFFT分别进行序列比对,使用HMMER 3.0分别建立隐马尔科夫模型序列谱(profile hidden Markov models,profile HMMs)。在茄科植物数据库(http://solgenomics.net/)下载普通烟草TN90蛋白序列数据库,分别使用建立的3个隐马尔科夫模型序列谱对普通烟草TN90蛋白序列数据库进行检索,选取E值为0.01,手工去除冗余序列,得到候选序列。最后针对所获得的候选序列,利用SMART和Pfam分析蛋白结构,剔除不含完整SBP结构域的蛋白序列。利用ExPASy Proteomics Server(http://expasy.org/)对所有SPB家族成员蛋白氨基酸序列进行分子量、等电点预测分析。
1.2 多序列比对和系统发育分析
将拟南芥和普通烟草TN90的SBP蛋白全长序列用MAFFT[15]在默认参数下进行多序列比对。选取保守的SBP结构域,利用Texshade将结果可视化。利用拟南芥、普通烟草SBP结构域蛋白序列的比对结果,使用MGEA5构建邻接树(Neighbor-Joining, NJ tree),参数设置为:Bootstrap检验次数设为1000次,并选择Poisson Model 和Pairwise Deletion进行分析。利用同样的比对结果,使用PhyML构建极大似然树[16](Maxumum Likeihood, ML tree)。Bootstrap设为100次,并利用ProtTest 2.4[17]对拟南芥和普通烟草SBP结构域氨基酸序列比对结果进行分析,选择LG+G模型构建极大似然树,并利用Tree View 1.4.0显示系统发生树。
1.3 基因结构和结构域分析
在茄科植物数据库(http://solgenomics.net/),下载普通烟草TN90基因组信息,通过Perl程序分析并提取SBP基因家族成员的基因结构信息,使用GSDS(http://gsds.cbi. pku.edu.cn/)工具对分析结果进行基因结构的可视化展示。利用Multiple Expectation Maximization for Motif Elicitation(MEME, http://meme.nbcr.net/meme/cgi-bin/mem e.cgi)[18]对SBP转录因子家族成员的蛋白序列基序(motif)进行分析。
1.4 普通烟草SBP转录因子家族基因表达分析及GO注释分析
在NCBI SRA(http://www.ncbi.nlm.nih.gov/sra)数据库检索并下载TN90转录组测序(RNA-seq)数据,提取表达数据,利用R语言对转录组表达数据进行分析并提取SBP基因家族成员的基因表达数据,将表达数据标准化后,使用R语言pheatmap程序绘制热图(heatmap),将表达数据可视化。使用普通烟草SBP转录因子家族蛋白序列,利用Blast2GO进行GO(Gene Ontology, GO)注释[19],WEGO工具[20]分析注释结果并进行可视化展示。
2 结 果
2.1 普通烟草SBP转录因子家族成员鉴定及理化特征分析
利用文献报道的拟南芥SBP转录因子家族成员蛋白序列以及PlantTFDB中番茄和马铃薯的SBP转录因子家族成员蛋白序列,分别构建了隐马尔可夫模型序列谱。利用构建的3个隐马尔科夫模型序列谱分别检索普通烟草TN90蛋白数据库,综合3个检索结果,人工去除冗余序列,获得候选蛋白序列。通过SMART和Pfam对候选蛋白序列进行结构域分析,剔除不含完整SBP功能域的蛋白序列,最终预测得到32个烟草SBP基因家族成员,命名为NtSPL1-NtSPL32(表1)。

表1 普通烟草SBP转录因子家族成员特征Table 1 Characteristics of SBP transcription factor family in N. tabacum
对普通烟草SBP转录因子家族成员进行蛋白质序列及理化性质分析发现,这些氨基酸的长度变化差异较大,在133~999 aa,蛋白分子量则介于15.27~110.97 kD。家族成员蛋白的理论等电点变化较小,在5.15~9.76,其中第I-V亚族(7.64~9.43)理论等电点都在碱性范围内,表明这些亚家族蛋白富含碱性氨基酸,而第VIII亚族(NtSPL6和NtSPL32)理论等电点较低(5.15~5.29),其蛋白含有较多的酸性氨基酸。所有SBP转录因子家族蛋白疏水性均为负值,表明此家族均属于亲水性蛋白,其中第Ⅶ亚族蛋白疏水性普遍较低,从-1.313到-1.084,说明第VII亚族蛋白均具有较强的亲水性。
2.2 普通烟草SBP结构域的鉴定和分析

图1 拟南芥、普通烟草SBP转录因子家族蛋白SBP结构域分析Fig. 1 Conserved SBP domain analysis of SBP family proteins in N. tabacum and A. thaliana
将文献报道的16条拟南芥SBP转录因子家族成员蛋白序列和预测的32条普通烟草SBP转录因子家族成员蛋白序列,使用MAFFT在默认参数下进行多序列比对,发现所有序列均含保守的SBP结构域。选取保守的SBP结构域,利用Texshade将结果可视化(图1),拟南芥和普通烟草的SBP结构域高度相似,均含79个氨基酸残基。利用核磁共振方法,拟南芥中两个SBP结构域的三维溶液构象已经被报道[21],两者都含2个锌指结构(Zinc finger)。AtSPL3的N端锌指结构(Zn-1)为Cys3His(C3H),C端锌指结构(Zn-2)为Cys2HisCys(C2HC)。AtSPL7的N端锌指结构为Cys4(C4),C端锌指结构与同样为C2HC。经过对拟南芥和普通烟草SBP结构域的分析,发现所有的AtSPL和NtSPL转录因子均包含两个锌指结构:AtSPL7、NtSPL6和NtSPL32的N段锌指结构为C4,其余成员的N端锌指结构均为C3H;所有的C端锌指结构均为C2HC。与拟南芥类似,普通烟草SBP转录因子家族成员蛋白SBP结构域C端存在由大量的碱性氨基酸残基组成的高度保守的核定位信号(nuclear localization signal, NLS)[21]。
2.3 拟南芥和普通烟草SBP转录因子家族的系统发育分析
为了揭示普通烟草SBP转录因子家族的进化关系,参照Guo等[2]的方法,使用拟南芥和普通烟草SBP转录因子家族成员SPB结构域氨基酸序列,利用MEGA和PhyML分别构建了邻接树(图2)和极大似然树(图3),两者具有高度相似的拓扑结构。构建的邻接树显示,拟南芥和普通烟草SBP转录因子家族成员被分为8个亚家族每一个亚家族都包含了两个物种中至少一个的SBP转录因子家族成员,这说明了SBP转录因子家族成员的分化时间可能早于两个物种的分化时间。其中亚家族I-VIII含有拟南芥SBP转录因子家族成员的个数分别为:1、2、2、3、1、3、3和1;含有普通烟草SBP转录因子家族成员的个数分别为:7、2、2、4、3、4、8和2。普通烟草SBP转录因子家族成员的数量约为拟南芥的两倍,但是拟南芥和普通烟草在不同亚家族中SBP转录因子家族成员的倍数关系差异较大,这说明在拟南芥和普通烟草分化后,SBP各个亚家族可能发生了不同的进化事件。此外,在亚家族I中普通烟草有7个SBP转录因子家族成员,而拟南芥只有一个SBP转录因子家族成员位于该分支中,说明在普通烟草物种形成后,该亚家族内SBP转录因子家族成员可能经历了多次复制事件。在8个亚家族中,亚家族VIII与其他亚家族的亲缘关系较远,形成一个较为独立的分支,这与以前的报道是一致的[22]。进一步分析发现,亚家族8中3个成员AtSPL7、NtSPL6和NtSPL32在SBP结构域中的N段锌指结构为C4,而其他亚家族所有成员的N端锌指结构均为C3H。

图2 普通烟草、拟南芥SBP转录因子家族成员邻接系统发育树Fig. 2 Neighbor-joining phylogenetic tree of SBP family in N. tabacum and A. thaliana
2.4 拟南芥和普通烟草SBP基因家族的基因结构分析
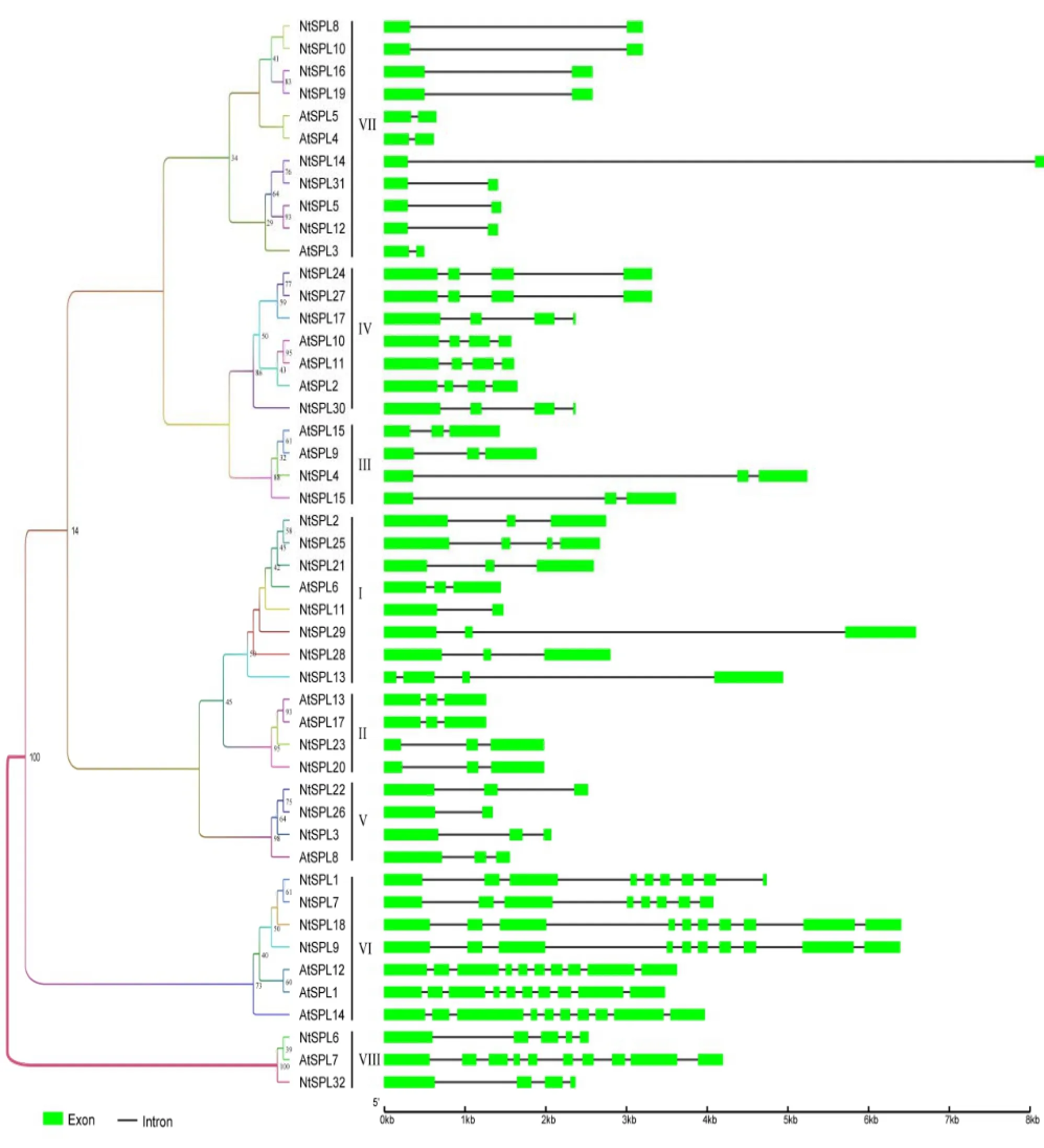
图3 拟南芥、番茄SBP转录因子家族极大似然系统发育树及基因结构Fig. 3 The Maximum-Likelihood phylogenetic tree and gene structure of SBP family members in N. tabacum and A. thaliana
为了分析普通烟草SBP基因的结构特征,利用GSDS构建了拟南芥和普通烟草SBP基因家族成员的基因结构图(图3)。内含子-外显子结构图表明,不同亚家族的SPL基因之间内含子数目变异比较大,最多的有9个内含子,最少的有1个内含子。在亚家族I、II、III和V中,大部分成员含有2个内含子;亚家族IV中,大部分成员含有3个内含子;亚家族VII中,大部分成员含有1个内含子;亚家族VI和VIII中,大部分成员含有9个内含子。同一亚家族内,拟南芥与普通烟草直系同源的SPL基因,其外显子、内含子的组织结构高度相似并区别于其他亚家族内的SPL基因,这同样说明了SBP基因家族成员的分化时间可能要早于拟南芥和普通烟草的物种分化时间。进一步分析发现,同一亚家族内,拟南芥和普通烟草的旁系同源SPL基因的内含子的组织结构也相对一致,这也从侧面反映了系统发育分析的可靠性。
2.5 拟南芥和普通烟草SBP转录因子家族的蛋白保守结构域分析
利用序列保守结构域识别工具MEME,对拟南芥SBP转录因子和预测的普通烟草SBP转录因子的蛋白序列的保守结构域进行分析(图4)。分析结果表明,所有的拟南芥和普通烟草SBP转录因子家族成员均含有79个氨基酸残基组成的SBP结构域(Motif 1)。除了SBP结构域,如Motif 2等一些新的保守结构域被鉴定出来,同一亚家族内的SBP转录因子家族成员的保守结构域的种类、数目和组织形式具有很强的一致性,同样从侧面反映了系统发育分析的可靠性。在亚家族VII内,拟南芥和普通烟草的SBP转录因子家族成员除了SBP结构域外没有其他的保守结构域,而亚家族VI内存在Motif2、Motif3、Motif5和Motif7等其他亚家族中没有或不典型的保守结构域,这说明亚家族VI的成员可能具有其他亚家族成员所不具有的功能。此外,亚家族I、II、III和IV中拟南芥有8个成员、普通烟草有13个成员有一个位于Motif 8中的保守基序ALSLLS,据文献报道,其对应的mRNA序列可能是mRNA156的靶序列[8]。
2.6 普通烟草SBP基因家族的基因表达分析
使用TN90转录组测序(RNA-seq)数据进行分析并提取SBP基因家族成员的基因表达数据,使用R语言将表达数据可视化。从图5中可以看出,SBP基因家族成员在根、茎、幼叶、成熟叶、衰老叶、幼花等6个组织中均有表达。进一步的分析发现,NtSPL基因的表达具有一定的组织特异性。所有NtSPL基因在幼花中都有表达,而且绝大多数成员还具有较高的表达量,这与拟南芥AtSPL基因在花中高度表达的报道相一致,表明SPL基因在植物的花发育中起重要作用[23]。进一步的分析发现在根、成熟叶、老叶等组织中,有些NtSPL基因表达量很低,如根中的第VII亚家族基因,成熟叶和老叶中的第I、III、V亚家族基因表达量普遍较低。同一亚家族的NtSPL基因在同一组织中的表达具有差异,成员较多的亚家族如第Ⅰ、第Ⅶ亚家族,在同一组织如茎、衰老叶中表达差异较大。同一亚家族的NtSPL基因在不同组织中的表达差异较为明显,只有第Ⅵ亚家族的NtSPL基因在所检测的6个组织中都有较高的表达量。此外,有些NtSPL基因随着组织的发育而呈规律性的表达,如NtSPL2随着叶片的衰老表达量逐渐升高,NtSPL10在衰老叶中表达量显著增加,初步推测它们可能与植物的衰老进程相关。
2.7 普通烟草SBP转录因子家族GO注释分析
通过对普通烟草SBP蛋白序列进行GO注释发现,经鉴定普通烟草SBP转录因子家族成员的细胞组成(Cellular Component)、分子功能(Molecular Function)和所参与的生物学过程(Biological Process)与拟南芥SBP转录因子家族成员相似。GO注释分析结果(图6)表明,拟南芥和普通烟草的SBP蛋白均在细胞核中,发挥DNA结合和转录调节相关的功能,这与SBP转录因子家族成员作为转录因子的功能相一致。在细胞生物学过程方面,SBP转录因子家族成员的注释呈多样化。所有的拟南芥和普通烟草SBP蛋白
被注释为参与到花器官发育,这与SBP成员在花器官中有较高的表达量是相吻合的。大部分蛋白还参与到叶片、茎和花粉管等植物器官的生长发育过程中,一些蛋白参与植物的防御响应等过程中,这与已有的文献报道是相符的[2]。

图4 拟南芥、番茄SBP转录因子家族蛋白保守结构域分析Fig. 4 Conserved domains within different subgroups of SBP family members in N. tabacum and A. thaliana

图5 普通烟草SBP基因家族成员表达模式分析Fig. 5 Expression patterns of the SBP gene family members in N. tabacum
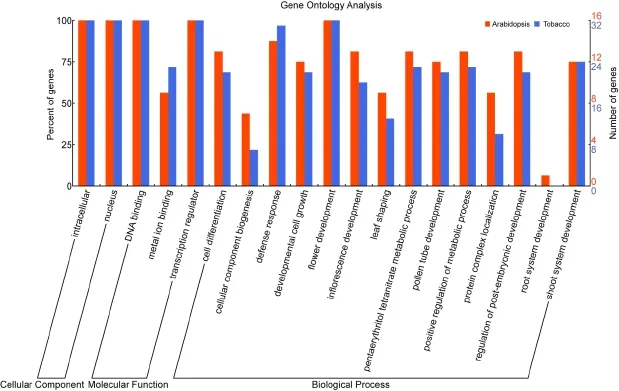
图6 拟南芥、普通烟草SBP蛋白GO注释Fig. 6 Gene ontology analysis of SBP proteins in N. tabacum and A. thaliana
3 讨 论
SBP基因家族所编码的具有植物特异性的转录因子具有许多重要的生物学功能,广泛参与植物生长发育调节、逆境胁迫响应及孢子、花和果实发育等诸多生命过程。本研究采用生物信息学手段,首次从普通烟草基因组中,鉴定出32个SBP转录因子家族成员,所有成员均含有保守的SBP结构域。通过对SBP结构域进行分析发现,所有的NtSPL均具有两个典型的锌指结构和一个位于C端的保守核定位信号。针对拟南芥和普通烟草SBP转录因子家族成员的蛋白序列,通过邻接法和极大似然法构建的系统发育树具有高度相似的拓扑结构。系统发育分析表明拟南芥和普通烟草的SBP转录因子家族成员被分成了8个亚家族,每一个亚家族都包含了至少一个拟南芥和普通烟草SBP转录因子家族成员,说明SBP转录因子家族成员的分化形成可能早于拟南芥和普通烟草的分化时间。通过对拟南芥和普通烟草SBP基因的结构特征分析发现,不同亚家族中拟南芥和普通烟草的SPL基因之间内含子数目变异较大。然而,通过比较拟南芥与普通烟草的直系同源SPL基因,发现其外显子、内含子的组织结构高度相似,这同样印证了SBP基因家族成员的分化形成时间可能要早于拟南芥和普通烟草的物种分化时间。通过对SBP转录因子家族成员结构域的分析发现,相较其他亚家族而言,亚家族VI内存在许多较为复杂的保守结构域。此外,普通烟草中有13个成员存在一个位于C端的保守基序ALSLLS,其对应的mRNA序列可能是mRNA156的靶序列。SBP基因家族表达分析发现,NtSPLs基因的表达具有一定的组织特异性,所有NtSPL基因在幼花中都有表达,而且绝大多数成员还具有较高的表达量。GO注释表明,普通烟草SBP转录因子家族成员注释为参与到花器官发育,这与烟草SBP基因家族成员在花器官中有较高的表达量是相吻合的。大部分SBP转录因子家族成员被注释为参与到叶片、茎和花粉管等植物器官的生长发育过程中,一些成员被注释为参与植物的防御响应等过程中,但具体功能有待于利用分子生物学等手段进行进一步的基因功能验证。
4 结 论
本研究利用普通烟草TN90基因组数据,通过生物信息学方法鉴定了普通烟草基因组编码的32个SBP转录因子家族成员,并将这些成员分为8个亚家族。通过分析转录组数据,发现SBP基因家族成员在花器官中的表达水平较高。GO注释结果表明,SBP家族在植物的器官发育、逆境防御等生物过程中发挥重要作用。
本研究通过生物信息学手段对SBP转录因子家族进行了鉴定和一些功能预测,但其具体的生物学功能以及调控机制,还需要通过基因克隆、表达分析及转基因等实验进行分析验证。
[1] Zhang S D, Ling L Z. Genome-wide identification and evolutionary analysis of the SBP-box gene family in castor bean[J]. PLoS One, 2014, 9(1): e86688.
[2] Guo A Y, Zhu Q H, Gu X, et al. Genome-wide identification and evolutionary analysis of the plant specific SBP-box transcription factor family[J]. Gene,2008, 418(1-2): 1-8.
[3] Cardon G H, Höhmann S, Nettesheim K, et al. Functional analysis of the Arabidopsis thaliana SBP-box gene SPL3: A novel gene involved in the floral transition[J]. The Plant Journal, 1997, 12(2): 367-377.
[4] Zhang Y, Schwarz S, Saedler H, et al. SPL8, a local regulator in a subset of gibberellin-mediated developmental processes in Arabidopsis[J]. Plant Molecular Biology, 2007, 63(3): 429-439.
[5] Unte U S. SPL8, an SBP-Box Gene That Affects Pollen Sac Development in Arabidopsis[J]. The Plant Cell Online, 2003, 15(4): 1009-1019.
[6] Stone J M, Liang X, Nekl E R, et al. Arabidopsis AtSPL14, a plant-specific SBP-domain transcription factor, participates in plant development and sensitivity to fumonisin B1[J]. The Plant Journal, 2005, 41(5): 744-754.
[7] Yamasaki K, Kigawa T, Inoue M, et al. A novel zincbinding motif revealed by solution structures of DNA-binding domains of Arabidopsis SBP-family transcription factors[J]. Journal of Molecular Biology,2004, 337(1): 49-63.
[8] Rhoades M W, Reinhart B J, Lim L P, et al. Prediction of plant microRNA targets[J]. Cell, 2002, 110(4): 513-520.
[9] Schwab R, Palatnik J F, Riester M, et al. Specific effects of microRNAs on the plant transcriptome[J]. Development Cell, 2005, 8(4): 517-527.
[10] Xie K, Wu C, Xiong L. Genomic organization,differential expression, and interaction of SQUAMOSA promoter-binding-like transcription factors and microRNA156 in rice[J]. Plant Physiology, 2006,142(1): 280-293.
[11] 陆莹,刘艳华,任民,等. 烟草CONSTANS同源基因的克隆与分析[J]. 中国烟草科学,2013,34(3):60-64
[12] 王倩,刘好宝. 烟草重要基因篇:2. 烟草钾吸收与转运相关基因[J]. 中国烟草科学,2014,35(2):139-142
[13] 刘贯山. 烟草重要基因篇:4. 烟草钙依赖蛋白激酶基因[J]. 中国烟草科学,2014,35(4):109-111.
[14] Jin J, Zhang H, Kong L, et al. PlantTFDB 3.0: a portal for the functional and evolutionary study of plant transcription factors[J]. Nucleic acids research, 2014, 42: D1182-D1187.
[15] Katoh K, Kuma K, Toh H, et al. MAFFT version 5: improvement in accuracy of multiple sequence alignment[J]. Nucleic Acids Research, 2005, 33(2): 511-518.
[16] Guindon S, Lethiec F, Duroux P, et al. PHYML Online—a web server for fast maximum likelihood-based phylogenetic inference[J]. Nucleic Acids Research,2005, 33(suppl 2): W557-W559.
[17] Abascal F, Zardoya R, Posada D. ProtTest: selection of best-fit models of protein evolution[J]. Bioinformatics,2005, 21(9): 2104-2105.
[18] Bailey T L, Boden M, Buske F A, et al. MEME SUITE: tools for motif discovery and searching[J]. Nucleic acids research, 2009, 37: W202-W208.
[19] Conesa A, Götz S, García-Gómez J M, et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 2005, 21(18): 3674-3676.
[20] Ye J, Fang L, Zheng H, et al. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research,2006, 34(suppl 2): W293-W297.
[21] Birkenbihl R P, Jach G, Saedler H, et al. Functional dissection of the plant-specific SBP-domain: overlap of the DNA-binding and nuclear localization domains[J]. Journal of Molecular Biology, 2005, 352(3): 585-596.
[22] Riese M, Höhmann S, Saedler H, et al. Comparative analysis of the SBP-box gene families in P. patens and seed plants[J]. Gene, 2007, 401(1): 28-37.
[23] 朱红霞,胡利宗,邓小莉,等. 大豆SBP基因家族的序列特征,表达及进化分析[J]. 东北农业大学学报,2012,43(7):26-33.
Genome-Wide Identification, Phylogenetic Analysis and Expression Profiling of the SBP Transcription Factor Family in Nicotiana tabacum
LIU Cheng1,2, LI Xiaoxu1,2, SU Yulong1,2, GUO Yongfeng1*
(1. Tobacco Research Institute of Chinese Academy of Agricultural Sciences, Qingdao 266101, China; 2. Graduate School of Chinese Academy of Agricultural Sciences, Beijing 100081, China)
The members of the SBP transcription factor family have been reported to play significant roles in regulating flower and fruit development as well as other biological processes. In this study, 32 SBP transcription factor family members were identified in Nicotiana tabacum with HMMER. Both Maximum-Likelihood phylogenetic tree and neighbor-joining tree were constructed and were found to possess similar topologies using the protein sequences of the SBP-domain. Results of phylogenetic analysis revealed that the SBP transcription factor family members could be classified into 8 subfamilies. The patterns of exon-intron structure and conserved domains in Arabidopsis and tobacco were consistent with the phylogenetic results. Transcriptome analysis showed that the expression patterns of NtSPLs were different in different tissue types and the expression level of NtSPLs in flower was found to be significantly higher than the other tissues. GO analysis suggested that as transcription factors, the SBP family members could be involved in a series of biological processes such as developmental regulation and defense response. The results of this study provide insight into the evolution of the SBP transcription factor family in Nicotiana tabacum and provide useful information for future research on these genes.
bioinformatics analysis; SBP transcription factor family; Nicotiana tabacum
S572.01
1007-5119(2015)04-0001-11
10.13496/j.issn.1007-5119.2015.04.001
农业部948项目“高通量植物衰老基因克隆技术(TASSEL-tagging系统)的引进、创新及利用”(2013-Z4)
刘 成(1988-),男,硕士研究生,研究方向为烟草分子育种。E-mail:liuch1221@163.com。*通信作者,E-mail:guoyongfeng@caas.cn
2015-03-20
2015-07-20

