海洋酸化对马氏珠母贝珍珠层形成的影响
董冰冰, 黄荣莲, 王庆恒, 郑 哲, 焦 钰, 邓岳文, 杜晓东
(广东海洋大学 水产学院 广东省珍珠养殖与加工工程技术研究中心, 广东 湛江 524088)
生态系统对地球环境的变化的应对, 以及对气候和人类社会的反作用是目前重大研究课题之一。陆生生物及生态系统应对环境变化的研究已有较多研究。例如应用开放式空气 CO2浓度增高(free-air CO2enrichment, FACE) 和开放式温度升高(free-air temperature increase, FATI)技术获得陆生性植物和群落应对环境变化的各种信息[1]。空气和海洋表面是紧密相关的, 过去观测到的大气变化与如今海洋中发生的变化类似。20世纪后期海洋表面无机碳水平以及温度比50~200年前显著升高[2]。当前海水表层的 CO2浓度(体积分数约 788×10–6)已达到工业革命前(280×10–6)的 2倍多, 相应的海水 pH 下降了 0.1;预计到21世纪末海水pH会继续下降0.3~0.4[3-5]; 随着大气CO2浓度升高, 海水pH预计在2300年可能还会降低 0.7[6]。CO2的增加影响海水的碳酸盐平衡系统, 减少CaCO3形成, 预测结果显示到2100年海洋钙沉积(钙化)将减少14%~30%[7]。研究表明, 矿化类群将遭受海洋环境变化的重大影响[8]。
生物矿化是在有机大分子(蛋白质、糖蛋白或多糖)调控下无机矿物在特定部位有序沉淀的过程, 其无机相受到有机相的精确调控[9]。贝壳是典型的生物矿化产物, 一般而言, 外套膜组织首先分泌角质层形成相对封闭的外套膜外液腔, 当腔内矿化相关的有机组分以及 Ca2+浓度达到一定的水平, 晶体开始形成并不断沉积形成贝壳。贝壳中的蛋白组分是形成不同的结晶及晶体形貌特征的关键要素[10]。目前已经鉴定获得贝壳基质蛋白有上百种, 如已经完成全基因组测序的长牡蛎(Crassostrea gigas)和帽贝(Lottia gigantea)分别鉴定获得239种[11]和569种贝壳蛋白[12]。马氏珠母贝是我国海水珍珠培育的主要品种, 养殖的“南珠”占我国海水珍珠产量的90%以上。马氏珠母贝贝壳有文石和方解石两种晶型, 分别构成了珍珠层和棱柱层两种结晶形态。普遍认为, 珍珠层和棱柱层分别由外套膜组织的中央膜区(mantle center, MC)和边缘膜区(mantle edge, ME)分泌形成[13]; 而人工育珠通过植入外套膜上皮组织小片,经细胞增殖形成封闭的珍珠囊, 从而分泌形成珍珠,因此珍珠也被称为“圆形贝壳”, 即贝壳中珍珠层与珍珠囊分泌的珍珠层晶体结构一致, 外套膜上皮与珍珠囊上皮组织的功能也被认为具有一定的相似性[14]。目前已经鉴定获得部分与珍珠层和棱柱层形成相关的特有功能蛋白, 如珍珠层相关的MSI60[15]、pif177[16]、nacrein[17]、pearlin[18]和棱柱层相关的 prismalin-14[19]、MSI30[20]、aspein[21]。RNA干扰实验证明Pif表达量的变化直接影响珍珠层晶体形貌[16,22]。Nacrein蛋白在矿物中的分布以及其高级结构显示其促进晶体的成核[23]。Pearlin属于文石层间基质蛋白, 能将钙离子呈递给正在成核的文石层, 参与珍珠层的形成[24]。
据报道, 海洋酸性环境变化对对马氏珠母贝的发育产生较大影响, 酸化实验组 D 型幼虫较对照组壳长和壳高明显偏小, 壳长增长缓慢[25]; 海水pH和温度变化对马氏珠母贝珍珠质形成相关基因nacrein表达具有显著影响[26]。以上研究都未对贝壳晶体微结构是否发生变化做进一步的分析。本研究模拟未来海洋酸化环境, 应用实时荧光定量 PCR(Real-time PCR)技术检测马氏珠母贝外套膜中央区pif177、nacrein及pearlin基因的表达, 结合扫描电子显微镜技术(SEM)观察贝壳珍珠层中晶体形态结构, 分析和探讨海洋酸化对马氏珠母贝珍珠层形成的影响以及在基因表达水平上的响应, 以期为揭示海洋环境变化对珍珠贝生物矿化的影响和珍珠养殖业的健康发展提供基础数据。
1 材料与方法
1.1 实验材料
实验用马氏珠母贝为项目组培育的马氏珠母贝选系F5, 于2013年7月取自广东湛江徐闻县西连镇承梧村的养殖基地(养殖海区pH 8.2, 盐度31), 清洗表面附着物后带回实验室, 在65 L水箱中驯养。实验海水为取自养殖海区的天然海水, 过滤后使用,驯养过程中每天定时换水, 驯养 2 d, 整个驯养过程水温为28℃±0.5℃, pH为8.20±0.1, 盐度为31。驯化结束后, 取规格一致, 壳高5~6 cm个体进行实验。
1.2 实验方法
1.2.1 实验设计
实验设对照组(CG)和2个实验组(E)。其中, CG以上述天然海水(pH 8.2)养殖马氏珠母贝; 实验组根据模型预测的 2100年与 2300年的 CO2的积累浓度[4-6,27], 分别调节海水 pH为 7.7(实验一组, E1)和7.4(实验二组, E2)。pH调节参考Reynaud等[28]和其他研究的方法[8,29], 通过向海水中充入高纯 CO2气体加以控制, 使用沃米特pH酸碱度监测仪实时监测每个水族箱中pH的变动情况, 以保持各组pH的稳定。每组用65 L收纳箱作为实验容器, 装入30 L曝气一天的海水; 每组10个个体, 养殖72 h后取样测定。实验温度为 28℃±0.5℃, 盐度为 31。实验设 3个平行组。
每个实验组中随机取 5个个体, 取外套膜组织的中央膜区, 液氮保存, 提取 RNA, 用于荧光定量PCR分析; 贝壳保存, 用于扫描电镜观察分析珍珠层结晶层形态特征。
1.2.2 实时荧光定量 PCR(Real-time PCR)
荧光定量引物采用引物设计软件Primer Premier 5.0设计, 设计原则跨一个内含子, 以避免模板中DNA 杂质对扩增结果产生干扰,pif177、nacrein、pearlin以及内参基因GAPDH的荧光定量引物设计参考NCBI已收录的马氏珠母贝相应的基因序列[30]。
马氏珠母贝中央膜总RNA的提取按照Invitrogen Trizol试剂盒说明书进行, 然后进行cDNA第一链的合成。实时荧光定量PCR按照SYBR Premix ExTaqTMⅡ(Tli RNaseH Plus) 说明书要求, 在ABI 7500 Real Time PCR System上进行, 其中每个样品的目的基因和内参基因分别进行 3 次重复, PCR反应体系:SYBR Premix ExTaqTM5 μL, 引物 F 0.5 μL, 引物 R0.5 μL, cDNA 0.5 μL, RNase-free H2O 3.5 μL, , 反应体系总体积为10 μL。反应程序采用三步法程序: 95℃预变性 7 min; 95 ℃ 15 s, 57 ℃ 15 s, 72 ℃ 35 s,40个循环, 采集荧光信号; 72~95 ℃获得熔解曲线。

表1 荧光定量PCR所用的引物序列Tab.1 Primer sequences used for Real-time PCR
利用SPSS 13.0软件采用t-检验分别比较两个实验组与对照组间的基因表达量差异, 显著性水平设为P<0.05。
1.2.3 扫描电镜样品制备
取上述经酸化海水培养的马氏珠母贝, 将软体部取出后用超纯水清洗贝壳, 在室温阴凉处干燥。然后用切割机将贝壳生长区部分(珍珠层与棱柱层的交界)切成1cm×1cm 大小的块, Hitachi E1010离子溅射仪镀膜, Hitachi S-4800扫描电子显微镜观察贝壳内表面珍珠层形貌。
2 结果与分析
2.1 贝壳珍珠层形态比较
利用扫描电镜观察三个组的贝壳内表面珍珠层差异, 观察结果如图1所示。
CG组(pH 8.2)贝壳珍珠层表面具有清晰的呈阶梯状规则排列的显微结晶层; 每层边沿为花边状,纹理间距平均值 18 μm; 在每层边沿均有不少游离的文石结晶, 晶体线性长度平均值为 2.1 μm; 结晶形态为六边形的薄板块或不规则多边形的扁平板块;结晶表面光滑平整, 晶体排列紧密, 粒间的界限清晰, 颗粒边缘平直(图1A-B)。E1组(pH 7.7)贝壳珍珠层的阶梯规则排列与板块大小与CG组没有差别, 近似六变形, 但边界较为圆滑, 板块边界有少量的不规则生长(图1C-D)。E2组(pH 7.4)贝壳珍珠层表面不具有阶梯状排列的显微结晶层; 边沿不清晰, 结晶形态极不规则, 结晶形态不完整, 晶体板块凹凸不平, 极少观察到板块间连成片(图1E-F)。

图1 贝壳珍珠层扫描电镜照片Fig.1 SEM images of the surface of the nacreous layer of the shell
2.2 海水酸化对pif177基因表达的影响
利用 Real-time qPCR技术检测了马氏珠母贝pif177基因在外套膜中央区的表达, 结果如图2所示,随海水pH下降,pif177基因相对表达量呈下降趋势,在 CG(pH 8.2)、E1(pH 7.7)和E2(pH 7.4)相对表达量的分别为 2.32±0.59、1.86±0.55和 1.19±0.29。其中, 与对照组比较, 实验二组 pif177基因的表达出现了显著下调(P<0.05)。

图2 pif177基因在马氏珠母贝外套膜中央区的表达Fig.2 Expression levels of pif177 in MC of P.martensii
2.3 海水酸化对nacrein基因表达的影响
如图3所示, 在海水pH8.2~7.4之间,nacrein基因在马氏珠母贝外套膜中央区的表达量随 pH降低而降低, 与pif177基因的变化趋势类似。CG(pH 8.2)、E1(pH 7.7)和E2(pH 7.4)nacrein基因相对表达量的具体数值分别为 16.49±2.00、15.06±4.05和 3.71±1.99。其中, 与CG比较, E2组的nacrein基因的表达出现了显著下调(P<0.05)。

图3 nacrein在马氏珠母贝外套膜中央区的表达Fig.3 Expression levels of nacrein in MC of the mantle of P.martensii
2.4 海水酸化对pearlin基因表达的影响
如图4所示, 随着海水pH的逐渐下降,pearlin基因在外套膜中央区的表达整体趋势与pif177和nacrein基因基本一致: E1(pH 7.7)与CG(pH 8.2)之间无显著性差异, E2相对于CG显著下调(P<0.05)。CG、E1和 E2(pH 7.4)nacrein基因相对表达量分别为7.33±1.77、8.24±1.85 和 4.49±0.94。
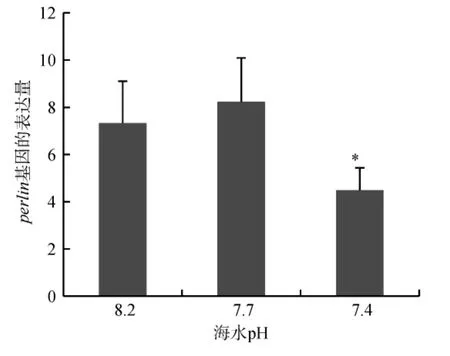
图4 pearlin在马氏珠母贝外套膜中央区的表达Fig.4 Expression levels of pearlin in MC of P.martensii.
3 讨论
3.1 酸性条件下珍珠层晶体形貌特征变化
海洋生物矿化形成的 CaCO3主要以文石和方解石结晶形式存在。而海洋酸化造成了文石和方解石饱和度的急剧下降, 这必然影响具有 CaCO3骨架(外壳)或生物钙化过程的海洋生物[31], 包括珊瑚虫[32]、软体动物[33-35]、棘皮动物[36]等。Talmage 和 Gobler通过实验室模拟海洋酸化发现, 生长于模拟工业革命前水体 CO2浓度(250×10–6)下的海湾扇贝(Argopecten irradians)和文蛤(Mercenaria mercenaria)具有生长速度快、变态速度快、贝壳厚而紧实的特点, 而生长在模拟 2100年 CO2浓度(>400×10–6)条件下的海湾扇贝和文蛤不仅存活率降低、生长速度减慢, 贝壳面积显著减小并呈现溶蚀状态[33]。Hahn等[36]研究了CO2等因素对地中海贻贝(Mytilus galloprovincialis)贝壳超微结构的影响, 发现随着贝壳的沉淀, 贝壳的超微结构在酸性的海水中出现了紊乱的结构; Melzner等[37]通过体视显微镜和扫描电镜观察在低浓度的藻类饵料和高浓度 CO2环境下的紫贻贝的贝壳内侧文石层发现, 贝壳内部珍珠层的形貌都有了一定程度的改变。
外套膜是软体动物参与贝壳形成和珍珠形成的重要器官。在正常生理状态下, 外套膜分泌有机质诱导珍珠层的沉积, 所形成的成熟珍珠板块的形貌特征为规则六边形, 生长端小板片堆砌规则, 小板块直径1.3~1.7 μm, 粒径均匀[38]。而在外界环境或人为干扰的情况下, 如水压、食性以及pCO2变化时, 珍珠层出现不规则生长或“溶解”(dissolution)现象, 微观的形貌特征表现为不规则的六边形或有“缺刻”(bitten crystal)现象[39-40]。本研究结果表明, 在海水pH值至 7.4时, 对马氏珠母贝的珍珠层的沉积和晶体的形貌有着显著影响。由此推测, 海水的酸化将极有可能会影响到海区养殖珍珠的质量。
3.2 酸性条件下与珍珠层形成相关的矿化基因的表达变化
Fabry[41]认为海洋生物体应对机体本身生理状态下产生的 CO2和氢离子具有相同的机制, 即(1)构建细胞内部和外部溶液的被动式缓冲体系(passive buffering); (2)运输和交换相关离子; (3)具有呼吸色素分子的种类通过血液运输CO2; (4)降低代谢。一些在高 CO2浓度下生存的物种通过长期进化, 体内的缓冲、离子交换和 CO2运输能力上都获得加强[42]。通常认为, 软体动物的有壳种类通过贝壳的溶解,Ca2+重新回到体液中, 从而缓冲由于过多 CO2而导致的体内pH的下降。值得注意的是, 从珍珠层的材料学性能而言, 体内或体液中pH下降并不是导致贝壳结晶形态变化的直接原因, 贝壳矿化相关的蛋白和酶的变化才是关键因素。本研究利用 Real-time PCR技术检测了与珍珠层形成相关的3个矿化基因:pif177、nacrein及pearlin, 其中pif177[16]和pearlin[18]与文石晶体成核相关;nacrein含碳酸酐酶结构域[17]。在外套膜的MC区,pearlin的表达均表现为先升高后下降的趋势。其中, 当养殖海水pH由8.2下降到7.7,pif177与nacrein在 MC区的表达呈下降的趋势,pearlin则略为上升, 但三者都与对照组无显著差异;下降至pH7.4时, 与空白对照相比,nacrein则出现显著性下调(P<0.05)。nacrein的表达变化与刘文广和何毛贤的研究结果相似[26]。在其他低等无脊椎动物中, 矿化基因的表达同样受海洋酸性环境变化的影响。Todgham和Hofmann通过分析比较正常海水和酸化海水培养的紫球海胆(Strongylocentrotus purpuratus)浮游幼体转录组发现, 在海洋酸化条件下(pH7.96; pH7.88), 紫球海胆浮游幼体中有23个参与生物矿化的相关基因的表达发生了显著变化[43]。
3.3 珍珠层形成过程中矿化体系的动态平衡与对酸化环境的响应
综合以上研究, 海洋酸化对马氏珠母贝贝壳珍珠层的形成的影响是十分明显的, 在一定程度上会影响珍珠产量和质量的提高。在pH 7.7处理组海水pH值接近我国南海海区的 pH正常变化范围(7.5~8.5)[44]中, 珍珠层的晶体形貌和矿化基因的表达未发生明显变化; 而 pH7.4处理组无论在珍珠层晶体形貌还是矿化基因的表达都发生了显著变化,说明在此酸性条件下, 马氏珍珠贝已经通过调节自身的矿化系统来应对这一环境变化。对于正常状态下稳定的矿化体系, 存在正向(CaCO3结晶)和反向(结晶 CaCO3溶解)调控体系[45]。当外界环境变化或人为干扰的情况下, 矿化正向被抑制, 如本研究中的碳酸酐酶家族成员nacrein表达下调, 从而降低了CO2向的转化效率, 珍珠质特有基质蛋白pif177和pearlin基因的下调, 对Ca2+的利用将减少,导致珍珠层出现不结晶或异常结晶现象, 以此来缓解体内pH的升高。另一方面, 已有报道显示, 水温在 11℃以下或是 29℃以上时, 人工培育的珍珠会出现溶解现象, 珍珠质的分泌停滞[46]。从珍珠层的材料学性能及能量学角度分析, 与珍珠层形成相似, 晶体溶解应当需要酶等相关蛋白的参与。遗憾的是矿化体系的反向机制的研究相对滞后。
如何提高珍珠贝的珍珠层质量是珍珠产业的关键环节。本实验的研究结果显示, 酸化条件下, 在短期内已对珍珠层的质量产生了一定的影响。对于海区养殖品种而言, 长期处于酸性环境下, 珍珠层所受影响以及生物体本身适应性等问题, 都将是未来关注的焦点。系统的工作已经在其他海洋低等生物中开展。Lohbeck等[47]报道, 海洋球石藻经过约500次无性繁殖后, 生存状态都变差, 但处于高浓度二氧化碳条件下的藻类却表现出更高的生长速率以及钙化物质的部分复原。Pespeni等[48]报道紫球海胆(S.purpuratus)生长状况良好, 并未出现不良状况, 经研究发现其基因组发生了大量变异, 说明其以进化的方式适应了海洋酸化所带了的环境变化。因此, 有必要更进一步系统深入研究海洋酸化对珍珠层产生影响的机理, 为珍珠养殖业乃至整个海洋贝类养殖业的健康发展提供必要的理论基础。
[1] Nijs I, Ferris R, Blum H, et al.Stomatal regulation in a changing climate: a field study using free air temperature increase (FATI) and free air CO2enrichment(FACE)[J].Plant, Cell & Environment, 1997, 20(8):1041-1050.
[2] Levitus S, Antonov J I, Boyer T P, et al.Warming of the world ocean[J].Science, 2000, 287(5461): 2225-2229.
[3] Solomon S, Qin D, Manning M, et al.The physical science basis[C]//Contribution of working group I to the fourth assessment report of the intergovernmental panel on climate change.Cambridge: Cambridge University Press, 2007: 235-337.
[4] Caldeira K, Wickett M E.Ocean model predictions of chemistry changes from carbon dioxide emissions to the atmosphere and ocean[J].Journal of Geophysical Research: Oceans , 2005, 110(C9): 1-12.
[5] Wikfors G H, Krome C.Ocean acidification and molluscan hemocytes: Basis and rationale for experimental studies[J].Journal of Shellfish Research,2009, 28(3): 658-659.
[6] Caldeira K, Wickett M E.Anthropogenic carbon and ocean pH[J].Nature, 2003, 425(6956): 365.
[7] Kleypas J A, Buddemeier R W, Archer D, et al.Geochemical consequences of increased atmospheric carbon dioxide on coral reefs[J].Science, 1999,284(5411): 118-120.
[8] Orr J C, Fabry V J, Aumont O, et al.Anthropogenic ocean acidification over the twenty-first century and its impact on calcifying organisms[J].Nature, 2005,437(7059): 681-686.
[9] 崔福斋.生物矿化[M].北京: 清华大学出版社.2007:54-61.
[10] Marin F, Le Roy N, Marie B.The formation and mineralization of mollusk shell[J].Frontiers in Biosciencs, 2012, 4(sup1y 799), 1099-1125.
[11] Zhang G F, Fang X D, Guo X M, et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature, 2012, 490(7418): 49-54.
[12] Mann K, Edsinger-Gonzales E, Mann M.In-depth proteomic analysis of a mollusc shell: acid-soluble and acid-insoluble matrix of the limpetLottia gigantea[J].Proteome Science, 2012, 10(1): 28.
[13] Takeuchi T, Endo K.Biphasic and dually coordinated expression of the genes encoding major shell matrix proteins in the pearl oysterPinctada fucata[J].Marine Biotechnology, 2006, 8(1): 52-61.
[14] Liu X, Li J, Xiang L, et al.The role of matrix proteins in the control of nacreous layer deposition during pearl formation[J].Proceedings: Biological Sciences, 2012,279(1730): 1000-1007.
[15] Sudo S, Fujikawa T, Nagakura T, et al.Structures of mollusk shell framework proteins[J].Nature, 1997,387(6633): 563-564.
[16] Suzuki M, Saruwatari K, Kogure T, et al.An acidic matrix protein, Pif, is a key macromolecule for nacre formation[J].Science, 2009, 325(5946): 1388-1390.
[17] Miyamoto H, Miyoshi F, Kohno J.The carbonic anhydrase domain protein nacrein is expressed in the epithelial cells of the mantle and acts as a negative regulator in calcification in the molluscPinctada fucata[J].Zoological science, 2005, 22(3): 311-315.
[18] Miyashita T, Takagi R, Okushima M, et al.Complementary DNA cloning and characterization of pearlin, a new class of matrix protein in the nacreous layer of oyster pearls[J].Marine Biotechnology, 2000,2(5): 409-418.
[19] Suzuki M, Murayama E, Inoue H, et al.Characterization of Prismalin-14, a novel matrix protein from the prismatic layer of the Japanese pearl oyster (Pinctada fucata)[J].Biochemical Journal, 2004,382(1): 205-213.
[20] Tsukamato D, Sarashina I, Endo K.Structure and expression of an unusually acidic martix protein of Pearl oyster shells[J].Biochemical Biophysis ResearchCommun, 2004, 320: 1175-1180.
[21] Hammond S M, Bernstein E, Beach D, et al.An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells[J].Nature, 2000,404(6775): 293e5.
[22] Xiaotong WANG, Xiaorui SONG, Tong WANG, et al.Evolution and functional analysis of the Pif97 gene of the Pacific oysterCrassostrea gigas[J].Current Zoology , 2012, 59 (1): 109-115.
[23] Norizuki M, Samata T.Distribution and function of the nacrein-related proteins inferred from structural analysis[J].Marine Biotechnology, 2008, 10(3): 234-241.
[24] Montagnani C, Marie B, Marin F, et al.Pmarg-Pearlin is a Matrix Protein Involved in Nacre Framework Formation in the Pearl OysterPinctada margaritifera[J].Chembiochem, 2011, 12(13): 2033-2043.
[25] 何盛毅, 林传旭, 何毛贤, 等.海洋酸化对马氏珠母贝胚胎和早期幼虫发育的影响[J].生态学杂志, 2011,30 (4): 747-751.
[26] W Liu, X Huang, J Lin, et al., Seawater acidification and elevated temperature affect gene expression patterns of the pearl oysterPinctada fucata[J].PloS One, 2012, 7(3): e33679.
[27] Guinotte J M, Fabry V J.Ocean acidification and its potential effects on marine ecosystems[J].Annals of the New York Academy of Sciences, 2008, 1134(1):320-342.
[28] Reynaud S, Leclercq N, RomaineLioud S, et al.Interacting effects of CO2partial pressure and temperature on photosynthesis and calcification in a scleractinian coral[J].Global Change Biology, 2003,9(11): 1660-1668.
[29] Rodolfo-Metalpa R, Martin S, Ferrier-Pagès C, et al.Response of the temperate coral Cladocora caespitosa to mid-and long-term exposure topCO2and temperature levels projected for the year 2100 AD[J].Biogeosciences, 2010, 7(1): 289-300.
[30] Dheda K, Huggett J F, Bustin S A, et al.Validation of housekeeping genes for normalizing RNA expression in real-time PCR[J].Biotechniques, 2004, 37(1): 112-119.
[31] Feely R A, Sabine C L, Lee K, et al.Impact of anthropogenic CO2on the CaCO3system in the oceans[J].Science, 2004, 305(5682): 362-366.
[32] Nakamura M, Ohki S, Suzuki A, et al.Coral larvae under ocean acidification: survival, metabolism, and metamorphosis[J].PLoS One, 2011, 6(1): e14521.
[33] Talmage S C, Gobler C J.Effects of past, present, and future ocean carbon dioxide concentrations on the growth and survival of larval shellfish[J].Proceedings of the National Academy of Sciences, 2010, 107(40):17246-17251.
[34] Nienhuis S, Palmer A R, Harley C D.Elevated CO2affects shell dissolution rate but not calcification rate in a marine snail[J].Proceedings of the Royal Society B:Biological Sciences, 2010, 277(1693): 2553-2558.
[35] Dupont S, Ortega-Martínez O, Thorndyke M.Impact of near-future ocean acidification on echinoderms[J].Ecotoxicology, 2010, 19(3): 449-462.
[36] Hahn S, Rodolfo-Metalpa R, Griesshaber E, et al.Marine bivalve geochemistry and shell ultrastructure from modern low pH environments[J].Biogeosciences Discussions, 2011, 8(5): 10351-10388.
[37] Melzner F, Stange P, Trübenbach K, et al.Food supply and seawaterpCO2impact calcification and internal shell dissolution in the blue musselMytilus edulis[J].PloS One, 2010, 6(9): e24223.
[38] 张刚生, 谢先德, 王德强, 等.我国主要育珠贝 (蚌)贝壳珍珠层的扫描电子显微镜研究[J].热带海洋学报, 2003, 22(1): 55-61.
[39] Kadar E, Checa A G, Oliveira A, et al.Shell nacre ultrastructure and depressurisation dissolution in the deep-sea hydrothermal vent musselBathymodiolus azoricus[J].Journal of Comparative Physiology B,2008, 178(1): 123-130.
[40] Melzner F, Stange P, Trübenbach K, et al.Food supply and seawaterpCO2impact calcification and internal shell dissolution in the blue musselMytilus edulis[J].Plos One, 2011, 6(9): e24223.
[41] Fabry V J, Seibel B A, Feely R A, et al.Impacts of ocean acidification on marine fauna and ecosystem processes[J].ICES Journal of Marine Science: Journal du Conseil, 2008, 65(3): 414-432.
[42] Seibel B A, Walsh P J.Potential impacts of CO2injection on deep-sea biota[J].Science, 2001,294(5541): 319-320.
[43] Todgham A E, Hofmann G E.Transcriptomic response of sea urchin larvaeStrongylocentrotus purpuratusto CO2-driven seawater acidification[J].Journal of Experimental Biology.2009, 212(16): 2579-2594.
[44] 黄自强, 张克, 许昆灿, 等.中华人民共和国国家标准海水水质标准 GB 3097-1997.1997.
[45] Wibur K M, Saleuddin A.Shell formation[M].New York: Academic Press, 1983, 235-287.
[46] 和田浩二.珍珠的研究[M].日本东京: 珍珠出版社.1999.
[47] Lohbeck K T, Riebesell U, Reusch T B H.Adaptive evolution of a key phytoplankton species to ocean acidification[J].Nature Geoscience, 2012, 5(5): 346-351.
[48] Pespeni M H, Sanford E, Gaylord B, et al.Evolutionary change during experimental ocean acidification[J].Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(17): 6937-6942.

