我国沿海大竹蛏(Solen grandis)野生群体及增殖放流群体遗传多样性的ISSR分析*
曹 奕 吴洪喜 陈爱华① 吴杨平 张 雨王 超 张志伟 姚国兴 蔡永祥
(1.江苏省海洋水产研究所 江苏省海洋经济贝类研究开发中心 南通 226007;2.浙江省海洋水产养殖研究所 浙江省近岸水域生物资源开发与保护重点实验室 温州 325005)
大竹蛏(Solen grandis)隶属瓣鳃纲、竹蛏科、竹蛏属,在渤海辽东湾与莱州湾、南海北部湾、黄海朝鲜湾、海州湾及南部海域均有分布。其个体较大,肉质鲜美,是我国重要的经济贝类。近年来,因大竹蛏市场需求扩大导致的过度采捕,以及海洋生态环境的恶化,造成了大竹蛏野生资源量的急剧减少,故需通过人工增养殖等手段来对大竹蛏资源进行保护。近十几年以来,江苏、山东等地均开展了对于大竹蛏人工繁育技术的研究(侯和要等,2004;陈爱华等,2009),并进行了规模不等的增殖放流活动;其中,江苏省海洋水产研究所以江苏启东大竹蛏群体为亲本育成幼苗放流至江苏蒋家沙海域的规模为最大,自 2007年来,共计放流4.5mm左右的苗种6.26×108ind,江苏大竹蛏资源量上升明显。然而,人工繁育群体的瓶颈效应和近交衰退现象较野生群体更为明显,在一定程度上会导致群体遗传多样性的改变。而遗传多样性水平与生长速度、抗病能力等生产性状密切相关(李俊清等,2006)。因此,对大竹蛏野生群体和增殖放流群体遗传多样性水平进行鉴定,一方面能够确保我国种质资源的可持续利用,另一方面能为后续的良种选育研究提供理论数据。
ISSR作为一种共显性、重复性好、稳定性高、价格低廉的分子标记,已被广泛运用在贝类遗传学研究中。目前国内对大竹蛏群体的研究主要采用SSR和AFLP等方法(乔洪金等,2012;张滔等,2013),本文通过该技术对我国沿海大竹蛏群体进行遗传多样性研究,评判增殖放流活动对野生群体遗传结构的影响,能够了解大竹蛏当前的种质资源状况,为合理保护该资源提供科学依据。
1 材料与方法
1.1 样品采集
2013年11月至2014年3月,通过实地采样获得广西北海、江苏启东、山东日照、辽宁营口、河北秦皇岛、江苏蒋家沙增殖放流区等处大竹蛏(采样信息见表1)。每个群体选取25个规格较为一致的健康个体,取其斧足,用于实验。
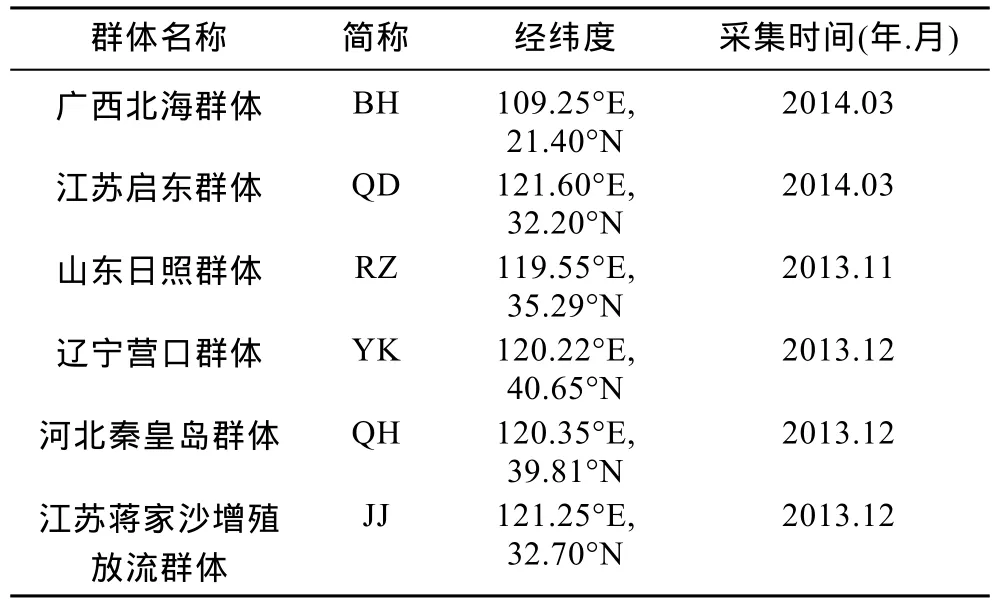
表1 大竹蛏样品采集时间及经纬度Tab.1 Sampling time,longitudes and latitudes of the S.grandis population
1.2 DNA提取
采用传统的酚-氯仿法进行提取。紫外分光光度计OD-1000测定样品DNA的浓度和纯度,并进行电泳验证,提取所得的DNA保存在–20°C环境下。
1.3 引物合成及筛选
由上海生工生物工程有限公司合成60条ISSR引物用于筛选,其序列均来自哥伦比亚大学(UBC)。PCR 反应体系含 50ng/μL 模板 DNA 1.0μL,10×PCR buffer (含 Mg2+)2.5μL,10mmol/L dNTP 1.0μL,10μmol/L引物 1μL,5U/μL Taq 酶 0.2μL,加 ddH2O 至 25μL;反应条件如下: 反应前94°C预变性4min,94°C变性45s,38—55°C 退火 45s,72°C 延伸 60s,共 35个循环,反应后72°C延伸7min。
1.4 ISSR-PCR
筛选得出10条结果较佳的引物对大竹蛏6个群体共 150个样品进行 ISSR-PCR扩增,产物进行2%琼脂糖电泳(120V,100mA,90min),EB染色后用Bio-Rad Doc XR凝胶成像系统拍照。对所得电泳图进行分析。电泳结果如图1所示。
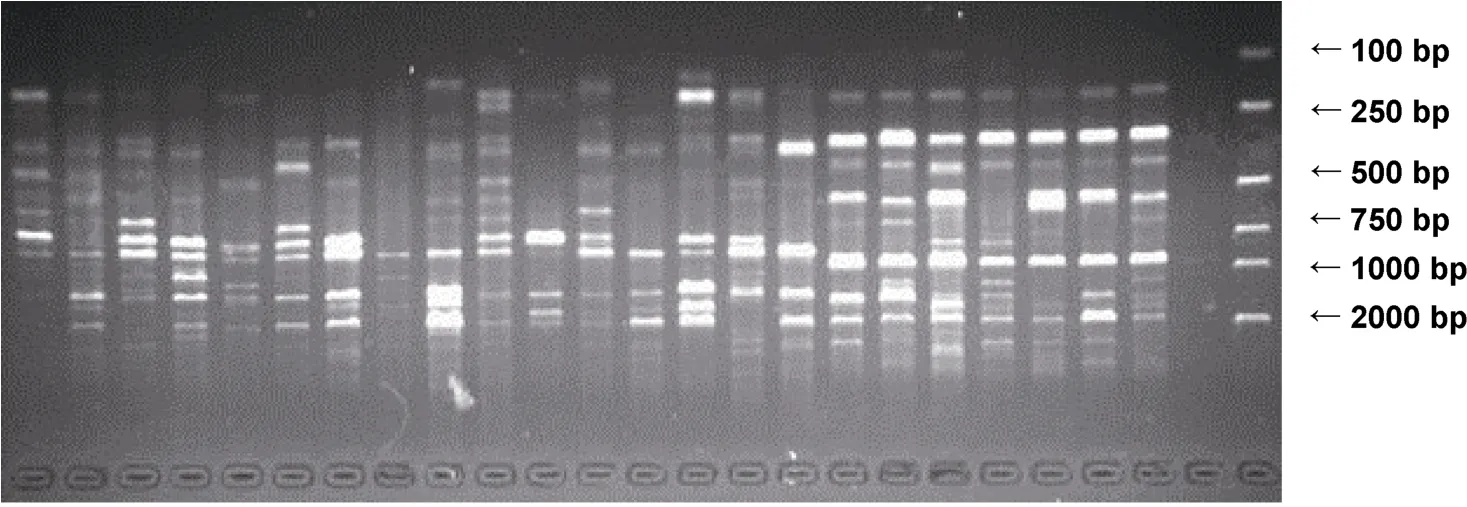
图1 UBC 824引物对BH群体的扩增结果Fig.1 The result of ISSR-PCR of primer UBC 824 to BH population
1.5 数据分析
获得ISSR电泳图谱后,用Quantity One软件进行分析,将所得结果进行归类。采用Popgene32软件进行遗传参数分析,并根据各群体遗传多样性参数计算了群体内基因多样性(Hs)、群体总基因多样性(Ht)、各群体间的遗传分化指数(Gst)(Gst=1–Hs/Ht)(Slatkin et al,1989)、遗传距离(D)(Nei,1978)。基于遗传距离,用Mega5.0软件构建6个群体及150个样品的 UPGMA聚类树。利用 Arlequin 3.5软件(Excoffier et al,2010)对所有群体间和群体内的遗传变异进行分子变异分析(AMOVA),计算群体间遗传分化系数(Φst)。
2 结果与分析
2.1 大竹蛏群体内的遗传分析
筛选得出的10条引物序列见表2,从所有6个群体中共扩增得到清晰位点 127个,其中多态性位点118个(PPB=92.91%)。5个野生群体的H值和I值分别在0.0982—0.1633和0.1603—0.2776之间(见表3),江苏蒋家沙增殖放流各项遗传多样性参数均低于亲本QD野生群体。
2.2 大竹蛏群体间的遗传分化
野生群体间 Gst=0.1065,增殖放流群体(JJ)与其亲本 QD群体的Gst=0.0235,所有群体的遗传分化系数 Gst=0.1370(见表 4)。野生群体两两间 Gst在0.0525—0.0857间,其中 BH群体与其余野生群体间Gst均在0.07以上(见表6)。

表2 筛选获得引物序列Tab.2 The nucleotide sequences of selected primer
而从AMOVA分析结果可以看出,Φst=0.1268,即在总变异中,12.68%的变异归因于大竹蛏群体间的变异,87.32%的变异归因于群体内的变异,群体间和群体内变异均显著(P<0.01)(见表5),该结果与分析所得的Gst值基本一致。
2.3 遗传距离与聚类分析
利用 Popgene32软件计算了大竹蛏各群体间的Nei’s遗传距离(见表6)。5个大竹蛏野生群体间遗传距离值变化范围为 0.0139—0.0387,而增殖放流群体(JJ)与其亲本QD群体的遗传距离仅为0.0088。
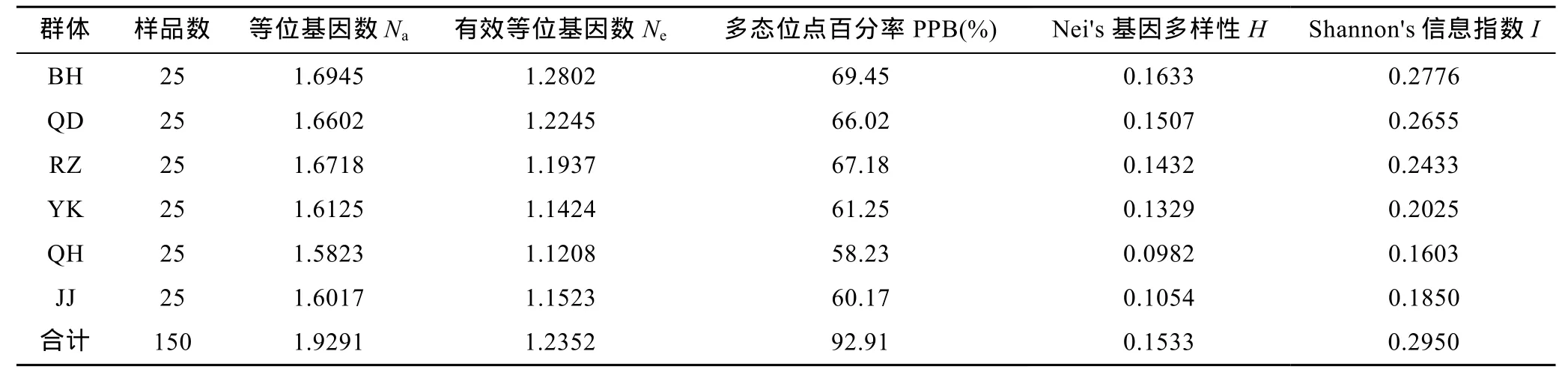
表3 大竹蛏6个群体遗传多样性参数Tab.3 Genetic diversity of the six S.grandis populations

表4 不同群体间的遗传分化分析Tab.4 Analysis of genetic differentiation among populations

表5 大竹蛏群体间和群体内分子变异的AMOVA分析结果Tab.5 Analysis of molecular variance (AMOVA)within/among S.grandis populations
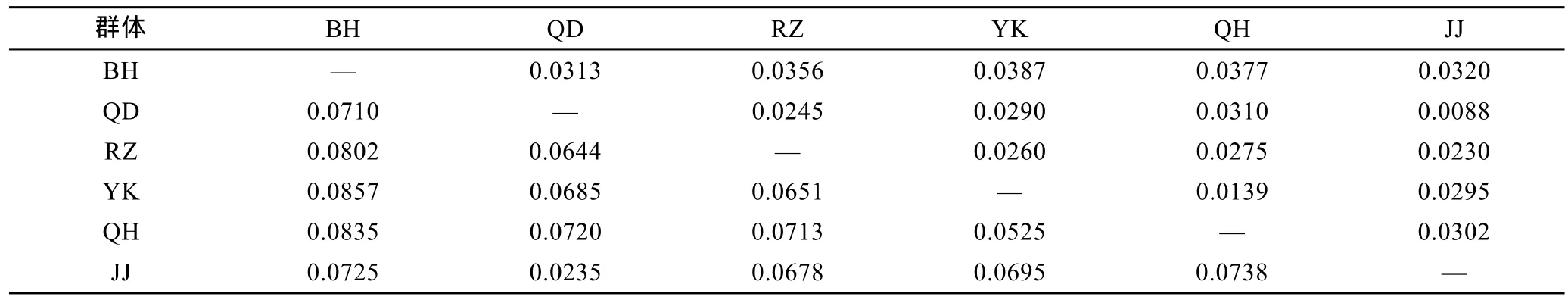
表6 大竹蛏6个群体间遗传距离和遗传分化系数Tab.6 Genetic distance and genetic diversity coefficient of the six S.grandis populations
根据实验结果计算所得的遗传距离,通过Mega5.0软件构建了UPGMA聚类树。6个大竹蛏群体的聚类结果见图2。QD群体与JJ群体首先聚为一支,接着与RZ群体聚为一支,QH群体与YK群体聚为另一支,最后与 BH群体聚在一起。150个个体的聚类结果见图3。由图3可见,6个群体中大部分个体各自独立汇聚成一支。只有少数个体出现在其它支系中,该情况QD、JJ群体中各有3个,YK、QH群体各有1个。从不同群体角度来看,个体间的聚类结果与群体间的聚类结果相一致。
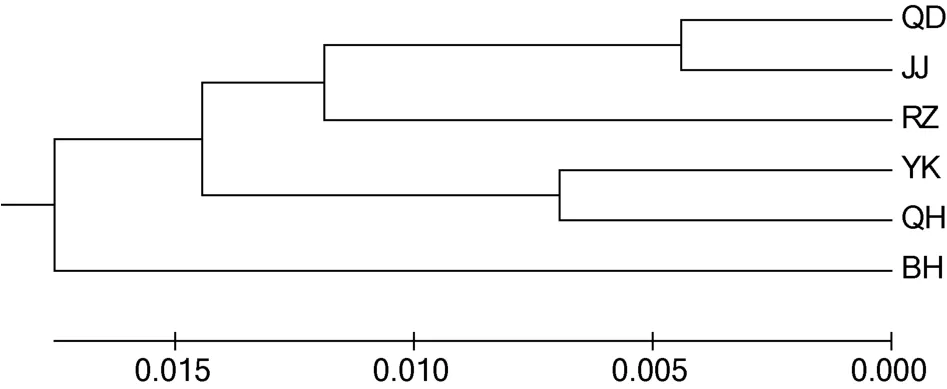
图2 大竹蛏野生群体和增殖放流群体的UPGMA聚类图Fig.2 The UPGMA phylogenetic tree of stock and wild S.grandis populations
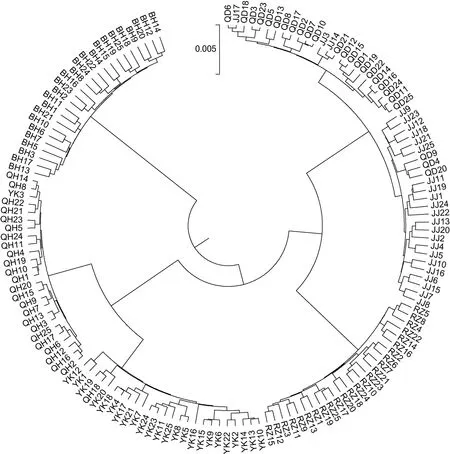
图3 大竹蛏150个个体的UPGMA聚类图Fig.3 The UPGMA dendrogram of 150 individuals from the six population of S.grandis
3 讨论
3.1 大竹蛏群体间的遗传多样性分析
表3显示,BH群体的PPB、H、I值分别为0.1633、0.2776,和69.45%,为5个野生群体的最高值,QH群体的相应指标则为 0.0982、0.1603和 58.23%,为 5个野生群体的最低值。QH群体遗传多样性水平的低下可能与所处海域有关。秦皇岛位于环渤海经济圈中心地带,陆域污染源较多,同时自身水动力条件较差决定了该海域自净能力有限,造成该区域种群长期遭受环境污染,从而使得种群数量减少或敏感性个体缺失,群体遗传多样性水平下降,大大降低了生物资源的利用率(Krane et al,1999)。
本研究还特别关注了江苏蒋家沙增殖放流群体,研究分析了增殖放流对群体的遗传结构的影响。结果表明蒋家沙增殖放流群体 Nei's基因多样性、Shannon's信息指数和多态位点百分率分别为0.1054、0.1850、60.17%,而其亲本群体即启东野生群体相应数值则为0.1507、0.2655、66.02%。两者相比而言,增殖放流群体的各项遗传多样性参数均有所下降。推测该结果与下列因素有关: 增殖放流群体亲本的选择区域狭窄,仅为启东吕四港附近约 100公顷的海域;且有效亲本使用量较低,每年使用亲本量大约为50kg。以上情况均可导致培育的苗种在人工繁殖过程中出现近交现象。为避免增殖放流群体的遗传多样性下降,可采取的措施有: 一是扩大亲本选择区域,二是提高亲本的数量和品质。
3.2 分子标记的选择及大竹蛏与其它贝类群体遗传多样性的比较分析
通过对几种基于 PCR技术的分子标记进行比较分析发现,与SSR标记相比,ISSR引物开发费用低,可以在不同物种间通用。与RAPD单引物相比,ISSR揭示的多态性百分率较高,而且其具有较高的退火温度,重复性好,稳定性高。相对于AFLP而言,ISSR费用较低,反应体系具有一定的通用性,操作简单,可广泛应用。综合考虑,本研究运用 ISSR标记进行研究相对较好。
表 7列举了相关海洋经济贝类分子标记的分析结果,将6个群体大竹蛏的多态位点百分率、Nei's 基因多样性指数与其比较,发现参数相近,说明在群体水平上大竹蛏具有较高的遗传多样性。
广布种贝类如缢蛏(S.constricta)、四角蛤蜊(M.veneriformis)、青蛤(Cyclina sinensis),和西施舌(Mactra antiquata)群体间遗传差异相对较大(Nei's多样性指数普遍相差 0.04以上)(表 7),而翡翠贻贝(Penera viridis)和马氏珠母贝(Pinctada martensi)等仅分布于东南沿海的贝类,其各个群体间遗传多样性差异相差甚微(马氏珠母贝各群体Nei's多样性指数仅相差0.0003)。由此可见,广布种比狭域分布的物种具有更多的遗传差异。本研究结果表明大竹蛏群体间遗传多样性水平差异较大(0.0651),符合其作为广布种的实际情况,为物种水平上的遗传变异提供了理论基础。
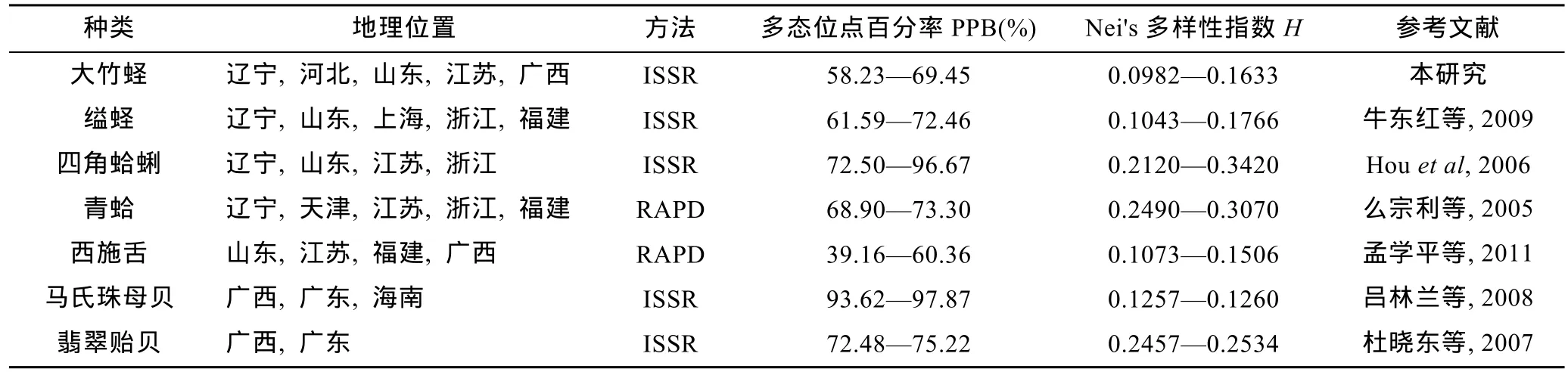
表7 大竹蛏与其它贝类的遗传多样性比较Tab.7 Comparison in genetic diversity between S.grandis and other clams
3.3 大竹蛏群体的遗传分化状况
分化系数在 0—0.05之间,群体间分化很弱,0.05—0.15之间表示中等分化,0.15—0.25之间表示分化大,大于 0.25表示分化极大(Wright,1978)。JJ群体与其亲本QD群体的Gst<0.05,表明增殖放流群体与其野生亲本群体并没有产生明显的遗传分化。而各野生群体间 Gst在 0.0525—0.0857间,均达到中等分化水平。所有群体间也达到了中等分化水平(0.1370)。该研究结果表明江苏启东大竹蛏种质状况较好,数年来的人工育苗、增殖放流没有使得启东群体产生明显的遗传变异,依然可以通过增殖放流手段实现对大竹蛏资源的保护。
AMOVA分析结果表明大竹蛏群体间和群体内分化均达到了极显著水平(P<0.01)。Φst=0.1268,即在总变异中,87.32%的变异来自群体内,该结果与分析所得的Gst值基本一致。
3.4 大竹蛏群体间的亲缘关系分析
遗传距离是衡量群体间亲缘关系的重要指标。Thorp研究认为,遗传距离D>0.15的两个群体,不可能是同一物种;同科属间 D=0.5—0.9;不同物种间D=0.2—0.8;同种不同群体D=0.03—0.2 (Thorp,1982)。6个大竹蛏群体的遗传距离大致在0.01—0.04范围之内,属于同种不同群体。聚类关系也反映了群体间亲缘关系的远近。本研究的聚类结果显示,南通的两个群体(QD,JJ)首先聚为一支,接着与同为南黄海区域的 RZ群体聚为一支,同为 渤海区域的 QH群体与YK群体则聚为另一支,最后与南海区域的 BH群体聚在一起。由此推断沿海大竹蛏群体间亲缘关系和地理距离存在相关性。而导致这一现象的原因是大竹蛏的浮游幼虫期只有 6—8d (陈爱华等,2009),自然水流的作用只能在较近范围内形成基因交流。这一原因同样能够解释个体的聚类结果中少数个体出现在邻近地理群体支系中的现象。
4 结论
本研究分析了我国沿海大竹蛏的遗传多样性状况,并对人工增殖放流活动对野生群体的影响情况进行了评价。结果表明,我国沿海大竹蛏在保持着高遗传多样性水平的同时,不同野生地理群体之间已经出现了显著的分化。开展的增殖放流活动会使得本地野生群体的遗传多样性有所下降,但不会产生明显的遗传变异。可以得出结论: 人工增殖放流仍然能够作为主要的大竹蛏野生资源恢复手段,但在苗种培育过程中,应当在保证亲本数量及品质的情况下尽量扩大选择范围;同时,注意对本地原种加大保护力度,避免异地繁养带来的遗传变异。相关监测工作仍需持续进行,以便对我国沿海大竹蛏资源的保护状况进行准确的评估。
么宗利,周 凯,来琦芳等,2005.我国五个青蛤地理群体遗传变异的RAPD分析.海洋渔业,27(2): 102—107
牛东红,李家乐,冯冰冰等,2009.缢蛏 6个群体遗传结构的ISSR分析.应用与环境生物学报,15(3): 332—336
吕林兰,杜晓东,王 嫣等,2008.马氏珠母贝 3个野生种群及种群间杂交后代遗传多样性的ISSR分析.水生生物学报,32(1): 26—32
乔洪金,刘相全,孙国华等,2012.大竹蛏(Solen grandis)cDNA文库中微卫星标记的筛选.海洋与湖沼,43(6):1128—1133
杜晓东,李 康,黄荣莲等,2007.翡翠贻贝 3个野生种群遗传多样性分析.热带海洋学报,26(4): 51—55
李俊清,李景文,2006.保护生物学.北京: 中国林业出版社
张 滔,刘相全,孙国华等,2013.大竹蛏(Solen grandis)不同地理群体遗传多样性的 AFLP 分析.海洋与湖沼,44(2):525—530
陈爱华,姚国兴,张志伟,2009.大竹蛏生产性人工繁育试验.海洋渔业,31(1): 66—72
孟学平,王 帅,高如承等,2011.西施舌群体遗传结构及分化的RAPD分析.海洋科学,35(2): 6—10
侯和要,牟乃海,宋全山等,2004.大竹蛏人工繁殖技术研究.齐鲁渔业,21(6): 32—35
Excoffier L,Lischer H E L,2010.Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows.Molecular Ecology Resources,10(3): 564—567
Hou L,Lü H L,Zou X Y et al,2006.Genetic characterizations of Mactra veneriformis (Bivalve)along the Chinese coast using ISSR-PCR markers.Aquaculture,261(3): 865—871
Krane D E,Sternberg D C,Burton G A,1999.Randomly amplified polymorphic DNA profile-based measures of genetic diversity in crayfish correlated with environmental impacts.Environmental Toxicology and Chemistry,18(3):504—508
Nei M,1978.Estimation of average heterozygosity and genetic distance from a small number of individuals.Genetics,89(3):583—590
Slatkin M,Barton N H,1989.A comparison of three indirect methods for estimating average levels of gene flow.Evolution,43(7): 1349—1368
Thorp J P,1982.The molecular clock hypothesis: biochemical evolution,genetic differentiation and systematics.Annual Review of Ecology and Systematics,13(1): 139—168
Wright S,1978.Evolution and the genetics of populations 4.Variability within and among natural populations.Chicago:University of Chicago Press,4

