啮齿目中RNase4基因的分子进化研究
郎大田, 王磊, 罗家刚, 祁岑, 王忻
(1.昭通学院 化学与生命科学学院,云南 昭通 657000;2.云南省粮油科学研究院,云南 昆明 650033)
现存的生物都需要靠自身的进化来适应变化的环境.基因型和基因数目的改变是适应性进化的重要机制,其中基因复制为生物进化提供了原材料,是生物进化的动力源泉.随着基因组学研究的不断深入,发现越来越多的生物表型与基因复制有着密切的关系[1-14].
RNASE A基因超家族是研究分子进化的最佳模型之一,仅在脊椎动物中存在,截至目前在人的基因组中检测到15个成员(RNase1-15)[15-17].其中RNase4基因是RNASE A基因超家族的一个重要成员,过去基于单基因水平(引物设计,PCR扩增,单基因测序)对RNase4基因的进化研究相对较少,但随着分子生物学和基因组学的迅速发展,越来越多的物种基因组陆续公布,RNase4基因的研究引起了越来越多学者的关注,在基因组水平对该基因的研究越来越多[15-17].特别是2013年Goo等[17]对20个哺乳动物(仅包含哺乳动物第一大类群啮齿目中的几内亚猪、裸鼹鼠、小家鼠、褐家鼠和仓鼠5个物种)基因组的RNase4基因进行研究,惊奇地在几内亚猪中检测到RNase4基因发生基因复制,而在另外的四个物种均为单拷贝[18].这一有趣的发现提示RNase4基因在啮齿目中可能具有独特的进化模式.但该研究仅包含啮齿目的5个物种, 那么RNase4基因在啮齿目的其他物种、属、科甚至亚目中发生基因复制的时间以及进化模式如何?因此,急需增加更多啮齿目代表物种对RNase4基因进行深入的进化分析,以揭示上述未解决的科学问题.因此,我们对2013年Goo等[18]研究的啮齿目 5个物种基因组及截至目前在NCBI上公布的所有啮齿目的11个物种基因组进行RNase4基因分析.
1 材料与方法
本次研究共包含啮齿目10科16个物种(包括2013年Goo等[18]研究的5个物种).这16个物种覆盖了啮齿目的三个进化枝:豪猪亚目、“与小家鼠相关的进化枝”和“与松鼠相关的进化枝”.
1.1 RNase4基因的鉴定
用小鼠RNase4基因的氨基酸序列和核酸序列分别作为查询序列,采用先前多数学者采用的TBlastN和BlastN方法[15-17],E值为10-10分别对每个物种的基因组搜索同源序列,进行分析.
1.2 序列比对和进化分析
使用ClustalX1.8.3[19]比对软件进行序列比对,并进行手工矫正.采用邻接法(Neighbor-Joining,NJ)和最大似然法(Maximum Likelihood,ML)进行系统树构建.选择兔形目(Lagomorpha)中的穴兔(Oryctolaguscuniculus)和北美鼠兔(Ochotonaprinceps)的RNase4基因(Oc_RNase4,Op_RNase4)作为外群.使用MEGA5.0软件构建NJ树[20-21],使用RaxML7.0.4软件构建ML树[22].
2 结果与讨论
2.1 啮齿目的RNase4基因序列
本次研究为啮齿目16个物种,22条RNase4基因序列,包括本次分析的11个物种(16条RNase4基因序列)和2013年Goo等.[17]研究的5个物种(6条RNase4基因序列).在这22条序列中,有5条序列的开放阅读框,因移码突变或者终止密码子提前变为假基因.白臀豚鼠和几内亚猪的RNase4基因有2个拷贝,鹿白足鼠和非洲跳鼠有1个真基因和2个假基因,奥氏更格卢鼠仅有1个假基因,其他11个物种的RNase4基因均为单拷贝(表1).这一结果揭示RNase4基因在白臀豚鼠、几内亚猪、鹿白足鼠和非洲跳鼠这四个物种发生基因扩张.
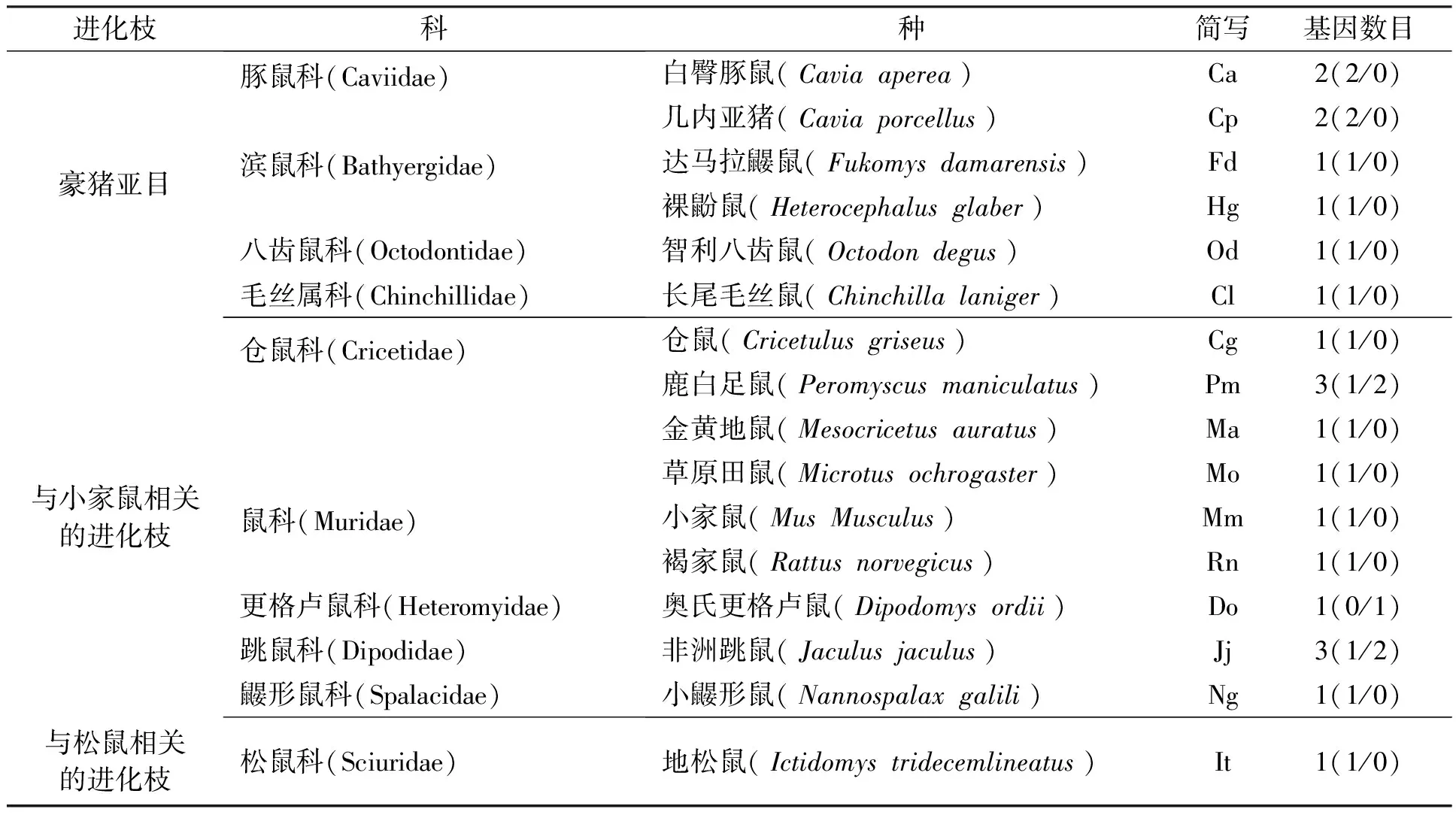
表1 本研究的物种信息和RNase4基因序列数
接下来对发生基因扩张的四个物种的旁系同源序列差异度进行计算.几内亚猪的两个基因(Cp_RNase4A,Cp_RNase4B)间仅有1个碱基和1个氨基酸的差异;白臀豚鼠中的RNase4A(Ca_RNase4A)基因在376-414 bp区域未测序到,用N替代,除去未测序到的这个区域,白臀豚鼠中2个基因(Ca_RNase4A,Ca_RNase4B)间仅有1个碱基和1个氨基酸的差异,这2个拷贝在白臀豚鼠基因组的同一个Contig上(AVPZ01000045.1),但位置相距约15 kb.几内亚猪和白臀豚鼠的拷贝间仅有1个氨基酸的差异,揭示RNase4基因这两个物种中发生基因复制事件是在最近一段时间内,这也正是在基因组水平对基因复制的进化模式的研究的优点之一(基于单基因水平研究,拷贝间氨基酸差异数小于3,视为同一拷贝[23-25]),当然不能排除是由于基因组组装错误所致,但我们认为豚鼠科的几内亚猪和白臀豚鼠两个基因组对RNase4基因的组装同时出现错误的可能性很小.仓鼠科中的鹿白足鼠和跳鼠科的非洲跳鼠的RNase4基因,因仅有1个功能基因,另外2个为假基因,只能对其核酸序列的差异度进行计算.鹿白足鼠3个拷贝(Pm_RNase4,Pm_RNase4psA和Pm_RNase4psB)中两两之间的碱基差异为12-14个.非洲跳鼠 3个拷贝(Jj_RNase4,Jj_RNase4psA和Jj_RNase4psB)中两两之间的碱基差异都在100个碱基左右,拷贝间的差异如此之大,且在系统发育树中非洲跳鼠3个拷贝的枝都相对较长(图2),于是对这3条序列分别在NCBI上进行在线同源序列搜索,结果仍然与RNase4基因的相似度最高,这就揭示非洲跳鼠3个拷贝间有如此大的差异很可能是RNase4基因发生基因复制后,其中2个拷贝假基因化,不再受到选择压力,进化速率快的结果.
啮齿目的22条序列的长度为423~537 bp,比对后的序列长度为566 bp,包含信号肽(1-84 bp)和成熟肽(85~566 bp).17条功能基因比对后的氨基酸序列为179个氨基酸,这些序列几乎都具有RNASE A基因超家族典型的序列特征:(1)有8个结构半胱氨酸,形成二硫键;(2)有3个催化氨基酸残基;(3)有序列标签“CKXXNTF”,但值得注意的是豪猪亚目中所有拷贝的苏氨酸(T)突变为丝氨酸(S)(图1).

图1 啮齿目RNase4真基因的氨基酸序列对比
2.2 啮齿目RNase4基因的系统发育分析
采用最大似然法(ML)和邻接法(NJ)对RNase4基因的功能基因和假基因一起构树,得到相似的拓扑结构(图2和图3).
由图2和图3可以看出,拓扑结构支持啮齿目分为三个进化枝:“与松鼠相关的进化枝”(ML BS=94%,NJ BS=54%)、“与小家鼠相关的进化枝”(ML BS=84%,NJ BS=59%)和豪猪亚目(ML BS=100%,NJ BS=99%),且支持率都比较高.这三支的进化关系为“与松鼠相关的进化枝”最先分歧,豪猪亚目和“与小家鼠相关的进化枝”形成姐妹群(ML BS=94%,NJ BS=54%),这一结果与2009年Shani等对啮齿目的系统发育研究结果一致[26].
另外,由图2和图3还可以看出,RNase4基因在豪猪亚目豚鼠科的白臀豚鼠和几内亚猪(ML BS=100%,NJ BS=100%)、“与小家鼠相关的进化枝”中的非洲跳鼠(ML BS=99%,NJ BS=99%)和鹿白足鼠(ML BS=100%,NJ BS=100%)中发生三次独立基因复制,发生基因复制的时间是在啮齿目中各科形成之后.在豚鼠科的白臀豚鼠和几内亚猪中均检测到2个RNase4功能基因,并且是以基因的形式相互混合,这一结果揭示RNase4基因在白臀豚鼠和几内亚猪中发生一次基因复制事件,且基因复制是在这两个物种形成之前,豚鼠科形成之后发生的.在非洲跳鼠和鹿白足鼠中均检测到1个功能基因和2个假基因,并且基因是以物种的形式聚集,这一结果揭示RNase4基因在非洲跳鼠和鹿白足鼠中均独立发生2次基因复制和2次假基因化事件,即“生与灭(birth and death)”的进化模式,这一进化模式在啮齿目的EAR基因(RNASE A超基因家族中的2个成员)和其他基因家族的研究中也有报道[27-29].

图2 基于啮齿目 RNase4基因构建的 ML 系统发育树
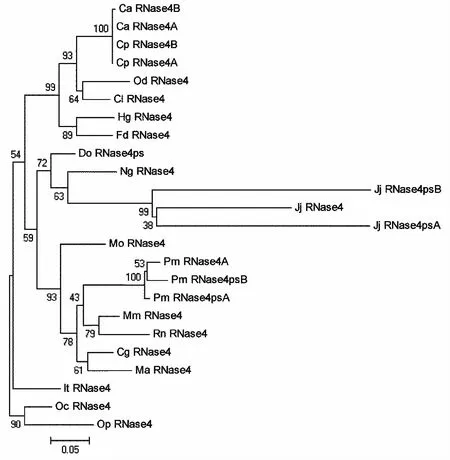
图3 基于啮齿目 RNase4基因构建的 NJ 系统发育树
3 展 望
RNase4基因作为RNASE A超基因家族中的一个重要成员,在过去的分子进化研究中相对较少,但随着越来越多的基因组公布,在基因组水平对RNase4基因的研究引起了越来越多学者的关注,特别是对啮齿目中的许多模式生物进行深入进化分析研究,能为后续的功能实验奠定基础.
由以上的研究可以看出RNase4基因在啮齿目中的进化模式比想象的复杂,在所研究的类群中发生了3次独立基因复制事件,并且在每一次独立的基因复制事件中有不同的进化模式.同时,该研究为后续对啮齿目RNase4基因的深入研究提供了线索:(1)在仓鼠科所分析的仓鼠、鹿白足鼠、金黄地鼠和草原田鼠这4个物种中,RNase4基因仅在鹿白足鼠中发生基因复制事件,而其他3个物种为单拷贝,这就提示后续研究需要增加仓鼠科代表物种,特别是增加与鹿白足鼠亲缘关系较近的代表物种.在单基因水平进行深入的分析研究,更加清晰地追溯RNase4基因在仓鼠科中发生基因复制的时间和进化模式,同时对RNase4基因发生复制的驱动力进行深入研究.(2)在跳鼠科中,本次研究仅包含非洲跳鼠,并且检测到RNase4基因发生2次基因复制和2次假基因化事件,这就带来疑问:RNase4基因是仅在跳鼠科的非洲跳鼠,还是在跳鼠科的某一个或者某几个属,甚至整个跳鼠科中发生基因复制事件?这同样需要增加跳鼠科代表物种来进行深入研究.
当然,对RNase4基因的研究并不局限于上述的进化分析,功能方面的研究也急需加入.为了对基因复制发生后,旁系同源基因的命运(新功能化、亚功能化和假基因化等)和活性进行深入探讨,需要对拷贝在不同组织的表达、酶活性和点突变等进行研究.
另外,随着基因组测序成本的降低和技术的成熟以及更多代表物种的基因组公布,在基因组水平对更多的生物类群进行RNase4基因研究,促进人们对该基因的认识,同时也为系统认识动物遗传机制做出贡献.
[1] RUBIN C J,ZODY M C,ERIKSSON J,et al.Whole-genome resequencing reveals loci under selection during chicken domestication[J].Nature,2010,464(7288):587-591.
[2] ALFOLDI J,DI PALMA F,GRABHERR M,et al.The genome of the green anole lizard and a comparative analysis with birds and mammals[J].Nature,2011,477(7366):587-591.
[3] KIM E B, FANG X, FUSHAN A A,et al.Genome sequencing reveals insights into physiology and longevity of the naked mole rat[J].Nature,2011,479(7372):223-227.
[4] LINDBLAD-TOH K,GARBER M,ZUK O,et al.A high-resolution map of human evolutionary constraint using 29 mammals[J].Nature,2011,478(7370):476-482.
[5] XU X,PAN S,CHENG S,et al.Genome sequence and analysis of the tuber crop potato[J].Nature,2011,475(7355):189-195.
[6] THE TOMATO GENOME CONSORTIUM.The tomato genome sequence provides insights into fleshy fruit evolution[J].Nature,2012,485(7400):635-641.
[7] ABECASIS G R,AUTON A,BROOKS L D,et al.An integrated map of genetic variation from 1,092 human genomes[J].Nature,2012,491(7422):56-65.
[8] D'HONT A,DENOEUD F,AURY J M,et al.The banana (Musa acuminata) genome and the evolution of monocotyledonous plants[J].Nature,2012,488(7410):213-217.
[9] GROENEN M A,ARCHIBALD A L,UENISHI H,et al.Analyses of pig genomes provide insight into porcine demography and evolution[J].Nature,2012,491(7424):393-398.
[10]MAYER K F,WAUGH R,BROWN J W,et al.A physical, genetic and functional sequence assembly of the barley genome[J].Nature,2012,491(7426):711-716.
[11]PASANIUC B,ROHLAND N,MCLAREN P J,et al.Extremely low-coverage sequencing and imputation increases power for genome-wide association studies[J].Nat Genet,2012,44(6):631-635.
[12]QIU Q,ZHANG G,MA T,et al.The yak genome and adaptation to life at high altitude[J].Nat Genet,2012,44(8):946-949.
[13]SCALLY A,DUTHEIL J Y,HILLIER L W,et al.Insights into hominid evolution from the gorilla genome sequence[J].Nature,2012,483(7388):169-175.
[14]ZHANG G,FANG X,GUO X,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54.
[15]CHO S,BEINTEMA J J,ZHANG J.The ribonuclease A superfamily of mammals and birds:identifying new members and tracing evolutionary histories[J].Genomics,2005,85(2):208-220.
[16]Cho S,Zhang J.Ancient expansion of the ribonuclease A superfamily revealed by genomic analysis of placental and marsupial mammals[J].Gene,2006,373:116-125.
[17]GOO S M,CHO S.The expansion and functional diversification of the mammalian ribonuclease a superfamily epitomizes the efficiency of multigene families at generating biological novelty[J].Genome Biol Evol,2013,5(11):2124-2140.
[18]GUPTA S K,HAIGH B J,GRIFFIN F J,et al.The mammalian secreted RNases: mechanisms of action in host defence[J].Innate Immun,2013,19(1):86-97.
[19]THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res,1997,25(24):4876-4882.
[20]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J].Mol Biol Evol,2011,28(10):2731-2739.
[21]KUMAR S,NEI M,DUDLEY J,et al.MEGA:a biologist-centric software for evolutionary analysis of DNA and protein sequences[J].Brief Bioinform,2008,9(4):299-306.
[22]STAMATAKIS A,HOOVER P,ROUGEMONT J.A rapid bootstrap algorithm for the RAxML Web servers[J].Syst Biol,2008,57(5):758-771.
[23]XU H,LIU Y,MENG F,et al.Multiple bursts of pancreatic ribonuclease gene duplication in insect-eating bats[J].Gene,2013,526(2):112-117.
[24]YU L,WANG X Y,JIN W,et al.Adaptive evolution of digestive RNASE1 genes in leaf-eating monkeys revisited: new insights from ten additional colobines[J].Mol Biol Evol,2010,27(1):121-131.
[25]YU L,ZHANG Y P.The unusual adaptive expansion of pancreatic ribonuclease gene in carnivora[J].Mol Biol Evol,2006,23(12):2326-2335.
[26]BLANGA-KANFI S,MIRANDA H,PENN O,et al.Rodent phylogeny revised: analysis of six nuclear genes from all major rodent clades[J].BMC Evol Biol,2009,9:71-83.
[27]OTA T,NEI M.Divergent evolution and evolution by the birth-and-death process in the immunoglobulin VH gene family[J].Mol Biol Evol,1994,11(3):469-482.
[28]NEI M,ROGOZIN I B,PIONTKIVSKA H.Purifying selection and birth-and-death evolution in the ubiquitin gene family[J].Proc Natl Acad Sci U S A,2000,97(20):10866-10871.
[29]ZHANG J,DYER K D,ROSENBERG H F.Evolution of the rodent eosinophil-associated RNase gene family by rapid gene sorting and positive selection[J].Proc Natl Acad Sci U S A,2000,97(9):4701-4706.

