基于辣椒基因组重测序的InDel标记开发及应用
郭广君, 孙 茜, 刘金兵, 潘宝贵, 刁卫平, 戈 伟, 高长洲, 王述彬
(江苏省农业科学院蔬菜研究所/江苏省高效园艺作物遗传改良重点实验室,江苏 南京 210014)
辣椒(Capsicum spp.)属于茄科 (Solanaceae) 辣椒属(Capsicum)作物,作为一种重要的蔬菜作物和调味品,广泛栽培于南北美、亚洲、欧洲和大洋洲等世界各地。随着消费者需求的多样性,利用传统方法在辣椒的遗传改良上取得了很大进展,但是分子标记[1]和第二代测序技术[2]的出现为辣椒的遗传改良提供了更大的机遇[3]。目前辣椒分子标记的开发和应用主要集中于利用公用数据或者基因组文库开发SSR标记[4-8]。随着高通量测序技术的发展和辣椒基因组数据的发布,使得辣椒分子标记的开发和应用进入了一个新的阶段[9-10],第三代分子标记SNP标记和InDel标记的开发和应用在辣椒中逐渐广泛[11-14]。
插入/缺失多态性(Insertion/deletion,InDel)标记是由于等位基因位点处的DNA序列在不同个体间发生了核苷酸片段的插入/缺失而产生的长度多态性变异[15]。根据目标位点两侧的序列设计特异引物进行PCR扩增,扩增片段的长度多态性即是InDel标记。在整个基因组中,InDel标记的多态性频率仅次于SNP标记,远高于SSR标记。InDel标记多态性可通过聚合酶链式反应(PCR)和琼脂糖凝胶电泳或非变性聚丙烯酰胺凝胶电泳等简单的步骤达到基因分型的目的[16-17],相对于SNP标记,其标记的设计和检测更为简易,应用也更为方便[18]。此外,InDel标记的另一优点是不可能在同1个基因组位点上发生同样长度的2个 InDel位点突变,所以具有共同InDel突变位点的2个物种间代表其具有血缘一致性(Identity-by-descent)[19]。综上所述,In-Del标记为共显性标记,具有较好的稳定性和较为丰富的多态性,是高分辨率图谱构建,关联分析和基因定位等方面最佳的候选标记[20]。但是,辣椒中已开发和应用的InDel标记数量非常少,本研究的目的在于基于辣椒基因组重测序数据开发InDel标记,利用不同遗传背景的辣椒种质对开发出的InDel标记进行验证,筛选获得有效的InDel标记,进一步探讨InDel标记在种质鉴定中的应用。
1 材料与方法
1.1 InDel位点的挖掘
本研究利用重测序数据与参考基因组进行比对查找InDel位点。参考基因组版本为pepper.V.1.55,下载网址:http://peppergenome.snu.ac.kr/ download.php。采用illumina HiSeqTM2500测序平台对一年生辣椒(Capsicum annuum)“G29”和灌木状辣椒(Capsicum.frutescents)“G108”进行测序。将测序得到的原始Reads进行质量评估并过滤得到Clean Reads。利用bwa软件比对定位Clean Reads在参考基因组上的位置。InDel的检测主要使用GATK软件工具包实现。根据Clean Reads在参考基因组的定位结果,使用 Samtools进行去重复(Mark duplicates),使用GATK软件进行局部比对(Local realignment)和碱基质量值校正(Base recalibration)等预处理,以保证检测结果的准确性,再使用GATK软件进行变异检测和校准,选取可靠的In-Del位点[21-22]。利用SnpEff软件根据InDel位点在参考基因组上的位置信息,对比参考基因组的基因、CDS位置等信息对其进行注释和预测其影响。
1.2 InDel引物设计和分析
根据重测序数据中预测到InDel位点,筛选出插入/缺失碱基数为5~10个碱基的位点。基于辣椒基因组序列,将筛选出的位点定位在基因组上,取InDel位点两翼各200 bp碱基长度,共401 bp长度进行引物设计。采用Primer 3.0进行引物设计,上游引物设计范围在1~195 bp,下游引物设计范围为205~401 bp,产物大小为150~250 bp,退火温度为52~60℃。
优化后的扩增反应采用10 μl反应体系,包括20 ng模板DNA、5 μl 2×Mix混合液(含Mg2+的10×PCR buffer、2.5 mmol/L的 dNTPs、0.5 U Taq DNA聚合酶)、每个引物终浓度为0.2 μmol/L。反应程序为94℃预变性4 min;94℃变性30 s,55℃复性30 s,72℃延伸40 s,循环30次;72℃延伸7 min,置于4℃下保存。扩增产物用10%非变性聚丙烯酰胺凝胶电泳,银染显色,统计分析条带类型。
1.3 供试辣椒种质及其DNA提取
用于检测标记有效性的辣椒材料来源于国外和国内多个科研单位,其中包括一年生辣椒(Capsicum annuum)21份、灌木状辣椒(Capsicum frutescents)2份,下垂辣椒(Capsicum baccatum)1份。除11份辣椒品种外,其余13份以G开头的材料为项目组引进的国外种质资源(表1)。采用改良CTAB法提取叶片的基因组DNA[23],经琼脂糖凝胶电泳和微量分光光度计检测,DNA浓度调至50 ng/μl备用。
2 结果与分析
2.1 辣椒InDel标记的鉴定
通过与参考基因组序列比对,一年生辣椒品种G29和灌木状辣椒品种G108的全基因组范围内共检测到的InDel数量分别为533 523个和1 664 770个,其中编码区的InDel数量分别为1 019个和2 515个,具体结果见表2。通过进一步比较分析,G29和G108之间的全基因组范围内 InDel数量为1 586 427个。
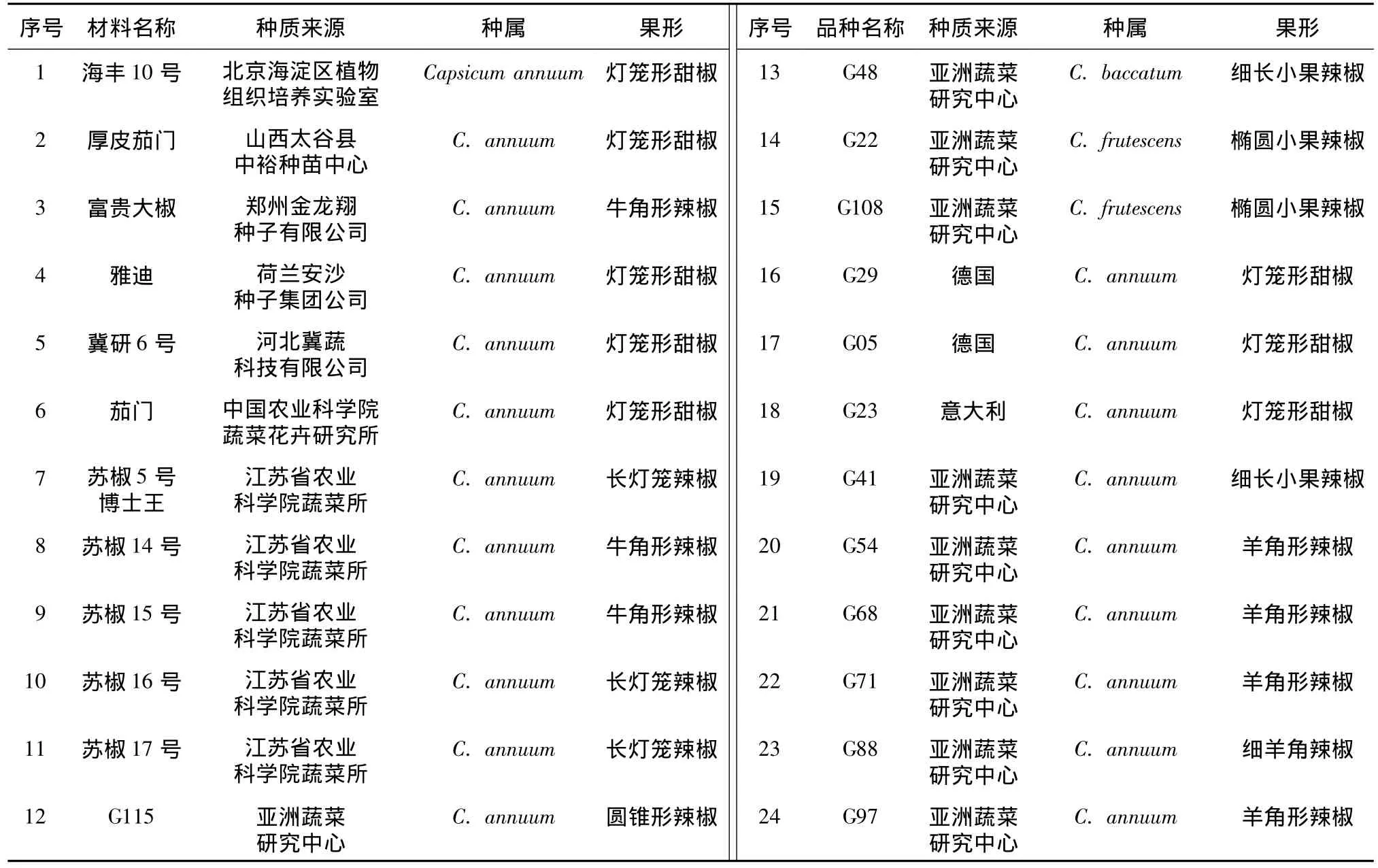
表1 24份供试辣椒种质基本信息Table 1 The information of 24 tested pepper ge rmplasm

表2 全基因组和编码区InDel位点信息Table 2 The information of InDel loci within the whole genome and CDS
2.2 InDel标记有效性验证
根据预测的InDel位点,在2号染色体上随机选择了40个InDel位点设计引物,引物信息见表3。以24份不同来源的辣椒材料验证标记的有效性。经过PCR和产物检测发现,40对引物在24份辣椒材料中均能扩增出条带,扩增条带的大小与预测产物大小基本相同。如图1为引物InDel-2-3对24份辣椒种质PCR扩增结果。经统计,在24份材料中共扩增出70个清晰可辨的位点,其中35个标记表现出多态性,5个无多态性,多态率达到87.5%。
2.3 InDel标记在品种和种质资源鉴定中的应用
利用Popgene32软件对40对引物在24份辣椒材料中的扩增结果进行分析发现,每对引物反映的基因多样性指数范围为 0.08~0.50,其均值为0.20;Shannon′s多样性信息指数分布在0.17至0.80之间,其均值为0.34(表3)。
利用NTSYSpc 2.10e中的UPGMA方法对24份材料进行聚类分析,由聚类结果(图2)可以看出,在遗传相似系数0.42处,24份辣椒种质 分为2个大类,一类为21份一年生辣椒,另一大类为2份灌木状辣椒(C.frutescens)和1份下垂辣椒(C.baccatum)。21份一年生辣椒中可以聚类为羊角形辣椒、牛角形(长灯笼形)辣椒和甜椒三大类别。同时,聚类结果显示牛角形(长灯笼形)辣椒和甜椒相似度更高,同一单位品种间相似度高于不同单位间相似度。

表3 本研究开发的辣椒InDel标记信息Table 3 The information of pepper InDel markers developed in the study
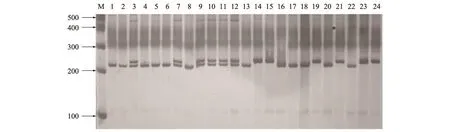
图1 引物InDel-2-3对24份辣椒种质的扩增结果Fig.1 Amplification of 24 pepper germplasm DNA with marker InDel-2-3

图2 基于Nei′s遗传距离的24份辣椒种质的UPMGA聚类图Fig.2 UPGMA dendrogram of 24 pepper germplasm based on the Nei′s genetic distance
3 讨论
InDel标记为共显性标记,具有较好的稳定性和较为丰富的多态性。虽然在整个基因组中,其多态性频率次于SNP标记[24],但是,相对于SNP标记,InDel标记在引物设计、多态性检测等方面更为简易,应用也更为方便[16-17,25]。但是,目前辣椒中In-Del标记的开发数量较少,应用较为有限,这一现状急待改善[11]。
本研究基于全基因组重测序数据,通过与参考基因组序列比对,在一年生辣椒品种G29和灌木状辣椒品种G108的全基因组范围内检测到的InDel数量分别为533 523个和1 664 770个。以上结果说明利用全基因组重测序技术可以在辣椒中更好地挖掘InDel位点,为全基因组范围内的InDel设计提供了更优选择。这一观点已经在包括辣椒在内的多个物种中得以验证[12,26-27]。
利用24份辣椒材料对2号染色体上的40个标记的有效性进行验证,发现24份材料在40个标记中共扩增出70个清晰可辨的位点,其中35个标记表现出多态性,多态率达到87.5%。这一结果很好地验证了本研究开发出的InDel标记的有效性和较高的多态性。目前辣椒分子标记的开发和应用主要集中于利用公用数据或者基因组文库开发SSR标记,但是大部分研究结果表明SSR标记的多态性及其应用率远低于InDel标记。如Huang等人基于数据库中的辣椒序列开发出12个SSR标记,其中多态性标记为5个,多态率为41.7%[5];Lee等构建辣椒分子遗传连锁图谱时开发出76个SSR标记,其中具有多态性的标记为46个,多态率为60.5%[4];Yi等开发出1 201个EST-SSR标记,其多态性标记只有150个,多态率仅为29.2%[6];Huang等依据EST序列开发出SSR标记755个,利用8份辣椒栽培种对其中的210个进行验证,其多态性标记为127个,多态率为60.5%[8];Shirasawa等基于辣椒EST序列设计了5 751个SSR标记,利用辣椒材料对其中77个进行验证,其中60个表现出多态性,多态率为77.9%[7]。但是,也有少数研究结果显示,InDel标记与SSR标记的多态率并无明显差异。Li等利用InDel标记构建辣椒种内遗传图谱时显示,1 000个InDel标记中251个标记可以成功应用于图谱构建,其多态率为25.1%[11]。随后,该团队(2015年)基于SSR和InDel标记构建辣椒种间遗传图谱时,分别筛选了1 038个InDel标记和674个SSR标记,其中多态性标记分别为140个和102个,多态率分别为13.5%和15.1%[12]。
利用NTSYSpc 2.10e中的UPGMA方法对24份材料进行聚类分析,结果表明35个InDel标记不仅可以有效区分出21份一年生辣椒、2份灌木状辣椒和1份下垂辣椒,而且21份材料中又可以聚类为不同种类,其聚类结果与表型特征非常吻合。这一结果表明InDel标记在种质资源的遗传分析、品种鉴定和品种保护方面具有极大的应用价值。但同一单位育成品种的相似度很高,说明可利用的辣椒种质的遗传背景较窄,对于品种改良极为不利,需要开发和应用新的种质进行品种的选育和改良。
总的来说,基于全基因组重测序数据开发InDel标记可以有效地应用于辣椒种质鉴定,遗传图谱的构建,关联分析和基因定位等方面,是辣椒种质资源开发利用和分子标记辅助育种的高效工具。
[1] 王述彬,刁卫平,刘金兵,等.辣椒胞质雄性不育相关基因的克隆及表达[J].江苏农业学报,2014,30(4):833-838.
[2] 徐婉莉,裴徐梨,荆赞革,等.辣椒actin基因电子克隆与生物信息学分析[J].江苏农业科学,2014,42(5):46-48.
[3] RAMCHIARY N,KEHIE M,BRAHMA V,et al.Application of genetics and genomics towards Capsicum translational research[J].Plant Biotechnology Reports,2014,8(2):101-123.
[4] LEE J M,NAHM S H,KIM Y M,et al.Characterization and molecular genetic mapping of microsatellite loci in pepper[J].Theoretical and Applied Genetics,2004,108(4):619-627.
[5] HUANG S W,ZHANG B X,MILBOURNE D,et al.Development of pepper SSR markers from sequence databases[J].Euphytica,117,2(2001):163-167.
[6] YI G,LEE J M,LEE S,et al.Exploitation of pepper EST-SSRs and an SSR-based linkage map[J].Theoretical and Applied Genetics,2006,114(1):113-130.
[7] SHIRASAWA K,ISHII K,KIM C,et al.Development of Capsicum EST-SSR markers for species identification and in silico mapping onto the tomato genome sequence[J].Molecular Breeding,2013,31(1):101-110.
[8] HUANG H H,ZHANG Z H,ZHANG Z H,et al.Analysis of SSRs information in Capsicum spp.from EST database[J].Agricultural Sciences in China,2011,10(10):1532-1536.
[9] QIN C,YU C,SHEN Y,et al.Whole-genome sequencing of cultivated and wild peppers provides insights into Capsicum domestication and specialization[J].Proceedings of the National Academy of Sciences,2014,111(14):5135-5140.
[10]KIM S,PARK M,YEOM S I,et al.Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species[J].Nature Genetics,2014,46(3):270-278.
[11]LI W,CHENG J,WU Z,et al.An InDel-based linkage map of hot pepper(Capsicum annuum)[J].Molecular Breeding,2015,35(1):219-319.
[12]TAN S,CHENG J W,ZHANG L,et al.Construction of an interspecific genetic map based on InDel and SSR for mapping the QTLs affecting the initiation of flower primordia in pepper(Capsicum spp.)[J].Plos One,2015,10(3):1-15.
[13]LU F H,KWON S W,YOON M Y,et al.SNP marker integration and QTL analysis of 12 agronomic and morphological traits in F^sub 8^RILs of pepper(Capsicum annuum L.)[J].Molecules and Cells,2012,34(1):25-34.
[14]LIU W Y,KANG J H,JEONG H S,et al.Combined use of bulked segregant analysis and microarrays reveals SNP markers pinpointing a major QTL for resistance to Phytophthora capsici in pepper[J].Theoretical and Applied Genetics,2014,127(11): 2503-2513.
[15]JANDER G,NORRIS S R,ROUNSLEY S D,et al.Arabidopsis map-based cloning in the post-genome era[J].Plant Physiology,2002,129(2):440-450.
[16]VÄLIÜ,BRANDSTRÖM M,JOHANSSON M,et al.Insertion-deletion polymorphisms(indels)as genetic markers in natural populations[J].BMC Genet,2008,9(1):1-8.
[17]VARSHNEY R.Gene-based marker systems in plants:high throughput approaches for marker discovery and genotyping[M].New York:Springer Netherlands,2009.
[18]HAYDEN M J,TABONE T,MATHER D E.Development and assessment of simple PCR markers for SNP genotyping in barley[J].Theoretical and Applied Genetics,2009,119(5):939-951.
[19]SHEDLOCK A M,OKADA N.SINE insertions:powerful tools for molecular systematics,Bioessays[J].2000,22(2):148-160.
[20]MILLS E R,LUTTIG T C,LARKINS E C,et al.An initial map of insertion and deletion(INDEL)variation in the human genome[J].Gemome Research,2006,16:1182-1190.
[21]MCKENNA A,HANNA M,BANKS E,et al.The genome analysis toolkit:A map reduce framework for analyzing next-generation DNA sequencing data[J].Genome Research,2010,20(9): 1297-1303.
[22]LI H,HANDSAKER B,WYSOKER A,et al.The sequence alignment/map format and SAMtools[J].Bioinformatics,2009,25(16):2078-2079.
[23]ZHANG X,WANG L,SHOU L.Modified CTAB method for extracting genomic DNA from wheat leaf[J].Agricultural Science&Technology,2013,14(7):946-949.
[24]BROOKES A J.The essence of SNPs[J].Gene,1999,234 (2):177-186.
[25]CHUNG W H,ISHII H,NISHIMURA K,et al.Genetic analysis and PCR-based identification of major Fusarium species causing head blight on wheat in Japan[J].Journal of General Plant Pathology:JGPP,2008,74(5):364-374.
[26]PǍCURAR D I,PǍCURAR M L,STREET N,et al.A collection of INDEL markers for map-based cloning in seven Arabidopsis accessions[J].Journal of Experimental Botany,2012,63(7): 2491-2501.
[27]LIU B,WANG Y,ZHAI W,et al.Development of InDel markers for Brassica rapa based on whole-genome re-sequencing[J].Theoretical and Applied Genetics,2013,126(1):231-239.

