铜绿假单胞菌耐药基因分析及多位点序列遗传分型
袁 梦,袁月明,陈宏彬,罗锦雁,俞慕华,段永翔
铜绿假单胞菌耐药基因分析及多位点序列遗传分型
袁 梦,袁月明,陈宏彬,罗锦雁,俞慕华,段永翔
目的 了解2011-2012年深圳市南山区医院病人,各医院、诊所内环境台面涂抹样、医护人员手涂抹样分离的铜绿假单胞菌,抗生素耐药基因分布及基因的遗传多样性。方法 采用聚合酶链反应技术检测铜绿假单胞菌的20种耐药基因:TEM、VEB、CARB、OXA、SHV、PER、GES、GTX、SPM、GIM、IMP、VIM、DHA、oprD、Aac(6′)-Ⅰ、Aac(6′)-Ⅱ、Aac(3′)-Ⅰ、Aac(2″)-Ⅰ、qacE1-sull及Ⅰ类整合子基因。采用多位点序列分子分型方法进行聚类和克隆分析。结果 检出11种耐药基因:TEM、SHV、IMP、DHA、Aac(6’)-Ⅰ、Aac(6′)-Ⅱ、Aac(3′)-Ⅰ、Aac(2″)-Ⅰ、qacE1-sull、Ⅰ类整合子及oprD基因,检出率分别为8.1%、6.4%、4.8%、9.7%、4.8%、14.5%、4.8%、56.5%,8.1%,8.1%,oprD基因缺失率为61.2%。52株铜绿假单胞菌检出耐药基因,形成19种耐药基因谱。多位点序列分型方法将62株铜绿假单胞菌,分为39个ST型,5个克隆群,1个优势克隆群CC244,1个优势独特型ST856。结论 不同类型样本分离菌株携带耐药基因存在差异,部分病人分离株携带多种耐药基因。本研究铜绿假单胞菌具有遗传多样性,存在优势克隆群。
铜绿假单胞菌;多重耐药;耐药基因;聚合酶链反应;多位点序列分子分型;克隆群
Supported by Shenzhen Science and Technology Plan Project (No. 201103296)
铜绿假单胞菌(Pseudomonasaeruginosa,PA)为革兰氏阴性杆菌,广泛存在于各类环境中。近年来,PA已成为医院感染最主要的条件致病菌,占世界范围内院内感染细菌的10%~15%[1]且耐药性逐年增加,多重耐药株不断出现[2]。PA的多重耐药机制十分复杂,本研究选择20种抗生素耐药基因,分别是β-内酰胺TEM、VEB、CARB、OXA、SHV、PER、GES、GTX、SPM、GIM,金属β-内酰胺酶IMP、VIM,AmpC酶DHA,膜通道蛋白oprD,氨基糖苷类修饰酶Aac(6′)-Ⅰ、Aac(6′)-Ⅱ、Aac(3′)-Ⅰ、Aac(2″)-Ⅰ,耐消毒基因qacE1-sull与Ⅰ类整合子基因,了解耐药基因携带情况。多位点序列分子分型(Multilocus Sequencing Typing,MLST)是一种通过直接测定多个管家基因的核苷酸序列,发现细菌变异的分型方法[3],由于可以准确记录细菌基因水平上的变异,并且序列可通过互联网方便传递,故MLST被用于研究细菌的流行病学、致病性和进化学[4]。本研究采用MLST分型方法对62株菌进行分子分型,扩增菌株7个管家基因acsA、aroE、guaA、mutL、nuoD、ppsA、trpE,聚合酶链反应(polymeras chain reaction,PCR)产物测序,测序结果上传数据库比对,了解南山PA菌的基因多态性分布,优势克隆群,病人与环境PA株之间是否具有同源性,建立分子分型数据库,为疾病耐药监测以及溯源提供依据。
1 材料与方法
1.1 材料
1.1.1 菌株 分离2011-2012年南山区医院病人、环境台面及医护人员双手涂抹样,共62株PA,其中37株分离自院内病人;25株分离自环境样及医护人员涂抹样。
1.1.2 主要仪器和试剂 基因扩增仪(英国Barloworl公司);电泳仪(美国Major Science公司);凝胶成像系统(美国Bio-Rad公司)。PCR相关试剂(大连宝生物工程有限公司);PCR产物测序由上海生工生物工程有限公司完成。
1.2 方法
1.2.1 菌株的分离鉴定 按照《消毒技术规范》(2002年版)进行PA菌株的分离培养。
1.2.2 DNA模板制备 从营养琼脂培养平皿上挑取单菌落,溶于100 μL纯水中煮沸5 min,冰浴5 min后,以12 000 r/min离心5 min,取上清即为提取的DNA。
1.2.3 引物 耐药基因(TEM,SHV,OXA,PER,VEB,GES,CARB,IMP,VIM,GTX,SPM,GIM,DHA,oprD,Aac(6′)-Ⅰ,Aac(6′)-Ⅱ,Aac(3′)-Ⅰ,Aac(2′′)-Ⅱ,qacE1-sull,Ⅰ类整合子)引物及MLST7个管家基因(acsA、aroE、guaA、mutL、nuoD、ppsA、trpE)引物序列见相关文献[5-7],由上海生物工程有限公司合成。
1.2.4 PCR体系和反应条件
1.2.4.1 耐药基因检测扩增体系 10×PCR Buffer 2.5 μL;10 mmol/L dNTPs 2.0 μL;10 μnol/L耐药基因上下游引物各1.2 μL;Taq酶(5 u/μL)0.2 μL;50 ng DNA模板5 μL,总体积25 μL。扩增条件:预变性93 ℃ 2 min;变性93 ℃ 1 min,退火55 ℃ 1 min,延伸72 ℃ 1 min,共35个循环;最后延伸为72 ℃ 5 min[5]。
1.2.4.2 MLST分型扩增体系 10×PCR Buffer 5.0 μL;10 mmol/L dNTPs 4.0 μL;10 μmol/L耐药基因上下游引物各2.0 μL;Taq酶(5 u/μL)0.25 μL;50 ng DNA模板3.0 μL,总体积50 μL。扩增条件:预变性96 ℃ 1 min;变性96 ℃ 1 min,退火55 ℃ 1 min,延伸72 ℃ 1 min,共35个循环;最后延伸为72 ℃ 10 min[7]。PCR扩增产物上样于1.2%琼脂糖凝胶中电泳,凝胶成像仪观察结果并拍照。
1.2.5 MLST扩增产物测序 测序结果提交至MLST数据库分析网站(www.mlst.net)分析,确定序列型(sequence type,ST)。
1.2.6 聚类以及克隆群分析 运用Bionumerics软件,采用Categorical方法计算不同ST型7个位点的差异,用非加权配对算术平均法(Unweighted Pair Group with Arithmetic Averages,UPGMA)进行聚类,构建聚类树。运用Bionumerics软件确定不同的克隆群(clonal complexes ,CCs),7个位点中有5个相同的,认为是同一克隆群[8]。
2 结 果
2.1 MLST分型 7个管家基因PCR产物扩增图谱,条带清晰,条带大小acsA(842 bp)、aroE(825 bp)、guaA(940 bp)、mutL(940 bp)、nuoD(1 042 bp)、ppsA(989 bp)、trpE(811 bp)。
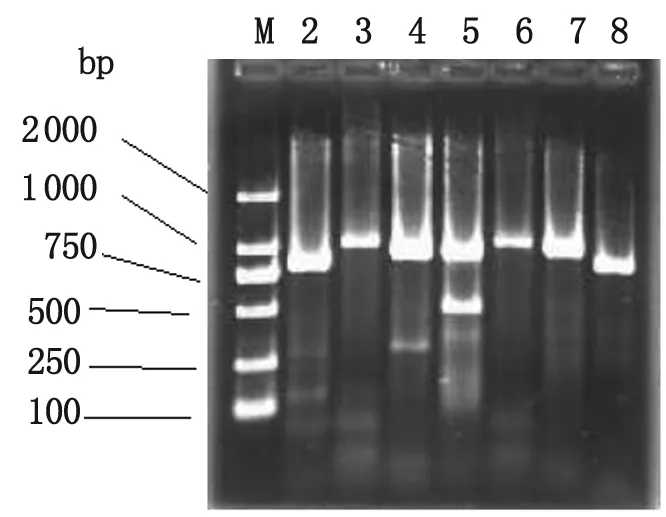
M:DNA marker 2000, 2:Positive fragment ofacsAgene, 3:Positive fragment ofaroEgene, 4:Positive fragment ofguaAgene, 5:Positive fragment ofmutLgene, 6:Positive fragment ofnuoDgene, 7:Positive fragment ofppsAgene, 8:Positive fragment oftrpEgene.
图1 7个管家基因扩增产物电泳图谱
Fig.1 Amplification products electrophoretogram of seven housekeeping genes
2.2 62株PA菌聚类分析图 不同菌株的科室来源、标本种类、ST型及7个管家基因号等详细信息见图2。New1-new13为新发现ST型别。聚类结果得到39种不同的ST型。有12种ST型别存在2株以上PA菌,分别为:ST856(7株),ST381(3株),ST274(2株),ST699(2株),ST260(3株),ST155(2株),ST645(2株),ST244(4株),new12(2株),ST357(4株),ST1752(2株),ST507(2株)。其余MLST型别较为分散。

图2 MLST分型64株PA菌的聚类结果
2.3 克隆复合体分析 采用Bionumerics软件分析得到5个不同的CCs:CC155(包括ST155、ST645)、CC244(包括ST244、ST1929、STnew3、STnew10、ST1623、ST408、ST699、ST1930、ST836)、CC274(包括ST274、STnew7)、CC357(包括ST357、ST1752)、CC1521(包括ST1521、STnew2)。ST155、ST244、ST274、ST357、ST1521分别为5个复合体的原始ST型。见图3。不能形成CCs的STs称为独特型。
2.4 耐药基因检出情况 本研究对62株PA菌进行20种耐药基因检测,检出11种耐药基因,分别为β-内酰胺类耐药基因:5株菌携带TEM基因(8.1%,5/62),4株菌携带SHV基因(6.5%,4/62);β-内酰胺酶类耐药基因:3株菌携带IMP基因(4.8%,3/62);Ampc酶类耐药基因:6株菌携带DHA基因(9.7%,6/62);外膜通道蛋白耐药基因:24株菌携带oprD基因(38.7%,24/62);氨基糖苷类耐药基因:3株菌携带Aac(6′)-Ⅰ基因(4.8%,3/62),9株菌携带Aac(6′)-Ⅱ基因(14.5%,9/62),3株菌携带Aac(3′)-Ⅰ基因(4.8%,3/62),35株菌携带Aac(2″)-Ⅰ基因(56.5%,35/62);5株菌携带qacE1-sull与Ⅰ类整合子基因(8.1%,5/62)。
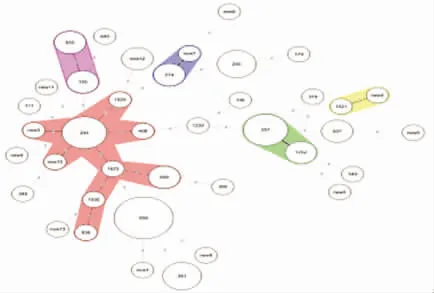
图3 62株PA菌最小生成树进化溯源图
Fig.3 Distribution of the STs and CCs of the 62 isolates of PA on a dendrogram built
2.5 检出较少耐药基因的菌株,见表1。共有10株PA菌未检出耐药基因,病人与环境各分离5株。病人分离株,仅有2株检出oprD基因,其余35株菌均存在oprD基因缺失。20株菌仅检出Aac(2″)-Ⅰ基因。
2.6 检出2种以上耐药基因的克隆群及ST型别 见表2。共有13种耐药基因谱,5株均为oprD+Aac(2″)-Ⅰ耐药基因谱,其余耐药基因谱分散。7株携带2种耐药基因,5株携带3种耐药基因,6株携带4种以上耐药基因。
3 讨 论
本研究中,5株携带Ⅰ类整合子与qacE1-sull基因,其中4株存在oprD基因缺失,5株菌均携带4种以上耐药基因包括β-内酰胺、β-内酰胺酶以及氨基糖苷类修饰酶基因,与相关报道相符[9],证明了在整合子介导的耐药机制中,Ⅰ类整合子起着非常重要的作用[10]。整合子具有捕获、切除、重排耐药基因的能力,目前已有超过130多种耐药基因盒可以整合在整合子上,可以编码临床上几乎所有抗生素的抗性基因[11]。Ⅰ类整合子,5′保守区含有表达被整合基因盒的增强子区域,3′保守区含有编码耐药消毒剂qacE1-sull基因[12]。qacE1基因表达产物可使多种化合物排出胞外,赋予细菌对季胺类、双胍类消毒剂耐药,在医院PA菌监测中,需加强对携带Ⅰ类整合子菌株的监测。

表1 未检出及检出1种耐药基因的ST型

表2 检出2种以上耐药基因PA菌的CCs与ST型别分布情况
注:⑴-TEM、⑵-SHV、⑶-IMP、⑷-DHA、⑸-oprD、⑹-Aac(6′)-Ⅰ、⑺-Aac(6′)-Ⅱ、⑻-Aac(3′)-Ⅰ、⑼-Aac(2″)-Ⅰ、⑽-qacE1-sull、⑾-int-Ⅰ
40株PA携带氨基糖苷类修饰酶基因,阳性率为64.5%(40/62),有20株菌仅携带Aac(2″)-Ⅰ基因。是本研究中,检出率最高的耐药基因,虽与之前研究表型耐药对氨基糖苷类药物100%耐药存在差异,亦表明产氨基糖苷类修饰酶是细菌对氨基糖苷类抗菌药物耐药的主要原因[13]。
本研究应用MLST分型方法结合Bionumerics软件进行聚类和克隆溯源分析。聚类结果将62株PA分为39种MLST型,型别较为分散,与文献报道一致,可能与PA的基因变异性较高有关,这种高变异性易导致PA适应性强和致病性,容易产生耐药[14]。最小生成树结果,主要有5个克隆群。存在多重耐药的型别(见表2),8种ST型检出2种耐药基因,5种ST型检出3种耐药基因,6种ST型检出4种以上耐药基因。
有研究表明CC235、CC111、CC175属于高风险克隆群,易携带β-内酰胺酶基因(VIM、IMP与SPM),全世界分布广泛,呈多重耐药,在很多国家为引起院内感染的优势型别[15-19],但在本研究中以及国内的报道中未分离到ST235、ST175型别[14,20],可能与地域差异以及标本来源的局限性有关。CC111为独特型,没有形成克隆群。本研究中62株PA形成5个克隆群CC244为较大的克隆群,包括9种ST型别。CC244克隆群中,分离自环境的菌株不携带或者携带较少的耐药基因,耐药基因谱以氨基糖苷类为主。分离自病人菌株,耐药基因谱较为复杂,为多重耐药,是南山区主要克隆群。本研究中ST244型携带IMP基因,与有关文献在中国的多个城市发现携带IMP耐药基因的ST244型的报道一致[18]。CC155、CC274与CC357克隆群国际上均有报道[15-16],CC155克隆群包括ST155与ST645,3株分离自环境,1株分离自病人,仅1株分离自环境的PA携带Aac(2″)-Ⅰ基因,其余3株耐药基因未检出。CC274克隆群包括ST274与STnew7,来自不同科室的不同样本,耐药谱较为单一,均仅携带1种耐药基因。CC1521包括ST1521与STnew2,此克隆群耐药谱复杂是本研究中携带耐药基因最多的克隆群,此克隆群需引起高度重视,避免发生院内感染。
通过聚类发现,ST856、ST381、ST260虽属于独特型,但每种型别都包括3株以上的PA菌,ST856包括7株PA携带耐药基因较少。环境与病人中均存在ST381与ST260型。
New1-new13为本研究新发现的ST型别,new1-new10分离自不同科室病人的不同标本,new11-new13分离自环境样。New3、new10属于CC244克隆群,new7属于CC274克隆群,new2属于CC1521克隆群,其余属于独特型。不同诊所出现了相同的ST型别(ST155、ST175、ST507),仅携带oprD基因,为何不同环境分离PA会出现相同型别,但型别较为分散,无集中优势,菌株之间是否有关联,有待进一步研究。
本研究中,院内病人分离株与环境分离株之间有同源关系的有4个型别:ST381、ST260、ST645、ST244,4个型别分离菌株携带耐药基因较少。医院的病人分离株与来自不同医院、诊所环境涂抹样的PA具有同源性,但无优势菌株,亦说明PA作为条件致病菌广泛存在于环境中。
(感谢中国疾病预防控制中心传染病预防控制所呼吸道传染病实验室热心提供技术指导。)
[1]Strateva T, Yordanov D.Pseudononasaeruginosa-a phenomenon of bacterial resistance[J]. Med Microbiol, 2009, 58(Pt9):1133-1148.
[2]Guo Y, Zhang Z. The research between gene cassette of integron and mutidrug-resistance ofPseudomonasaeruginosa[J]. Chin J Lab Diagn, 2008, 12(8):1069-1072. (in Chinese) 郭宇,张正.整合子基因盒系统与铜绿假单胞菌多重耐药的研究进展[J].中国实验诊断学,2008,12(8):1069-1072.
[3]Zhang SM, Xu JG. Multilocus sequence typing and its application[J]. Dis Surveill, 2008, 23(10):648-650. (in Chinese) 张少敏,徐建国.多位点序列分型及其应用[J].疾病监测,2008,23(10):648-650.
[4]Maiden MC, Bygraves JA, Feil E, et al. Multilocus sequence typing:A portable approach to the identification of clones within populations of pathogenic microorganisms[J]. Proc Natl Acad Sci U S A, 1998, 95(6):3140-3145.
[5]Shi WF, Wang YY, Jiang QB, et al. Resistant genes and cluster analysis in multidrug-resistantPseudomonasaeruginosa[J]. Chin J Nosocomiol, 2007, 17(9):1057-1060. (in Chinese) 史伟峰,王玉月,姜庆波,等.多重耐药性铜绿假单胞菌耐药基因及菌株聚类分析[J].中华医院感染学杂志,2007,17(9):1057-1060.
[6]Mao JF, Wang W, Xu WZ, et al. Disinfectant-resistant gene of qacE△1-sull and drug-resistant genes as sociated β-lactams inPseudomonasaeruginosa[J]. Zhejiang J Lab Med, 2007, 5(1):27-29. (in Chinese) 毛剑锋,王伟,徐伟珍,等.铜绿假单胞菌耐消毒剂-磺胺基因及β内酰胺类抗生素耐药相关基因研究[J].浙江检验医学,2007,5(1):27-29.
[7]Curran B, Jonas D, Grundmann H, et al. Development of a multilocus sequence typing scheme for the pathogenPseudomonasaeruginosa[J]. J Clin Microbiol, 2004, 42(12):5644-5649.
[8]Cholley P, Ka R, Guyeux C, et al. Population structure of clinicalPseudomonasaeruginosafrom west and central Afican countries[J]. PLoS ONE, 2014(9):1-9.
[9]Zhu YY, Yi Y, Yang X, et al. Discovery of new structure of class 1 integron in MDRPseudomonasaeruginosaand its association with drug-resistance[J]. Acta Microbiologica Sinica, 2013, 53(9):927-932. (in Chinese) 朱玉莹,易勇,杨犀,等.多重耐药铜绿假单胞菌中Ⅰ型整合子新结构的发现及其与耐药的相关性[J].微生物学报,2013,53(9):927-932.
[10]Zhao ZX, Chen HL, Wei SQ, et al. Reasearch on the part of resistant genes and resistance to antibiotics of multi - drug resistantPseudomonasaeruginosa[J]. Chin J Lab Diagn, 2011, 1(7):1159-1162. (in Chinese) 赵祝香,陈惠玲,魏树全,等.多药耐药铜绿假单胞菌耐药性及相关耐药基因分析[J].中国实验诊断学,2011,15(7):1159-1162.
[11]Partridge SR, Tsafant G, Coeira E, et al. Gene cassettes and cassette arrays in mobile resistance integrons[J]. FEMS Microbiol Rev, 2009, 33(4):757-763.
[12]Biskri L, Bouvier M, Guerout AM, et al. Comparative study of classⅠintegron andVibriocholeraesuper integron integrase activities[J]. J Bacteriol, 2005, 187(5):1740-1750.
[13]Chen L, Zha ZH, Leng YR, et al. The drug resistance and drug resistance genes ofPseudomonasaeruginosa[J]. Chin J Nosocomiol, 2014, 24(8):1837-1839. (in Chinese) 陈璐,查筑红,冷应蓉,等.铜绿假单胞菌的耐药性及耐药基因研究[J].中华医院感染学杂志,2014,24(8):1837-1839.
[14]Wang D, Liang QF, Wang ZQ, et al. Genetic typing and antibiotic susceptibility testing of strains fromPseudomonasaeruginosakeratitis patients[J]. Ophthalmol Chin, 2007, 16(3):179-183. (in Chinese) 王丹,梁庆丰,王智群,等.铜绿假单胞菌性角膜炎的细菌基因分型与体外药物敏感性研究[J].眼科,2007,16(3):179-183.
[15]Cho HH, Kwon KC, Sung JY, et al. Prevalence and genetic analysis of multidrug-resistantPseudomonasaeruginosaST235 isolated from a hospital in Korea, 2008-2012[J]. Ann Clin Lab Sci, 2013, 34(4):414-419.
[16]Wright LL, Turton JF, Livermore DM, et al. Dominace of international ‘high-risk clones’ among netallo-β-lactamase-producingPseudomonasaeruginosain the UK[J]. J Antimicrob Chemother, 2014, 10:1-8.
[17]Fazeli H, Sadighian H, Esfahani BN, et al. Molecular epidemiology and mechanisms of antimicrobial resistance inPseudomonasaeruginosaisolates causing burn wound infection in Iran[J]. J Chemother, 2014, 2(4):222-228.
[18]Chen Y, Sun M, Wang M, et al. Dissemination of IMP-6-producingPseudomonasaeruginosaST244 in multiple cities in China[J]. Eur J Clin Microbiol Infect Dis, 2014, 33:1181-1187.
[19]Liakopoulos A, Mavroidi A, Katsifas EA, et al. Carbapenemase-producingPseudomonasaeruginosafrom central Greece:molecular epidemiology and genetic analysis of classⅠintegrons[J]. BMC Infect Dis, 2013, 29(13):505-512.
[20]Chen SH, Chen RY, Chen HT, et al. Multilocus sequence typing scheme ofPseudomonasaeruginosaisolates from naval divers[J]. Acad J Sec Mil Med Univ, 2012, 11(33):1241-1244. (in Chinese) 陈双红,陈锐勇,陈海庭,等.潜水人群铜绿假单胞菌基因多位点序列遗传分型[J].第二军医大学学报,2012,11(33):1241-1244.
Antibiotic-resistant genes and multilocus sequencing typing ofPseudomonasaeruginosa
YUAN Meng,YUAN Yue-ming,CHEN Hong-bin,LUO Jin-yan,YU Mu-hua,DUAN Yong-xiang
(MicrobiologicalLaboratoryDepartment,NanshanDistrictCenterforDiseaseControlandPrevention,Shenzhen518054,China)
We investigated the antibiotic-resistant genes and genetic diversity ofPseudomonasaeruginosafrom patients in hospital, the smear samples from hospital and clinic environment, and from medical staff’ hands respectively in 2011-2012 in Nanshan District of Shenzhen. Polymerase chain reaction were used to detect the 20 kinds of antibiotic-resistant genes (TEM,VEB,CARB,OXA,SHV,PER,GES,GTX,SPM,GIM,IMP,VIM,DHA,oprD,Aac(6′)-Ⅰ,Aac(6′)-Ⅱ,Aac(3′)-Ⅰ,Aac(2″)-Ⅰ,qacE1-sullandint-Ⅰ). Multilocus sequencing typing was used to analyze the clonal complexes. The 11 kinds resistant genesTEM,SHV,IMP,DHA,Aac(6′)-Ⅰ,Aac(6′)-Ⅱ,Aac(3′)-Ⅰ,Aac(2″)-Ⅰ,qacE1-sull,int-ⅠandoprDwere detected, for the positive rates respectively, and which were 8.1%, 6.4%, 4.8%, 9.7%, 4.8%, 14.5%, 9.7%, 56.5%, 8.1%, and 8.1%; the loss rate ofoprDgene was 61.2%. The 19 antibiotic resistance gene profiles existed in 52Pseudomonasaeruginosastrains. Multilocus sequencing typing found 39 sequence types and 5 clonal complexes in 62Pseudomonasaeruginosastrains, CC244 and ST856 were dominant. There were some differences of antibiotic resistance gene profiles between different samples, thePseudomonasaeruginosastrains from patients carried multiple resistant genes. In our research, thePseudomonasaeruginosahad the genetic diversity and the dominant clonal complexes existed.
Pseudomonasaeruginosa; multi-drug resistance; antibiotic-resistant genes; polymeras chain reaction; multilocus sequencing typing; clonal complexes
深圳市南山区疾病预防控制中心微生物检验科,广东 518054;Email:yuanmeng726@163.com
10.3969/j.issn.1002-2694.2015.10.013
R378.99
A
1002-2694(2015)10-0957-06
2014-12-02;
2015-06-20
深圳市科技计划项目(201103296)

