长江刀鲚体内菌群PCR-DGGE指纹图谱及多样性比较分析
聂志娟徐钢春杜富宽沈芬华黄敏康顾若波
(1. 中国水产科学研究院淡水渔业研究中心, 农业部淡水渔业和种质资源利用重点实验室, 无锡 214128;2. 江苏省宜兴市新庄街道农业服务中心, 宜兴 214266; 3. 南京农业大学渔业学院, 无锡 214128)
长江刀鲚体内菌群PCR-DGGE指纹图谱及多样性比较分析
聂志娟1徐钢春1杜富宽1沈芬华2黄敏康3顾若波1
(1. 中国水产科学研究院淡水渔业研究中心, 农业部淡水渔业和种质资源利用重点实验室, 无锡 214128;2. 江苏省宜兴市新庄街道农业服务中心, 宜兴 214266; 3. 南京农业大学渔业学院, 无锡 214128)
以长江刀鲚(Coilia nasus)洄游前幼鱼和洄游后成鱼为对象, 通过 PCR-DGGE 指纹技术探讨长江刀鲚菌群多样性及受洄游路径周围环境影响之后的稳定性。结果显示, PCR-DGGE 指纹谱带丰富, 共显示出 70条可鉴别条带, 其中长江水体谱带数(28)高于洄游后刀鲚鳃(26)、胃(26)、肠道壁(20)、肠道内容物(21)和洄游前刀鲚鳃(21)、胃(20)、肠道壁(11)、肠道内容物(13), 洄游后刀鲚成鱼体内各对应部位菌群数显著高于洄游前刀鲚幼鱼。UPGMA 聚类和PCA结果显示不同样品之间差异显著, 虽长江水体与洄游后刀鲚鳃、胃及肠道内容物样品在聚类图上聚为一簇, 但其菌群结构的相似度较低, 分别为43%、35%和28%。成功克隆测序其中 43条条带, 主要包含 α-变形菌(25.6%)、β-变形菌(7%)、γ-变形菌(16.3%)、放线菌(25.6%)、厚菌门(9.3%)、拟杆菌(7%)、柔膜菌门(4.6%)、绿弯菌(2.3%)和未定义菌(2.3%)。以上结果表明长江刀鲚体内不同部位及其在洄游前后不同阶段, 菌群结构存在显著差异, 并受环境和宿主双层因素影响。
PCR-DGGE; 长江刀鲚; 洄游; 香农指数; 细菌菌落
微生物个体微小, 结构简单, 但数量庞大, 种类丰富在生命活动中扮演重要的角色, 对宿主生物体健康、营养和免疫等具有重要作用。关于鱼类微生物研究, 人类早在 1953 年就有研究报道[1], 随后有关水产微生物生态生理研究报道越来越多, 研究技术愈来愈成熟。不仅如此, 基于16S rDNA的PCR-DGGE基因指纹技术是一种不依赖分离纯化培养且菌群结构原位探究的有效方法, 目前已经成为微生物群落研究强有力工具之一[2], 可以较快速揭示某一环境样品中的微生物结构组成和动态变化[3]。数据表明, 该技术可鉴定出许多无法培养的菌种[4],广泛运用于土壤[5]、海洋湖泊[6]、食品微生物[7]及动物消化道[8]等不同生态环境菌群结构调查和种群动态变化分析。事实证明, 由于食性、基因等因素的影响, 不同种类鱼的肠道细菌组成和结构也不尽相同。Ward等[9]在研究得出杂食性的南极岩斑鳕鱼(Notothenia coriiceps)比肉食性的头带冰鱼(Chaenocephalus aceratus)肠道微生物丰富, 也已有研究报道指出不同生长环境下鱼类的肠道细菌群落有明显的差异[10]。尽管如此, 关于洄游性鱼在不同洄游路径阶段体内菌群结构的报道较少。
刀鲚(Coilia nasus) 隶属鲱形目(Clupeiformes)鳀科(Engraulidae) 鲚属(Coilia), 又名长颌鲚, 俗称刀鲚[11]。刀鲚主要分布在中国、日本和朝鲜半岛, 包括洄游、淡水定居及海水定居 3种生态类型, 其中洄游生态型[12]刀鲚生活史中经历两种不同的水域环境, 春季性成熟后从近海口溯江产卵繁殖, 秋季幼鱼沿水返海肥育。其肉质鲜美, 营养丰富, 与河豚(Tetraodontidae fasciatus)、鲥(Tenualosa reevesii)并称为中国长江三鲜, 为名特优珍稀鱼类。
本试验通过 PCR-DGGE 技术检测刀鲚微生物区系组成, 对比探究洄游前后刀鲚菌群结构差异及多样性分析, 探讨长江刀鲚菌群受洄游路径周围环境影响之后的稳定性, 将有助于刀鲚洄游路径中菌群变化特征及长江刀鲚肠道固有菌群的确定, 为肠道细菌的功能定位提供基础依据及刀鲚养殖专用益生菌的开发奠定了基础。
1 材料与方法
1.1 样品采集
本实验样品鱼采集位于长江靖江段, 采集时间为: 2012年9月随机抽取10尾新鲜幼鱼(洄游前刀鲚), 体重为(4.94±2.57) g, 体长(106.2±14.4) mm;2014年 4月采集 10尾长江刀鲚成鱼(洄游后刀鲚),体重为(114.51±27.91) g, 体长(280.1±63.2) mm, 同时在刀鲚捕捞处采集水深1.5 m左右水样1 L, 放置于冰上运输至实验室。
1.2 样品处理
在超净工作台上处理实验鱼, 先用75 %酒精喷洒体表、无菌的解剖剪刀分别取出长江刀鲚鳃、胃、肠及肠内容物, 将同一批次的采集样品的相同组织合并于–30℃保存备用。
水样处理在超净工作台上操作, 用0.22 µm 的无菌滤膜和真空抽滤装置过滤水样, 在无菌条件下将滤膜上的过滤物用无菌水在无菌培养皿中冲洗下来, 在 12000 r/min离心 5min, 收集沉淀物保存于–30℃。
1.3 总DNA提取
所有样品在提取 DNA 之前需完全解冻。DNA提取参照Yu等[13]的方法, 并结合传统的CTAB法稍作修改。称取约 0.25—0.5 g样品到灭菌的2 mL离心管, 加入1 mL 细胞裂解液(1.4 mol/L NaCl, 2% CTAB, 100 nmol/L Tric-HCl, 50 mmol/L EDTA), 20 µL 20 mg/mL蛋白酶k, 20 µL 20 mg/mL 溶菌酶, 漩涡震荡5min; 37℃ 250 r/min摇床振动 1h, 加入100 µL的 20% SDS, 漩涡混匀 3min, 65℃水浴 40min, 13000 r/min 4℃离心5min, 将上清吸入2 mL离心管中; 加入等体积的酚∶氯仿∶异戊醇抽提, 12000 r/min离心 5min, 取上清; 加入等体积酚∶氯仿∶异戊醇重复抽提纯化一次; 取上清液加入等体积氯仿∶异戊醇去酚纯化; 上清液吸入至新的无菌离心管加入0.6倍体积的异丙醇沉淀, –20℃, 放置 30min;12000 r/min离心10min, 弃上清; 用70%预冷乙醇洗涤沉淀, 无菌风吹干, 加适量无菌水溶解 DNA。样品各取 5 µL DNA, 0.7 %琼脂糖凝胶电泳进行检测。
1.4 PCR-DGGE分析
PCR具体扩增引物和条件参考文献[14]进行。将具有清晰条带PCR的产物进行变形梯度凝胶电泳(DGGE)分析。凝胶浓度为8%的聚丙烯酰胺凝胶[化学变性剂为100%尿素7 mol/L和40%(v/v)的丙烯酰胺];变形梯度为35%—55%。电泳条件: 60℃, 在1×TAE缓冲液中150 V下电泳10h。银染步骤参考郁二蒙等[15]的方法进行。在银染后, 在Vilber凝胶成像扫描系统中照像, 并用BIO-RAD Quantity One 软件对DGGE指纹图谱进行分析, 通过Biodap软件对样品泳道灰度值进行分析计算得出一些比较不同样品微生物多样性的基本指标。电泳条带的数量可以代表样品群落的丰富度指数; 香农多样性指数(Shannon diversity index)反应群落多样性, Evenness 代表样品群落的均匀度; Berger-Parker Index 用来表示优势度[16]。
1.5 DGGE图谱中部分优势条带的回收与测序分析
用灭菌的手术刀切割待回收DGGE条带, 加入30 μL无菌水4℃静置过夜。以DNA浸出液为模板, 338F/518R为引物进行PCR扩增。PCR扩增体系和程序参考文献[17]。将有扩增结果为200 bp左右的PCR产物进行 TA克隆, 并挑选阳性克隆子寄至上海博尚生物技术有限公司进行序列测定, 测序结果去载体并NCBI进行BLAST同源性比对分析。
2 结果
2.1 DGGE指纹图谱建立及分析
采用 PCR-DGGE 技术分析刀鲚洄游前后体内微生物群落多样性, 结果显示(图 1), 指纹图谱上共产生了70条可鉴别的条带, 每一条泳道代表一种来源样品的 DGGE 指纹图谱, 不同位置的条带代表不同的菌群, 长江水体、鳃(洄游后)、鳃(洄游前)、胃(洄游后)、胃(洄游前)、肠道壁(洄游后)、肠道壁(洄游前)、肠道内容物(洄游后)、肠道内容物(洄游前)分别具有可鉴别的条带为28、26、21、26、20、20、11、21和13条, 其中, 长江水体的条带数量为最多。条带的数量的多少反应了该样品细菌的多样性程度,充分显示了刀鲚及其生存环境样品的菌群多样性。各泳道含有数目、迁移率及信号强度各异的条带,不同样品的菌落带型有明显差别, 其中数量、位置和亮度的差异表明长江刀鲚洄游路径体内菌群组成是不同的, 反映了由江入海的环境差异影响长江刀鲚体内菌群结构组成。指纹图谱带型的差别可以很好地反映出长江洄游细菌种群组成的差异与相同之处。指纹图谱UPGMA聚类分析图(图2)。长江水体与洄游后刀鲚鳃、胃及肠道内容物样品在聚类图上聚为一簇, 菌群结构的相似度为分别 43%、35%和28%。表明同一水体环境内刀鲚体内物质交替频繁样品间菌落结构具有一定的相关性, 由于此类样品在空间位置上紧密相连、相互影响所致。

图1 菌群 16S rDNA变性梯度凝胶电泳(DGGE) 指纹图谱及模式图(1—70: 条带数)Fig. 1 The DGGE fingerprint profiles on 16S ribosomal DNA (rDNA) of the bacterial communities

图2 菌群的UPMGA 聚类分析Fig. 2 The UPMGA clustering analysis of the bacterial communities
根据DGGE图谱各泳道的灰度值计算基本参数进行多样性比较分析(表 1), 长江水体丰富度(条带数)27及香农指数为3.23, 明显高于洄游前后长江刀鲚体内菌群种类, 同时刀鲚鳃(洄游后)、鳃(洄游前)、胃(洄游后)、胃(洄游前)、肠道壁(洄游后)、肠道壁(洄游前)、肠道内容物(洄游后)、肠道内容物(洄游前)香农多样性指数分别为2.96、2.83、3.10、2.92、2.85、2.17、2.87和2.47, 可以代表各个样品细菌多样性。从均匀度指数比较, 样品之间均匀度无较大差别,洄游前长江刀鲚肠道壁均匀度最差(0.91), 而菌落优势度指数较大(0.164); 长江水体、洄游后刀鲚鳃、胃、肠道壁及肠道内容物均匀度最高(0.97), 反映优势度较小, 与优势度指数(都小于 0.097)结果显示一致。PCA结果显示(图 3): 不同样品之间距离较远,存在明显差异; 第一轴贡献率 23.2%, 第二轴贡献率17.9%, 在轴一上清晰显示不同样品之间差异。
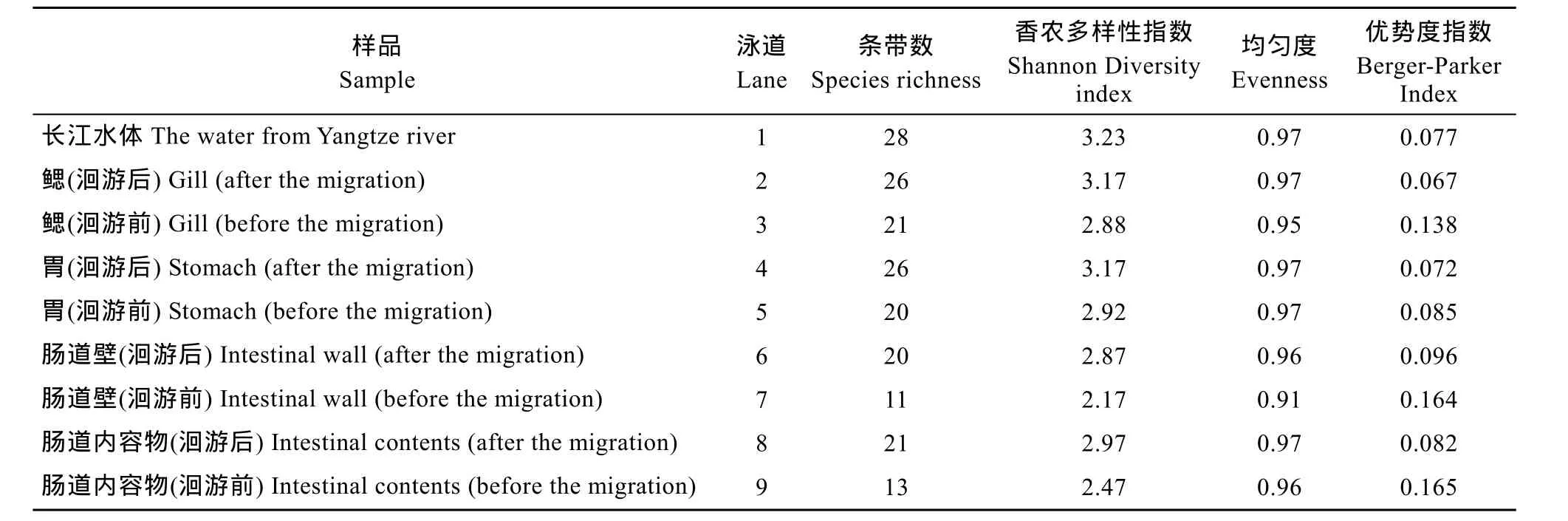
表1 DGGE指纹图谱微生物多样性指数Tab. 1 The microbial diversity indices calculated from the DGGE banding patterns

图3 DGGE指纹图谱PCAFig. 3 PCA of DGGE profiles
2.2 部分优势条带的序列比对与系统发育树分析
对DGGE指纹图谱中70条带全部切胶回收, 其中 43条条带被成功克隆测序, 测序结果比对制表分析(表2)。在43个测序结果中, 与GenBank数据库中微生物的同源性为 94%以上, 67%菌同源性达到了100%, 主要包含α-变形菌(25.6%)、β-变形菌(7%)、γ-变形菌(16.3%)、放线菌(25.6%)、厚菌门(9.3%)、拟杆菌(7%)、柔膜菌门(4.6%)、绿弯菌(2.3%)和未定义菌(2.3%)。养殖水体菌群最丰富, 共显示28条带, 不同条带浓度及显示的灰度各异, 条带band2、12、20、24、25、28、31、37、46、55和67为养殖水体的相对优势菌群, 成功测序结果中包含厚壁菌、柔膜菌门、α-变形菌、β-变形菌、γ-变形菌和放线菌。洄游前后刀鲚鳃和胃菌群种类较丰富、条带数 20以上,水体与洄游前后刀鲚鳃相似度相对较高, band26、28、41、42、48、49、51和59为其共有带菌, 为变形菌和放线菌, 序列比对相似度为 99%以上; band33检测为唯一绿弯菌, 存在于长江水体、洄游前刀鲚鳃胃部及洄游后刀鲚胃肠道壁样品中。

表2 DGGE谱带回收获得的16S rDNA序列分析比对结果Tab. 2 Sequence similarities to closest relatives of 16S rDNA recovered from the DGGE gel
3 讨论
目前对于水产动物菌群的研究较多, 较早的研究采用传统的可培养方法, 16S rDNA 基因文库构建法分析群落结构组成[18,19], 但因在自然条件下多为不可培育菌及克隆法费时费力不直观, 之后则发展出如PCR-DGGE、T-RFLP、PCR-SSCP等多种高效的监视微生物群落动态的分子生物学方法[20]。DGGE技术依据16S rDNA可变区核酸物质的高分辨分离提供微生物群落多样性的指纹图谱被广泛应用, 但DGGE和其他方法一样也有它的缺点。DGGE通常只能检测到群落中含量超过1%优势菌种的16S rDNA 片段[21], 较多研究结果揭示某些单一菌种16S rDNA存在多拷贝或者异源双链, 序列相同但因碱基构象不同, 显示在图谱的不同位置[22—24], 本研究结果也再次佐证这一结论, 如图谱中条带47、48、54和57序列相同; 条带8和65序列一致, 与Clostridium aurantibutyricum (NR_044841.2) 相似度为100%。尽管DGGE 技术与理论预期存在一定的差距, 但倪加加等[25]对 DGGE凝胶条带内 DNA回收测序评估DGGE 可靠性得出DGGE 技术基本能够反映微生物群落的多样性。
本文采用香浓多样性指数反映刀鲚菌群多样性,由表 1 可以看出洄游前后刀鲚各部位菌群丰富,多样性指数高 (2.17—3.17), 远远高于锯缘青蟹(Scylla serrata)(0.897—1.309)[26]; 此外, 多样性指数差异明显, 洄游后刀鲚鳃、胃、肠道壁和肠道内容物比洄游前刀鲚对应部位香浓多样性指数值都高,及可能由洄游后刀鲚经历海洋生境生长、生存环境更为复杂状况所导致的。DGGE图谱聚类分析得出,虽长江水体与洄游后刀鲚鳃、胃及肠道内容物样品在聚类图上聚为一簇, 但其菌群结构的相似度较低,分别为 43%、35%和 28%; 洄游型刀鲚不同时期的相似性不高, 菌群结构差异明显, 同时洄游前刀鲚体内各部位样品之间菌群结构相似度更低(<29%),充分显示宿主本身对菌群结构的分布起到决定性作用。Li等[27]探究四种同一养殖条件下的白鲢(Hypophthalmichthys molitrix)、鳙(Hypophthalmichthys nobilis)、草鱼(Ctenopharyngodon idellus)和团头鲂(Megalobrama amblycephala)的仔鱼肠道细菌群落结构, 证明 4种仔鱼宿主显著地影响着其肠道菌群结构。不同生长时期(洄游前和洄游后), 刀鲚鳃部菌群结构相似度为 33.8%, 而同一养殖条件下三疣梭子蟹(Portunus trituberculatus)与锯缘青蟹[28]鳃组织内黏附菌群结构相似度为 75%, 相似度差异如此明显, 直接显示环境变更也将较大程度影响菌群结构。刀鲚从开口摄食, 体内不同部位就开始黏附并寄生菌群, 逐渐形成特有的优势菌种, 从而造成了与外部环境菌群结构的差异, 但同时又受制于外部环境的影响。
DGGE图谱序列测定结果显示, 刀鲚菌群鉴定主要分类于变形菌、放线菌、厚壁菌、拟杆菌、柔膜菌、绿弯菌和少量未定义菌种, 菌群类别丰富, 其中变形菌类群又包含 α、β、γ亚群, 占测定菌群48.9%, 为刀鲚的绝对优势菌群。Kim等[29]分析虹鳟(Oncorhynchus mykiss)肠道菌群, 发现大于 70%的细菌属于变形菌门; 也有许多研究发现这些细菌多为正常健康水产动物中普遍存在的菌群类别, 发挥着辅助消化, 增强免疫, 拮抗外来病原菌群提供营养物质等功效[30], 其中 Sugita等[31]研究得出, 专性厌氧菌如拟杆菌(Bacteroides)、梭菌属(Clostridium)是维生素B12的主要产生菌; Rawls等[32]发现, 斑马鱼(Barchydanio rerio var)消化道内菌群能够调控肠道 212个基因, 其中部分菌群能够刺激上皮细胞增殖和先天性免疫应答与促进营养物质代谢。鱼类寄生菌群作为微生物的一个资源库, 富含许多不同特性的功能细菌, 亟需深入研究及开发。本研究中菌群指纹图谱显示丰富的菌群结构, 但较多优势菌群未成功鉴定, 需结合其他研究方法进一步深入探究刀鲚优势菌群的功能特性。
[1] Margolis L. The effect of fasting on the bacterial flora of the intestine of fish [J]. Journal of the Fisheries Board of Canada, 1953, 10(2): 62—63
[2] Muyzer G, Smalla K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology [J]. Antonie van Leeuwenhoek, 1998, 73(1): 127—141
[3] Muyzer G, De Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reactionamplified genes coding for 16S rRNA [J]. Applied and Environmental Microbiology, 1993, 59(3): 695—700
[4] Mccracken V J, Simpson J M, Mackie R I ,et al. Molecular ecological analysis of dietary and antibiotic-induced alterations of the mouse intestinal microbiota [J]. TheJournal of Nutrition, 2001, 131(6): 1862—1870
[5] Nakatsu C H, Torsvik V, Øvreås L. Soil community analysis using DGGE of 16S rDNA polymerase chain reaction products [J]. Soil Science Society of America Journal, 2000, 64(4): 1382—1388
[6] Li Z Y, He L M, Wu J, et al. Bacterial community diversity associated with four marine sponges from the South China Sea based on 16S rDNA-DGGE fingerprinting [J]. Journal of Experimental Marine Biology and Ecology, 2006, 329(1): 75—85
[7] Hovda M B, Sivertsvik M, Tore Lunestad B, et al. Characterisation of the dominant bacterial population in modified atmosphere packaged farmed halibut (Hippoglossus hippoglossus) based on 16S rDNA-DGGE [J]. Food Microbiology, 2007, 24(4): 362—371
[8] Hovda M B, Lunestad B T, Fontanillas R, et al. Molecular characterisation of the intestinal microbiota of farmed Atlantic salmon (Salmo salar L.) [J]. Aquaculture, 2007, 272(1): 581—588
[9] Ward N L, Steven B, Penn K, et al. Characterization of the intestinal microbiota of two Antarctic notothenioid fish species [J]. Extremophiles, 2009, 13(4): 679—685
[10] Spanggaard B, Huber I, Nielsen J, et al. The microflora of rainbow trout intestine: a comparison of traditional and molecular identification [J]. Aquaculture, 2000, 182(1): 1—15
[11] Zhuang P, Wang Y K, Li S F, et al. The Fish of Yangtze Estuary [M]. Shanghai: Shanghai Scientific and Technical Publishers. 2006, 153—157 [庄平, 王幼槐, 李圣法, 等.长江口鱼类. 上海: 上海科学技术出版社. 2006, 153—157]
[12] Huang R S. The biological characteristics, current status and protection measures of Coilia nasus [J]. Reservoir Fisheries, 2005, 25(2): 33—34 [黄仁术. 刀鲚的生物学特性及资源现状与保护对策, 水利渔业, 2005, 25(2): 33—34]
[13] Yu Z, Morrison M. Improved extraction of PCR-quality community DNA from digesta and fecal samples [J]. Biotechniques, 2004, 36(5): 808—813
[14] Ruamkuson D, Tongpim S, Ketudat-Cairns M. A model to develop biological probes from microflora to assure traceability of tilapia [J]. Food Control, 2011, 22(11): 1742—1747
[15] Yu E M, Yu D G, Bi X M, et al. Fingertprinting and diversity of the intestinal bacterial community of crisp grass carp and Dang-zai Grass Carp (Ctenopharyngodon idellus) with PCR-DGGE [J]. Journal of Agricultural Biotechnology, 2012, 20(10): 1184—1191 [郁二蒙, 余德光, 毕香梅, 等. 脆化草鱼与 氹仔草鱼的肠道细菌群落PCR-DGGE 指纹图谱及多样性分析. 农业生物技术学报, 2012, 20(10): 1184—1191]
[16] Hill T C J, Walsh K A, Harris J A, et al. Using ecological diversity measures with bacterial communities [J]. FEMS Microbiology Ecology, 2003, 43(1): 1—11
[17] Zhou Z G, Shi P J, Yao B, et al. Comparison of the predominant bacterial community structure in the gastrointestinal wall between Lutjanus sebae and Ephippus orbis based on 16s r DNA PCR-DGGE fingerprint [J]. Acta Hydrobiologica Sinica, 2007, 31(5): 682—688 [周志刚, 石鹏君, 姚斌, 等. 基于 PCR-DGGE 指纹图谱川纹笛鲷及圆白鲳消化道壁优势菌群结构比较分析. 水生生物学报, 2007, 31(5): 682—688]
[18] Zhao Q X. An analysis of intestinal microflora of cyprinidae [J]. Micobiology, 2001, 21(2): 18—20 [赵庆新. 鲤科鱼肠道菌群分析. 微生物学杂志, 2001, 21(2): 18—20]
[19] Ling Z C, Yang H L, Sun Y Z, et al. Cultivablemicrobiota in the gut of juvenile grouper Epinephelus coioides and in aquaculture water [J]. Journal of Dalian Fisheries University, 2009, 24(6): 497—503 [凌泽春, 杨红玲, 孙云章, 等. 斜带石斑鱼幼鱼消化道与养殖水体中可培养菌群的研究.大连水产学院学报, 2009, 24(6): 497—503]
[20] Liang W, Wu S Q, Wu Z B. The application of molecular techniques to characterize the microbial communities in constructed wetland [J]. Ecology and Environmental Sciences, 2010, 4: 42 [梁威, 吴苏青, 吴振斌. 分子技术在湿地微生物群落解析中的应用. 生态环境学报, 2010, 4: 42]
[21] Ma Y X, Holmstrm C, Webb J, et al. Application of denaturing gradient gel electrophoresis (DGGE) in microbial ecology [J]. Acta Ecologica Sinica, 2003, 23(8): 1561—1569 [马悦欣, Holmstrm C, Webb J, 等. 变性梯度凝胶电泳(DGGE) 在微生物生态学中的应用. 生态学报, 2003, 23(8): 1561—1569]
[22] Muyzer G, De Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reactionamplified genes coding for 16S rRNA [J]. Applied and Environmental Microbiology, 1993, 59(3): 695—700
[23] Xing D F, Ren N Q. Common problems in the analyses of microbial community by denaturing gradient gel electrophoresis (DGGE) [J]. Acta Microbiologica Sinica, 2006, 46(2): 331—335 [邢德峰, 任南琪. 应用DGGE 研究微生物群落时的常见问题分析. 微生物学报, 2006, 46(2): 331—335]
[24] Wintzingerode F V, Göbel U B, Stackebrandt E. Determination of microbial diversity in environmental samples: pitfalls of PCR-based rRNA analysis [J]. FEMS Microbiology Reviews, 1997, 21(3): 213—229
[25] Ni J J, Yi Y H, Wu H H, et al. Effects generated by different band extracting methods in the analysis of DGGE profile [J]. Acta Hydrobiologica Sinica, 2012, 36(5): 1009—1011 [倪加加, 余育和, 吴含含, 等. 不同DGGE 谱带信息提取方法对分析结果的影响. 水生生物学报, 2012, 36(5): 1009—1011]
[26] Cai L P. Xu H S, He L, et al. Analysis of the intestinalbacterial communities in wild Scylla serrata from different districts by PCR-DGGE [J]. Acta Agriculturae Zhejiangensis, 2011, 23(2): 278—282 [蔡丽萍, 徐海圣, 何琳, 等. PCR-DGGE 技术分析不同地区野生锯缘青蟹肠道菌群多样性. 浙江农业学报, 2011, 23(2): 278—282]
[27] Li X, Yu Y, Feng W, et al. Host species as a strong determinant of the intestinal microbiota of fish larvae [J]. The Journal of Microbiology, 2012, 50(1): 29—37
[28] Wang F, Yang J F, Cheng J G, et al. Comparison of the bacterial community structure in the crab seawater cultured and the outside environment by PCR-DGGE fingerprint technique: Portunus trituberculatus and Scylla serrata [J]. Journal of Marine Sciences, 2010, 28(4): 59—64 [王芳, 杨季芳, 陈吉刚, 等. 海水养殖蟹体内与外部环境中菌群结构的 PCR-DGGE 比较——以三疣梭子蟹和锯缘青蟹为例. 海洋学研究, 2010, 28(4): 59—64]
[29] Kim D H, Brunt J, Austin B. Microbial diversity of intestinal contents and mucus in rainbow trout (Oncorhynchus mykiss) [J]. Journal of Applied Microbiology, 2007, 102(6): 1654—1664
[30] Song Z F, Wu T X. Review on intestinal normal microflora in fish [J]. Fisheries Science, 2007, 26(8): 471—474 [宋增福, 吴天星. 鱼类肠道正常菌群研究进展. 水产科学, 2007, 26(8): 471—474]
[31] Sugita H, Miyajima C, Deguchi Y. The vitamin B12-producing ability of the intestinal microflora of freshwater fish [J]. Aquaculture, 1991, 92: 267—276
[32] Rawls J F, Samuel B S, Gordon J I. Gnotobiotic zebrafish reveal evolutionarily conserved responses to the gut microbiota [J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(13): 4596—4601
PCR-DGGE FINGERPRINTING AND DIVERSITY ANALYSIS OF THE PREDOMINANT BACTERIAL COMMUNITY IN COILIA NASUS
NIE Zhi-Juan1, XU Gang-Chun1, DU Fu-Kuan1, SHENG Fen-Hua2, HUANG Min-Kang3and GU Ruo-Bo1
(1. Key Laboratory of Freshwater Fisheries and Germplasm Resources Utilization, Ministry of Agriculture, Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214128, China; 2. Xinzhuang street agricultural service center, Yixing 214266; 3. College of Fisheries, Nanjing Agriculture University, Wuxi 214128, China)
In this study we investigated the diversity and stability of the bacterial community structure in the juvenile (before the migration) and adult (after the migration) Coilia nasus using PCR-DGGE. The DGGE fingerprint bands were abundant and there were 70 detectable bands with different signal intensities. The number of bands in the water (28) was higher than that in the gill, the stomach, the intestinal wall, and the intestinal contents of Coilia nasus before and after the migration. And the numbers of the bands in the tested organs in adult Coilia nasus were higher than those in juvenile Coilia nasus. The UPGMA clustering and PCA analysis of the DGGE fingerprint showed significant differences between samples. Between the water sample and the post-migration Coilia nasus, the similarities of the bacteria structures in the fish gill, the stomach and the intestinal contents were only 43%, 35% and 28% respectively. Forty-three DGGE bands were successfully cloned including α-Proteobacteria (25.6%), β-Proteobacteria (7%), γ-Proteobacteria (16.3%), Actinobacteria (25.6%), Firmicutes (9.3%), Bacteroidetes (7%), Tenericutes (4.6%), Chloroflexi (2.3%), and some unclassified bacteria (2.3%). These results revealed that the bacterial community varied significantly at different migration stages, and in different bacterial parasitic parts of Coilia nasus. Therefore the external environment and the host should be the main factors affecting the composition of a bacterial community.
PCR-DGGE; Coilia nasus; Migration; Shannon index; Bacterial community
Q938.8
A
1000-3207(2015)05-1019-08
10.7541/2015.133
2014-10-17;
2015-03-26
公益性行业科研专项(No.201203065); 国家科技支撑计划项目(No.2012BAD26B05); 中央级公益性科研院所基本科研业务费专项资金项目(No.2013JBFT04); 农业部东海与远洋渔业资源开发利用重点实验室开放课题资助
聂志娟(1985—), 女, 江西新干人; 助理研究员; 研究方向为水产微生物学。E-mail: niezj@ffrc.cn
顾若波, E-mail: gurb@ffrc.cn

