规模化引种场进口奶牛隐孢子虫种类、基因亚型鉴定及其生物安全评价
王臣荣,齐 萌,李俊强,张振杰,刘鹏飞,余复昌,曹建科,宁长申,张龙现
规模化引种场进口奶牛隐孢子虫种类、基因亚型鉴定及其生物安全评价
王臣荣,齐 萌,李俊强,张振杰,刘鹏飞,余复昌,曹建科,宁长申,张龙现
目的 为了解进口奶牛寄生的隐孢子虫对引种场污染情况及是否影响隐孢子虫种类和基因型在当地的分布。方法 采用PCR方法检测河南省某规模化引种场奶牛隐孢子虫感染情况。结果 基于18S rRNA基因位点进行PCR检测,奶牛隐孢子虫总感染率为17.8%(90/507),鉴定出4种隐孢子虫,分别为微小隐孢子虫、牛隐孢子虫、芮氏隐孢子虫和安氏隐孢子虫,隐孢子虫感染率随牛年龄增长而呈递减趋势,差异有统计学意义(P<0.01)。断奶前犊牛以微小隐孢子虫为优势感染种,断奶前犊牛腹泻与微小隐孢子虫感染呈正相关性,差异有统计学意义(P<0.01)。基于gp60基因位点,43份微小隐孢子虫阳性样品成功扩增出35份,序列分析显示35个微小隐孢子虫均为人兽共患基因亚型IIdA19G1, 而奶牛引种地(澳大利亚)的牛源微小隐孢子虫均为IIa亚型家族存在显著不同。结论 证实微小隐孢子虫为犊牛腹泻病原之一,IId亚型为中国独特分布的微小隐孢子虫亚型,其基因亚型存在地理隔离遗传特征,引种青年牛和成年牛不影响当地重要人兽共患虫种微小隐孢子虫基因亚型分布,从这个角度考虑不具有生物安全重要性。
隐孢子虫;种类;基因亚型;鉴定;奶牛
隐孢子虫是重要的人兽共患原虫之一,宿主谱包括人在内的260多种脊椎动物,可致以腹泻为主要临床症状的隐孢子虫病[1]。隐孢子虫多通过“粪-口”途径传播,牛被认为是人类隐孢子虫病的重要传染源,牛粪污染水源和食物是导致人体隐孢子虫病暴发的主要因素[2-3]。寄生于牛的隐孢子虫主要有4种:微小隐孢子虫(Cryptosporidiumparvum)、安氏隐孢子虫(Cryptosporidiumandersoni)、牛隐孢子虫(Cryptosporidiumbovis)和芮氏隐孢子虫(Cryptosporidiumryanae)[1]。微小隐孢子虫是最重要的人兽共患隐孢子虫虫种,世界范围内人体隐孢子虫病例约45%由该虫引起,而其它3个寄生于牛的虫种均具有宿主特异性[1,4]。研究表明,冠状病毒(coronavirus)、轮状病毒(rotaviruses)、肠毒素性大肠杆菌(enterotoxicEscherichiacoli)和微小隐孢子虫是引起犊牛腹泻的4个主要病原,以轮状病毒和微小隐孢子虫更为常见[5]。
微小隐孢子虫具有广泛的宿主范围,为阐明该虫种动力学传播和人兽共患的潜在威胁性,基因亚型分析工具常用于该虫群体遗传结构鉴定和人兽共患风险评价[6-7]。研究表明,微小隐孢子虫群体遗传存在较大的种内变异,基于60-kDa 糖蛋白(gp60)基因分为IIa-i,IIk-o共14个亚型家族,其中IIa和IId亚型家族可在人和反刍动物之间传播,而IIc和IIe亚型家族仅通过人-人途径传播,其它亚型家族多具有宿主特异性[4,6,8-12]。
我国已成为世界上最大种牛进口国,2008-2013年从澳大利亚、新西兰和乌拉圭等国进口奶牛46万头。在我国与主要进口国双边签署的动物检疫条款中,隐孢子虫未作为检查项目。
1 材料方法
1.1 样品采集 调查样品来自河南省某规模化引种奶牛场,该场于2013年4月20日从澳大利亚引进1 000头荷斯坦成年奶牛,采用人工授精方式进行配种繁育。2014年6月至2014年12月对该场随机经直肠采集507头奶牛新鲜粪便样品,其中有腹泻症状的断奶前犊牛85头,每份10~30 g,装入洁净自封袋中,记录信息,带回实验室,置4 ℃保存。奶牛年龄划分参照中华人民共和国农业部《奶牛标准化规模养殖生产技术规范(试行)》,<60 d为断奶前犊牛,61~180 d为断奶后犊牛,181~450 d为育成牛,>450 d 为成年牛。
1.2 DNA提取 取200 mg粪便样本,用美国OMEGA Bio-Tek公司生产的粪便DNA提取试剂盒(E.Z.N.A.®Stool DNA Kit,D4015-02),购于郑州科贸生物技术有限公司,按说明书步骤操作,提取的DNA置-20 ℃保存。
1.3 PCR扩增 基于18S rRNA基因对所有样品进行隐孢子虫检测和虫种鉴定,引物及反应程序参照Xiao等[13]的方法。基于gp60基因对微小隐孢子虫进行基因亚型鉴定,引物及反应程序参照Sulaiman等[14]的方法。引物由生工生物工程(上海)股份有限公司合成。反应体系中使用的酶为KOD-plus酶0.5 μL (日本TOYOBO公司生产,购于郑州科贸生物技术有限公司),每次PCR扩增均设阳性对照样品和阴性对照样品。电泳结束后在电泳凝胶成像系统仪中观察拍照。
1.4 测序 PCR产物测序委托北京诺赛基因有限公司完成,采用双脱氧终止法进行双向测序,测序仪为ABI PRISMTM®3730 XL DNA Analyzer (Applied Biosystems,USA)。在GenBank上用Blast进行同源序列搜索,应用Clustal X 1.83进行比对分析。
1.5 统计学分析 统计学分析采用χ2检验。
2 结 果
2.1 隐孢子虫感染情况 基于18S rRNA基因位点对507份样品进行PCR扩增和测序,90份鉴定为隐孢子虫阳性,总感染率为17.8%。鉴定出4种隐孢子虫,以微小隐孢子虫为优势感染虫种,所占阳性样品比例为47.8%,牛隐孢子虫、赖氏隐孢子虫和安氏隐孢子虫所占比例分别为41.1%、5.6%和5.6%。见表1。隐孢子虫感染率随牛年龄增长而呈递减趋势,断奶前犊牛感染率达38.8%,断奶后犊牛、育成牛和成年牛的感染率分别为15.6%、3.8%和1.0%。见表1。不同年龄段奶牛隐孢子虫感染率差异有统计学意义(P<0.01)。断奶前犊牛以微小隐孢子虫为优势感染虫种,断奶后犊牛、育成牛和成年牛均以牛隐孢子虫为优势感染虫种。见表1。
2.2 不同临床症状断奶前犊牛感染情况 本次调查中,断奶前犊牛多在7~25 d出现腹泻症状。断奶前腹泻和未腹泻犊牛隐孢子虫的感染率分别为44.7%和32.5%,无统计学差异(P>0.05)。见表2。断奶前犊牛微小隐孢子虫为优势虫种,与腹泻呈正相关关系,断奶前腹泻与未腹泻犊牛微小隐孢子虫感染率差异有统计学意义(P<0.01)。断奶前犊牛感染牛隐孢子虫与腹泻呈负相关关系,断奶前腹泻与未腹泻犊牛的牛隐孢子虫感染率差异有统计学意义(P<0.01)。见表2。
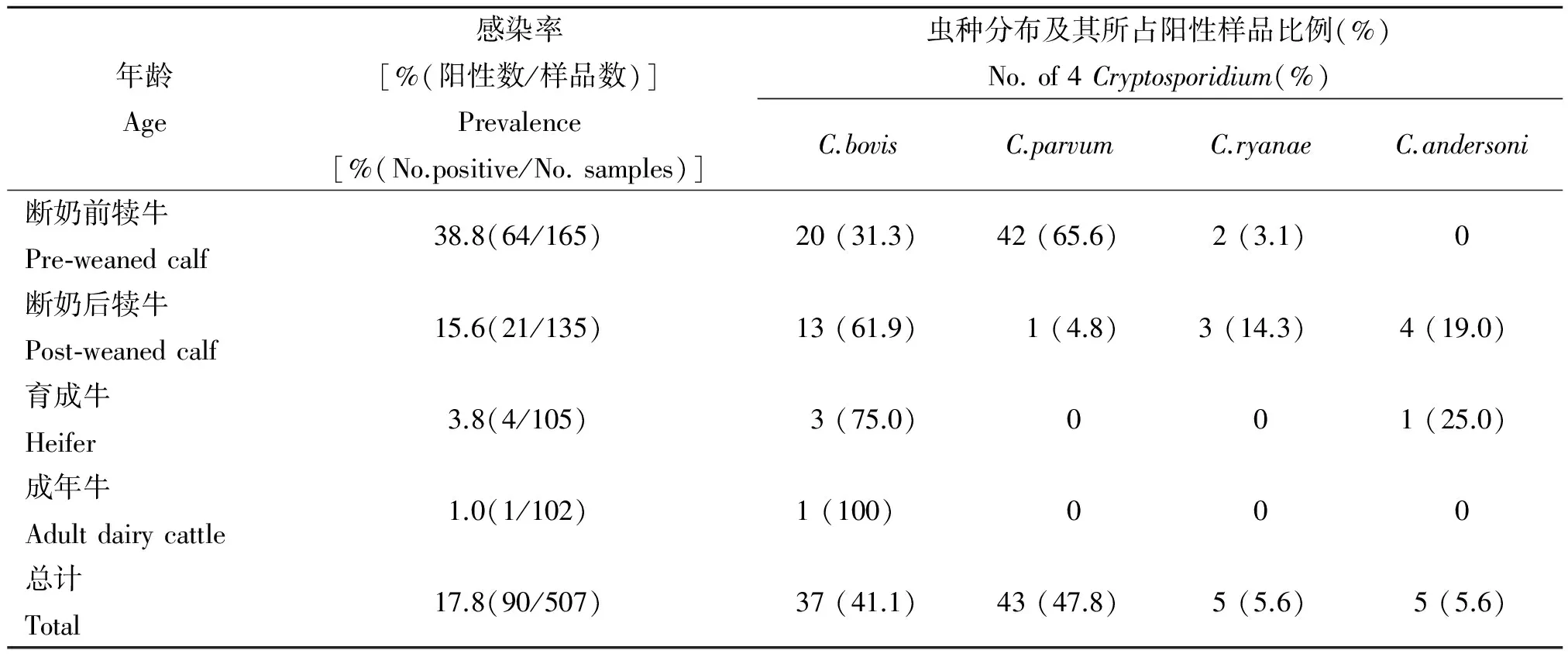
表1 不同年龄段奶牛隐孢子虫感染情况Tab.1 Infection of Cryptosporidium in dairy cattle in different age

表2 不同临床症状断奶前犊牛隐孢子虫感染情况Tab.2 Infection of Cryptosporidium in different clinical signs in pre-weaned calves
2.3 断奶前犊牛隐孢子虫种类分布 0~7 d、8~14 d、15~21 d、22~28 d、29~35 d、36~42 d、43~49 d和50`56 d犊牛隐孢子虫感染分别为17.4%、66.7%、42.1%、38.5%、52.0%、29.4%、18.2%和5.6%,统计学差异有统计学意义(P<0.01)。不同周龄断奶前犊牛隐孢子虫虫种分布存在显著差异,仅在<35 d犊牛中发现微小隐孢子虫感染,4 d即可检查到该虫感染,8~14 d 犊牛该虫感染率最高,为64.1%;牛隐孢子虫则主要感染4~7周龄犊牛,29~35 d 犊牛该虫感染率最高,为40.0%;芮氏隐孢子虫偶见感染4周龄后犊牛;未发现有C.andersoni感染。见图1。
2.4 微小隐孢子虫基因亚型序列分析 基于gp60基因位点对43份微小隐孢子虫进行基因亚型鉴定,成功扩增出35条序列。经Blast搜索和Clustal X 1.83 比对,所得35条序列均与微小隐孢子基因亚型IIdA19G1(GenBank登录号:HQ009809)序列同源性为100%,该基因亚型为河南省奶牛源微小隐孢子虫优势亚型。
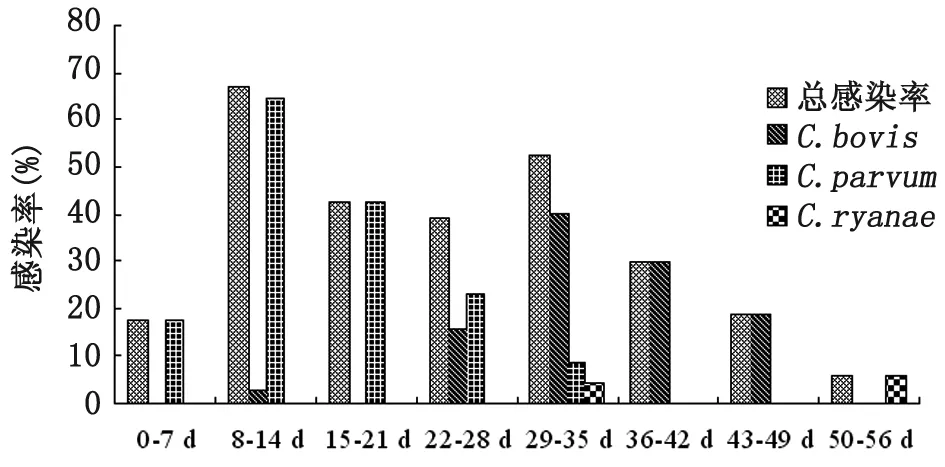
图1 不同周龄断奶前犊牛隐孢子虫虫种分布
Fig.1 Distribution ofCryptosporidiumspecies in different week ages of pre-weaned calves
3 讨 论
不同国家和地区,牛隐孢子虫病流行率存在差异较大,与其饲养管理、样本数量、年龄段分布以及检测方法等因素有关。本研究中奶牛隐孢子虫总感染率为17.8%,其中断奶前犊牛感染率最高,为38.8%,高于河南省、陕西省和宁夏回族自治区断奶前犊牛隐孢子虫感染率,分别为21.5%(172/801)、20.2%(52/258)和10.2%(19/186)[9,15-16],低于黑龙江省断奶前犊牛隐孢子虫感染率47.7%(72/151)[17]。断奶后犊牛感染率为15.6%,高于黑龙江省(5.5%,8/145)和河南省(11.3%,86/758)断奶后犊牛隐孢子虫感染率[18-19]。在澳大利亚西部,犊牛隐孢子虫感染率为48%(26/54)[20],东南部新南威尔士州断奶前犊牛隐孢子虫感染率为22.3%(81/364)[21]。美国一项调查研究显示,<8周龄的断奶前犊牛隐孢子虫感染率最高,为45.8%,依次是3~12月龄断奶后犊牛(18.5%)和1~2岁的母牛(2.2%)[22];国内的研究数据也显示幼龄奶牛隐孢子虫感染率高于成年牛[23]。本研究结果发现隐孢子虫感染率随牛年龄增长而呈递减趋势,与先前的国内外报道相一致。
感染牛的隐孢子虫虫种较多,除4种自然宿主为牛的隐孢子虫虫种外,还有7种隐孢子虫和一个基因型可感染牛科动物[24]。本研究发现该引种场奶牛感染4种主要感染牛隐孢子虫虫种,以微小隐孢子虫为优势感染虫种,与大多数发达国家研究报道相一致,微小隐孢子虫几乎是感染断奶前犊牛的唯一虫种[4]。在中国河南省、黑龙江省和陕西省的调查结果显示,牛隐孢子虫是感染断奶前犊牛的主要虫种,而在宁夏回族自治区的调查结果显示微小隐孢子虫是感染断奶前犊牛的优势虫种,表明犊牛感染微小隐孢子虫与地理区域分布有一定关系[9,12,15-17]。同样在澳大利亚的研究也表明犊牛感染微小隐孢子虫与地理分布有关,在澳大利亚西部断奶前犊牛微小隐孢子虫感染率为17.6%,而在维多利亚州感染率为46.3%[24]。在美国、丹麦、瑞士等国家的研究发现,牛隐孢子虫是断奶后牛主要的感染虫种[1,4],而在中国的大多数调查发现安氏隐孢子虫是断奶后牛主要的感染虫种[19],本次调查发现牛隐孢子虫是该场断奶后牛感染的优势虫种,或与该引种场建场时间较短相关。
不同的隐孢子虫可导致牛表现出不同的临床症状,安氏隐孢子虫多感染青年牛和成年牛,虽不表现出明显的临床症状,但可引起泌乳牛产奶量下降[19,23]。牛隐孢子虫和芮氏隐孢子虫一般不会导致牛表现任何临床症状[17,21]。微小隐孢子虫则主要感染断奶前犊牛,可引起犊牛水样腹泻、脱水,甚至死亡[6]。犊牛腹泻是一个复杂的,多因素的疾病,与宿主对病原体抵抗力相关,微小隐孢子虫是最常见的犊牛腹泻病原之一[5]。先前国内有关微小隐孢子虫引起犊牛腹泻的报道较少,近来在中国西北地区某奶牛场暴发由微小隐孢子虫引起的隐孢子虫病,犊牛群体出现严重的水样腹泻并导致死亡,调查发现2~3周龄的断奶前犊牛感染率最高,达83.3%(40/48)[12]。在澳大利亚的一项调查显示,断奶前腹泻犊牛的隐孢子虫感染率为73.5%(144/196),其中以微小隐孢子虫为主要虫种,所占阳性比列为59.4%[24]。本次调查发现,断奶前犊牛多在7~25 d出现腹泻症状,且主要由微小隐孢子虫引起。因此,应加强对犊牛粪便的无害化处理,尤其是防止粪便污染水源。
牛源微小隐孢子虫可引起人体急性或慢性腹泻,导致免疫功能低下者水样腹泻甚至死亡,但并非所有牛源微小隐孢子虫均可人兽共患[7-8]。近年来,国内有关奶牛源微小隐孢子种类和基因亚型鉴定的报道较多,基于gp60基因位点,奶牛源微小隐孢子虫分离株的基因亚型均为IId亚型家族,存在人兽共患风险[9,12,17]。本研究中,35个微小隐孢子分离株的基因亚型均为IIdA19G1,与Wang等[9]报道的河南省牛源微小隐孢子分离株的基因亚型相一致。目前,在中国已有11个微小隐孢子基因亚型被鉴定,在奶牛体内仅见2个IId亚型家族感染,分别为IIdA15G1和IIdA19G1。而在澳大利亚,14个奶牛源微小隐孢子虫基因亚型则均属于IIa亚型家族,分别为II aA15G2R1、IIaA16G3R1、IIa-A17G2R1、IIaA18G2R1a、IIaA18G2R1b、IIaA18G3R1、IIaA18G4R1、II aA19G3R1a、IIaA19G3R1b、IIaA20G2R1、IIaA20G3R1、IIaA20G4R1、IIaA21G3R1和II aA23G3R1。微小隐孢子虫主要感染幼龄牛,本次调查的养殖场所引进种牛均为成年牛,一定程度上隔离了不同微小隐孢子虫基因亚型的扩散和传播。大量群体遗传学研究表明微小隐孢子虫的存在独特的地理遗传隔离特征[6-8],本研究结果进一步支持微小隐孢子虫的基因亚型与地理分布存在明显相关性。
国内有关微小隐孢子虫对动物致病性研究资料较少,本研究证实微小隐孢子虫是断奶前犊牛腹泻的主要病原之一,IId亚型为中国独特分布的微小隐孢子虫亚型,其基因亚型存在地理隔离遗传特征,引种青年牛和成年牛不影响当地重要人兽共患虫种微小隐孢子虫基因亚型分布,从这个角度考虑不具有生物安全重要性,为防控隐孢子虫病传播奠定了基础。
[1]Ryan U, Fayer R, Xiao L.Cryptosporidiumspecies in humans and animals: current understanding and research needs[J]. Parasitology, 2014, 141(13): 1667-1685. DOI: 10.1017/S0031182014001085
[2]Blackburn BG, Mazurek JM, Hlavsa M, et al. Cryptosporidiosis associated with ozonated apple cider[J]. Emerg Infect Dis, 2006, 12(4): 684-686.
[3]Baldursson S, Karanis P. Waterborne transmission of protozoan parasites: review of worldwide outbreaks-an update 2004-2010[J]. Water Res, 2011, 45(20): 6603-6614. DOI: 10.1016/j.watres.2011.10.013
[4]Xiao L. Molecular epidemiology of cryptosporidiosis: an update[J]. Exp Parasitol, 2010, 124(1): 80-89. DOI: 10.1016/j.exppara.2009.03.018
[5]Meganck V, Hoflack G, Piepers S, et al. Evaluation of a protocol to reduce the incidence of neonatal calf diarrhoea on dairy herds[J]. Prev Vet Med, 2015, 118(1): 64-70. DOI: 10.1016/j.prevetmed.2014.11.007
[6]Wang R, Zhang L, Axen C, et al.CryptosporidiumparvumIId family: clonal population and dispersal from Western Asia to other geographical regions[J]. Sci Rep, 2014, 4: 4208. DOI: 10.1038/srep04208
[7]De Waele V, Van den Broeck F, Huyse T, et al. Panmictic structure of theCryptosporidiumparvumpopulation in irish calves: influence of prevalence and host movement[J]. Appl Environ Microbiol, 2013, 79(8): 2534-2541. DOI: 10.1128/AEM.03613-12
[8]Widmer G, Sullivan S. Genomics and population biology ofCryptosporidiumspecies[J]. Parasite Immunol,2012,34(2/3): 61-71. DOI: 10.1111/j.1365-3024.2011.01301.x
[9]Wang R, Wang H, Sun Y, et al. Characteristics ofCryptosporidiumtransmission in preweaned dairy cattle in Henan, China[J]. J Clin Microbiol, 2011, 49(3): 1077-1082. DOI: 10.1128/JCM.02194-10
[10]Amer S, Zidan S, Adamu H, et al. Prevalence and characterization ofCryptosporidiumspp. in dairy cattle in Nile River delta provinces, Egypt[J]. Exp Parasitol, 2013, 135(3): 518-523. DOI: 10.1016/j.exppara.2013.09.002
[11]Santin M. Clinical and subclinical infections withCryptosporidiumin animals[J]. N Z Vet J, 2013, 61(1): 1-10. DOI: 10.1080/00480169.2012.731681
[12]Cui Z, Wang R, Huang J, et al. Cryptosporidiosis caused byCryptosporidiumparvumsubtype IIdA15G1 at a dairy farm in Northwestern China[J]. Parasit Vectors, 2014, 7(1): 529.
[13]Xiao L, Escalante H, Yang CF, et al. Phylogenetic analysis ofCryptosporidiumparasites based on the small-subunit rRNA gene locus[J]. Appl Environ Microbiol, 1999, 65(4): 1578-1583.
[14]Sulaiman IM, Hira PR, Zhou L, et al. Unique endemicity of cryptosporidiosis in children in Kuwait[J]. J Clin Microbiol, 2005, 43(6): 2805-2809.
[15]Qi MZ, Fang YQ, Wang XT, et al. Molecular characterization ofCryptosporidiumspp. in pre-weaned calves in Shaanxi Province, Northwestern China[J]. J Med Microbiol, 2015, 64(Pt1): 111-116. DOI: 10.1099/jmm.0.079327-0
[16]Huang J, Yue D, Qi M, et al. Prevalence and molecular characterization ofCryptosporidiumspp. andGiardiaduodenalisin dairy cattle in Ningxia, northwestern China[J]. BMC Vet Res, 2014, 10(1): 292.
[17]Zhang W, Wang R, Yang F, et al. Distribution and genetic characterizations ofCryptosporidiumspp. in pre-weaned dairy calves in Northeastern China’s Heilongjiang Province[J]. PLoS One, 2013, 8(1): e54857. DOI: 10.1371/journal.pone.0054857
[18]Liu A, Wang R, Li Y, et al. Prevalence and distribution ofCryptosporidiumspp. in dairy cattle in Heilongjiang Province, China[J]. Parasitol Res, 2009, 105(3): 797-802. DOI: 10.1007/s00436-009-1457-2
[19]Wang R, Ma G, Zhao J, et al.Cryptosporidiumandersoniis the predominant species in post-weaned and adult dairy cattle in China[J]. Parasitol Int, 2011, 60(1): 1-4. DOI: 10.1016/j.parint.2010.09.002
[20]Becher KA, Robertson ID, Fraser DM, et al. Molecular epidemiology ofGiardiaandCryptosporidiuminfections in dairy calves originating from three sources in Western Australia[J]. Vet Parasitol, 2004, 123(1/2): 1-9.
[21]Ng J, Yang R, McCarthy S, et al. Molecular characterization ofCryptosporidiumandGiardiain pre-weaned calves in Western Australia and New South Wales[J]. Vet Parasitol, 2011, 176(2/3): 145-150. DOI: 10.1016/j.vetpar.2010.10.056
[22]Santin M, Trout JM, Fayer R. A longitudinal study of cryptosporidiosis in dairy cattle from birth to 2 years of age[J]. Vet Parasitol, 2008, 155(1/2): 15-23. DOI: 10.1016/j.vetpar.2008.04.018
[23]Lu QB, Qiu SX, Ru BR, et al. Identification of 5 types of Cryptosporidiosis in dairy calves in some prefectures of Henan Province[J]. Chin Vet Sci, 2008, 38(3): 261-267. (in Chinese) 卢庆斌,仇书兴,茹宝瑞,等.河南省部分地区乳牛隐孢子虫病的流行病学调查[J].中国兽医科学,2008,38(3):261-267
[24]Ng JS, Eastwood K, Walker B, et al. Evidence ofCryptosporidiumtransmission between cattle and humans in northern New South Wales[J]. Exp Parasitol, 2012, 130(4): 437-441. DOI: 10.1016/j.exppara.2012.01.014
Species and subtype identification ofCryptosporidiumspp.from dairy cattle in an imported farm and its biosafety evaluation
WANG Chen-rong,QI Meng,LI Jun-qiang,ZHANG Zhen-jie,LIU Peng-fei,YU Fu-chang,CAO Jian-ke,NING Chang-shen,ZHANG Long-xian
(CollegeofAnimalScienceandVeterinaryMedicine,HenanAgriculturalUniversity/InternationalJointResearchLaboratoryforZoonoticDiseasesofHenanProvince,Zhengzhou450002,China)
We investigated the effect ofCryptosporidiumfrom imported cow in the local area and the distribution effect ofCryptosporidiumspecies and genotypes in the local area. Using polymerase chain reaction (PCR), we studied the infection ofCryptosporidiumfrom dairy cattle in an imported farm in Henan Province. The overall infection rate ofCryptosporidiumin dairy cattle was 17.8% (90/507) based on 18SRNA gene locus.Cryptosporidiumparvum,Cryptosporidiumbovis,CryptosporidiumryanaeandCryptosporidiumandersoniwere found. Significant differences were observed between the infection rate of sampling and age (P<0.01). The infection rate ofCryptosporidiumdecreased with age increasing in dairy cattle.Cryptosporidiumparvumwere the dominatingCryptosporidiumspp. in pre-weaning calves. We found a positively correlation between the infection ofCryptosporidiumand diarrhea of pre-weaning calves (P<0.01). Thirty-fiveCryptosporidiumparvumwere successfully amplified from 43 positive samples atgp60 gene locus, the subtype of the amplifiedCryptosporidiumparvumwere IIdA19G1, the subtype was different with IIa subtype which were found in imported country (Australia). This study confirmed thatCryptosporidiumparvumis a kind of diarrhea pathogens in dairy calves, the subtype ofCryptosporidiumparvumin China is only IId and the subtype ofCryptosporidiumparvumexist geographical isolation genetic characteristics. It does not have the importance of biological safety from the point that imported heifers and adults do not affect the distribution ofCryptosporidiumspecies and genotypes in the local area.
Cryptosporidium; species; subtype; identification; dairy cattle Support by the Key Program of the National Natural Science Foundation of China (No. 31330079), the Innovation Scientists and Technicians Troop Construction Projects of Henan Province (No. 134200510012), and the Program for Science and Technology Innovative Research Team at the University of Henan Province (No. 012IRTSTHN005)
Zhang Long-xian, Email: zhanglx8999@henau.edu.cn
国家自然科学基金重点项目(No.31330079), 河南省科技创新人才计划(No.134200510012), 河南省高校科研创新团队项目(No. 012IRTSTHN005)联合资助
张龙现,Email: zhanglx8999@henau.edu.cn
河南农业大学牧医工程学院,河南省人兽共患病国际联合实验室,郑州 450002
10.3969/j.issn.1002-2694.2015.11.004
S855.9
A
1002-2694(2015)11-1005-05
2015-04-13;
2015-07-21

