大豆耐低磷相关基因的定位与克隆
张丹,宋海娜,程浩,喻德跃
大豆耐低磷相关基因的定位与克隆
张丹1,宋海娜2,程浩3,喻德跃3
1. 河南农业大学农学院,河南省粮食作物协同创新中心,郑州 450002;2. 平顶山学院低山丘陵区生态修复重点实验室,平顶山 467000;3. 南京农业大学国家大豆改良中心,作物遗传与种质创新国家重点实验室, 南京 210095
大豆是食用油和植物蛋白的主要来源,土壤有效磷含量低是限制当前大豆生产的重要因素之一。鉴定优异耐低磷种质资源、快速发掘利用优异耐低磷基因、通过分子育种培育磷高效品种、改善大豆应对低磷胁迫的能力,是解决土壤有效磷含量低这一问题的有效途径。近年来,国内外开展了一些大豆磷效率相关基因的定位与克隆研究,但由于QTL连锁作图的精度较低,难以准确地分离候选基因,而大豆基因组的复杂性及相关功能基因分子机制的不明确阻碍了人们对大豆耐低磷特性本质的认识。文章综述了大豆耐低磷相关基因的定位、克隆及功能验证等方面的研究进展,分析了大豆耐低磷相关基因研究中存在的问题,以期为快速有效地分离目的基因、验证基因功能、解析大豆磷高效分子机制提供参考。
大豆;耐低磷;基因定位;分子育种
大面积土壤缺磷是目前限制我国农作物产量提高和品质改良的主要因素之一。据统计,我国耕地约有1亿hm2,缺磷(有效磷≤10 mg/kg)的耕地面积约占81.5%,其中严重缺磷(有效磷≤5 mg/kg)的耕地占50.5%左右[1]。实际上土壤中的总磷含量并不低,只是能被植物直接吸收利用的有效磷含量很低,因此作物仍表现缺磷,这就是特有的“遗传学缺磷”现象[2, 3]。为保证作物获得或维持较高产量,只能不断施用磷肥[4]。近来,我国每年在土壤中施用1100多万吨磷肥,约占全球磷肥消费总量的35%。而当季磷肥利用效率仅为10%~25%,这意味着每年近1000万吨磷素累积于土壤[5]。另外,过量施磷会造成土壤中有害元素的积累、环境污染增加[6]。更为严重的是,磷矿是不可再生资源,研究表明全球磷素资源有可能在未来30~100年间枯竭[7, 8]。由此将导致磷肥市场供不应求,进一步危及全球粮食安全。有研究表明,未来世界面临的主要危机不是水危机,也不是石油危机,而是磷危机[7]。我国易开采的富磷矿储量只占总储量的8.4%,仅能维持我国10~15年时间的使用需求,磷素危机已经对我国粮食生产构成了严重威胁。因此,面对土壤磷素利用率低及磷资源枯竭的危机,发掘作物自身潜力和高效利用土壤潜在的磷资源是解决供磷与磷流失矛盾和防止磷污染的有效途径。
大豆[(L). Merr.]是世界上最重要的粮油兼用作物,是植物蛋白、食用油和动物饲料的主要来源。作为需磷量较大的农作物,大豆籽粒含磷量远远高于水稻、小麦和玉米[9]。由于大豆的根瘤菌具有固氮作用,磷素已成为大豆生长及产量最重要的限制性元素[10]。磷的缺乏不仅影响大豆的生长发育,增加花荚脱落,还会抑制根瘤的形成,降低固氮效率,最终严重影响其产量和品质[9]。因此,低磷胁迫已经成为当前我国大豆生产的重要限制因素。尽管大豆对低磷胁迫的耐性受数量性状基因控制,是环境和遗传因素共同作用的结果,但已有研究表明,不同大豆品种对低磷胁迫和磷肥效应有显著的遗传差异[11~13]。磷效率本身的遗传特性是不同大豆品种间耐低磷能力存在差异的主要原因,而耐低磷能力的强弱是影响大豆磷效率及产量的关键因素,因此挖掘与大豆耐低磷能力相关的关键基因,通过分子标记辅助选择等手段选育磷高效的优良品种,对提高大豆产量及高效利用土壤难溶态磷具有重要意义。
本文综述了近年来大豆耐低磷相关基因的定位、克隆及功能验证等方面的研究进展,介绍了目前大豆中已克隆的磷效率相关基因及其克隆方法,比较各种方法的优缺点和应用特点,并对这些候选基因的功能及其潜在育种利用价值进行了归纳。同时,分析了当前研究存在的主要问题,期望能为快速准确地选择合适的目标基因及大豆磷高效分子机制的解析研究提供参考。
1 大豆耐低磷相关基因的QTL定位
大豆磷效率是受多基因控制的复杂数量性状,基于家系的连锁分析是研究复杂数量性状遗传基础的经典方法。近年来,国内外通过连锁分析对大豆磷效率相关性状进行了广泛的QTL定位研究(表1)。2005年Li等[14]首先利用科丰1号与南农1138-2杂交衍生的116个重组自交系在F连锁群上检测到7个与耐低磷相关QTL;同样,耿雷跃等[15]利用该群体的184个家系在高磷和低磷水平下分别检测到3个和12个加性的QTL,可解释4.0%~13.8%的表型变异;另外在两种磷水平下还检测到19对互作QTL,可解释3.3%~19.9%的表型变异。2007年,崔世友等[16]利用耐低磷品种南农94-156和低磷敏感品种波高衍生的152个重组自交系通过盆栽试验对大豆耐低磷相关性状进行QTL分析,结果检测到7个QTL,对表型变异的解释率为4.8%~17.0%。Zhang等[17]利用同一群体进一步在低磷和正常供磷条件下调查大豆苗期5个磷效率相关性状,定位到34个加性QTL与耐低磷性状相关。这些结果有助于人们初步认识大豆耐低磷的遗传基础,为分子标记辅助育种提供依据。
上述研究中所采用的筛选指标主要涉及生物量、磷含量、根相关性状等,且均集中在苗期进行。虽然大豆在苗期对磷缺乏较为敏感,但是笔者前期实验中发现,大豆对低磷胁迫的另一个敏感期是开花结荚期,此时是大豆对磷素吸收的高峰时期,约占整个时期吸磷量的90%,尤其是结荚鼓粒期吸磷量约占一半以上,此时缺磷显著减少花和荚的形成,增加花荚的脱落,原因是磷是大豆花和荚的形成所必须的营养元素[18]。因此,在花荚期选择耐低磷相关性状来评价大豆的耐低磷特性及进行耐低磷QTL的定位研究更具合理性和代表性。2010年,Zhang等[19]利用南农94-156和波高衍生的重组自交系群体尝试在花荚期利用花荚脱落率、酸性磷酸酶活性和荚叶磷含量来评价大豆耐低磷性,取得良好效果,除了利用条件定位法在该时期检测到耐低磷QTL的净效应,发现该位点对表型的贡献达19.3%,还通过多年多点试验利用相对酶活性和荚叶磷含量定位到一个磷效率主效QTL,并在后期图位克隆了控制该位点的基因[20](表1)。2010年,Liang等[21]在花期利用磷效率敏感基因型BD2和磷高效基因型BX10衍生的重组自交系对磷效率相关性状进行QTL分析,结果在 5个连锁群上检测到31个QTL位点,可解释9.1%~31.1%的表型变异。此外,2013年,King等[22]利用92个F2:4家系定位大豆籽粒磷含量相关的QTL,共定位到3个QTL分别位于12、7和17号染色体上,其中两个位点包括磷酸盐转运相关基因,一个位点包含一个ZIP转运蛋白,为进一步研究磷酸盐代谢途径奠定了基础。
基于大豆耐低磷相关QTL定位结果,可以看出大豆磷效率除了受少数主效QTL作用外,还受大量微效多基因控制,而这些基因除了表现为加性效应,还具有一定上位性作用。如位于大豆D1b+W连锁群上Satt274标记附近的QTL同时存在着加性效应和上位性效应,且在该位点还检测到耐低磷QTL的净遗传效应,对表型变异的解释率也很大[19]。这些结果不仅说明大豆磷效率的遗传基础比较复杂,还表明磷效率存在主效QTL,为精细定位及图位克隆主效QTL提供了理论依据。由于目前定位的磷效率QTL均是基于分离群体的家系连锁分析所得,而分离群体又受杂交或自交次数的限制,缺乏足够的重组事件,置信区间一般较大,导致QTL作图的精度一般较低,因此被精细定位、图位克隆的QTL非常少。
2 大豆耐低磷相关基因的克隆与功能分析
大豆是古四倍体自花授粉作物,基因组复杂且重复序列多。一些重要的功能基因,尤其是控制复杂数量性状的基因,其克隆存在较大困难。目前,已克隆的大豆耐低磷相关基因主要是通过同源克隆和电子克隆获得,也有少数基因是通过图位克隆、芯片技术、抑制差减杂交及功能克隆等方法获得。
2.1 图位克隆获得的大豆耐低磷相关基因
图位克隆法(Map-based cloning)是分离和克隆植物基因的有效方法[24],也是常用的基因克隆技术之一。近年来,尽管植物磷效率相关的QTL定位研究发展较快,但是通过图位克隆法分离得到的磷效率相关基因却很少。2012年,Gamuyao等[25]在水稻中通过经典的图位克隆法分离得到一个耐低磷相关基因(),该基因编码一个蛋白激酶,能够显著增强水稻早期根系的生长,从而获得更多的磷及其他营养元素,并最终增加产量。2014年,Zhang等[19]在连锁分析与关联分析相结合方法的基础上,在大豆中图位克隆了一个磷效率相关基因,该基因编码一个酸性磷酸酶,其分离克隆过程不同于在水稻中的经典图位克隆方法。首先利用全基因组关联分析对前期连锁分析定位一个主效耐低磷QTL()进行验证,然后在该区域开发高密度的分子标记,通过关联分析局部扫描将目标QTL限定在250 kb的范围内,并结合大豆基因组信息、生物信息学及诱导表达分析等方法分离并克隆了控制该位点的候选基因()。进一步通过诱导表达分析、原核表达、遗传转化等实验证明在低磷条件下表达量显著升高,导致酸性磷酸酶活性升高从而提高大豆耐低磷能力。通过候选基因关联分析鉴定与大豆耐低磷相关的有利单倍型,开发了一个功能分子标记[19],为大豆磷高效分子育种提供新的基因信息和技术支持。
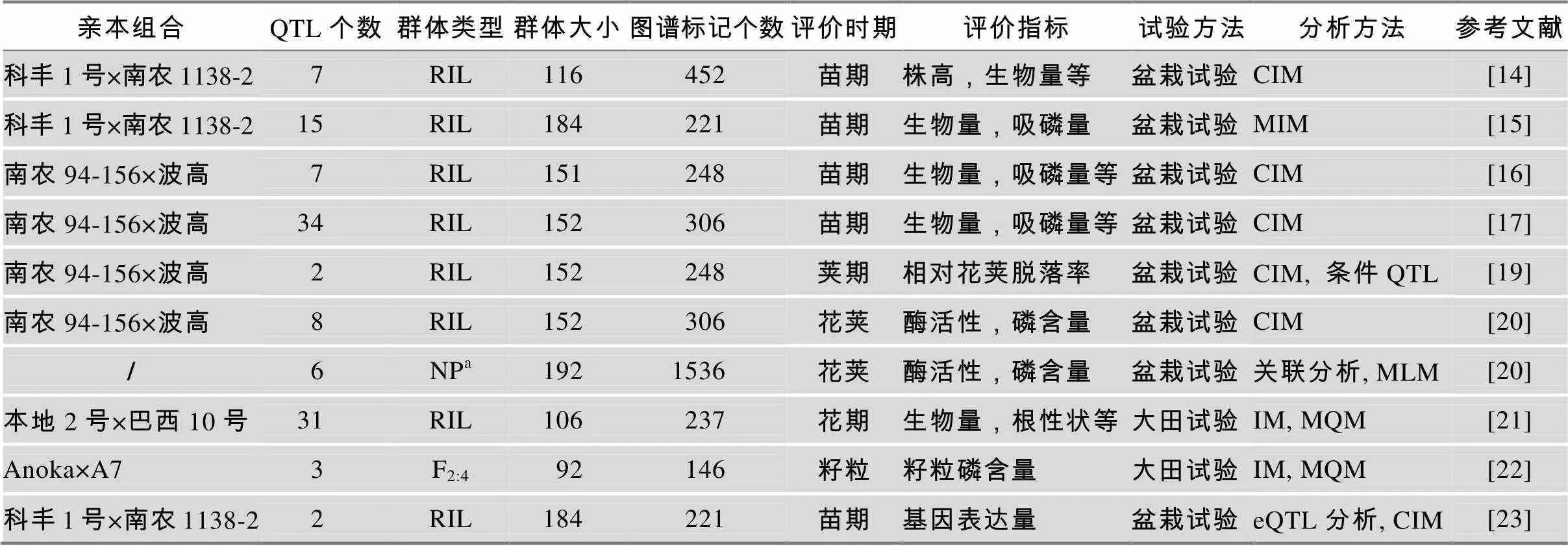
表1 已定位的大豆磷效率相关QTL
注:aNP表示自然群体。
2.2 同源克隆与电子克隆获得的大豆耐低磷相关基因
同源克隆法(Homology-based cloning)是大豆基因克隆中常用的一种方法。不同物种具有相同或相似功能的基因及相对保守的区域,根据保守结构域设计引物进行PCR扩增得到目的基因片段,然后利用cDNA末端快速扩增技术(Rapid amplification of cDNA end, RACE)和染色体步移技术分别得到全长cDNA和全长基因[26]。这种方法相对于其他方法比较简单,尤其是随着对拟南芥、水稻等模式植物的深入研究,生物信息学迅猛发展,公共数据库及辅助设计软件日趋完善,不同物种具有相同或相似功能基因之间的基因组比较分析成为可能。因此,同源克隆也是目前大豆磷效率相关基因克隆中应用最多的方法。近年来,国内外研究者利用该方法克隆到的大豆磷效率相关基因见表2。如2009年Wang等[3]通过同源克隆的方法将拟南芥紫色磷酸酶基因转入大豆,在酸性土壤中生长的3个转基因株系磷效率及产量相关性状均明显优于野生型,其中磷效率提高了117.8%、56.5%、57.8%,单株荚数提高35.9%、41.0%、59.0%,单株粒数也提高46.0%、48.3%、66.7%。2013年,Liang等[34]通过拟南芥基因的保守区域设计引物在大豆中扩增得到一个苹果酸转运基因(),结果表明苹果酸分泌是大豆在酸性土壤中的重要适应机制,土壤中pH值、铝和磷三因素共同作用,影响苹果酸转运基因的表达量进而调节其功能,并最终影响苹果酸的分泌提高大豆对酸性土壤的适应性。2014年,Kong等[35]通过搜索比对拟南芥数据库克隆了一个大豆紫色酸性磷酸酶基因,该基因具有植酸酶活性,在植酸作为唯一磷源时,该基因的过量表达能提高拟南芥对有机磷的利用能力。另外,笔者所在实验室也利用同源克隆方法克隆到一个耐低磷相关基因,该基因编码一个类受体蛋白激酶,初步验证该基因能够提高大豆在低磷环境中的耐低磷能力(未发表)。
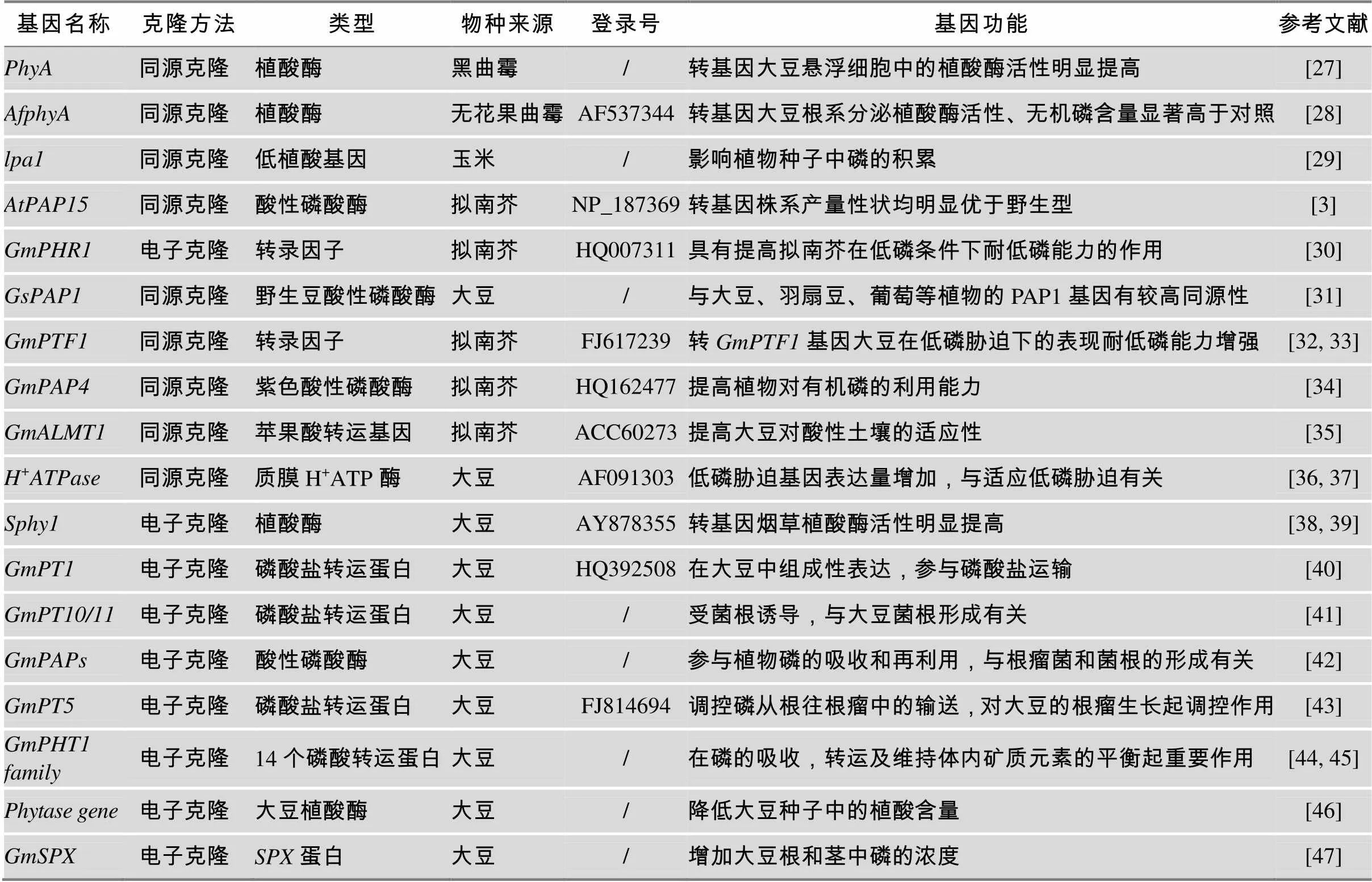
表2 利用同源克隆与电子克隆法获得的大豆磷效率相关基因
电子克隆法是基于表达序列标签(Expressed sequence tag, EST)和基因组数据库发展起来的基因克隆新型技术,利用生物信息学知识和计算机技术对EST 或基因组数据库中进行同源性比较分析、整理拼接出新基因的编码序列,确认完整后根据序列设计引物进行RT-PCR验证获得全长基因[48]。随着大豆EST数据库中的基因片段不断增加及大豆全基因组测序的完成,该方法在大豆磷效率相关基因克隆中应用也较多(表2)。2012年,Qin等[43]以拟南芥与水稻中磷酸转运蛋白基因基因为查询序列搜索大豆基因组网站序列(http://www.phytozome.net/soybean.php),克隆了一个高亲和力的磷酸转运蛋白基因,该基因调控磷从根向根瘤中的输送,对大豆的根瘤及植株生长起着重要的调控作用。2013年,Fan等[45]基于大豆基因组数据库(Phytozome V8.0)获得14个基因家族的序列,通过RT-PCR电子克隆了这些基因,进化分析发现这些基因起源于4个不同的祖先,实验表明基因家族各成员在维持细胞乃至整个植株体内磷的平衡具有协同作用。2014年,Yao等[47]通过搜索大豆基因组数据库获得了9个成员,系统发育分析将这9个成员分为3组,每个成员对氮、磷、钾和铁的缺乏有不同的响应,其中在高磷处理条件下,过表达基因能够增加植株根和茎中磷的浓度,同时在大豆毛状根中,能导致其他7个磷饥饿相关基因的上调表达。这些结果为大豆耐低磷胁迫和磷高效利用的功能基因组和分子设计育种打下了基础。
尽管同源克隆法在发掘磷效率相关基因时相对于传统方法具有一定的优越性。但是通过这种方法获得真正有功能的磷效率基因仍存在一定的风险。比如克隆得到的同源序列可能与目的磷效率基因无关;也可能只是与目的基因紧密连锁等。因此,应用这种方法要注意一些问题。首先,在设计引物时应尽可能考虑到出现的问题和目的基因的类别,如进行PCR检验时所设计引物是否会扩增出非目的片段。其次,磷效率基因在植物中多成簇或以基因家族的形式存在,难以判断克隆产物是否为目的基因片段。第三,一些新的磷效率相关基因可能不具有保守域结构,而是新的编码蛋白,用此方法不能得到目的基因。对于电子克隆而言,尽管这种方法在理论上已没有什么障碍,且具有效率高、成本低、对实验条件要求低等特点,但是由于生物大分子结构和功能的复杂性,许多分析软件的输出结果存在较大的偏差,因此利用生物信息学进行的“虚拟”克隆的结果尚需回到实验室进行验证。
2.3 抑制性差减杂交与基因芯片等方法获得的大豆磷效率相关基因
抑制性差减杂交(Suppression subtractive hybridization , SSH)是用于分离在特定组织、特定发育阶段或特定环境下表达的基因的方法。以其假阳性低、敏感性高、速度快、效率高等优点广泛应用于基因转录水平上的差异表达。SSH技术不需要目的基因的序列信息和位置信息,已在植物基因克隆中得到了广泛应用。Guo等[49]和Zhou等[50]从低磷诱导SSH文库中筛选得到一个β-扩张蛋白基因,该基因在低磷诱导的大豆根系中高效表达,且参与根尖的形态建成,超表达转基因拟南芥的根系细胞分裂与伸长明显增加,从而使得转基因拟南芥的生长与磷素吸收也得到明显改善。转大豆缺磷处理使基因在根系和下胚轴中的表达量增加;缺磷条件下,基因通过参与植株根毛的伸长过程,从而影响植株的生长和磷吸收量。SSH技术也有局限性,只能用于两个样本的比较,只能筛选在对照样本中不表达的基因,不能反应表达量的差别。
基因芯片技术是建立在基因探针和杂交测序技术上的一种高效快速的核酸序列分析手段。2014年,Song等[23]根据基因芯片结果,发现一个在叶片大量表达的磷酸转运蛋白基因(),并对该基因的表达模式及功能进行了研究。结果发现基因在老叶中具有较高的表达量,其中在低磷胁迫处理15 d的大豆幼苗成熟叶、幼叶和侧根中表达量极显著增强,表明该基因可能参与地上部磷酸盐的活化再转移过程。进一步在烟草中过表达该基因,发现转基因植株在低磷胁迫条件下显著提高了植株的总干重、磷的利用效率和籽粒重量。虽然芯片技术本身也具有局限(如假阳性高、得到的cDNA片段也比较短),但它是一种可以在大豆基因克隆中广泛应用且有效的方法。
2.4 其他方法获得的大豆磷效率相关基因
除了以上主要方法外,近年来在大豆磷效率相关基因克隆和功能研究中还利用了一些其他的基因克隆方法如功能克隆方法,并且取得了一些研究进展。
传统的功能克隆方法属于正向遗传学的范畴,是根据已知基因表达的蛋白质产物反推其核苷酸序列,再根据此序列设计探针或引物从cDNA文库或者基因组文库中筛选克隆,也可以制备产量抗体,从表达载体构建的cDNA文库中筛选相应的克隆,进而分离目的基因。如2001年Hegeman等[51]利用功能克隆方法从萌发的大豆子叶中得到了大豆中的一个植酸酶基因,分析发现该基因编码的氨基酸序列与其他植物中已知的紫色酸性磷酸酶基因序列的同源性较高,而与已报道的玉米或者微生物来源的植酸酶基因蛋白序列同源性较低;能够在大豆幼苗子叶中特异表达,该基因转入大豆悬浮细胞后,获得的转基因细胞系具有较高的植酸酶活性。表明该基因可分解贮存在植株子叶中的植酸磷,为幼苗后期正常生长提供磷源,转基因大豆细胞的植酸酶活性明显提高。这种方法的局限性是只适用于克隆那些蛋白质产物及其相应功能都已清楚的基因,无法克隆蛋白质产物还不清楚的基因。
随着测序技术的发展,表达谱测序技术成为功能基因发掘的重要手段。2013年,Xu等[52]利用小RNA和降解组测序技术对磷充分和磷缺乏条件下获得的大豆叶片和根部的4个小RNA文库和磷缺乏大豆根部的一个降解组文库进行测序。结果在低磷胁迫条件下,检测到25个miRNAs被诱导和11个miRNAs受抑制。其中miR399受低磷胁迫诱导上调,作用于靶基因和,作用位点在的5¢非编码区和的编码区。研究结果为阐明miRNAs在磷信号传导中发挥的作用以及磷代谢调控网络的构建提供了有用的信息。
3 大豆磷效率基因研究的问题与展望
目前我国大豆生产形势十分严峻,需求缺口及进口量逐年增大,危及国家的粮食安全。仅靠土壤增施磷肥求高产的传统途径已不适应当今农业可持续发展的要求。发掘磷高效基因,提高大豆自身活化吸收土壤无效态磷的能力是生态农业发展的一个重要方向。采用常规育种手段与现代转基因技术相结合的方法来实现磷高效基因的转育,具有速度快、育种进程短等优点,因而越来越受到重视。然而,要实现转基因育种与常规育种的有效结合,分离和发掘新的、有功能的磷效率关键基因无疑是开展上述研究的重要前提。近10年来,大量磷效率相关QTL的定位为大豆磷高效分子育种奠定了良好的基础,然而相对于模式植物来说,大豆磷效率相关基因的图位克隆及育种应用却进展缓慢,主要有以下几个方面的原因:(1)缺少统一的磷效率评价指标和筛选时期,难以准确鉴定磷效率特异基因型;(2)定位研究所用的材料及群体遗传基础相对狭窄,难以找到可供育种利用的最有利等位基因;(3)QTL在染色体上的范围较大,且基因组庞大,构建高密度的分子标记图谱困难,QTL精细定位及图位克隆难以实现;(4)大豆遗传转化率相对较低,导致基因的功能验证比较困难;(5)大多试验在温室和实验室内进行,缺少大田试验的验证,很少考虑表型与环境的互作,应用于生产仍有一定距离。
因此,为克服上述问题,加速大豆磷高效育种进程,需要快速准确地获得磷高效主效基因,充分了解磷效率关键基因的功能和分子机制,明确其作用机理,筛选出可用于育种实践的磷高效功能基因,加快这些候选基因的转化应用及育种进程。首先,应在大豆苗期磷效率QTL连锁分析的基础上,对田间大豆花荚期耐低磷性开展更为广泛的定位研究,鉴定QTL的稳定性和与环境的互作效应;其次,要拓宽种质资源遗传基础,如加入野生豆和地方性种质资源等,利用基于自然群体进行的全基因组关联分析或连锁分析与关联分析相互结合,快速实现主效QTL的精细定位与图位克隆,发掘蕴藏于种质资源基因组中最有利的耐低磷等位基因;最后,基于对所克隆基因功能的认识,采用分子标记辅助选择将优良等位基因聚合到优良遗传背景中,通过全基因组设计创造出集中众多优良基因、性状得到明显改良的新种质或新品种。将来会有多种类型的磷高效基因应用于生产,打破土壤中磷的“遗传学缺乏”,低磷胁迫对大豆乃至其他作物生产的影响有望得到有效解决。
[1] 曾宪坤. 磷的农业化学 (Ⅱ). 磷肥与复肥, 1999, 14(2): 63–69.
[2] 尹逊霄, 华珞, 张振贤, 滑丽萍, 高娟. 土壤中磷素的有效性及其循环转化机制研究. 首都师范大学学报(自然科学版), 2005, 26(3): 95–101.
[3] Wang XR, Wang YX, Tian J, Lim BL, Yan XL, Liao H. Overexpressingenhances phosphorus efficiency in soybean., 2009, 151(1): 233–240.
[4] Yan XL, Wu P, Ling HQ, Xu GH, Xu FS, Zhang QF. Plant nutriomics in China: an overview., 2006, 98(3): 473–482.
[5] 方陵生. 磷肥过度使用之警示. 世界科学, 2010, (11): 6, 5.
[6] Behrendt H, Boekhold A. Phosphorus saturation in soils and ground waters., 1993, 4(4): 233–243.
[7] Oelkers EH, Valsami–Jones E. Phosphate Mineral Reactivity and Global Sustainability., 2008, 4(2): 83–87.
[8] Mihelcic JR, Fry LM, Shaw R. Global potential of phosphorus recovery from human urine and feces., 2011, 84(6): 832–839.
[9] 李喜焕, 常文锁, 张彩英. 中国大豆磷素营养及磷高效品种筛选最新进展. 大豆科学, 2011, 30(2): 322–327.
[10] 任海红, 刘学义, 李贵全. 大豆耐低磷胁迫研究进展. 分子植物育种, 2008, 6(2): 316–322.
[11] 丁洪, 李生秀, 郭庆元, 张学江, 徐巧珍. 酸性磷酸酶活性与大豆耐低磷能力的相关研究. 植物营养与肥料学报, 1997, 3(2): 123–128.
[12] 徐青萍, 罗超云, 廖红, 严小龙, 年海. 大豆不同品种对磷胁迫反应的研究. 大豆科学, 2003, 22(2): 108–114.
[13] Wang LD, Liao H, Yan XL, Zhuang BC, Dong YS. Genetic variability for root hair traits as related to phosphorus status in soybean., 2004, 261(1–2): 77–84.
[14] Li YD, Wang YJ, Tong YP, Gao JG, Zhang JS, Chen SY. QTL mapping of phosphorus deficiency tolerance in soybean (L. Merr.)., 2005, 142(1–2): 137–142.
[15] 耿雷跃, 崔士友, 张丹, 邢邯, 盖钧镒, 喻德跃. 大豆磷效率QTL定位及互作分析. 大豆科学, 2007, 26(4): 460–466.
[16] 崔世友, 耿雷跃, 孟庆长, 喻德跃. 大豆苗期耐低磷性及其QTL定位. 作物学报, 2007, 33(3): 378–383.
[17] Zhang D, Cheng H, Geng LY, Kan GZ, Cui SY, Meng QC, Gai JY, Yu DY. Detection of quantitative trait loci for phosphorus deficiency tolerance at soybean seedling stage., 2009, 167(3): 313–322.
[18] Mengel K, Kirkby EA,Kosegarten H, Appel T. Principles of Plant Nutrition. International Potash. Institute, Berne, Switzerland: Kluwer Academic Publishers, 2001.
[19] Zhang D, Liu C, Cheng H, Kan G, Cui S, Meng Q, Gai J, Yu D. Quantitative trait loci associated with soybean tolerance to low phosphorus stress based on flower and pod abscission., 2010, 129(3): 243–249.
[20] Zhang D, Song H, Cheng H, Hao D, Wang H, Kan G, Jin H, Yu D. The Acid Phosphatase-Encoding GeneContributes to Soybean Tolerance to Low-Phosphorus Stress., 2014, 10(1): e1004061.
[21] Liang Q, Cheng XH, Mei MT, Yan XL, Liao H. QTL analysis of root traits as related to phosphorus efficiency in soybean., 2010, 106(1): 223–234.
[22] King KE, Lauter N, Lin SF, Scott MP, Shoemaker RC. Evaluation and QTL mapping of phosphorus concentration in soybean seed., 2013, 189(2): 261–269.
[23] Song HN, Yin ZT, Chao MN, Ning LH, Zhang D, Yu DY. Functional properties and expression quantitative trait loci for phosphate transporterin soybean.,, 2014, 37(2): 462–472.
[24] Peters JL, Cnudde F, Gerats T. Forward genetics and map-based cloning approaches., 2003, 8(10): 484–491.
[25] Gamuyao R, Chin J H, Pariasca-Tanaka J,Pesaresi P, Catausan S, Dalid C, Slamet-Loedin I, Tecson-Mendoza EM, Wissuwa M, Heuer S. The protein kinasefrom traditional rice confers tolerance of phosphorus deficiency., 2012, 488(7412): 535–539.
[26] 李子银, 陈受宜. 水稻抗病基因同源序列的克隆、定位及其表达. 科学通报, 1999, 44(7): 727–733.
[27] Li J, Hegeman CE, Hanlon RW, Lacy GH, Denbow DM, Grabau EA. Secretion of active recombinant phytase from soybean cell-suspension cultures., 1997, 114(3): 1103–1111.
[28] Li GL, Yang SH, Li MG, Qiao YK, Wang JH. Functional analysis of an Aspergillus ficuum phytase gene inand its root-specific, secretory expression in transgenic soybean plants., 2009, 31(8): 1297–1303.
[29] Gillman JD, Pantalone VR, Bilyeu K. The low phytic acid phenotype in soybean line CX1834 is due to mutations in two homologs of the maize low phytic acid gene., 2009, 2(2): 179–190.
[30] Wu B, Li XH, Liu C, Wang YJ, Li WL, Chang WS, Zhang CY. Genetic transformation and function analysis of transcription factorin soybean., 2013, 32(3): 302–305.
[31] 王军卫, 侯立江, 李登, 程春明, 王瑞珍, 赵现伟, 赵朝森. 野生大豆紫色酸性磷酸酶基因的克隆及分析. 大豆科学, 2013, 32(5): 596–600.
[32] Li XH, Wu B, Kong YB, Zhang CY., a novel transcription factor gene, is involved in conferring soybean tolerance to phosphate starvation., 2014, 13(1): 926–937.
[33] 钟磊, 乔亚科, 乔潇, 李桂兰, 王林红, 刘晨光. 转基因大豆在低磷胁迫下的表现. 核农学报, 2013, 27(7): 1041–1047.
[34] Liang CY, Piñeros MA, Tian J, Yao ZF, Sun LL, Liu JP, Shaff J, Coluccio A, Kochian LV, Liao H. Low pH, aluminum, and phosphorus coordinately regulate malate exudation throughto improve soybean adaptation to acid soils., 2013, 161(3): 1347–1361.
[35] Kong YB, Li XH, Ma J, Li WL, Yan GJ, Zhang CY., a novel purple acid phosphatase gene isolated from soybean (), enhanced extracellular phytate utilization in., 2014, 33(4): 655–667.
[36] Shen H, Chen JH, Wang ZY, Yang CY, Sasaki T, Yamamoto Y, Matsumoto H, Yan XL. Root plasma membrane H-ATPaseis involved in the adaptation of soybean to phosphorus starvation., 2006, 57(6): 1353–1362.
[37] Shen H, He LF, Sasaki T, Yamamoto Y, Zheng SJ, Ligaba A, Yan XL, Ahn SJ, Yamaguchi M, Sasakawa H. Citrate secretion coupled with the modulation of soybean root tip under aluminum stress. Up-regulation of transcription, translation, and threonine-oriented phosphorylation of plasma membraneH-ATPase., 2005, 138(1): 287–296.
[38] 郭丽. 大豆苗期磷代谢生理参数和大豆新型植酸酶基因 ()克隆[学位论文].保定: 河北农业大学, 2008.
[39] 郭丽, 赵玉新, 张树华, 张海娜, 肖凯. 超表达大豆植酸酶基因对转基因烟草植株利用有机态磷能力的影响. 中国科技论文在线, 2009.
[40] Wu ZY, Zhao JM, Gao RF, Hu GJ, Gai JY, Xu GH, Xing H. Molecular cloning, characterization and expression analysis of two members of thefamily of phosphate transporters in., 2011, 6(6): e19752.
[41] Tamura Y, Kobae Y, Mizuno T, Hata S. Identification and expression analysis of arbuscular mycorrhiza-inducible phosphate transporter genes of soybean.,, 2011, 76(2): 309–313.
[42] Li CC, Gui SH, Yang T, Walk T, Wang XR, Liao H. Identification of soybean purple acid phosphatase genes and their expression responses to phosphorus availability and symbiosis.,2012, 109(1): 275–285.
[43] Qin L, Zhao J, Tian J, Chen LY, Sun ZA, Guo YX, Lu X, Gu M, Xu GH, Liao H. The high-affinity phosphate transporterregulates phosphate transport to nodules and nodulation in soybean., 2012, 159(4): 1634–1643.
[44] Qin L, Guo YX, Chen LY, Liang RK, Gu M, Xu GH, Zhao J, Walk T, Liao H. Functional characterization of 14family genes in yeast and their expressions in response to nutrient starvation in soybean., 2012, 7(10): e47726.
[45] Fan CM, Wang X, Hu RB, Wang YH, Xiao CW, Jiang Y, Zhang XM, Zheng CY, Fu YF. The pattern ofgenes evolutionary divergence inL., 2013, 13(1): 48.
[46] Singh P, Punjabi M, Jolly M, Rai RD, Sachdev A. Characterization and expression of codon optimized soybean phytase gene in., 2013, 50(6): 537–547.
[47] Yao ZF, Tian J, Liao H. Comparative characterization ofmembers reveals thatGmSPXis involved in phosphate homeostasis in soybean., 2014, 114 (3): 477–488.
[48] 王冬冬, 朱延明, 李勇, 李杰, 柏锡. 电子克隆技术及其在植物基因工程中的应用. 东北农业大学学报, 2006, 37(3): 403–408.
[49] Guo WB, Zhao J, Li XX, Qin L, Yan XL, Liao H. A soybean β-expansin geneintrinsically involved in root system architecture responses to abiotic stresses., 2011, 66(3): 541–552.
[50] Zhou J, Xie JN, Liao H, Wang XR. Overexpression of β-expansin geneimproves phosphorus efficiency in soybean., 2014, 150(2): 194–204.
[51] Hegeman CE, Grabau EA. A novel phytase with sequence similarity to purple acid phosphatases is expressed in cotyledons of germinating soybean seedlings., 2001, 126(4): 1598–1608.
[52] Xu F, Liu Q, Chen LY, Kuang JB, Walk T, Wang JX, Liao H. Genome-wide identification of soybean microRNAs and their targets reveals their organ-specificity and responses to phosphate starvation., 2013, 14(1): 66.
(责任编委: 吴为人)
Mapping and cloning of low phosphorus tolerance genes in soybeans
Dan Zhang1, Haina Song2, Hao Cheng3, Deyue Yu3
Soybean is a major source of edible oil and phytoprotein. Low phosphorus available in soil is an important factor limiting the current soybean production. Effective ways to solve the problem include identification of germplasms and genes tolerant to low-phosphorus stress, and cultivation of soybean varieties with high phosphorus efficiency. Recently many researches have been carrying out investigations to map and clone genes related to phosphorus efficiency in soybeans. However, due to the complexity of the soybean genome and little knowledge of functional genes, it has been difficult to understand the mechanism of soybean tolerance to low phosphorus. Although quantitative trait locus (QTL) mapping related to low phosphorus tolerance has made some progress, it remains elusive to obtain accurate candidate genes for molecular breeding applications, due to the limited accuracy of QTL. Even for the cloned soybean low phosphorus tolerance genes, the molecular mechanisms are largely unknown, further limiting the application to breeding. In this review, we summarize the progresses on mapping, cloning and functional characterization of soybean low phosphorus tolerance genes.
soybean; low phosphorus tolerance; gene mapping; molecular breeding
2014-07-18;
2014-09-17
国家自然科学基金项目(编号:31301336, 31370034)资助
张丹,博士,副教授,研究方向:植物分子遗传与育种。E-mail: zhangdan8006@163.com
喻德跃,博士,教授,研究方向:植物分子遗传与育种。E-mail: dyyu@njau.edu.cn
10.16288/j.yczz.14-244
2014-12-24 9:50:27
http://www.cnki.net/kcms/detail/11.1913.R.20141224.0950.001.html

