土壤生物与土壤污染研究前沿与展望
陈保冬, 赵方杰, 张 莘,伍松林,乔 敏,欧阳纬莹,朱永官,,*
1 中国科学院生态环境研究中心城市与区域生态国家重点实验室,北京 100085 2 南京农业大学资源与环境科学学院作物遗传与种质创新国家重点实验室,南京 210095 3 中国科学院生态环境研究中心土壤环境研究室,北京 100085 4 中国科学院城市环境研究所城市环境与健康研究中心重点实验室,厦门 361021
土壤生物与土壤污染研究前沿与展望
陈保冬1, 赵方杰2, 张 莘1,伍松林1,乔 敏3,欧阳纬莹4,朱永官3,4,*
1 中国科学院生态环境研究中心城市与区域生态国家重点实验室,北京 100085 2 南京农业大学资源与环境科学学院作物遗传与种质创新国家重点实验室,南京 210095 3 中国科学院生态环境研究中心土壤环境研究室,北京 100085 4 中国科学院城市环境研究所城市环境与健康研究中心重点实验室,厦门 361021
随着社会经济发展,人类生产活动对自然环境产生越来越广泛深刻的影响,土壤污染已成为危及生态系统稳定、农产品质量安全和人体健康的突出环境问题之一。重金属、有机污染化合物、病原菌及抗性基因等各类污染物大量进入土壤后,对土壤生物系统造成毒害作用,影响到土壤生态功能;另一方面,土壤生物如细菌、真菌、土壤动物等在一定程度上能够适应土壤污染,深刻影响着污染物在土壤中的迁移转化过程,在土壤污染修复中具有潜在重要作用。从土壤污染的生态毒理效应、土壤生物对土壤污染的响应与适应机制、污染土壤修复原理与技术等三方面综述了土壤生物与土壤污染相关研究前沿,展望了重点研究方向。
土壤污染;土壤生物;生态毒理效应;适应机制;生物修复
随着社会经济的发展,人类工业或农业生产过程中产生的各种污染物通过不同途径进入土壤环境,最终超过土壤自净能力,使土壤质量与功能发生变化,危及人类及其他生物的生存和发展,即为土壤污染。根据2014年全国土壤污染状况调查公报(http://www.zhb.gov.cn/gkml/hbb/qt/201404/t20140417_270670.htm),我国土壤环境状况总体不容乐观,部分地区土壤污染较重,耕地土壤环境质量堪忧,工矿业废弃地土壤环境问题突出,土壤污染治理迫在眉睫。
按照污染物属性土壤污染可区分为无机污染、有机污染以及生物污染等。土壤无机污染以重金属或类金属(如镉、砷、汞、铜、铅、铬)污染为主。值得注意的是,近年来随着纳米技术和纳米材料的大规模应用,大量的人工纳米颗粒物如纳米金属氧化物、含碳纳米颗粒物及量子点(一种由有限数目的原子组成且三个维度尺寸均在纳米数量级的新型纳米材料,因其独特的物理化学性质在生命科学、材料等领域得到广泛应用)等进入到土壤中,其生态安全性和潜在健康风险也日益受到关注。土壤有机污染以六六六、滴滴涕、多氯联苯、多环芳烃等有机化合物污染为主,而近年来一些新兴有机污染物,如全氟化合物、多溴联苯醚、短链氯化石蜡、五氯苯等也逐渐引起关注[1]。土壤生物污染则是指病原体和有害生物种群从外界侵入土壤,破坏土壤生态系统平衡,引起土壤质量下降的现象[2]。土壤中往往含有一些病原微生物,如果大量繁衍,将会破坏原有土壤生态平衡。同时,很多外源病原微生物(如大肠杆菌、沙门氏菌、脊髓灰质炎病毒等)随着生活污水、污泥或畜禽粪便等进入土壤,也造成土壤生物污染[2]。近年来因抗生素的滥用而导致抗生素抗性基因(Antibiotic resistance genes,ARGs)的环境扩散和积累也被归为生物污染,并已成为全世界关注的一个环境问题[3]。
各种污染物进入土壤环境后会对土壤生态系统造成深刻影响。一方面污染物往往具有致毒作用,会直接影响土壤生物活性,改变土壤生物系统结构,进而影响土壤生态功能;另一方面,土壤生物通过一系列代谢途径消纳、转化或富集污染物,影响污染物的环境行为和归趋。系统深入研究土壤生物与土壤污染物之间的相互作用及其机制,不仅对于全面认识土壤污染的生态效应,以及土壤生物对土壤污染的适应策略具有重要意义,同时也是发展污染土壤生物修复技术的重要基础。
1 土壤生物与土壤污染研究前沿
土壤污染会影响土壤生物系统的结构和功能,而土壤生物则对土壤污染产生适应、反馈调控以及消解清除作用,对两者相互作用的研究有利于发展土壤生物修复技术。基于土壤污染与土壤生物的相互作用(图1),相关科学研究主要分为三个方面:(1)土壤污染的生态毒理效应;(2)土壤生物对土壤污染的响应与适应机制;(3)污染土壤修复原理与技术。
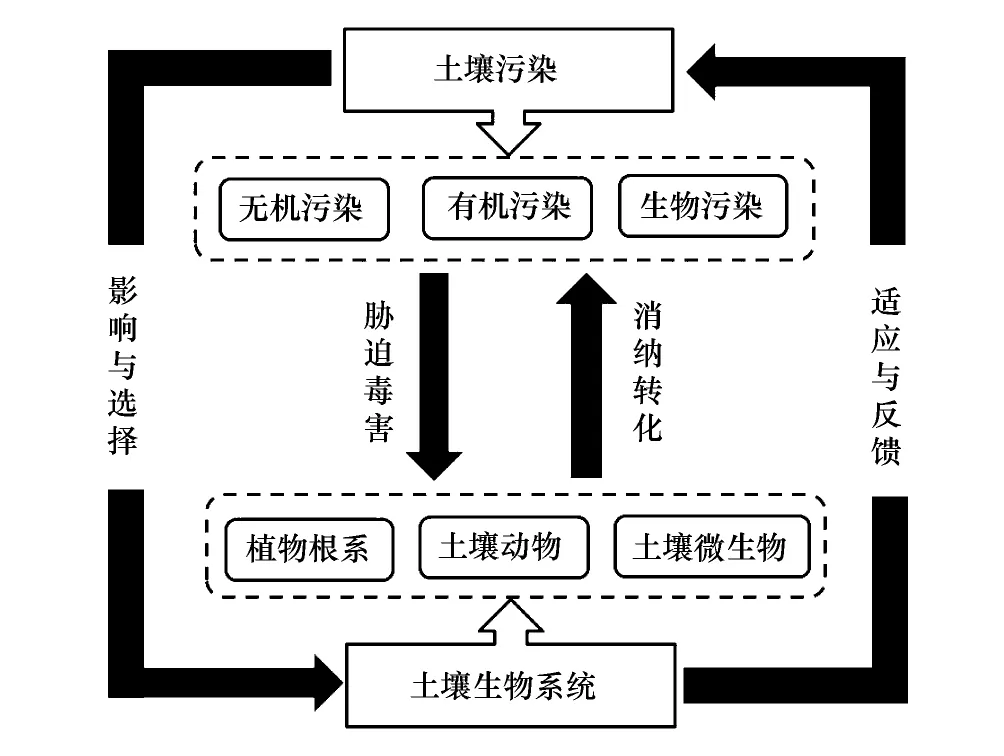
图1 土壤污染与土壤生物系统相互作用Fig.1 Interactions between soil pollutants and soil organisms
1.1 土壤污染的生态毒理效应
土壤污染的生态毒理效应研究主要是考察污染物对生物个体生理、生物种群、群落结构乃至生态系统结构和功能的影响[4]。当前的研究多集中在生物个体层次,如以典型模式生物个体或细胞为考察对象,通过死亡率和生长发育相关指标(如无脊椎动物的繁殖率、产卵率以及植物的种子发芽、根伸长抑制和生物量等)来反映污染物的毒性效应[5-6],或采用基因组学及蛋白组学等研究有毒污染物的毒性机制,在基因、细胞和个体水平上揭示典型污染物和生物相互作用的位点及其作用方式[7],以及采用代谢组学等手段解析有毒污染物对生物代谢过程影响的机理和调控途径[8]。针对生物个体毒理效应的研究被广泛用来评价单一污染物或复合污染物的直接毒性,但由于这类研究不能反映土壤中物种间的相互作用,对物种敏感性差异考虑不足,因而在将污染物毒性外推到种群或群落水平时具有很多局限性。显而易见,生物个体的变化必然会引起种群数量改变, 使种群衰落直至灭亡,从种群/群落乃至生态系统水平探讨土壤污染的生态毒理效应,研究其所致的生态毒理效应演化过程,正逐渐形成生态毒理学新的学科增长点。
近年来有学者提出利用多物种测试系统来构建简化的土壤食物网,从而在一定程度上反映物种间相互作用[9]。陆生微宇宙(Microcosm)系统被认为是最接近自然生态系统的试验系统,一般由处理后的土壤(采集并过筛)或是未经扰动的土体(Soil core)及引入的多种物种组成,能够在生态系统水平上模拟研究污染物的迁移转化和归趋,以及评价污染物对生态系统整体结构和功能的影响[10-11]。不过,由于微宇宙中生物群落结构比较复杂,对毒物反应的终点不够明确,使得微宇宙中观测到结果往往不易解释,试验可再现性也有待加强。相对而言,针对群落水平的毒性研究,以微生物为评价对象在技术上是最容易实现的。土壤微生物在生态系统中具有特殊的生态地位,对元素的生物地球循环具有不可替代的重要作用,因而选择具有代表性的土壤微生物作为受体测试环境污染的生态效应已成为近年生态毒理研究领域的热点。
重金属是土壤中最为常见的污染物。无论是对生物体必需还是非必需的重金属元素,达到一定浓度时都会损坏生物细胞膜、改变生物酶活性、影响细胞内正常的生理功能、并破坏生物体的DNA结构[12]。譬如,Hg2+、Cd2+、Ag+等生物体非必需元素能与巯基基团(-SH)结合,进而抑制生物体的酶活性,Cr与S,以及As与P在结构上类似导致这些元素很容易取代生物体内的必需元素[13]而导致生理功能紊乱。土壤中的微生物数量庞大并具有高的比表面积,为重金属与微生物细胞壁的相互作用提供了广阔场所,而微生物细胞膜具有的网状负电荷也使得其更易从周围环境中积累金属离子,且这种传输不能区分必需元素还是有毒重金属[14]。已有大量研究报道了土壤重金属污染对微生物生物量、土壤呼吸、底物利用、氮转化、酶活性以及微生物多样性的影响[15-19]。例如,Chander等[20]研究发现受重金属胁迫的土壤微生物需要消耗更多能量,导致对底物的利用效率降低,引起土壤微生物生物量的降低;Gans等[21]研究发现在原始土壤样品中存在超过100万种不同的细菌基因组,而重金属污染会降低其99.9%的多样性。Singh等[22]研究也发现在重金属长期胁迫下,土壤微生物群落多样性显著降低,并导致一些特定功能如对污染物的矿化能力丧失。
对外源有机污染物而言,土壤微生物是有机污染物降解的主要驱动力,然而有机污染物也深刻影响着土壤微生物的生长、丰度及降解能力。很多研究发现有机污染物能显著抑制微生物的生长和生理活动,如敌草隆的降解产物对亚硝酸细菌和硝酸细菌有抑制作用,苯氧淡酸类除草剂可通过影响寄主植物而抑制共生固氮菌的生长和活动,2,4-D和甲基氯苯氧乙酸对土壤中蓝细菌的光合作用有毒性作用[23]。不过,也有研究发现,有机污染物胁迫下功能微生物类群的丰度会相应提高[24], 并且在某些环境中,其丰度与有机污染物的浓度成正比[25]。当这些功能微生物再次受到有机污染物胁迫时,因经过前期的适应过程,其生长速率明显增加[26]。这些研究结果的差异可能与有机污染物的类型和土壤中污染物浓度水平有关。此外,有研究发现有机物存在时还会改变重金属移动性及土壤微生物活性,例如溴代阻燃剂BDE209的适当加入会加速Pb转化为更稳定的形态进而对土壤微生物的生物量、呼吸和代谢产生不同影响[27]。
对于新型污染物抗性基因而言,其可随畜禽粪便进入土壤系统,并可能将抗性基因转移到土著微生物中[28-29]。抗生素选择压力也会诱导抗性基因的产生,有报道土壤重复施加含磺胺嘧啶的猪粪后,sul1和sul2两种抗性基因的丰度显著增加[30]。同时,由于抗生素进入土壤后能直接杀死土壤中某些微生物或抑制相关微生物的生长,因而能改变微生物群落结构及微生物活性。Haller等[31]研究发现磺胺类(磺胺嘧啶和磺胺甲恶唑)和四环素类(土霉素)抗生素明显抑制了土壤细菌和放线菌生长,使土壤微生物生物量明显下降,但土壤真菌生物量增加。Hammesfahr等[32]也证实磺胺嘧啶污染的粪便使土壤细菌/真菌比例下降。Boleas等[33]则发现土壤中四环素浓度达到1 mg/kg即可显著抑制土壤脱氢酶和磷酸酶的活性。最近研究发现,即使施用不含抗生素的有机肥,也可以显著富集土壤中抗生素抗性微生物和抗性基因,其原因是土壤养分的投入可以改变土壤微生物群落结构,特别是一些通常含有抗性基因的微生物类群,如假单胞杆菌等[34]。
尽管有关污染物对土壤生物的影响已有大量研究报道,但目前还非常缺乏真正意义上的土壤污染生态毒理学研究:很少有研究考虑物种间的相互作用,更少有真正在生态系统层次开展的研究。此外,自然条件下土壤污染通常是多种污染物共存的复合污染,但已有研究多以单种或单类污染物作为研究对象,考察其在环境介质中的迁移转化过程,但很少考虑复合污染的生态效应。客观地讲,在群落水平乃至生态系统水平开展毒性测试还存在很多难点,如在户外自然光照和降雨条件下进行的中宇宙测试,与真实环境高度接近,但由于测试系统中的组分是动态变化和相互依赖的,这种复杂性使得建立污染物和群落变化间的剂量-效应关系非常困难,同时成本较高导致实验重复少,也限制了实际应用。今后还需进一步发展针对多物种的半野外测试方法来评价群落/生态系统水平的效应。
1.2 土壤生物对环境污染的响应及其适应机制
污染物对土壤生物具有毒害作用,但另一方面土壤生物对各类污染物也有一定的抗性和解毒机制。对于重金属而言,土壤生物的解毒及耐性机制包括:(1)泌出、吸附以及细胞外的沉淀作用,如许多微生物通过胞外聚合物将重金属固持在胞外[35];(2)对过量重金属的区隔化作用,如重金属进入植物细胞后往往聚集在液泡中[36],而在菌根共生体系中重金属会被固持在根内真菌结构中[37];(3)细胞内解毒作用,植物或微生物摄入过量重金属离子可诱导金属硫蛋白、植物络合素、谷胱甘肽等物质的大量合成,并与重金属离子相结合,从而减轻重金属毒害,如铜或镉胁迫能够上调丛枝菌根真菌中金属硫蛋白基因(GintMT1)的表达[38];(4)通过氧化、还原、甲基化和去甲基化作用对重金属进行转化,将重金属转化为无毒、低毒或易排出体外的形态。如一些微生物能够将砷甲基化,从而降低砷毒害,同时也帮助植物减轻毒害[39-40],有些厌氧微生物可将汞甲基化,再将甲基汞排出细胞外达到解毒作用[41]。
一些土壤微生物与植物在长期适应环境过程中形成了协同抵抗重金属机制。例如,在重金属污染环境中,AM真菌即可通过多种机制对宿主植物起到保护作用。概括起来,AM真菌主要通过间接和直接两条途径增强植物重金属耐性,间接途径主要通过促进植物对矿质养分(尤其是磷)的吸收,改善植物营养状况,促进植物生长,进而增强植物重金属耐性;直接途径主要指丛枝菌根对重金属环境行为的直接影响,如根外菌丝对重金属的直接固持作用等[42]。此外,值得注意的是,一些微生物或植物甚至能够在重金属污染下获益,如一些化能自养型砷氧化微生物能够将三价砷氧化为五价砷,并且利用其产生的能量同化二氧化碳,供自身生长[43-44]。虽然土壤生物消纳重金属的过程及机制已有较多研究,但过去还是以重金属迁移转化的化学过程为主,相关生理及分子机制方面的深入研究相对较少,而这方面的研究对于从本质上认识重金属的地球化学循环过程无疑具有重要意义。
对于有机污染物而言,土壤生物在其降解过程中具有不可替代的重要作用。土壤有机污染物的降解关键过程如脱氯、开环、脱氮、氧化及还原等往往在微生物作用下完成。微生物降解有机污染物主要分为两种方式,生长代谢和共代谢。生长代谢是指微生物将有机污染物作为碳源和能源物质加以分解和利用的代谢过程[45]。以多环芳烃为例,微生物在单加氧酶或双加氧酶的催化作用下,将氧加到苯环上,形成C—O键,再通过加氢、脱水等作用使C—C键断裂,苯环数减少,形成一些中间产物,如邻苯二酚等,这些中间产物进而被微生物利用合成自身生长所需物质[46]。共代谢则是指微生物能够降解或改变有机污染物化学结构,但并不以此作为碳源和能源而是从其它底物获取碳源和能源的代谢过程,如一些微生物如不动杆菌等能够在以苯为碳源的条件下降解三氯苯酚和四氯苯酚[47]。通常一些高分子多环芳烃及其类似物的降解要依赖共代谢作用,一些持久性难降解有机污染物如多氯联苯等,也能够在特定菌种及合适条件发生脱氯反应[48]。总体来说,土壤生物对有机污染的响应和适应机理研究还比较少,尚待通过分子生物学手段研究微生物解毒基因,针对不同结构的有机物污染,研究其解毒途径及降解产物。
对于生物污染而言,研究其在土壤中的传播与演化机制才刚刚起步。土壤颗粒对病原微生物具有较强的吸附作用,同时存在对流-弥散、沥滤等过程,并受到病原微生物自身性质、土壤性质和环境条件等因素的影响。另一方面,不同病原微生物在土壤中的存活状态也不尽相同[49],并受到诸多因素如土壤pH、有机质、土壤温度及湿度、离子强度、外界紫外线强度、土著微生物区系以及根际效应等的影响[50]。多数病原微生物进入土壤后会很快消亡,但仍有小部分病原微生物长时间内仍能保持感染活力,如弓蛔虫(Toxocara)在土壤中存活时间达3—4年,炭疸杆菌(Bacillusanthracis)在土壤中存活时间甚至长达几十年。土壤病原菌还可以通过污灌、畜禽粪便施用、昆虫传播等多种方式进入到植物体内产生二次污染[51]。一些抗生素抗性细菌进入土壤后,其携带的抗性基因甚至可通过水平转移传播到土著微生物中[52],从而对土壤生态系统造成破坏。揭示病原微生物致毒基因在不同土壤生物类群之间的转移机制及毒力基因残留效应,探究抗生素抗性菌及抗性基因和病原微生物的复合污染以及两者相互作用是土壤生物污染研究领域的重点内容。
1.3 污染土壤修复原理与技术
研究土壤污染的生态毒理效应及土壤生物对土壤污染的响应与适应机制,一方面能够为建立土壤环境质量标准提供依据,另一方面也为解决土壤污染问题奠定了重要基础。事实上,污染土壤修复原理和技术一直也是土壤学领域的重要研究内容。传统的污染土壤修复主要以客土、土壤淋洗、施加稳定剂等物理化学方法为主。近年来,也有一些新型的环境功能材料应用于污染土壤修复,如近年迅速发展起来的纳米零价铁(Nanoscale zero-valent iron,nZVI)修复技术。nZVI因其比表面积大、强还原性,且具有量子效应、比表面效应、体积效应等特性,对重金属、无机盐及有机污染物都具有较好的去除效果,逐渐被广泛用于重金属(如六价铬)或有机化合物(多环芳烃等)污染土壤的修复[53-54]。传统物理化学修复技术对于治理严重污染土壤具有时间短、见效快等优点,但往往伴随着高能耗、高费用、二次污染等风险,因而不适用于大规模污染土壤的修复。相应地,近年来发展起来的利用特定植物或微生物修复污染土壤的生物修复方法因其绿色环保、高效、成本低等优点而受到广泛关注。
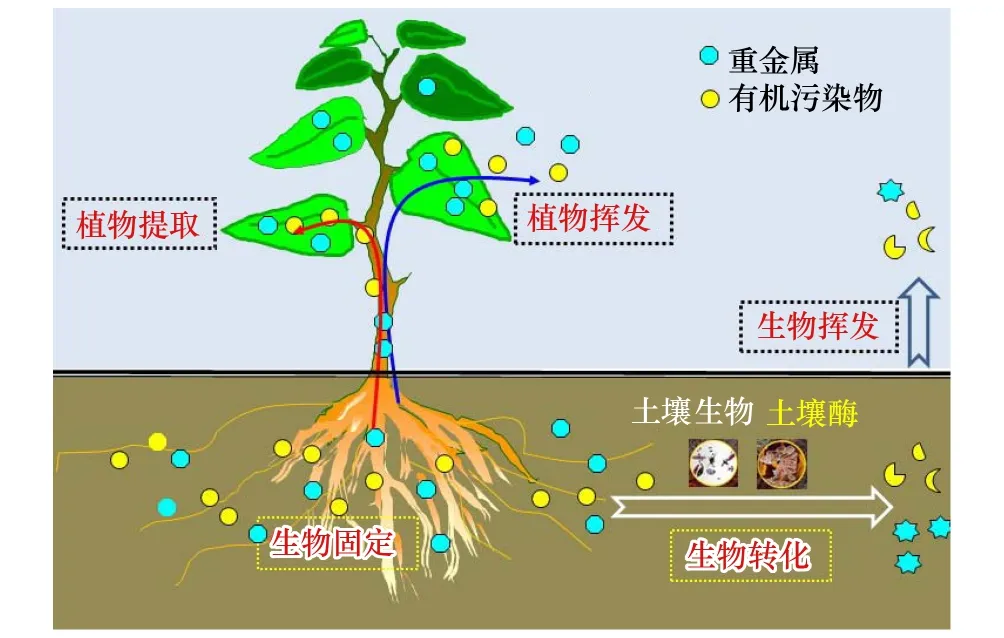
图2 植物-微生物联合修复重金属和有机污染土壤示意图 Fig.2 Bioremediation of contaminated soil by plant and soil microorganisms植物根系、真菌菌丝等可以直接固持土壤中的重金属(生物固定);超积累植物能将重金属从土壤中吸收积累至植物地上部分(植物提取),亦或将可挥发性金属元素通过气孔释放到大气中(植物挥发)。另一方面,土壤生物通过直接吸收或共代谢作用转化降解土壤中的有机污染物(生物转化),有机物降解产物可直接释放到环境介质当中(生物挥发)
在概念上,生物修复是指利用生物的代谢活动及其代谢产物富集、降解或固定土壤中的污染物,从而恢复被污染土壤的生产或景观价值的一个受控或自发进行的生物学过程(图2)。生物修复主要以植物修复和微生物修复为主。对于不同类型的污染物,其修复方法往往不同。对于重金属来说,其结构简单,性质较为稳定,因而常常采取植物萃取和植物固定方法进行修复[55]。植物萃取主要是利用超积累植物吸收、富集重金属,从而减少乃至清除土壤中的重金属,如利用砷超积累植物蜈蚣草进行砷污染土壤的修复[56]。植物固定则主要利用特定耐性植物将污染物固定在根际或根系中以降低重金属可迁移性,如利用一些矿区中天然存在的耐性植物修复重金属污染土壤[57]。此外,也可利用某些具有特殊功能的微生物类群的代谢活动来降低重金属活性及毒性以达到土壤修复的目的(即微生物修复),如利用六价铬还原细菌高效还原土壤中六价铬以降低铬毒性从而修复铬污染土壤[58]。对于有机污染物而言,其结构和性质较为复杂,且生物降解难易程度不同,因而要根据污染物的特性选取适宜的修复方法。土壤中多数有机污染物能够被微生物降解转化,最终变成无害形态。土壤微生物转化有机污染物主要涉及氧化还原、水解、集团转移、酯化、缩合、氨化等过程,且分为生长代谢和共代谢途径两种类型。基于土壤微生物能够降解有机污染物的特性,微生物修复已成为土壤有机污染修复的主要技术[59]。此外,一些植物也能够吸收转运有机物,并将小分子有机物通过叶片挥发至大气当中(植物挥发),而一些难挥发性有机物能够通过酶促反应被无害化降解(植物降解),一些难降解有机污染物也能够被特定植物吸收累积(植物富集)。因而,利用植物修复有机污染土壤也具有一定应用前景[60-61]。
值得注意的是,自然环境中植物与微生物之间往往具有密切的相互作用,不仅影响着彼此的污染耐受性,同时也对污染物迁移转化产生重要影响。将植物修复和微生物修复技术有机结合起来,对于提高生物修复效率具有重要意义。例如,通过菌根共生体系通常能够增强植物对污染土壤的耐受性并促进植物修复[62]。在实际情况下,由于污染土壤往往存在复合污染、物理结构不良、水土保持能力弱等限制性因素,导致单一修复措施无法实现,因而需要根据实际情形将生物修复与物理化学修复有机结合,以获得最佳修复效果。此外,对于需要持续生产的农业用地或林地而言,可考虑生产与修复相结合的方式,如利用一些高生物量的能源植物进行修复,在达到污染土壤修复目的的同时产生一定的经济效益。
2 未来研究动向分析
基于国内外土壤污染与土壤生物研究前沿动态和我国土壤污染基本状况,这一研究领域的关键科学问题可凝练为:(1)土壤污染如何影响土壤生物群落结构与功能?(2)土壤消纳污染物的生物学机理?(3)生物污染在土壤中的传播与演化机制?围绕这些问题,以下几方面将成为未来研究的重点方向:
2.1 土壤污染背景下微生物群落结构和多样性的演变及生态效应
过去有关土壤污染的生态效应研究主要在细胞和生物个体水平开展,研究对象主要是动物和植物,但随着微生物分子生态学的发展,越来越多的研究开始关注土壤污染对微生物群落的影响,包括在功能基因、功能群乃至在土壤生态系统水平研究不同污染物及复合污染对土壤微生物多样性和功能的影响及其机制,重点是将生物多样性(群落结构)与相关生态功能/过程结合起来进行研究,从生物多样性和生态系统稳定性(抵抗力和恢复力)的关系角度理解土壤污染对土壤生态系统影响机制。
随着分子生物学及生物信息学的迅速发展,利用高通量测序、核酸杂交技术、DNA指纹图谱技术和基因芯片等分子生物学方法来分析土壤微生物多样性和群落结构,以及应用宏基因组学、宏蛋白组学及代谢组学等系统生物学的方法研究土壤微生物活性及功能的演变规律已成为土壤生物学领域的趋势。这些新方法的应用会大大促进我们对土壤污染与土壤生物系统相互作用的全面认识,有助于理解在土壤污染胁迫下土壤生物群落结构及功能的演变规律及响应适应过程。在上述研究的基础上,未来应加强基于长期定位实验的研究,也就是结合现代组学技术、大数据科学和模型等手段在较长的时间尺度上认识土壤生物系统在土壤污染胁迫下的演变规律和机制。将来土壤生态毒理学研究应更多地拓展到实际环境,从更大的时间和空间尺度上进行研究,考察的对象应从土壤生物群落层次上升到食物网层次,同时要将生物多样性演变与生态功能变化联系起来,从而进一步提高研究的科学性和实用性。
2.2 土壤污染的生物转化和清除机制
土壤生物可通过氧化、还原和甲基化等过程直接改变重金属化学形态,或通过改变铁、硫等元素的氧化还原状态、分泌有机络合物、细胞壁吸附和固定等过程影响重金属的生物有效性[12]。土壤生物同样可以通过特异性或非特异性酶脱毒系统,或通过一些特定的代谢途径降解有机污染物。通过野外调查及传统室内模拟试验,发现并分离特定功能微生物,进一步通过同位素示踪技术、原位荧光杂交技术、电子显微技术,以及近年来迅速发展起来的同步辐射技术(如X射线荧光微分析技术(XRF)和X射线吸收精细结构分析技术(XAFS))、次生离子质谱技术,可深层次揭示污染物迁移转化的界面化学过程[63]。另一方面,生物体内的生理代谢及分子调控过程是驱动污染物化学转化的内在动力,未来的研究也需要将分子生物学技术(如实时荧光定量PCR、基因克隆及功能验证、蛋白质结构解析、稳定性同位素核酸探针DNA/RNA-SIP等技术)与先进的物理化学技术(如高精度质谱、电镜分析及同步辐射等技术)相结合,从分子层次上深入探索土壤生物介导的污染物转化机制,为土壤污染的生物修复奠定基础。此外,一些污染物的生物代谢及消纳过程往往与生物自身代谢过程相耦合,如微生物驱动的有机污染物降解可以耦合铁还原、硝酸盐还原、硫酸盐还原及甲烷产生过程,这些耦合作用在元素生物地球化学循环过程中扮演着重要的角色。具体研究过程中,需要综合考虑生物自身的代谢过程及污染物的转化特点,全面理解污染物在生物体内的代谢过程及调控机制。
2.3 病原菌和抗生素抗性基因在土壤中的分布规律与传播机制
病原菌和抗生素抗性菌及抗性基因一方面会通过土壤-空气、土壤-植物、或土壤-地下水途径直接进入人体,危害人体健康,另一方面则会造成农作物病害,抑制农作物的生长发育,造成农业减产或农产品品质降低。因而,进行土壤病原菌及抗生素抗性基因污染防御及治理迫在眉睫。系统深入研究病原菌和抗生素抗性菌及抗性基因在土壤中的分布传播过程与机制是进行污染治理的前提。
土壤中存在大量的抗生素产生菌和同化菌,是天然的抗性基因储库[64],而抗生素的大规模生产和使用则进一步加速了土著抗性微生物和抗性基因的扩散,增加了抗生素耐药性的发生频率[65]。目前研究者通过实时荧光定量PCR、宏基因组以及高通量测序等现代分子生物学技术探究了不同土壤类型中抗性基因的分布规律,在远古冻土和不受人类活动影响的土壤中发现了多样性很高的抗性基因[66-67],发现在未受抗生素污染的农业和草地土壤中抗性基因结构由微生物群落组成决定[68],证明了土壤中抗性基因的自然广泛存在,同时也揭示了中水灌溉土壤、施用禽畜粪便土壤等典型污染土壤中抗性基因富集的现象[69-70]。
基于已有关于土壤生物污染现状的调查,进一步的研究应围绕抗性基因污染的来源、迁移转化机制及生态风险展开:(1)建立土壤抗性基因与潜在宿主菌之间的关系,明晰污染来源;(2)探究不同类型土壤环境中抗性基因发生水平转移的实际频率;(3)结合实验室模拟及长期实地监测,研究病原菌及抗生素抗性菌及抗性基因在土壤中的迁移过程,重点解析其通过植物吸收进入食物链以及通过地下水淋洗进入水环境的风险;(4)评估抗性基因在土壤微生物和致病菌之间的交叉转移,探究土壤抗性基因库的潜在生态风险。
2.4 基于活体微生物的土壤污染监测技术
传统的化学分析方法监测土壤污染的优点是能够精确测定污染物的浓度和形态,但却往往不能确切反映污染物的生物毒性强弱。化学分析本身不能反映生物暴露污染物的途径和环境条件的影响(即缺乏生态相关性),也不能区别急性毒性和慢性毒性(即缺乏时间相关性)。以活的生物体或群体来测试化学物质毒性效应的生物监测方法能够有效克服化学分析方法的缺点,反映环境污染对生物产生的综合效应或专性效应,以及污染物在土壤中的动态转化,因而越来越受到重视[71-72]。根据测试对象的类型,土壤生物监测主要分为三大类,即基于个体或组织层次的生物监测,基于功能基因或基因组学层次的生物监测,以及基于群落水平的抵抗力和恢复力测试。目前研究的重点是通过“组学”研究方法不断发展新型生物标志物(包括功能基因)、生物芯片和新型全细胞专性的生物传感器。污染物进入土壤后形态会发生一系列变化,如何确定具体是哪种形态致毒是今后的研究热点和难点。比如,通过基因重组和突变等手段发展能够区分无机砷和有机砷的全细胞微生物传感器,可以原位监测砷形态在转化过程[73]。在生物监测中测试终点的选择需要综合考虑生态相关性、暴露途径、敏感性、管理目标及终点可测性等因素,根据具体情况选择适宜的指标。
2.5 污染土壤生物修复原理与技术
污染土壤生物修复已有一定理论基础,但实践应用方面还有很长的路要走,许多技术瓶颈尚待突破。对于不同污染情形,往往需要构建不同的修复技术体系,如针对某一特定污染种类,选用专性耐受的植物和微生物种类,或利用转基因技术修饰改造植物或微生物,使之适应污染土壤并发挥修复功效[74]。同时,需要对特定功能的微生物或植物的污染物耐受及转化机制进行深入研究,明确其分子机理和遗传稳定性,并在此基础上发展基于功能基因的生物技术。在实际应用中,需要特别考虑生物的环境适应性问题,明确微生物修复污染土壤的适用条件和范围,研究提高修复效率的关键参数及工程设计。现有的生物修复技术多以基础理论研究为主,而相关的技术推广及实践应用方面研究较为稀少。今后研究需要更加关注生物修复技术的实地应用,使其逐渐成熟和完善。
作为土壤生物与土壤污染相关基础研究的应用目标,污染土壤生物修复技术势必越来越受到关注。而为了实现这一应用目标,则需要进一步系统深入地研究土壤污染对土壤生物的生态毒理效应,揭示土壤生物对土壤污染物的适应及消纳机制。在以往研究工作基础上,借助于研究方法技术的发展,未来的土壤生物与土壤污染研究,将向微观和宏观两个方向拓展,微观方向将不断深入至基因、分子乃至信号水平,宏观方向则向生态系统水平推进。传统单因素的研究将转变为双因素和多因素研究,结构或功能隔离研究将转向结构功能关系研究,离散的时间和空间节点观察,将转向空间分异和时间动态特征,乃至时空演变特征系统研究。总体上,至广大、至精微、多因素、多尺度的研究已经成为显而易见的学科发展趋势。
[1] 王亚韡, 王宝盛, 傅建捷, 阮挺, 曲广波, 汪畅, 曾力希, 刘倩, 袁博, 江桂斌. 新型有机污染物研究进展. 化学通报, 2013. 76(1): 3-14.
[2] Bradford S A, Morales V L, Zhang W, Harvey R W, Packman A I, Mohanram A, Welty C. Transport and fate of microbial pathogens in agricultural settings. Critical Reviews in Environmental Science and Technology, 2013, 43(8): 775-893.
[3] Martínez J L. Antibiotics and antibiotic resistance genes in natural environments. Science, 2008, 321(5887): 365-367.
[4] van Gestel C A M. Soil ecotoxicology: state of the art and future directions. ZooKeys, 2012, 176: 275-296.
[5] Christiansen K S, Borggaard O K, Holm P E, Vijver M G, Hauschild M Z, Peijnenburg W J G M. Experimental determinations of soil copper toxicity to lettuce (Lactucasativa) growth in highly different copper spiked and aged soils. Environmental Science and Pollution Research International, 2015, 22(7): 5283-5292.
[6] Zortéa T, Baretta D, Maccari A P, Segat J C, Boiago E S, Sousa J P, Da Silva A S. Influence of cypermethrin on avoidance behavior, survival and reproduction of Folsomia candida in soil. Chemosphere, 2015, 122: 94-98.
[7] Chen G Q, De Boer T E, Wagelmans M, van Gestel C A M, Van Straalen N M, Roelofs D. Integrating transcriptomics into triad-based soil-quality assessment. Environmental Toxicology and Chemistry, 2014, 33(4): 900-909.
[8] Baylay A J, Spurgeon D J, Svendsen C, Griffin J L, Swain S C, Sturzenbaum S R, Jones O A H. A metabolomics based test of independent action and concentration addition using the earthwormLumbricusrubellus. Ecotoxicology, 2012, 21(5): 1436-1447.
[9] Schnug L, Jensen J, Scott-Fordsmand J J, Leinaas H P. Toxicity of three biocides to springtails and earthworms in a soil multi-species (SMS) test system. Soil Biology and Biochemistry, 2014, 74: 115-126.
[10] Burrows L A, Edwards C A. The use of integrated soil microcosms to predict effects of pesticides on soil ecosystems. European Journal of Soil Biology, 2002, 38(3/4): 245-249.
[12] Bruins M R, Kapil S, Oehme F W. Microbial resistance to metals in the environment. Ecotoxicology and Environental Safety, 2000, 45(3): 198-207.
[13] Olaniran A O, Balgobind A, Pillay B. Bioavailability of heavy metals in soil: impact on microbial biodegradation of organic compounds and possible improvement strategies. International Journal of Molecular Sciences, 2013, 14(5): 10197-10228.
[14] Gall J E, Boyd R S, Rajakaruna N. Transfer of heavy metals through terrestrial food webs: a review. Environmental Monitoring and Assessment, 2015, 187: 201.
[15] Anderson G L, Williams J, Hille R. The purification and characterization of arsenite oxidase from Alcaligenes faecalis, a molybdenum-containing hydroxylase. Journal of Biological Chemistry, 1992, 267(33): 23674-23682.
[16] Giller K E, Witter E, McGrath S P. Toxicity of heavy metals to microorganisms and microbial processes in agricultural soils: a review. Soil Biology and Biochemistry, 1998, 30(10-11): 1389-1414.
[17] Giller K E, Witter E, McGrath S P. Heavy metals and soil microbes. Soil Biology and Biochemistry, 2009, 41(10): 2031-2037.
[18] Macdonald C A, Clark I M, Zhao F J, Hirsch P R, Singh B K, McGrath S P. Long-term impacts of zinc and copper enriched sewage sludge additions on bacterial, archaeal and fungal communities in arable and grassland soils. Soil Biology and Biochemistry, 2011, 43(5): 932-941.
[19] Boshoff M, De Jonge M, Dardenne F, Blust R, Bervoets L. The impact of metal pollution on soil faunal and microbial activity in two grassland ecosystems. Environmental Research, 2014, 134: 169-180.
[20] Chander K, Klein T, Eberhardt U, Joergensen R. Decomposition of carbon-14-labelled wheat straw in repeatedly fumigated and non-fumigated soils with different levels of heavy metal contamination. Biology and Fertility of Soils, 2002, 35(2): 86-91.
[21] Gans J, Wolinsky M, Dunbar J. Computational improvements reveal great bacterial diversity and high metal toxicity in soil. Science, 2005, 309(5739): 1387-1390.
[22] Singh B K, Quince C, Macdonald C A, Khachane A, Thomas N, Abu Al-Soud W, Sørensen S J, He Z L, White D, Sinclair A, Crooks B, Zhou J Z, Campbell C D. Loss of microbial diversity in soils is coincident with reductions in some specialized functions. Environmental Microbiology, 2014, 16(8): 2408-2420.
[23] 周启星, 王美娥. 土壤生态毒理学研究进展与展望. 生态毒理学报, 2006, 1(1): 1-11.
[24] O′Connell M, Baldwin D S, Robertson A I, Rees G. Release and bioavailability of dissolved organic matter from floodplain litter: influence of origin and oxygen levels. Freshwater Biology, 2000, 45(3): 333-342.
[25] Sei K, Sugimoto Y, Mori K, Maki H, Kohnol T. Monitoring of alkane-degrading bacteria in a sea-water microcosm during crude oil degradation by polymerase chain reaction based on alkane-catabolic genes. Environmental Microbiology, 2003, 5(6): 517-522.
[26] Johnsen A R, Karlson U. Diffuse PAH contamination of surface soils: environmental occurrence, bioavailability, and microbial degradation. Applied Microbiology and Biotechnology, 2007, 76(3): 533-543.
[27] Zhang W, Chen L, Zhang R, Lin K F. Effects of decabromodiphenyl ether on lead mobility and microbial toxicity in soil. Chemosphere, 2015, 122: 99-104.
[28] Chee-Sanford J C, Mackie R I, Koike S, Krapac I G, Lin Y F, Yannarell A C, Maxwell S, Aminov R I. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste. Journal of Environmental Quality, 2009, 38(3): 1086-1108.
[29] Esiobu N, Armenta L, Ike J. Antibiotic resistance in soil and water environments. International Journal of Environmental Health Research, 2002, 12(2): 133-144.
[30] Heuer H, Solehati Q, Zimmerling U, Kleineidam K, Schloter M, Müller T, Focks A, Thiele-Bruhn S, Smalla K. Accumulation of sulfonamide resistance genes in arable soils due to repeated application of manure containing sulfadiazine. Applied and Environmental Microbiology, 77(7): 2527-2530.
[31] Haller M Y, Müller S R, McArdell C S, Alder A C, Suter M J -F. Quantification of veterinary antibiotics (sulfonamides and trimethoprim) in animal manure by liquid chromatography-mass spectrometry. Journal of Chromatography A, 2002, 952(1/2): 111-120.
[32] Hammesfahr U, Heuer H, Manzke B, Smalla K, Thiele-Bruhn S. Impact of the antibiotic sulfadiazine and pig manure on the microbial community structure in agricultural soils. Soil Biology and Biochemistry, 2008, 40(7): 1583-1591.
[33] Boleas S, Alonso C, Pro J, Fernández C, Carbonell G, Arazona J V. Toxicity of the antimicrobial oxytetracycline to soil organisms in a multi-species-soil system (MS•3) and influence of manure co-addition. Journal of Hazardous Materials, 2005, 122(3): 233-241.
[34] Udikovic-Kolic N, Wichmann F, Broderick N A, Handelsman J. Bloom of resident antibiotic-resistant bacteria in soil following manure fertilization. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(42): 15202-15207.
[35] Li W W, Yu H Q. Insight into the roles of microbial extracellular polymer substances in metal biosorption. Bioresource Technology, 2014, 160: 15-23.
[36] Liu J, Duan C Q, Zhang X H, Zhu Y N, Hu C. Subcellular distribution of chromium in accumulating plantLeersiahexandraSwartz. Plant and Soil, 2009, 322(1/2): 187-195.
[37] Nayuki K, Chen B D, Ohtomo R, Kuga Y. Cellular imaging of cadmium in resin sections of arbuscular mycorrhizas using synchrotron micro X-ray fluorescence. Microbes and Environments, 2014, 29(1): 60-66.
[38] González-Guerrero M, Cano C, Azcón-Aguilar C, Ferrol N.GintMT1 encodes a functional metallothionein inGlomusintraradicesthat responds to oxidative stress. Mycorrhiza, 2007, 17(4): 327-335.
[39] Lomax C, Liu W J, Wu L Y, Xue K, Xiong J B, Zhou J Z, McGrath S P, Meharg A A, Miller A J, Zhao F J. Methylated arsenic species in plants originate from soil microorganisms. New Phytologist, 2012, 193(3): 665-672.
[40] Zhu Y G, Yoshinaga M, Zhao F J, Rosen B P. Earth abides arsenic biotransformations. Annual Review of Earth and Planetary Sciences, 2014, 42: 443-467.
[41] Parks J M, Johs A, Podar M, Bridou R, Hurt R A Jr, Smith S D, Tomanicek S J, Qian Y, Brown S D, Brandt C C, Palumbo A V, Smith J C, Wall J D, Elias D A, Liang L Y. The genetic basis for bacterial mercury methylation. Science, 2013, 339(6125): 1332-1335.
[42] 伍松林, 张莘, 陈保冬. 丛枝菌根对土壤——植物系统中重金属迁移转化的影响. 生态毒理学报, 2013, 8(6): 847-856.
[43] Oremland R S, Stolz J F. Arsenic, microbes and contaminated aquifers. Trends in Microbiology, 2005, 13(2): 45-49.
[44] Zhang J, Zhou W X, Liu B B, He J, Shen Q R, Zhao F J. Anaerobic arsenite oxidation by an autotrophic arsenite-oxidizing bacterium from an arsenic-contaminated paddy soil. Environmental Science & Technology, 2015, 49(10): 5956-5964.
[45] 石成春. 有机污染物微生物共代谢降解及其动力学研究. 化学工程与装备, 2010, (7): 164-167, 121-121.
[46] Juhasz A L, Naidu R. Bioremediation of high molecular weight polycyclic aromatic hydrocarbons: A review of the microbial degradation of benzo[a]pyrene. International Biodeterioration & Biodegradation, 2000, 45(1/2): 57-88.
[47] Kim M H, Hao O J. Cometabolic degradation of chlorophenols byAcinetobacterspecies. Water Research, 1999, 33(2): 562-574.
[48] 毕新慧, 徐晓白. 多氯联苯的环境行为. 化学进展, 2000, 12(2): 152-160.
[49] Crane S R, Moore J A. Modeling enteric bacterial die-off: A review. Water, Air, & Soil Pollution, 1986, 27(3/4): 411-439.
[50] Ongeng D, Geeraerd A H, Springael D, Ryckeboer J, Muyanja C, Mauriello G. Fate ofEscherichiacoliO157:H7 andSalmonellaentericain the manure-amended soil-plant ecosystem of fresh vegetable crops: A review. Critical Reviews in Microbiology, 2015, 41(3): 273-294.
[51] Wachtel M R, Charkowski A O. Cross-contamination of lettuce withEscherichiacoliO157:H7. Journal of Food Protect, 2002, 65(3): 465-470.
[52] Forsberg K J, Reyes A, Wang B, Selleck E M, Sommer M O A, Dantas G. The shared antibiotic resistome of soil bacteria and human pathogens. Science, 2012, 337(6098): 1107-1111.
[53] Chen C F, Binh N T, Chen C W, Dong C D. Removal of polycyclic aromatic hydrocarbons from sediments using sodium persulfate activated by temperature and nanoscale zero-valent iron. Journal of the Air & Waste Management Association, 2015, 65(4): 375-383.
[54] Di Palma L, Gueye M T, Petrucci E. Hexavalent chromium reduction in contaminated soil: A comparison between ferrous sulphate and nanoscale zero-valent iron. Jounal of Hazardous Materials, 2015, 281: 70-76.
[55] Chaney R L, Malik M, Li Y M, Brown S L, Brewer E P, Angle J S, Baker A J M. Phytoremediation of soil metals. Current Opinion in Biotechnology, 1997, 8(3): 279-284.
[56] Xie Q E, Yan X L, Liao X Y, Li X. The arsenic hyperaccumulator fernPterisvittataL. Environmental Science & Technology, 2009, 43(22): 8488-8495.
[57] Pérez-López R, Márquez-García B, Abreu M M, Nieto J M, Córdoba F.EricaandevalensisandEricaaustralisgrowing in the same extreme environments: Phytostabilization potential of mining areas. Geoderma, 2014, 230-231: 194-203.
[58] Smith W L, Gadd G M. Reduction and precipitation of chromate by mixed culture sulphate-reducing bacterial biofilms. Journal of Applied Microbiology, 2000, 88(6): 983-991.
[59] 滕应, 骆永明, 李振高. 污染土壤的微生物修复原理与技术进展. 土壤, 2007, 39(4): 497-502.
[60] Gerhardt K E, Huang X D, Glick B R, Greenberg B M. Phytoremediation and rhizoremediation of organic soil contaminants: Potential and challenges. Plant Science, 2009, 176(1): 20-30.
[61] Cunningham S D, Anderson T A, Schwab A P, Hsu F C. Phytoremediation of soils contaminated with organic pollutants. Advances in Agronomy, 1996, 56: 55-114.
[62] Dong Y, Zhu Y G, Smith F A, Wang Y S, Chen B D. Arbuscular mycorrhiza enhanced arsenic resistance of both white clover (TrifoliumrepensLinn.) and ryegrass (LoliumperenneL.) plants in an arsenic-contaminated soil. Environmental Pollution, 2008, 155(1): 174-181.
[63] Zhao F J, Moore K L, Lombi E, Zhu Y G. Imaging element distribution and speciation in plant cells. Trends in Plant Science, 2014, 19(3): 183-192.
[64] Dantas G, Sommer M O A, Oluwasegun R D, Church G M. Bacteria subsisting on antibiotics. Science, 2008, 320(5872): 100-103.
[65] Knapp C W, Dolfing J, Phillip A I E, Graham D W. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940. Environmental Science & Technology, 2009, 44(2): 580-587.
[66] Allen H K, Moe L A, Rodbumrer J, Gaarder A, Handelsman J. Functional metagenomics reveals diverse β-lactamases in a remote Alaskan soil. The ISME Journal, 2009, 3(2): 243-251.
[67] Lang K S, Anderson J M, Schwarz S, Williamson L, Handelsman J, Singer R S. Novel florfenicol and chloramphenicol resistance gene discovered in Alaskan soil by using functional metagenomics. Applied and Environmental Microbiology, 2010, 76(15): 5321-5326.
[68] Forsberg K J, Patel S, Gibson M K, Lauber C L, Knight R, Fierer N, Dantas G. Bacterial phylogeny structures soil resistomes across habitats. Nature, 2014, 509(7502): 612-616.
[69] Zhu Y G, Johnson T A, Su J Q, Qiao M, Guo G X, Stedtfeld R D, Hashsham S A, Tiedje J M. Diverse and abundant antibiotic resistance genes in Chinese swine farms. Proceedings of the NationalAcademy of Sciences of the United States of America, 2013, 110(9): 3435-3440.
[70] Wang F H, Qiao M, Su J Q, Chen Z, Zhou X, Zhu Y G. High throughput profiling of antibiotic resistance genes in urban park soils with reclaimed water irrigation. Environmental Science & Technology, 2014, 48(16): 9079-9085.
[71] Hund-Rinke K, Kördel W, Hennecke D, Eisenträger A, Heiden S. Bioassays for the ecotoxicological and genotoxicological assessment of contaminated soils (results of a round robin test) part I: Assessment of a possible groundwater contamination: Ecotoxicological and genotoxicological tests with aqueous soil extracts. Journal of Soils and Sediments, 2002, 2(1): 43-50.
[72] Maliszewska-Kordybach B, Smreczak B. Ecotoxicological activity of soils polluted with polycyclic aromatic hydrocarbons (PAHs)-effect on plants. Environmental Technology, 2000, 21(10): 1099-1110.
[73] Chen M J, Ogunseitan O A. Removing As, Ba, Cu and Zn from waste plasma display panel glass by electrokinetics//Li J H, Hu H L, eds. Selected Proceedings of the Eighth International Conference on Waste Management and Technology. Shanghai: ICWMT, 2014: 393-398.
[74] Vangronsveld J, Herzig R, Weyens N, Boulet J, Adriaensen K, Ruttens A, Thewys T, Vassilev A, Meers E, Nehnevajova E, Van Der Lelie D, Mench M. Phytoremediation of contaminated soils and groundwater: lessons from the field. Environmental Science and Pollution Research, 2009, 16(7): 765-794.
Soil pollution and soil organisms: an overview of research progress and perspectives
CHEN Baodong1, ZHAO Fangjie2, ZHANG Xin1, WU Songlin1, QIAO Min3, OUYANG Weiying4, ZHU Yongguan3,4,*
1StateKeyLaboratoryofUrbanandRegionalEcology,ResearchCenterforEco-EnvironmentalSciences,ChineseAcademyofSciences,Beijing100085,China2StateKeyLaboratoryofCropGeneticsandGermplasmEnhancement,CollegeofResourcesandEnvironmentalSciences,NanjingAgriculturalUniversity,Nanjing210095,China3DepartmentofSoilEnvironmentScience,ResearchCenterforEco-EnvironmentalSciences,ChineseAcademyofSciences,Beijing100085,China4KeyLaboratoryofUrbanEnvironmentandHealth,InstituteofUrbanEnvironment,ChineseAcademyofSciences,Xiamen361021,China
With social and economic development, anthropogenic activities have been exerting profound effects on the natural environment. Soil pollution has become one of the most pressing environmental problems threatening ecosystem stability, agricultural product safety, and human health. Soil ecosystem is a complex system consisting of plant roots, soil animals, and soil microorganisms, which play important roles in energy transfer and material cycling. Numerous pollutants such as heavy metals, organic compounds, and pathogens and antibiotic resistant genes, etc., can greatly influence soil ecosystem functions through their detrimental effects on soil organisms. On the other hand, soil organisms can evolve various strategies to adapt to the contaminated environment, mediate translocation and transformation of soil contaminants, and may have the potential for soil remediation. In this review, we summarize recent progresses in research on the interactions of soil organisms and soil pollutants, focusing on (1) eco-toxicological effects of soil pollution; (2) the mechanisms underlying the responses and adaptation of soil organisms to soil pollution; and (3) the principles and technologies for the bioremediation of contaminated soils. We also propose perspectives for future research, including changes in soil microbial community structure upon soil pollution, biotransformation and detoxification of pollutants by soil organisms, biological monitoring of soil pollution, and distribution and transmission of pathogens and antibiotic resistant genes in soil.
soil pollution; soil organisms; ecotoxicity; adaptation mechanisms; bioremediation
国家自然科学基金面上项目(41471219); 城市与区域生态国家重点实验室自主方向项目(SKLURE2012-1-03)
2015-06-23;
2015-08-17
10.5846/stxb201506231259
*通讯作者Corresponding author.E-mail: ygzhu@rcees.ac.cn
陈保冬, 赵方杰, 张莘,伍松林,乔敏,欧阳纬莹,朱永官.土壤生物与土壤污染研究前沿与展望.生态学报,2015,35(20):6604-6613.
Chen B D, Zhao F J, Zhang X, Wu S L, Qiao M, Ouyang W Y, Zhu Y G.Soil pollution and soil organisms: an overview of research progress and perspectives.Acta Ecologica Sinica,2015,35(20):6604-6613.

