基于酵母产孢的syntaxin家族蛋白SNARE区域功能分析
王丽萍, 邵侃凯, 高晓冬, 中西秀树
(江南大学 生物工程学院,江苏 无锡214122)
基于酵母产孢的syntaxin家族蛋白SNARE区域功能分析
王丽萍, 邵侃凯, 高晓冬, 中西秀树*
(江南大学 生物工程学院,江苏 无锡214122)
真核细胞中大部分膜融合过程由SNARE蛋白介导,但其功能和调节机制尚未完全清楚。酵母孢子形成是研究囊泡融合机制包括SNARE蛋白的理想模型系统。在该过程中涉及t-SNARE蛋白Sso1,它是突触囊泡融合所需蛋白syntaxin 1A的同源物,两者SNARE区域有51%的同源性。尽管如此,将SSO1的SNARE区域完全替换成syntaxin 1A,而构建的嵌合体却无法回补sso1△突变的产孢缺陷。为了确定哪些残基为Sso1功能所必须,作者进行了嵌合体和突变分析,发现Sso1/syntaxin 1A嵌合体中syntaxin 1A的SNARE区域的220位丙氨酸换成谷氨酸后获得产孢功能。另外,Sso1发生相应的突变-218位谷氨酸突变成丙氨酸后失去其功能。因此,218位谷氨酸残基为Sso1产孢功能所必须。
SNARE;人源化;酿酒酵母;Sso1p;syntaxin1A
在真核生物中,囊泡融合机制是高度保守的,SNARE(soluble N-ethylmaleimide-sensitive factor attachment protein receptors;可溶性NSF附着蛋白受体)蛋白是融合蛋白中的重要成员[1-2]。SNARE蛋白是保守性很强的蛋白家族,经常被分为两组:QSNARE蛋白和R-SNARE蛋白[3]。SNARE蛋白位于特定的膜上,只有当同源的Q-SNARE蛋白和RSNARE蛋白结合时,泡膜才能融合。
在神经元的突触囊泡融合过程中,syntaxin-1[4]和SNAP-25扮演Q-SNARE角色,synaptobrevin则扮演着R-SNARE角色。当囊泡膜上的synaptobrevin与syntaxin-1和 SNAP-25结合在一起时,突触囊泡中的神经递质就会释放出来,传递给突触后膜[1,3,5-7]。SNARE蛋白不仅和神经递质释放有关,也和粘蛋白及血小板升高引起的人类疾病有关。研究表明,抑制SNARE蛋白功能可导致神经退行性疾病[8-9]。SNARE融合过程受到complexin[10]、Tomosyn[11-12]、Munc18/nSec-1[13]等多种蛋白质的调控,但调控机制尚不明确。
酵母细胞中同样存在 SNARE融合系统,syntaxin-1在突触囊泡融合过程中的作用机制可利用酵母的SNARE融合系统来研究。
酵母中有两个 syntaxin蛋白——Sso1p和Sso2p,他们是哺乳动物syntaxin1的同源物且彼此高度相似。Sso1p和Sso2p在酵母营养细胞中共同执行基本功能——与SNARE蛋白Sec9p,Snc1p和Snc2p形成质膜复合体[14]。在孢子形成过程中则特异性地要求Sso1p存在[15-16]。在该过程中Spo20p代替Sec9p,与Sso1p、Snc1p和Snc2p形成复合体,促使纺锤体极点上prospore membrane(PrM)的扩展。Sso1p缺失的酵母因PrM无法扩展而不能形成孢子。
在SNAREs参与的孢子形成过程中,SSO1和SPO20是PrM形成所必需的基因[16]。Hideki Nakanishi等人已研发出监测PrM形成过程中囊泡融合的技术。这类现象也可通过荧光显微镜观察到,并且在活细胞中可看到PrM的延伸[17-18]。因此,PrM的形成过程是研究囊泡融合机理的一个理想操作模型系统。SNARE蛋白基因在进化过程中比较保守,可以将 Sso1p的 SNARE区域替换成 syntaxin1的SNARE区域从而构建人源化的膜融合体系。根据改造得到的菌株不仅可用于在酵母细胞中研究人类突触囊泡融合机理,还可用于医疗和工业用途,其中包括建立神经肉毒素的检测系统。而且,它们也是筛选参与突触囊泡融合新基因的非常有用的工具。
作者通过嵌合体的构建对Sso1p的SNARE区域的产孢功能区进行了定位,并成功构建了sso1△sso2△突变株,为筛选与syntaxin1A作用的蛋白或药物提供了更为简便的方法。也为后续人源化SNARE膜融合系统的构建及相关理论研究奠定了基础。
1 材料与方法
1.1 材料与仪器
1.1.1 菌株和质粒酿酒酵母出发菌株 AN120、HJ3(sso1△spo20△)、质粒pFA6a-kanMX6、质粒pFA6a-his3MX6、质粒pRS314、质粒pRS304、质粒pRS316、质粒pRS426:由作者所在实验室保存。
1.1.2 主要试剂和仪器KOD-Plus-Neo高保真DNA聚合酶:东洋纺公司;Taq DNA聚合酶、限制性内切酶及T4-DNA Ligase:TaKaRa公司;PCR产物纯化试剂盒、DNA琼脂糖凝胶回收试剂盒及质粒小提试剂盒:上海生工生物公司;其他试剂:均为进口分装或国产分析纯。
1.1.3 培养基
1)YPAD培养基:每升含酵母膏10 g,蛋白胨20 g,葡萄糖20 g,腺嘌呤30 mg;
2)YNB选择培养基:每升含酵母基础氮源6.7 g,葡萄糖20 g,补加适量必须元素;
3)YPAce培养基:每升含酵母膏10 g,蛋白胨20 g,乙酸钾20 g,腺嘌呤30 mg;
4)产孢培养基:每升含乙酸钾20 g;
5)5-FOA(5-Fluoroorotic Acid)[19]培养基:每升含酵母基础氮源6.7 g,5-氟乳清酸1 g,葡萄糖20 g,补加适量必须元素;
6)LB-氨苄培养基:每升含酵母膏5 g,胰蛋白胨10 g,NaCl 10 g,氨苄100 mg;
7)G418培养基:每升含酵母膏10 g,蛋白胨20 g,葡萄糖20 g,腺嘌呤30 mg,G418 500 mg。固体培养基添加2 g/dL的琼脂粉。
1.2 基因敲除
基因敲除方法见文献 [20]。其中质粒pFA6akanMX6为敲除SSO1的模板,质粒pFA6a-his3MX为敲除SSO2的模板,PCR引物见表1。
表1PCR引物
Table 1 Primers

?
1.3 产孢分析
将含质粒的酿酒酵母在YNB选择培养基于30℃培养过夜,取500 μL培养液转接至4.5 mL YPAce培养基中培养20~24 h,收集细胞,无菌水洗后重悬于3 mL产孢培养基中继续培养30 h,观察记产孢率。每种质粒转化两次,每次选3个单菌落计数,细胞数不少于500。
1.4 PrM的观察
将以pRS304为载体构建的质粒单酶切,回收所得线性质粒转化sso1△缺陷株,色氨酸缺陷平板筛选阳性克隆。用pRS426-G20转化所得菌株,色氨酸、尿嘧啶缺陷平板筛选阳性克隆,按产孢分析方法培养,产孢培养基中培养6、7、8 h时各取500 μL培养液收集细胞,水洗后重悬于50 μL无菌水,加450 μL甲醛,30℃摇床处理30 min。收集细胞,无菌水洗两次后重悬于100 μL无菌水,取5 μL细胞悬液与5 μL DAPI溶液混合,制片观察PrM。
1.5 营养细胞生长分析
以pRS316SSO1pr-SSO1转化sso1△单倍体缺陷株,以筛选所得阳性克隆为出发菌株敲除SSO2,得sso1△sso2△双缺陷型菌株。将待验证质粒转入sso1△sso2△,在YNB选择性平板上筛选出阳性克隆,随后划线到5-FOA平板上观察生长状况。
2 结果与讨论
2.1 Sso1p与syntaxin1A的嵌合体不能使酵母产孢
Syntaxin含有具三个短螺旋的 N端结构域、SNARE区域及带有极短亲水尾的C端跨膜结构域[3]。膜融合过程中,syntaxin的 SNARE区域与其他SNARE蛋白结合形成四螺旋束结构,将两侧膜拉近从而进行随后的融合。Syntaxin的SNARE区域为自身N端结构及多种其他调控蛋白的作用部位,故在本研究中考虑将 Sso1p的 SNARE区域替换成syntaxin1A的SNARE区域。
以 pRS314为载体构建含syntaxin1A(sytx1A)与Sso1p的嵌合体Sso1p/sytx1A(SNARE区域)的质粒,转化sso1△后观察其能否产孢。在减数分裂Ⅱ期,含不同质粒的酵母细胞均能形成四个新的细胞核;含Sso1p的酿酒酵母可形成PrM,进而形成孢子,嵌合体则只在纺锤体处形成点状结构而无法扩张,说明其缺乏Sso1p在孢子形成过程中的功能,意味着不能将 Sso1p的 SNARE区域全部替换成sytx1A的SNARE区域,见图1。
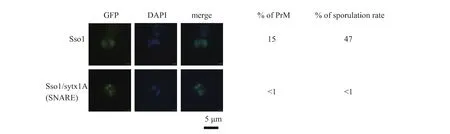
图1 含重组质粒的sso1△在产孢培养基中培养7 h后PrM形态Fig.1 Shape of PrM of sso1△containing recombinant plasmid which cultured for 7 h in spore medium
2.2 A220为嵌合体无产孢功能的原因
Syntaxin1A与sso1p的SNARE区域有51%的同源性,将其替换sso1p的SNARE区域后却使其失去产孢功能 (本研究中氨基酸编号为其原本在Sso1p与syntaxin1A中的编号),为何如此高的同源性功能却有如此大的差异?故设计了一系列嵌合体对Sso1p产孢功能区进行定位,见图2。产孢分析结果表明:保留Sso1p第214~221位氨基酸,其余SNARE区域全部替换成sytx1A,嵌合体仍能产孢,意味着 Sso1p的 214~221位氨基酸为其形成SNARE复合体所必须。
根据该结果,我们大胆假设当位于sytx1A与其他SNARE蛋白α螺旋的接触表面上的与Sso1p有显著性质差异的氨基酸发生突变后,可能使嵌合体具产孢功能。随后在Sso1p/sytx1A(SNARE)基础上设计了三个定点突变:I(209)M,HDM (213-215)TQL,AM(220,221)EE;结果sytx1A的SNARE区域发生AM(220,221)EE突变后能使sso1△产孢。进一步定点突变A(220)E、M(221)E,最终发现sytx1A发生A(220)E突变后具产孢功能,说明A220是Sso1p/sytx1A(SNARE)无产孢功能的原因。Sso1p/ sytx1A嵌合体仅将A220这一个氨基酸突变成E后便具有产孢活性,该嵌合体可用于与sytx1A相作用的蛋白质或药物的筛选。

图2 利用Sso1p与sytx1A的嵌合体定位Sso1p产孢功能区Fig.2 Mapping of the sporulation-specific functions in Sso1p using fusions with sytx1A
2.3 Sso1p功能的实现需要E218
由于Sso1p/sytx1A发生点突变A(220)E后具产孢功能,A220为Sso1p/sytx1A不能产孢的原因,在Sso1p中进行相应的突变E(218)A后则可以验证Sso1p的产孢功能是否特异性需要E的存在。
将Sso1p的E218突变成A后观察PrM状态并进行产孢分析。结果含野生型Sso1p的菌株能形成4个PrM,突变体则只能形成点状结构。结果表明,野生型的Sso1p发生该突变后无法形成PrM,见图3。失去产孢功能,进一步证实该位点氨基酸E对产孢功能的重要性。
酿酒酵母中Sso1p为产孢所必须,其在营养细胞中功能与Sso2p相同[21]。在单倍体酵母中敲除SSO1和SSO2后,细胞将无法存活。于是构建了sso1△sso2△双缺陷型单倍体菌株,可利用营养细胞实验验证嵌合体的功能。

图3 含重组质粒的sso1△在产孢培养基中培养7 h后PrM形态Fig.3 Shape of PrM of sso1△containing recombinant plasmid which cultured for 7 h in spore medium
将上述嵌合体转入sso1△sso2△突变株,所得转化株转移至含5-FOA的平板上使其丢失含野生型SSO1的质粒,嵌合体将成为细胞中惟一的Sso蛋白[19];观察转化株生长状况。在不含5-FOA的YNB选择性(缺乏色氨酸、尿嘧啶)平板上,各转化株生长状况无明显差别;转接到含5-FOA的YNB选择性(缺乏色氨酸)平板上后Sso1p/sytx1A(SNARE)-A(220)E能在 5-FOA平板上生长,Sso1p/sytx1A(SNARE)、Sso1p E(218)A则不能生长,见图4。当Sso1p的E218突变成非极性氨基酸A后无法进行膜融合,因而不能存活。与上述结果一致,Sso1p的E218为其形成SNARE复合体所必须。利用生长分析所得结果和产孢分析一致,该系统可用于随后的高通量筛选与sytx1A作用的基因或药物,大大简化筛选工作。
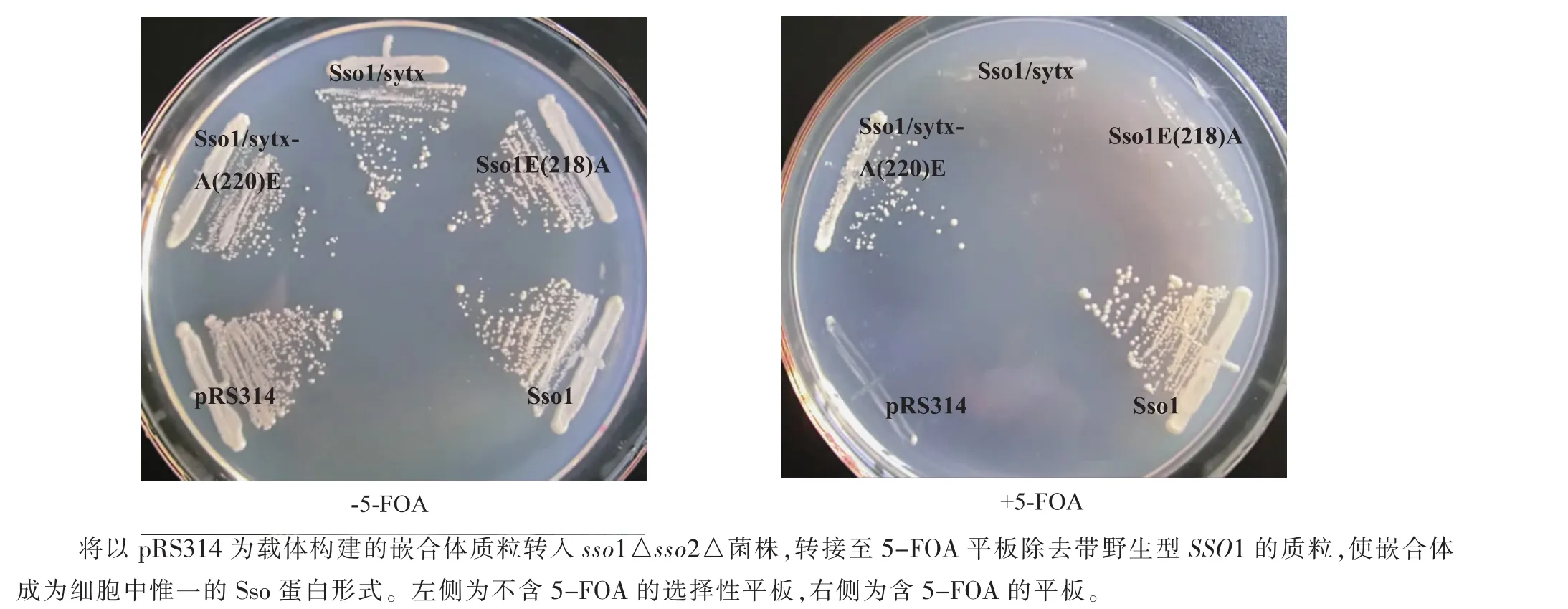
图4 营养细胞实验Fig.4 Vegetative cell experiments
3 结语
作者发现将酵母中Sso1p的SNARE区域替换成Sytx1A的SNARE区域而构建的嵌合体Sso1p/ sytx1A无法使酿酒酵母产生孢子,将嵌合体的A220突变成E后则具有产孢活性;而将Sso1p的E218突变成A后则失去了产孢活性,因此E218为Sso1p功能所必须。真核细胞中存在多种与syntaxin家族蛋白作用的蛋白质。Complexin与Tomosyn只存在于哺乳动物神经元细胞中,通过与syntaxin1A的SNARE区域结合调控神经递质的释放,具体的调控机制尚不明确。实验构建的嵌合体Sso1p/sytx1A-A(220)E为利用酵母细胞研究这些蛋白质与sytx1A的作用机制提供了新的思路。Munc18/Sec-1也存在于酿酒酵母细胞中,与SNARE复合体结合从而调节膜融合过程。由于Munc18/Sec-1与SNARE复合体结合时,E218处于结合位点上,因此该氨基酸发生突变后可能影响这两种蛋白质的结合。此外,形成SNARE核心复合体时,来自于4个螺旋的侧链相接触形成16个片层,位于片层上的氨基酸直接控制SNARE复合物的形成。而E218位于片层之外,发生点突变后使Sso1p失去活性,因此E218有可能调控SNARE复合体的形成,为酿酒酵母中膜融合机制的研究提供了参考。
[1]Hughson F M.Chaperones that SNARE neurotransmitter release[J].Science,2013,339(6118):406-407.
[2]Shi L,Shen Q-T,Kiel A,et al.SNARE proteins:one to fuse and three to keep the nascent fusion pore open[J].Science,2012,335(6074):1355-1359.
[3]Chen Y A,Scheller R H.SNARE-mediated membrane fusion[J].Nature Reviews Molecular Cell Biology,2001,2(2):98-106.
[4]Zhou P,Pang Z P,Yang X,et al.Syntaxin-1 N-peptide and Habc-domain perform distinct essential functions in synaptic vesicle fusion[J].The EMBO Journal,2013,32(1):159-171.
[5]Stein A,Weber G,Wahl M C,et al.Helical extension of the neuronal SNARE complex into the membrane[J].Nature,2009,460(7254):525-528.
[6]Südhof T C,Rothman J E.Membrane fusion:grappling with SNARE and SM proteins[J].Science,2009,323(5913):474-477.
[7]Ungermann C,Langosch D.Functions of SNAREs in intracellular membrane fusion and lipid bilayer mixing[J].Journal of Cell Science,2005,118(17):3819-3828.
[8]Garcia-Reitbock P,Anichtchik O,Bellucci A,et al.SNARE protein redistribution and synaptic failure in a transgenic mouse model of Parkinson’s disease[J].Brain,2010,133(7):2032-2044.
[9]Rendó n W O,Martí nez-Alonso E,Tomá s M,et al.Golgi fragmentation is Rab and SNARE dependent in cellular models of Parkinson’s disease[J].Histochemistry and Cell Biology,2013,139(5):671-684.
[10]Maximov A,Tang J,Yang X,et al.Complexin controls the force transfer from SNARE complexes to membranes in fusion[J]. Science,2009,323(5913):516-521.
[11]Yamamoto Y,Fujikura K,Sakaue M,et al.The tail domain of tomosyn controls membrane fusion through tomosyn displacement by VAMP2[J].Biochemical and Biophysical Research Communications,2010,399(1):24-30.
[12]Carr C M,Rizo J.At the junction of SNARE and SM protein function[J].Current Opinion in Cell Biology,2010,22(4):488-495.
[13]Rathore S S,Bend E G,Yu H,et al.Syntaxin N-terminal peptide motif is an initiation factor for the assembly of the SNARE-Sec1/Munc18 membrane fusion complex[J].Proceedings of the National Academy of Sciences,2010,107(52):22399-22406.
[14]Jantti J,Aalto M K,Oyen M,et al.Characterization of temperature-sensitive mutations in the yeast syntaxin 1 homologues Sso1p and Sso2p,and evidence of a distinct function for Sso1p in sporulation[J].Journal of Cell Science,2002,115(2):409-420.
[15]Burri L,Lithgow T.A complete set of SNAREs in yeast[J].Traffic,2004,5(1):45-52.
[16]Neiman A M.Sporulation in the budding yeast Saccharomyces cerevisiae[J].Genetics,2011,189(3):737-765.
[17]Yang H J,Nakanishi H,Liu S,et al.Binding interactions control SNARE specificity in vivo[J].The Journal of Cell Biology,2008,183(6):1089-1100.
[18]Nakanishi H,Morishita M,Schwartz C L,et al.Phospholipase D and the SNARE Sso1p are necessary for vesicle fusion during sporulation in yeast[J].Journal of Cell Science,2006,119(7):1406-1415.
[19]Boeke J D,Trueheart J,Natsoulis G,et al.5-Fluoroorotic acid as a selective agent in yeast molecular genetics[J].Methods in Enzymology,1987,154:164-175.
[20]Longtine M S,McKenzie III A,Demarini D J,et al.Additional modules for versatile and economical PCR-based gene deletion and modification in Saccharomyces cerevisiae[J].Yeast,1998,14(10):953-961.
[21]Yuan Q,Jantti J.Functional analysis of phosphorylation on Saccharomyces cerevisiae syntaxin 1 homologues Sso1p and Sso2p[J]. PloS One,2010,5(10):e13323.
Functional Analysis of the SNARE Domain of Syntaxin Family Proteins Using Yeast Sporulation
WANG Liping, SHAO Kankai, GAO Xiaodong, NAKANISHI Hideki*
(School of Biotechnology,Jiangnan University,Wuxi 214122,China)
In eukaryotic cells,most of membrane fusion processes are mediated by SNARE proteins. However,its functional and regulatory mechanisms have not been fully understood.Previous study have shown that yeast sporulation was an ideal model system to manipulate and investigate vesicle fusion machinery including SNARE proteins.The t-SNARE protein Sso1 involved in this process is an orthologue of syntaxin 1A which is required for the synaptic vesicle fusion,both share 51% homology at the SNRE domains.However,although their functional and amino acid sequence are similar,Sso1/syntaxin 1A chimera cannot complement the sporulation deficiency of sso1△mutant when SNARE domain of Sso1 is replaced completely by syntaxin 1A.To determine which residues are critical for Sso1 function,chimera and mutational analyses are further conducted.The resultsshowed that the SSO1/syntaxin 1A chimera gain the functionality by replacing Ala220 residue to Glu in the syntaxin 1A SNARE domain.By contrast,Sso1 lose its function by mutating the corresponding amino acid residue,Glu218 into Ala.Thus,Glu218 residue is specifically required for Sso1 function.
SNARE,humanization,Saccharomyces cerevisiae,Sso1p,syntaxin1A
Q 784
A
1673—1689(2015)05—0530—06
2014-00-00
国家 “111计划”项目 (111-2-06);教育部科学研究项目 (313027);高等学校博士学科点专项科研基金项目(20120093110001);江南大学自主科研项目(JUSRP311A02)。
*通信作者:中西秀树(1973—),男,日本人,农学博士,教授,博士研究生导师,主要从事分子生物学、酵母遗传学、细胞生物学和糖生物学方面的研究。E-mail:hideki@jiangnan.edu.cn

