选择性剪接基因RBFOX1在食管鳞癌中的研究
复旦大学附属肿瘤医院放疗科,复旦大学上海医学院肿瘤学系,上海 200032
选择性剪接基因RBFOX1在食管鳞癌中的研究
邓家营,赵快乐
复旦大学附属肿瘤医院放疗科,复旦大学上海医学院肿瘤学系,上海 200032
背景与目的:选择性剪接是基因表达中的重要调控机制,异常的剪接可导致细胞周期异常、癌基因转录因子激活及抑癌基因转录因子失活;异常剪接与肿瘤发生、发展息息相关。DNA甲基化是表观遗传修饰的重要组成部分,基因启动子的异常甲基化可导致基因沉默,抑癌基因和DNA修复基因的高甲基化参与多种肿瘤的发生;另外DNA甲基化还是选择性剪接的关键参数,DNA异常甲基化影响选择性剪接的平衡。本研究通过对食管鳞癌组织标本中RBFOX1(RNA binding protein, fox-1 homolog 1)选择性剪接基因的甲基化水平和表达进行检测,探讨其临床应用价值。方法:在149例配对的食管鳞癌及癌旁组织中,运用MassARRAY对RBFOX1基因的甲基化水平进行检测,并从同一批样品中选取42对组织采用RT-PCR进行RBFOX1基因的mRNA表达分析,统计甲基化水平与食管鳞癌主要临床病理特征的关系。结果:在食管鳞癌组织标本中,RBFOX1的甲基化水平为41.8%,明显低于对应癌旁组织的68.3%。差异有统计学意义(P<0.01);RBFOX1的甲基化水平与患者的性别、年龄、吸烟、饮酒以及肿瘤的分化、分期等无明显相关。依据癌组织中甲基化水平阈值(33.6%)=Mean(癌旁组织)-2.5SD(标准差),将研究对象分为两组,低于阈值的一组定义为组1,高于阈值的为组2。组1和组2的5年总体生存率(overall survival,OS)为57.0%和35.7%。差异无统计学意义(P=0.06)。组1和组2的5年无进展生存率(progression-free survival,PFS)为48.7%和28.9%。差异有统计学意义(P=0.03)。多因素分析结果显示仅TNM分期为生存的独立预测因子。结论:选择性剪接基因RBFOX1在食管鳞癌组织中的甲基化水平和表达水平均低于癌旁组织,RBFOX1启动子区的甲基化水平不能作为生存分析的预测因子。
食管鳞癌;选择性剪接;甲基化;表达;RBFOX1
流行病学资料显示,我国食管癌发病率和死亡率分别占全部恶性肿瘤的第6位和第4位[1]。在肿瘤诊断、分期及治疗方面的研究取得了进展,使患者的生存率有了一定的提高,但20%~30%的5年生存率尚不令人满意,所以迫切需要探究食管癌的确切发病机制以寻找更加有效的治疗方法。肿瘤的发生、发展与癌基因的表达密切相关,选择性剪接(alternative splicing)和DNA甲基化是调控基因表达的重要生物学过程。
选择性剪接是真核生物基因表达中的一种高效、精准的调控机制,通过在pre-mRNA上的识别定位,剪接体能够准确地切除内含子序列,将不同的外显子序列拼接到一起,以产生不同的成熟mRNA[2]。选择性剪接现象广泛存在于超过90%的人类基因表达过程中,在细胞生长和组织分化等方面发挥关键作用[3]。选择性剪接产物的自然平衡是维持正常机体功能的前提,异常的剪接过程将导致平衡破坏,进而导致多种疾病[4]。研究表明基因选择性或异常剪接所产生的蛋白异构体可以参与细胞周期、细胞凋亡、转录因子激活或失活等生命过程,与肿瘤发生、发展息息相关[5]。
DNA甲基化是一种重要的表观遗传修饰。在哺乳动物基因组中,DNA甲基化的主要位点是CpG二核苷酸,尤其是基因启动子区域的DNA甲基化,可引起基因转录水平沉默。抑癌基因和DNA修复基因的高甲基化、重复序列DNA的低甲基化与多种肿瘤的发生有关[6]。已证明维甲酸受体β2启动子的异常甲基化与食管鳞癌的发生、T分期和淋巴结转移有关[7]; F-box蛋白32 (F-box protein 32,FBXO32)的基因启动子异常甲基化促进食管鳞癌的发生并产生不良预后[8]。因此,对基因异常甲基化的研究可探求食管鳞癌治疗的新靶点,丰富食管鳞癌的治疗手段。另外,有研究表明DNA甲基化还是剪接过程中的一个关键参数,它影响选择性剪接的类型、剪接的位点,进而影响外显子的剪接潜能[9]。DNA甲基化既能通过DNA结合蛋白来影响剪接结果,还能通过改变组蛋白的修饰来调控选择性剪接过程[10]。
RNA结合蛋白同源体1(RNA binding protein, fox-1 homolog 1,RBFOX1)基因位于16p13.3,大小约1694 246 bp。编码蛋白通过与5’-UGCAUGU-3’元件结合来调节组织特异性的外显子剪接。研究表明RBFOX1的靶基因包括降钙素基因、纤连蛋白基因和上皮特异性纤维母细胞生长因子受体基因等[11-12]。RBFOX1编码的蛋白质可作用于肌动蛋白、肌凝蛋白、驱动蛋白和微管结合蛋白等,参与细胞构架、变形和迁移[13]。有研究报道RBFOX1基因在结肠癌组织中存在缺失突变,突变频率约为1.5%,缺失突变的发生与结肠癌的发病呈正相关[14];研究还证明RBFOX1基因是恶性胶质瘤的组织特异性选择剪切基因,RBFOX1基因不仅通过编码的蛋白质影响细胞的生长和运动,还通过影响网格蛋白轻链B (clathrin light chain B,CLTB)进而控制细胞迁移[15]。RBFOX1基因在肿瘤发生、发展等生物学过程中的作用值得关注,截止目前,尚无RBFOX1在食管鳞癌中的研究报道。
1 材料和方法
1.1 研究对象和标本来源
组织标本均来自复旦大学附属肿瘤医院2007—2010年间的食管癌手术患者,共149例。其中男性134例,女性15例,年龄37~76岁,平均年龄58.4岁。每例患者均取癌组织原发灶及其距离癌组织边缘5 cm以上的癌旁正常组织(adjacent normal tissue,ANT)。全部患者术前均未经化疗和放疗。术中立即取材,标本放入液氮中速冻后-80 ℃条件下保存以提取DNA及RNA。术后常规病理诊断证实癌组织均为鳞状细胞癌。按照国际抗癌联盟(International Union Against Cancer,UICC)7th标准进行TNM分期,149例肿瘤患者中Ⅰ期9例(6.0%),Ⅱ期26例(17.4%),Ⅲ期114例(76.6%)。肿瘤组织学分级为:高分化14例(9.4%),中分化88例(59.1%),低分化47例(31.5%)。标本获得了伦理委员会批准并签署知情同意书。
1.2 方法
1.2.1 DNA抽提和DNA的亚硫酸盐处理
本研究采用按QIAmp DNA迷你试剂盒(德国Qiagen公司)试剂盒进行DNA 提取,每份标本(癌组织/癌旁组织)取25 mg。应用 EpiTect亚硫酸氢盐试剂盒(德国Qiagen公司)试剂盒进行亚硫酸盐处理,癌组织和癌旁组织DNA各400~500 ng。均按照试剂盒说明书进行操作。
1.2.2 甲基化水平检测
PCR反应体系参照甲基化PCR试剂盒(Sequenom)进行加样:加入经亚硫酸盐修饰的DNA模板1 μL,10×缓冲液0.5 μL,20 mmol/L的MgCl20.2 μL,20 mmol/L的dNTPs 0.04 μL,5 nmol/L的上、下游引物各0.4 μL,Hotstar Taq DNA聚合酶0.04 μL,用双蒸水调整体系,使最终体积为5 μL。反应条件:94 ℃ 4 min,1个循环;94 ℃ 20 s,56 ℃ 30 s,72 ℃ 1 min,共45个循环;72 ℃ 3 min。反应结束后加入2 μL稀释10倍的SAP酶。反应条件:37 ℃,20 min;85 ℃,5 min。反应结束后取2 µL反应产物加入到5 μL的T-cleavage反应体系中:5×缓冲液0.89 μL,T-cleavage Mix 0.22 μL,DTT 0.22 μL,TRase DNA聚合酶0.4 μL,RNase A 0.06 μL;反应条件37 ℃,3 h;反应结束后加水20 μL,上机准备完成。用MassARRAY(美国Sequenom公司)检测样品的甲基化水平,设定MassARRAY的检测阈值为1%,ΔMean定义为RBFOX1在癌旁组织和癌组织中甲基化水平的差值。甲基化特异性PCR正向引物为5’-AGGAAGAGAGTTGATTTTAGAGATTTGTTGTGAGAA-3’;反向引物为5’-CAGTAATACGACTCACTATAGGGAGAAGGCTAAAAAAACCAAAAAACCAAATAACA-3’。
1.2.3 RT-PCR检测RBFOX1 mRNA的表达
按TRIzol试剂说明书提取总RNA(美国Invitrogen公司),并参照Superscript Ⅲ逆转录试剂盒(美国Invitrogen公司)说明书的比例加样,将RNA逆转录成cDNA。RT-PCR参照FastStart DNA Master SYBR Green aster Mix(Roche公司)试剂盒加样,反应体系使用ABI ViiA TM7 system(美国Life Technologies公司)进行扩增,所有样品均重复检测3次。RBFOX1上游引物为5’-AGCTAGTTTTGCACCCTGCTA-3’,下游引物为5’-TTGGTCCGTGTTATTGGCACC-3’。GAPDH作为内参基因,上游引物为5’- GGAGCGAGATCCCTCCAAAAT-3’,下游引物为5’- GGCTGTTGTCATACTTCTCATGG -3’。本实验通过比较ΔCt的方法对RBFOX1基因表达量进行相对分析。
1.3 随访
通过门诊及电话方式随访,随访时间自手术日起截止至2013年12月31日。随访时间为2~72个月,中位随访时间为20个月。常规行胸片、食管钡餐及内镜检查等,酌情行CT、PET/CT等影像学检查,明确有无局部的复发和转移。
1.4 统计学处理
所有数据均采用SPSS 20.0统计软件进行分析。食管鳞癌组织或配对癌旁组织基因甲基化水平差异采用t检验;各基因甲基化情况与食管鳞癌主要临床病理特征之间的关系运用Fisher精确检验;Kaplan-Meier法进行生存分析,Cox回归进行多因素分析;所有检验均为双侧,P<0.05为差异有统计学意义。
2 结 果
2.1 RBFOX1基因启动子异常甲基化
利用Vector NTI Advance 11(美国Invitrogen公司),结合MassARRAY芯片的要求:所分析的区域分布有5~10个CpG岛时,芯片的检测效果最佳。本研究比较了RBFOX1基因启动子区域不同位置的CpG岛密度,选择了chr16:6014214-6014616 ([GRCh37/hg19])进行分析(图1),该区域包含6个CpG位点(CpG1、CpG2、CpG3、CpG4、CpG5和CpG6)。结果发现6个CpG位点均被检测到,且发现癌组织中所有CpG位点的甲基化水平显著低于癌旁组织(表1;图2A),癌组织中的平均甲基化水平显著低于癌旁组织。两者差异有统计学意义(41.8% vs 68.3%,P<0.000 1)。

图1 RBFOX1基因启动子区域CpG位点示意图Fig.1 The schematic diagram of the CpG sites in the RBFOX1 promoter
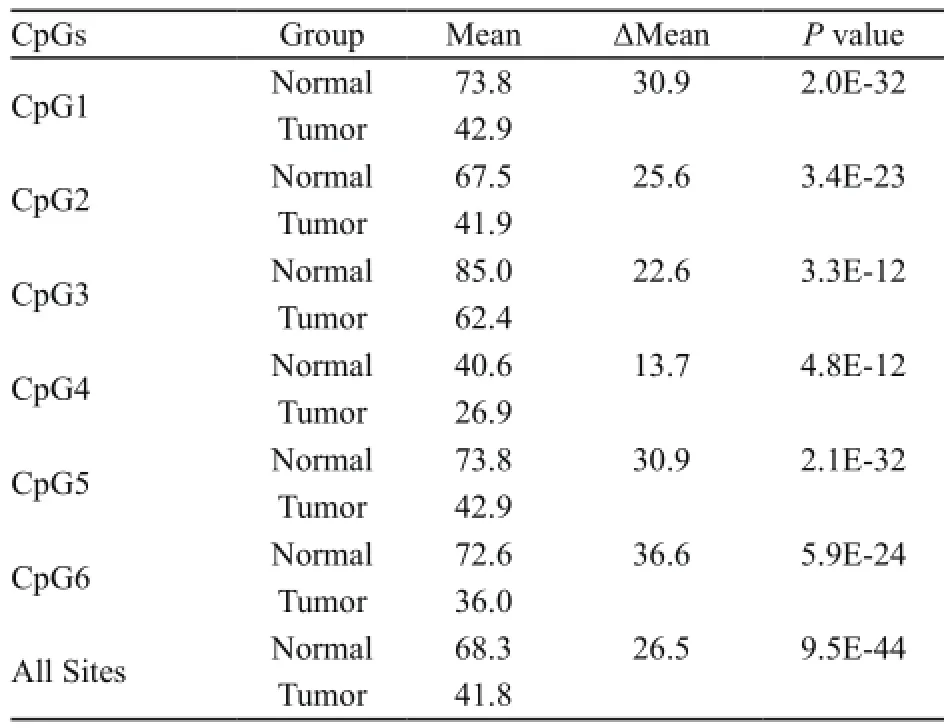
表1 RBFOX1基因启动子区各CpG位点的甲基化水平Tab. 1 Methylation status (%) of the detected sites of RBFOX1 promoter
2.2 RBFOX1基因在癌组织中的表达
为探究RBFOX1基因在食管鳞癌中的表达情况,本研究从149例配对的癌组织和癌旁组织中选取了42例配对的组织进行mRNA表达分析。结果发现相对于癌旁组织,癌组织中RBFOX1基因表达显著降低(约为癌旁组织中表达量的19%),两者差异有统计学意义(P<0.000 1,图2B)。
2.3 RBFOX1启动子甲基化与临床病理参数的关系
Δmean定义为RBFOX1在癌旁组织和癌组织中甲基化水平的差值,分析Δmean与性别、年龄、吸烟、饮酒、神经侵犯、血管侵犯、淋巴结转移、分期、分化和病灶位置的相关性。结果显示所分析的启动子区域RBFOX1所有位点的平均甲基化水平差异与上述临床病理参数无明显相关(P>0.05,表2),对各个位点分析发现CpG6位点的甲基化水平差异与淋巴结转移呈临界负相关(P=0.05,r=-0.16);发生淋巴结转移的癌组织甲基化水平差异较大(36.9% vs 26.3%);其他位点无明显相关。
2.4 RBFOX1启动子甲基化与预后的关系
参照文献中的方法[16],在本研究中设定癌组织中甲基化水平cut-off(阈值)=Mean(癌旁组织)-2.5SD(标准差),Mean(癌旁组织)=68.3%, SD=0.139,cut-off=33.6%。依据cut-off值将研究对象分为两组,低于阈值的一组定义为组1(Group 1);高于阈值的为组2(Group 2)。两组1、3和5年的总生存率(overall survival,OS)分别为90.7% vs 79.7%、66.1% vs 50.6%和57.0% vs 35.7%,差异无统计学意义(P=0.06);无进展生存率(progression-free survival,PFS)分别为81.9% vs 70.4%、56.9% vs 36.8%和48.7% vs 28.9%,差异有统计学意义(P=0.03)。单因素分析显示TNM分期是影响总体生存的因子;TNM分期、血管侵犯和甲基化水平是影响无进展生存的因子(表3);将单个危险因素纳入Cox回归进行多因素分析,发现TNM分期是总体生存和无进展生存的独立预测因子,甲基化水平不是总体生存和无进展生存的独立预测因子(P=0.316,P=0.121;表3)。

图2 RBFOX1基因CpG位点甲基化水平及RBFOX1基因的相对表达量Fig. 2 Methylation level at CpG sites and relative expression of RBFOX1 gene.

表2 RBFOX1甲基化水平与临床病理特征的关系Tab. 2 Association between methylation difference (%) of RBFOX1 and clinicopathological parameters

图3 RBFOX1基因可能的靶点和通路

表3 总体生存率和无进展生存的单因素和多因素分析Tab. 3 Univariate and multivariate analysis of OS and PFS
3 讨 论
已发现许多疾病与mRNA前体选择性剪接有关。选择性剪接更是以多种方式参与肿瘤的发生:选择性剪接影响癌基因的活化[17];选择性剪接导致肿瘤抑制基因的失活;选择性剪接还参与肿瘤转移和浸润[18]。研究选择性剪接基因可使人们更好地理解基因转录表达过程中的多样化选择,并为肿瘤的病因学研究提供新的思路。
RBFOX1基因的表达情况在肿瘤研究中有所报道。有研究表明在结肠癌中RBFOX1基因存在缺失突变,RBFOX1基因在癌组织中的表达水平明显低于癌旁组织中的表达水平[14];在卵巢癌和乳腺癌中RBFOX1基因的mRNA水平仍然低于癌旁组织中的水平[19],这些研究在一定程度上支持本研究的发现:RBFOX1基因在食管鳞癌中的表达水平比癌旁组织中的表达水平显著降低。基因的表达受多种因素的影响,基因缺失突变和基因启动子异常甲基化都是其中的影响因素。基因启动子的高甲基化往往导致转录沉默进而降低基因的表达,两者常呈负相关[20-21]。本研究中RBFOX1基因在癌组织中甲基化水平和表达量均低于癌旁组织,造成这种现象可能的原因有:①鳞癌中存在RBFOX1基因缺失突变,相对于缺失突变,甲基化可能不是影响RBFOX1基因表达的最关键的因素;②本研究中6个CpG位点对RBFOX1基因的表达影响较小。有观点认为,甲基化CpG位点的密度会影响甲基化对基因表达的作用[22]。有研究报道,基因甲基化与表达之间的关系,所选定的区域分布有25个CpG位点[23]。③RBFOX1基因的表达与细胞特异性有关。当然在食管鳞癌中是不是存在RBFOX1基因缺失突变还需要测序分析来证实。
由于RBFOX1基因调节的基因和靶点较多[11-12,24],可以看出,RBFOX1基因既参与肿瘤生成过程也参与抑制肿瘤的过程。目前尚不明确RBFOX1基因在食管鳞癌中的特异性靶点,所以需要进一步的实验来探究RBFOX1对食管鳞癌发生、发展的影响,以及RBFOX1基因启动子低甲基化对食管鳞癌功能的影响。
基因的异常甲基化多与临床病理的特征相关,在食管鳞癌中有研究表明基因异常甲基化与分化、肿瘤侵犯和淋巴结转移等相关[7-8,25]。在本研究所分析的6个CpG位点中,CpG6位点甲基化水平差异与淋巴结转移呈负临界相关,甲基化水平差异较大的肿瘤易发生淋巴结转移,这与异常甲基化促进恶性表型发生的研究结果相一致[16]。本研究中RBFOX1基因其他位点的异常甲基化与各临床特征无明显相关性,可能的原因与所选取的分析区域有关;也可能与RBFOX1基因对于食管鳞癌分化、淋巴结转移等影响较小有关;还可能是研究的样本量的影响。比较了不同甲基化水平下患者的预后:在单因素分析中,甲基化水平高于33.6%的患者5年生存率和无进展生存率低于甲基化水平低于33.6%的患者;但在多因素分析中,仅TNM分期是总体生存和是无进展生存率的独立预测因子,所分析的启动子区域的甲基化水平不能作为生存分析的预测因子。
总之,本研究结果表明,在食管鳞癌中RBFOX1基因甲基化水平和表达水平都低于正常食管组织,所分析的启动子区域chr16:6014214-6014616([GRCh37/hg19])的甲基化水平不能作为预后的独立预测因子。
[1] 陈万青, 张思维, 郑荣寿, 等. 中国肿瘤登记地区2007年肿瘤发病和死亡分析[J]. 中国肿瘤, 2011, 20(3): 162-169.
[2] 赵金璇, 王芳, 徐峥嵘, 等. 表观遗传调控pre-mRNA的选择性剪接[J]. 遗传, 2014, 36(3): 248-255.
[3] PAN Q, SHAI O, LEE L J, et al. Deep surveying of alternative splicing complexity in the human transcriptome by highthroughput sequencing[J]. Nat Genet, 2008, 40(12): 1413-1415.
[4] COOPER T A, WAN L, DREYFUSS G. RNA and disease[J]. Cell, 2009, 136(4): 777-793.
[5] ZHU N, GU L, FINDLEY H W, et al. An alternatively spliced survivin variant is positively regulated by p53 and sensitizes leukemia cells to chemotherapy[J]. Oncogene, 2004, 23(45): 7545-7551.
[6] LEWANDOWSKA J, BARTOSZEK A. DNA methylation in cancer development, diagnosis and therapy--multiple opportunities for genotoxic agents to act as methylome disruptors or remediators[J]. Mutagenesis, 2011, 26(4): 475-487.
[7] LI R N, YU F J, WU C C, et al. Methylation status of retinoic acid receptor beta2 promoter and global DNA in esophageal squamous cell carcinoma[J]. J Surg Oncol, 2014, 109(6): 623-627.
[8] GUO W, ZHANG M, SHEN S, et al. Aberrant methylation and decreased expression of the TGF-beta/Smad target gene FBXO32 in esophageal squamous cell carcinoma[J]. Cancer, 2014, 120(16): 2412-2423.
[9] SHUKLA S, KAVAK E, GREGORY M, et al. CTCF-promoted RNA polymerase Ⅱpausing links DNA methylation to splicing[J]. Nature, 2011, 479(7371): 74-79.
[10] MAUNAKEA A K, CHEPELEV I, CUI K, et al. Intragenic DNA methylation modulates alternative splicing by recruiting MeCP2 to promote exon recognition[J]. Cell Res, 2013, 23(11): 1256-1269.
[11] ZHOU H L, BARANIAK A P, LOU H. Role for Fox-1/Fox-2 in mediating the neuronal pathway of calcitonin/calcitonin gene-related peptide alternative RNA processing[J]. Mol Cell Biol, 2007, 27(3): 830-841.
[12] BARANIAK A P, CHEN J R, GARCIA-BLANCO M A. Fox-2 mediates epithelial cell-specific fibroblast growth factor receptor 2 exon choice[J]. Mol Cell Biol, 2006, 26(4): 1209-1222.
[13] VENABLES J P, KLINCK R, KOH C, et al. Cancer-associated regulation of alternative splicing[J]. Nat Struct Mol Biol, 2009, 16(6): 670-676.
[14] SENGUPTA N, YAU C, SAKTHIANANDESWAREN A, et al. Analysis of colorectal cancers in British Bangladeshi identifies early onset, frequent mucinous histotype and a high prevalence of RBFOX1 deletion[J]. Mol Cancer, 2013, 12: 1.
[15] CHEUNG H C, BAGGERLY K A, TSAVACHIDIS S, et al. Global analysis of aberrant pre-mRNA splicing in glioblastoma using exon expression arrays[J]. BMC Genomics, 2008, 9: 216.
[16] CASTELO-BRANCO P, CHOUFANI S, MACK S, et al. Methylation of the TERT promoter and risk stratification of childhood brain tumours: an integrative genomic and molecular study[J]. Lancet Oncol, 2013, 14(6): 534-542.
[17] LUKAS J, GAO D Q, KESHMESHIAN M, et al. Alternative and aberrant messenger RNA splicing of the mdm2 oncogene in invasive breast cancer[J]. Cancer Res, 2001, 61(7): 3212-3219.
[18] LEE J H, SEO Y W, PARK S R, et al. Expression of a splice variant of KAI1, a tumor metastasis suppressor gene, influences tumor invasion and progression[J]. Cancer Res, 2003, 63(21): 7247-7255.
[19] GE X, YAMAMOTO S, TSUTSUMI S, et al. Interpreting expression profiles of cancers by genome-wide survey of breadth of expression in normal tissues[J]. Genomics, 2005, 86(2): 127-141.
[20] SOEJIMA H, NAKAGAWACHI T, ZHAO W, et al. Silencing of imprinted CDKN1C gene expression is associated with loss of CpG and histone H3 lysine 9 methylation at DMR-LIT1 in esophageal cancer[J]. Oncogene, 2004, 23(25): 4380-4388.
[21] YANG X, HAN H, DE CARVALHO D D, et al. Gene body methylation can alter gene expression and is a therapeutic target in cancer[J]. Cancer Cell, 2014, 26(4): 577-590.
[22] WU C, MORRIS J R. Genes, genetics, and epigenetics: a correspondence[J]. Science, 2001, 293(5532): 1103-1105.
[23] CASTELO-BRANCO P, CHOUFANI S, MACK S, et al. Methylation of the TERT promoter and risk stratification of childhood brain tumours: an integrative genomic and molecular study[J]. Lancet Oncol, 2013, 14(6): 534-542.
[24] JIN Y, SUZUKI H, MAEGAWA S, et al. A vertebrate RNA-binding protein Fox-1 regulates tissue-specific splicing via the pentanucleotide GCAUG[J]. EMBO J, 2003, 22(4): 905-912.
[25] 缪珑昇, 相加庆, 张亚伟, 等. 食管鳞癌中hMLH1、E-cadherin、p16INK4a基因启动子甲基化及其意义[J].中国癌症杂志, 2009, 19(5): 340-346.
Research of the alternative splicing gene RBFOX1 in esophageal squamous cell carcinoma
DENG Jiaying, ZHAO Kuaile (Department of Radiation Oncology, Fudan University Shanghai Cancer Center, Department of Oncology, Shanghai Medical College, Fudan University, Shanghai 200032, China)
ZHAO Kuaile E-mail: kuaile_z@sina.com
Background and purpose:Alternative splicing is an important regulation mechanism of gene expression. Aberrant alternative splicing is associated with dysregulation of the cell cycle, activation of oncogenes and inactivation of the tumor suppressor genes. Thus, it is closely correlated with the pathogenesis and progression of various tumors. DNA methylation is an important part of epigenetic phenomena. Aberrant methylation of the gene promoter can result in gene silencing. Hypermethylation of tumor suppressor genes and DNA repair genes correlates with the onset of many different cancers. Additionally, DNA methylation acts as a pivotal factor for alternative splicing. Aberrant methylation disrupts the stabilization of the alternative splicing. This study investigated the promoter methylation and expression of RNA binding protein, fox-1 homolog 1 (RBFOX1) gene in esophageal squamous cell carcinoma (ESCC), and to elucidate its role in ESCC.Methods:MassARRAY approach and RT-PCR were used respectively to examine the methylation level of RBFOX1 gene and its expression at mRNA level in tumors and corresponding adjacent normal tissues. The correlation between methylation level and clinicopathological features was analyzed.Results:RBFOX1 methylation level and mRNA expression in tumor tissues were significantly lower than those in corresponding adjacent normal tissues (41.8% vs 68.3%, P<0.01). No significant correlation was observed between methylation level and clinicopathological features. The cut-off (33.6%) was calculated as the mean of the normal samples to which we applied 2.5 SD. According to the cut-off value, the object of the study was divided into two groups. The methylation level lower than the cut-off was defined as group 1; methylation level higher than the cut-off was defined as group 2. The 5-year overall survival rates of the two groups were 57.0% and 35.7%, respectively (P=0.06); 5-year progression-free survival rates were 48.7% and 28.9%, respectively (P=0.03). However, the multivariate analysis results indicated that TNM stage was the independent factor of prognosis.Conclusion:The methylation level and mRNA expression of RBFOX1 in tumor specimens are significantly lower than those in corresponding adjacent normal tissues. The methylation level of the RBFOX1 promoter is not an independent factor of prognosis.
Esophageal squamous cell carcinoma; Alternative splicing; Methylation; Expression; RBFOX1
10.3969/j.issn.1007-3969.2015.06.001
R735.1
A
1007-3639(2015)06-0401-08
2015-02-06
2015-04-15)
国家自然科学基金资助项目(21172043)。
赵快乐 E-mail:kuaile_z@sina.com

