CRISPR/Cas9的应用及脱靶效应研究进展
郑武,谷峰温州医科大学附属眼视光医院,眼视光学和视觉科学国家重点实验室培育基地,卫生部视觉科学研究重点实验室,温州 325027
CRISPR/Cas9的应用及脱靶效应研究进展
郑武,谷峰
温州医科大学附属眼视光医院,眼视光学和视觉科学国家重点实验室培育基地,卫生部视觉科学研究重点实验室,温州 325027
CRISPR/Cas9基因编辑技术在生命科学领域掀起了一场全新的技术革命,该技术可以对基因组特定位点进行靶向编辑,包括缺失、插入、修复等。CRISPR/Cas9比锌指核酸酶 (ZFNs)和转录激活因子样效应物核酸酶(TALENs)技术更易于操作,而且更高效。CRISPR/Cas9系统中的向导RNA(Single guide RNA, sgRNA)是一段与目标DNA片段匹配的RNA序列,指导Cas9蛋白对基因组进行识别。研究发现,设计的sgRNA会与非靶点DNA序列错配,引入非预期的基因突变,即脱靶效应(Off-target effects)。脱靶效应严重制约了 CRISPR/Cas9基因编辑技术的广泛应用。为了避免脱靶效应,研究者对影响脱靶效应的因素进行了系统研究并提出了许多降低脱靶效应的方法。文章总结了 CRISPR/Cas9系统的应用及脱靶效应研究进展,以期为相关领域的工作提供参考。
基因组编辑;CRISPR/Cas9;脱靶效应;向导RNA
CRISPR/Cas9(Clustered regulatory interspaced short palindromic repeat/Cas9)系统是目前基因组编辑领域最受欢迎的新方法。CRISPR/Cas9来源于简单的细菌免疫系统,经过人为改造后,可在真核细胞中实现高度灵活且特异的基因组编辑(图1)。该系统通过设计特异性向导 RNA(Single guide RNA, sgRNA)序列与靶序列进行碱基配对,从而引导Cas9蛋白结合到靶序列处,行使DNA切割功能,然后利用细胞的非同源性末端连接(Non-homologous end joining, NHEJ)或同源重组(Homologous recombination, HR)修复机制对断裂的DNA进行插入缺失(Indel)、修复(Repair)或替换(Replacement)[1]。
1 CRISPR/Cas9系统概述
CRISPR /Cas9系统是一种后天免疫防御系统,用以保护细菌或古细菌免受外来质粒或噬菌体的侵入。这类细菌或古细菌基因组的 CRISPR序列能表达与入侵者基因组序列相识别的RNA。当噬菌体等入侵时,该防御系统的 CRISPR序列会表达这类RNA并通过互补序列结合识别入侵者的基因组序列,然后CRISPR相关酶(Cas)在序列识别处切割外源基因组DNA,达到抵制入侵的目的。
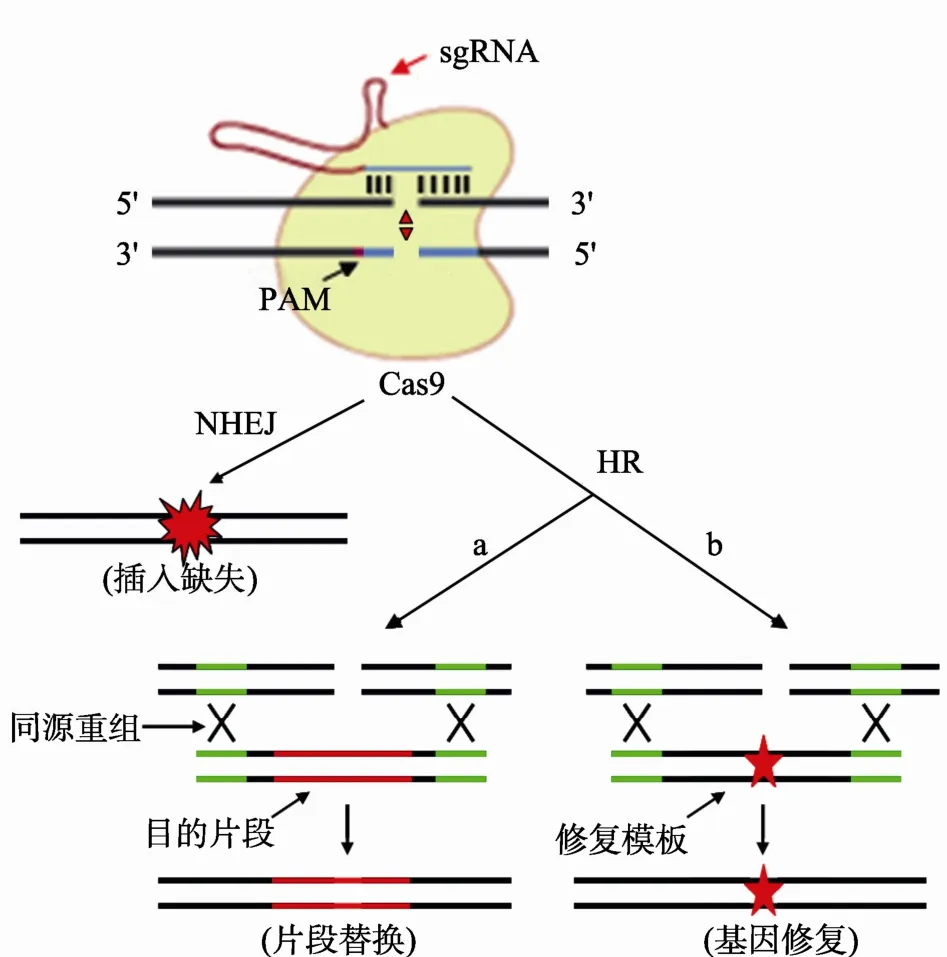
图1 CRISPR/Cas9系统介导基因编辑示意图
1.1 CRISPR/Cas9系统发展史
CRISPR/Cas9系统的发现可以追溯到1987年,研究者在大肠杆菌的基因组中首次发现了这一奇特的重复间隔序列[2],但当时并未引起很大的反响。随后,在其他细菌和古细菌中也发现了这一特殊的序列[3]。2005年,研究者发现这些间隔序列和噬菌体的基因序列匹配度很高,暗示着 CRISPR可能参与了微生物的免疫防御[4~6],之后有研究者提出了一种假设机制:CRISPR通过一种类似于真核生物的RNAi机制来行使免疫功能[7]。直到 2011年,才揭示了CRISPR/Cas系统的分子机制:当病毒首次入侵时,细菌会将外源基因的一段序列整合到自身的CRISPR的间隔区;病毒二次入侵时,CRISPR转录生成crRNA前体(pre-crRNA),pre-crRNA经过加工形成含有与外源基因匹配序列的crRNA,该crRNA与病毒基因组的同源序列识别后,介导 Cas蛋白结合并切割,从而保护自身免受入侵[8~10]。2013年,研究者报道了CRISPR/Cas9系统可高效地编辑基因组。其中分别在人类细胞和小鼠细胞中成功对部分基因实现了编辑[11,12]。至此开始,CRISPR/Cas9系统在生命医学研究领域掀起了一场革命。
1.2 CRISPR/Cas9系统的应用
在医学领域,利用CRISPR/Cas9技术,有多项革新式的报道。Fah基因突变会导致遗传性酪氨酸血症,研究者利用CRISPR/Cas9在肝细胞中成功修复了该基因,减缓了疾病小鼠体重下降的临床表现[13]。这一研究证明了该技术在人类遗传病治疗上的前景。此外,将sgRNA和Cas9注射到带有白内障致病基因CRYGC突变的小鼠受精卵中,发现有1/3小鼠的白内障得到了治愈[14]。在此基础上,以精原干细胞为材料进行基因修复,随后也得到基因修复的小鼠[15]。研究者利用CRISPR/Cas9基因修复技术,首次成功地在感染有 HIV-1病毒的细胞中灭活HIV-1病毒基因,该研究为HIV的治疗探索了新的临床治疗方法[16]。
肿瘤细胞基因组测序可以发现大量与癌症相关的候选基因。但是,要从大量的候选基因中筛选到真正与肿瘤发生、发展相关的基因,却是一项艰巨的工程。而利用CRISPR/Cas9系统,在小鼠体内实现了这些特定基因的敲出。相对于过去通过逐个敲除小鼠特定基因的方法来说,该方法大大加速了研究的进程[17]。CRISPR/Cas9技术在规模化研究基因功能等方面正发挥着重要作用[18]。笔者所在团队利用AAV-CRISPR/Cas9对人类胚胎干细胞的基因组进行了定点编辑,将RFP基因成功插入到视网膜光感受器特异表达的基因NRL位点,该基因敲入的人类胚胎干细胞系将对干细胞介导的人类视网膜疾病的治疗具有重要意义(文章待发表)。
在动物科学领域,CRISPR/Cas9技术已经成功对小鼠(Mus musculus)、大鼠(Rattus norvegicus)以及秀丽线虫(Caenorhabditis elegans)等基因组进行编辑[19~22],需要指出的是利用CRISPR/Cas9技术成功对食蟹猴(Macaca fascicularis)基因组的编辑,并培育出个体[23]。这一工作打破了长期以来无法实现高效地对灵长类生物基因组进行修饰的桎梏,为脑科学的进一步发展奠定了重要基础。利用CRISPR/Cas9系统,也可以实现约氏疟原虫(Plasmodium yoelii)基因组的编辑[24]。
在植物科学领域,CRISPR/Cas9系统也已成功应用到拟南芥(A. thaliana)、烟草(Nicotiana tabacum L.)和水稻(O. sativa)等植物中[25, 26]。对水稻基因组的定向编辑,通过后续的全基因组重测序及检测与靶序列高度同源的序列,仅在只有一个碱基不同的潜在脱靶位点检测到了脱靶效应。该研究为作物品种改良提供了新的思路[27]。
在微生物领域,利用该技术对酿酒酵母(Saccharomyces cerevisiae)的基因组进行编辑[28]。另外,发现CRISPR/Cas9系统还与微生物的耐药性相关[29]。
2 CRISPR/Cas9系统的脱靶效应
CRISPR/Cas9系统虽然能够高效快速地进行基因编辑,为基础研究提供了强大的平台。但是随着研究的深入,人们发现该技术的重要缺点是脱靶效应。因此CRISPR/Cas9系统脱靶的影响因素和如何解决成为该技术研究的热点。
CRISPR/Cas9基因编辑系统的特异性决定于sgRNA上的识别序列(~20nt)。然而,在复杂的生物基因组中,sgRNA的识别序列可能会与非靶点DNA发生局部匹配(Partial match)。这种局部匹配,目前可归结为两类:(1) sgRNA与脱靶位点DNA序列长度相同,但存在碱基错配。针对U2OS.EGFP细胞的VEGFA基因的3个不同位点以及EMX1、FANCF和RNF2基因的一个位点,共设计了6条sgRNA,经筛选检测后发现,除了FANCF和RNF2基因外,其他对应的sgRNA均导致不同数量的脱靶效应[30]。另外,通过体外筛选与高通量测序相结合的方法来检测CRISPR/Cas9系统在HEK293T细胞的脱靶情况,发现Cas9系统在4个靶位点切割中在基因组其他位置有5个脱靶位点[31]。(2) 脱靶位点 DNA 序列长度比sgRNA多或少几个碱基,通过形成DNA凸起(DNA bulge) 或RNA凸起(RNA bulge)来完成其他碱基的正确配对。sgRNA的识别序列与脱靶位点DNA序列长度相差5 bp时,仍能通过形成多个 bulge来进行配对并介导CRISPR/Cas9系统进行切割[32]。为降低CRISPR/Cas9系统的脱靶效应,研究者对脱靶的影响因素进行了探讨,并提出了一系列弱化或消除脱靶效应的对策。
2.1 CRISPR/Cas9系统脱靶效应的影响因素
2.1.1 前间区序列邻近基序(Protospacer adjacent motif, PAM)
PAM序列区是CRISPR/Cas9系统行使切割功能的基本条件。如果靶序列3’端没有PAM序列,即使靶序列与sgRNA序列完全匹配,Cas9蛋白也不会切割该序列位点[33]。PAM 序列主要影响 CRISPR/Cas9 的 DNA切割效率。有研究表明,在细胞水平上,NGG(既定PAM序列,canonical PAMs)介导的切割效率是最高的,而其他的15个非NGG序列(非既定PAM序列,non-canonical PAMs)中,NAG的切割效率要高于NGA。然而笔者所在团队利用绿色荧光报告系统,系统地研究了 PAM序列对切割效率的影响。研究发现,在HEK-293细胞中,NGA的切割效率在部分位点反而高于NAG[34]。另外,基于非既定 PAM的双切口酶在CRISPR/Cas9介导的DNA切割效率仍然很高(文章待发表)。这些都提示,研究者需要更多地关注由于非既定 PAM 序列所导致CRISPR/ Cas9的脱靶。
2.1.2 向导RNA(Single guide RNA, sgRNA)序列
sgRNA与目标基因组相结合的20nt序列区决定着 CRISPR/Cas系统的靶向特异性。但是有研究指出,CRISPR/Cas9与靶位点识别的特异性其实主要依赖于sgRNA与靠近PAM区的10~12 bp的碱基配对,而其余远离PAM序列8~10 bp碱基的错配对靶位点识别的影响并不明显[11,12,35]。目前研究结果均提示,可能靠近PAM的8~14 bp序列是决定特异性的关键,其他序列也均在不同程度上影响脱靶效应。
此外,sgRNA的长度也与特异性密切相关。有研究表明,sgRNA的20nt靶标序列缩短为17nt后,脱靶效应大幅度降低,但切割效率没有受到明显影响[36]。但是另有研究指出,在设计的 sgRNA的 5’端额外增加两个鸟嘌呤核苷酸后能够显著提高CRISPR/Cas9系统的特异性[37]。这两个观点看似相互矛盾,但实质上是从不同的角度来提高sgRNA的结合特异性。
2.1.3 细胞类型
研究人员发现在HeLa S3细胞中检测到的19个脱靶结合位点中,大多数位点都与在HEK293T细胞中检测到的脱靶结合位点不同,这提示Cas9系统的脱靶效应可能具有细胞特异性[38]。此外,DNA甲基化一直是限制ZFNs和TALENs应用的重要因素[39,40],而体外和体内实验表明 CRISPR/Cas9系统介导的DNA切割功能并不受甲基化的影响[41]。
2.2 CRISPR/Cas9系统脱靶效应的检测
脱靶效应造成了研究中的许多不确定性,这无疑限制了该技术的应用。因此,研究者希望能开发有效的方法来检测脱靶效应。染色体免疫共沉淀和高通量测序相结合(ChIP-seq),可以对CRISPR/ Cas9的可能脱靶位点进行分析,这可以较好预测脱靶位点[38,42]。另外,还有一种方法为 GUIDE-seq,其原理是利用一种短的双链寡聚核苷酸标记CRISPR/Cas诱导的脱靶断裂,然后对标签所在的基因组区域进行测序,从而确定脱靶位点。利用该技术可以在基因组范围内检测 CRISPR脱靶效应,从而改变了以往先预测假定脱靶位点再检测的思路;而且,与ChIP-seq相比较,GUIDE-seq更精确、更灵敏[43]。也有报道,利用整合酶缺陷的慢病毒载体(IDLV)来检测CRISPR/Cas9和TALENs对基因组造成的脱靶效应,利用IDLV捕获切割位点[44]。最近,一种名为Digenome-seq的技术也被用于检测脱靶,其原理是利用Cas9核酸酶对人类基因组DNA进行消化,然后进行全基因组测序。这种体外消化会根据脱靶情况产生不同的特定模式,然后利用计算机进行模式分析来检测脱靶效应[45]。
2.3 降低CRISPR/Cas9系统脱靶的策略
2.3.1 提高sgRNA序列设计的特异性
只要保证sgRNA序列的特异性,就可以大大降低脱靶的发生。目前,在设计sgRNA时需要遵守以下标准[41]:(1)尽量减少 sgRNA与预测的脱靶位点序列的配对碱基数;(2)设计的sgRNA与预测的脱靶位点在靠近 PAM 区至少有 2个碱基不配对;(3)sgRNA与预测的脱靶位点序列要避免有连续的或间隔的4个碱基配对。
为了使sgRNA的设计更加便捷,科学家们又研究出了专门用于sgRNA设计的软件。用户在使用的过程中,只要根据自己的需要来限定相关的参数即可迅速获得符合要求的 sgRNA。第一款在线的sgRNA设计软件:http://www.genome-engineering.org/;此后,其他研究者也陆续推出了一系列设计软件,并对参数和界面等进行了优化,如 CasOT,CHOPCHOP等[46,47]。
2.3.2 采用双切口措施(Double-nicking strategy)策略
为降低CRISPR/Cas9系统的脱靶效应,科学家们从 Cas9蛋白改造入手,得到了其突变型 D10A Cas9切口酶(Cas9n)(图2)[48,49]。野生型的Cas9蛋白有两个内切酶活性结构域,Cas9n的其中一个结构域被失活,因此Cas9n只能切割DNA单链,产生一个切口(Nick)。这一改造的系统中,成对sgRNA的设计是最为关键的,两条sgRNA结合不同的DNA链,其 PAM 序列要以尾对尾的形式出现,两个sgRNA结合的位点之间的距离要适中。Cas9n会在每个sgRNA结合的地方造成单链缺口,两个相邻的单链切口会形成一个DNA双链断裂(DSB),DSB的形成会激起细胞以非同源末端连接(NHEJ)的方式进行修复,进而引入插入、缺失或者突变。而在脱靶位点处,单个sgRNA只能造成单链缺口,无法形成DSB,只能通过同源模板修复方式精确修复该区域。利用改造过的CRISPR/Cas9系统,在不降低基因切割效率的前提下,可大幅度降低CRISPR/Cas9系统在小鼠基因组上的脱靶效应。该方法也被其他研究组验证[50]。另有研究指出,将双切口法和适当缩短sgRNA的靶标序列相结合,能够进一步降低脱靶效应[36]。2.3.3 fCas9系统策略(FokⅠ-dCas9)
将 Cas9改造为失去催化活性的 Cas(dCas9)(图3);然后将dCas9与FokⅠ核酸酶结合形成融合蛋白(FokⅠ-dCas9),只有当两个融合蛋白单体彼此靠近形成二聚体时才能行使切割功能。在应用中,需要针对靶点序列的两条链设计1对sgRNA序列,两条sgRNA会引导两个FokⅠ-dCas9蛋白复合体结合到靶标位点并形成二聚体。这一策略大幅度降低了非靶向位点的DNA切割[51,52]。
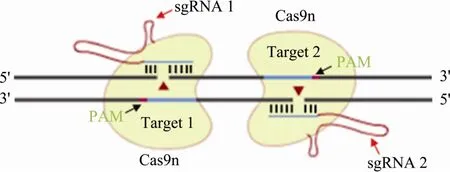
图2 双切口酶策略示意图
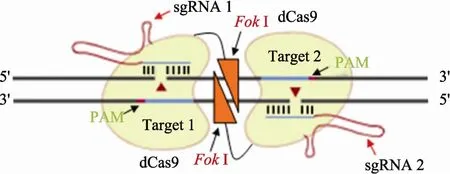
图3 fCas9策略示意图
3 展 望
CRISPR/Cas9系统被称为第三代基因编辑技术。相比于ZFNs和TALENs系统,它有着无可比拟的优点。首先,CRISPR/Cas9系统的可操作性更强。理论上基因组中每8个碱基就能找到一个可以用CRISPR/Cas9进行编辑的位置(PAM序列),几乎可以对所有基因进行编辑[11];其次,CRISPR/Cas9系统更具有可控性。例如可以通过对Cas9蛋白的修饰,让它不切断DNA双链,而只是切开单链,这样可以大大降低切开双链后带来的 NHEJ造成的染色体变异风险;第三,CRISPR/Cas9系统的使用极为方便,只需要简单的几步就能完成,几乎任何分子生物学实验室都可以开展。正是这些优势,使其成为科学界的新宠。在CRISPR/Cas9的基础上,衍生出了许多新的技术。例如:CRISPRi可通过对位点的识别来可逆地阻止转录延伸、RNA聚合酶的结合或转录因子的结合[53];一种名为 CRISPRm的技术则应用于促进生物分子的定向改造[54]。此外,对CRISPR系统进行改造,研究者可以在活细胞中有效激活任何基因。这一改造促进了人类对整个基因组的功能筛选以及特定疾病的基因鉴定[55]。
从CRISPR/Cas9技术的研究进程来看,解决脱靶效应的策略有以下几点:(1)改良sgRNA的结构。设计特异性的sgRNA是解决脱靶的最直接的手段。(2)改造Cas9蛋白。来自麻省理工学院的科学家们发现来自于金黄色酿脓葡萄球菌(Staphylococcus aureus)的Cas9蛋白(SaCas9)也具有良好的基因编辑功能,且体外实验表明其具有很好的特异性[56]。目前对该蛋白的认识还处于发展阶段,相信随着对其结构和功能研究的不断深入,科学家们可以找到特异性更强的 Cas蛋白。(3)研究其他类型的 CRISRP/Cas系统。CRISPR/Cas9属于Ⅱ型,是目前最为简便、最易操作的系统,而其他 2种类型还知之甚少,也许随着认识的加深,研究者可能找到更为特异的系统来代替CRISPR/Cas9。
目前,科学家们利用CRISPR/Cas9技术正在对不同的细胞和物种进行基因组改造。近期,中山大学的研究者首次对人类胚胎的基因组进行编辑,引起广泛的伦理讨论[57]。CRISPR/Cas9的脱靶效应并没有得到完美的解决,因此许多人担心该技术一旦过早地应用于人类非治疗性的基因编辑,可能会造成无法预测的后果。美国再生医学联盟主席Edward Lanphier等建议研究者目前不要对人类生殖细胞/胚胎进行基因编辑[58]。而有些人则认为该技术在人类生殖细胞的应用研究有利于遗传病的基因治疗,总体来讲是利大于弊。因此,这就要求相关监管部门制定切实可行的法律法规,在不影响该技术发展进步的基础上,严格监控其应用的范围和程度,杜绝出现违规滥用现象。
[1] Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096.
[2] Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol, 1987, 169(12): 5429–5433.
[3] Mojica FJ, Díez-Villaseñor C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. Mol Microbiol, 2000, 36(1): 244–246.
[4] Bolotin A, Quinquis B, Sorokin A, Ehrlich SD. Clusteredregularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(Pt 8): 2551–2561.
[5] Mojica FJM, Díez-Villaseñor C, García-Martínez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol, 2005, 60(2): 174–182.
[6] Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005, 151(Pt 3): 653–663.
[7] Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biol Direct, 2006, 1: 7.
[8] Makarova KS, Aravind L, Wolf YI, Koonin EV. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biol Direct, 2011, 6: 38.
[9] Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nat Rev Microbiol, 2011, 9(6): 467–477.
[10] Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao YJ, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature, 2011, 471(7340): 602–607. [11] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819–823.
[12] Jiang WY, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(3): 233–239.
[13] Yin H, Xue W, Chen SD, Bogorad RL, Benedetti E, Grompe M, Koteliansky V, Sharp PA, Jacks T, Anderson DG. Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype. Nat Biotechnol, 2014, 32(6): 551–553.
[14] Wu YX, Liang D, Wang YH, Bai MZ, Tang W, Bao SM, Yan ZQ, Li DS, Li JS. Correction of a genetic disease in mouse via use of CRISPR-Cas9. Cell Stem Cell, 2013, 13(6): 659–662.
[15] Wu YX, Zhou H, Fan XY, Zhang Y, Zhang M, Wang YH, Xie ZF, Bai MZ, Yin Q, Liang D, Tang W, Liao JY, Zhou CK, Liu WJ, Zhu P, Guo HS, Pan H, Wu CL, Shi HJ, Wu LG, Tang FC, Li JS. Correction of a genetic disease by CRISPR-Cas9-mediated gene editing in mouse spermatogonial stem cells. Cell Res, 2015, 25(1): 67–79.
[16] Hu WH, Kaminski R, Yang F, Zhang YG, Cosentino L, Li F, Luo B, Alvarez-Carbonell D, Garcia-Mesa Y, Karn J, Mo XM, Khalili K. RNA-directed gene editing specifically eradicates latent and prevents new HIV-1 infection. Proc Natl Acad Sci USA, 2014, 111(31): 11461–11466.
[17] Sánchez-Rivera FJ, Papagiannakopoulos T, Romero R, Tammela T, Bauer MR, Bhutkar A, Joshi NS, Subbaraj L, Bronson RT, Xue W, Jacks T. Rapid modelling of cooperating genetic events in cancer through somatic genome editing. Nature, 2014, 516(7531): 428–431.
[18] Findlay GM, Boyle EA, Hause RJ, Klein JC, Shendure J. Saturation editing of genomic regions by multiplex homology-directed repair. Nature, 2014, 513(7516): 120–123. [19] Qiu ZW, Liu MZ, Chen ZH, Shao YJ, Pan HJ, Wei GG, Yu C, Zhang L, Li X, Wang P, Fan HY, Du B, Liu B, Liu MY, Li DL. High-efficiency and heritable gene targeting in mouse by transcription activator-like effector nucleases. Nucleic Acids Res, 2013, 41(11): e120.
[20] Li DL, Qiu ZW, Shao YJ, Chen YT, Guan YT, Liu MZ, Li YM, Gao N, Wang LR, Lu XL, Zhao YX, Liu MY. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 681–683.
[21] Hu XL, Chang NN, Wang XL, Zhou FY, Zhou XH, Zhu XJ, Xiong JW. Heritable gene-targeting with gRNA/Cas9 in rats. Cell Res, 2013, 23(11): 1322–1325.
[22] Friedland AE, Tzur YB, Esvelt KM, Colaiácovo MP, Church GM, Calarco JA. Heritable genome editing in C. elegans via a CRISPR-Cas9 system. Nat Methods, 2013, 10(8): 741–743.
[23] Niu YY, Shen B, Cui YQ, Chen YC, Wang JY, Wang L, Kang Y, Zhao XY, Si W, Li W, Xiang AP, Zhou JK, Guo XJ, Bi Y, Si CY, Hu B, Dong GY, Wang H, Zhou ZM, Li TQ, Tan T, Pu XQ, Wang F, Ji SH, Zhou Q, Huang XX, Ji WZ, Sha JH. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 2014, 156(4): 836–843.
[24] Zhang C, Xiao B, Jiang YY, Zhao YH, Li ZK, Gao H, Ling Y, Wei J, Li SN, Lu MK, Su XZ, Cui HT, Yuan J. Efficient Editing of Malaria Parasite Genome Using the CRISPR/ Cas9 System. MBio, 2014, 5(4): e01414–14.
[25] Li JF, Norville JE, Aach J, McCormack M, Zhang DD, Bush J, Church GM, Sheen J. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9.Nat Biotechnol, 2013, 31(8): 688–691.
[26] Shan QW, Wang YP, Li J, Zhang Y, Chen KL, Liang Z, Zhang K, Liu JX, Xi JJ, Qiu JL, Gao CX. Targeted genome modification of crop plants using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 686–688.
[27] Zhang H, Zhang JS, Wei PL, Zhang BT, Gou F, Feng ZY, Mao YF, Yang L, Zhang H, Xu NF, Zhu JK. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation. Plant Biotechnol J, 2014, 12(6): 797–807.
[28] DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Res, 2013, 41(7): 4336–4343.
[29] Sampson TR, Napier BA, Schroeder MR, Louwen R, Zhao J, Chin CY, Ratner HK, Llewellyn AC, Jones CL, Laroui H, Merlin D, Zhou P, Endtz HP, Weiss DS. A CRISPR-Cas system enhances envelope integrity mediating antibiotic resistance and inflammasome evasion. Proc Natl Acad Sci USA, 2014, 111(30): 11163–11168.
[30] Fu YF, Foden JA, Khayter C, Maeder ML, Reyon D, Joung JK, Sander JD. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol, 2013, 31(9): 822–826.
[31] Pattanayak V, Lin S, Guilinger JP, Ma E, Doudna JA, Liu DR. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol, 2013, 31(9): 839–843.
[32] Lin, YN, Cradick TJ, Brown MT, Deshmukh H, Ranjan P, Sarode H, Wile BM, Vertino PM, Stewart FJ, Bao G. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Res, 2014, 42(11): 7473–7485.
[33] Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 2014, 507(7490): 62–67.
[34] Zhang Y,L Ge XL, Yang FY, Zhang LP, Zheng JY, Tan XF, Jin ZB, Qu J, Gu F. Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells. Sci Rep, 2014, 4: 5405
[35] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816–821.
[36] Fu YF, Sander JD, Reyon D, Cascio VM, Joung JK. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol, 2014, 32(3): 279–284.
[37] Cho SW, Kim S, Kim Y, Kweon J, Kim HS, Bae S, Kim J-S. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res, 2014, 24(1): 132–141.
[38] Duan JZ, Lu GQ, Xie Z, Lou ML, Luo J, Guo L, Zhang Y. Genome-wide identification of CRISPR/Cas9 off-targets in human genome. Cell Res, 2014, 24(8): 1009–1012.
[39] Bultmann S, Morbitzer R, Schmidt CS, Thanisch K, Spada F, Elsaesser J, Lahaye T, Leonhardt H. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res, 2012, 40(12): 5368–5377.
[40] Valton J, Dupuy A, Daboussi F, Thomas S, Maréchal A, Macmaster R, Melliand K, Juillerat A, Duchateau P. Overcoming transcription activator-like effector (TALE) DNA binding domain sensitivity to cytosine methylation. J Biol Chem, 2012, 287(46): 38427–38432.
[41] Hsu PD, Scott DA, Weinstein JA, Ran FA, Konermann S, Agarwala V, Li YQ, Fine EJ, Wu XB, Shalem O, Cradick TJ, Marraffini LA, Bao G, Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013, 31(9): 827–832.
[42] Kuscu C, Arslan S, Singh R, Thorpe J, Adli M. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol, 2014, 32(7): 677–683.
[43] Tsai SQ, Zheng Z, Nguyen NT, Liebers M, Topkar VV, Thapar V, Wyvekens N, Khayter C, Iafrate AJ, Le LP, Aryee MJ, Joung JK. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol, 2015, 33(2): 187–197.
[44] Wang XL, Wang YB, Wu XW, Wang JH, Wang YJ, Qiu ZJ, Chang T, Huang H, Lin RJ, Yee J K. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol, 2015, 33(2): 175–178.
[45] Kim D, Bae S, Park J, Kim E, Kim S, Yu HR, Hwang J, Kim J I, Kim J S. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods, 2015, 12(3): 237–243.
[46] Montague TG, Cruz JM, Gagnon JA, Church GM, Valen E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Res, 2014, 42(W1): W401–W407.
[47] Xiao A, Cheng ZC, Kong L, Zhu ZY, Lin S, Gao G, Zhang B. CasOT: a genome-wide Cas9/gRNA off-target searching tool. Bioinformatics, 2014, 30(8): 1180–1182.
[48] Ran FA, Hsu PD, Lin C-Y, Gootenberg JS, Konermann S, Trevino AE, Scott DA, Inoue A, Matoba S, Zhang Y,Zhang F. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380–1389.
[49] Trevino AE, Zhang F. Chapter eight–Genome editing using Cas9 nickases. Methods Enzymol, 2014, 546: 161–174.
[50] Shen B, Zhang WS, Zhang J, Zhou JK, Wang JY, Chen L, Wang L, Hodgkins A, Iyer V, Huang XX, Skarnes WC. Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects. Nat Methods, 2014, 11(4): 399–402.
[51] Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nat Biotechnol, 2014, 32(6): 577–582.
[52] Tsai SQ, Wyvekens N, Khayter C, Foden JA, Thapar V, Reyon D, Goodwin MJ, Aryee MJ, Joung JK. Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing. Nat Biotechnol, 2014, 32(6): 569–576.
[53] Qi LS, Larson MH, Gilbert LA, Doudna JA, Weissman JS, Arkin AP, Lim WA. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173–1183.
[54] Ryan OW, Skerker JM, Maurer MJ, Li X, Tsai JC, Poddar S, Lee ME, DeLoache W, Dueber JE, Arkin AP, Cate JHD. Selection of chromosomal DNA libraries using a multiplex CRISPR system. eLife, 2014, 3: e03703.
[55] Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 2014, 517(7536): 583–588.
[56] Ran FA, Cong L, Yan WX, Scott DA, Gootenberg JS, Kriz AJ, Zetsche B, Shalem O, Wu X, Makarova KS, Koonin EV, Sharp PA, Zhang F. In vivo genome editing using Staphylococcus aureus Cas9. Nature, 2015, 520(7546): 186–191.
[57] Liang PP, Xu YW, Zhang XY, Ding CH, Huang R, Zhang Z, Lv J, Xie XW, Chen YX, Li YJ, Sun Y, Bai YF, Songyang Z, Ma WB, Zhou CQ, Huang JJ. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell, 2015, 6(5): 363–372.
[58] Lanphier E, Urnov F, Haecker SE, Werner M, Smolenski J. Don’t edit the human germ line. Nature, 2015, 519(7544): 410–411.
(责任编委: 张博)
Progress of application and off-target effects of CRISPR/Cas9
Wu Zheng, Feng Gu
State Key Laboratory Cultivating Base of Optometry and Visual Science, Key Laboratory of Vision Science, Ministry of Health, The Eye Hospital of Wenzhou Medical University, Wenzhou 325027 China
The clustered regulatory interspaced short palindromic repeat/Cas9 (CRISPR/Cas9) system mediates genome editing and is revolutionizing genetic researches. Scientists are able to manipulate the gene of interest from any organism with CRISPR/Cas9. Compared with zinc finger nucleases (ZFNs) and transcription activator-like effector nucleases (TALENs) technologies, the CRISPR/Cas9 technology provides an easy and efficient approach to manipulate the genome. In this system, sgRNA (Single guide RNA), a short RNA matching the targeted DNA fragment, guides the CRISPR/Cas9 to interrogate the genome. Because sgRNA can tolerate certain mismatches to the DNA targets and thereby promote undesired off-target mutagenesis, the key limit of this technology is off-target effects. To eliminate the off-target effects, different strategies have been adopted. In this review, we summarize the application of CRISPR/Cas9 and different strategies for addressing off-target effects.
genome editing; CRISPR/Cas9; off-target effects; sgRNA
2015-02-09;
2015-04-10
国家重点基础研究发展计划(973计划)项目(编号:2013CB967502),国家自然科学基金项目(编号:81201181/H1818),浙江省卫生厅省部共建项目(编号:WKJ2013-2-023)和温州医科大学人才启动项目(编号:QTJ 12011 和KYQD141106)资助
郑武,博士,助理研究员,研究方向:基因修复研究。E-mail: zheng123wu@163.com
谷峰,博士,研究员,研究方向:基因修复研究。 E-mail: gufenguw@gmail.com
10.16288/j.yczz.15-070
时间:2015-6-10 15:57:06
URL:http://www.cnki.net/kcms/detail/11.1913.R.20150610.1557.002.html

