ChIP-seq比较分析人体心脏和脾脏的H3K9三甲基化差异
薛 雯,曹翠辉,2,眭维国,车文体,陈洁晶,郭 丽,戴 勇
(1.中国人民解放军第181医院 肾脏科 广西代谢性疾病研究重点实验室, 广西 桂林 541002;2.广西师范大学生命科学学院,广西 桂林 541004; 3.深圳市人民医院 暨南大学 第二临床医学院 临床医学研究中心, 广东 深圳 518020)
ChIP-seq比较分析人体心脏和脾脏的H3K9三甲基化差异
薛 雯1,曹翠辉1,2,眭维国1,车文体1,陈洁晶1,郭 丽1,戴 勇3*
(1.中国人民解放军第181医院 肾脏科 广西代谢性疾病研究重点实验室, 广西 桂林 541002;2.广西师范大学生命科学学院,广西 桂林 541004; 3.深圳市人民医院 暨南大学 第二临床医学院 临床医学研究中心, 广东 深圳 518020)
目的分析人体器官心脏和脾脏的H3K9me3的全基因图谱,以期发现H3K9三甲基化的修饰与组织特异性的表达、功能和发育具有相关性。方法通过染色质免疫共沉淀的方法获取各标本DNA,通过qPCR验证ChIP结果,然后构建ChIP Sequencing文库,与目的基因组序列比对,获取比对Reads,接着进行全基因组的Peak分析,GO功能富集分析peak相关基因的生物学功能。结果心脏和脾脏间有169个基因有显著地H3K9me3差异,其中心脏中H3K9me3的基因中有64个特殊基因,脾脏中H3K9me3的基因中有87个特殊基因。从这些基因中,挑选出8个表现H3K9me3 明显的基因,然后再从这8个基因中挑选出两个心脏和脾脏间表现H3K9me3差异最明显的基因,分别是PTPN3和RBMS。结论H3K9me3差异可作为一个潜在的生物标志物或是表观遗传疾病的治疗靶点。
H3K9me3;ChIP-seq;表观遗传学
在哺乳动物基因组中,组蛋白则可以有很多修饰形式。包括组蛋白末端的乙酰化,甲基化,磷酸化,泛素化以及ADP核糖基化等等,这些修饰都会影响基因的转录活性。在不同的组织和生物中已广泛研究并证实组蛋白H3K9三甲基化(H3K9me3)与异染色质形成,基因的表达和转录伸长率具有关联性[1]。在各种不同的组蛋白赖氨酸甲基化模式中,由于H3K9的甲基化和抑制的染色质相关而备受关注[2]。H3K9三甲基化(H3K9me3)是一个翻译后修饰并且与异染色质的形成和转录抑制相关联[3]。
ChIP-seq是染色质免疫共沉淀后并结合高通量测序的方法,它能够在全基因组中识别转录因子(TFs)和DNA结合蛋白的结合位点[4]。随着高通量测序平台如Illumina公司的基因组分析以及SOLiD和芯片级别的抗体出现,ChIP-seq已成为用于确定基因组中的功能元件的最广泛使用的方法[5]。与ChIP-chip相比,ChIP-seq具有更高的信噪比,更便宜以及更少量的样本优势[6]。
目前,只有极少数研究比较分析哺乳动物或人体组织的H3K9me3。因此,本研究采用ChIP-seq技术比较分析人体器官心脏和脾脏之间在全基因组中H3K9me3的变化及差异情况,了解组织间的表观遗传差异,并且寻找潜在的生物标志物或是表观遗传疾病的治疗靶点。
1 材料与方法
1.1 样本
采用两组标本,分别是两个心脏和两个脾脏,均来自中国广西桂林第181医院脑死亡患者自愿捐赠的器官。标本放置于-80 ℃保存。标本收集的方案与执行均获得广西代谢性疾病重点实验室和第181医院伦理委员会的批准。器官捐赠者均签署了知情同意书。
1.2 染色质免疫共沉淀
将组织样本先研磨匀浆并用1%甲醛处理细胞,使 DNA-protein 的相互结合作用被交联固定,然后裂解细胞,得到全细胞裂解液,超声处理,将基因组 DNA 打断至100~500 bp碎片,在细胞裂解液中加入特定的实验所需的三甲基化赖氨酸9的抗体和beads,并进行4℃孵育。采用合适的实验条件进行洗脱,并解交联,经过交联反转和蛋白酶K处理后,DNA被沉淀提取出,再构建DNA文库前,先用QIA快速PCR纯化试剂盒将DNA进一步纯化。通过qPCR 对ChIP结果进行验证,准备好的ChIP后的DNA样品可以用于 ChIP Sequencing 建库。
1.3 ChIP Sequencing文库构建流程
DNA 片段末端修复,3′端加A碱基,连接测序接头(详细步骤请参考Illumina公司Paired-End DNA Sample Prep kit)。PCR扩增及DNA产物的片段大小选择(一般为 100~300 bp,包括接头序列在内),合格的文库用于上机测序。
1.4 ChIP-seq分析
ChIP DNA 末端修复,接头连接以及扩增等均按之前的进行[7]。从琼脂糖凝胶中分离出大小约为49 bp的片段,用Solexa/Illumina 2G基因分析仪分析测序。与目的物种基因组序列(参考基因组版本为Hg19)进行比对,比对软件为SOAP 2.21[8]。采用软件MACS 1.4.0全基因组Peak扫描,将基因组上的候选peak区延伸,得到一定长度的建模区域,根据此区域中所有唯一比对reads的情况,使用Poisson分布模型进行检验,计算候选peak区域的P-value,若P-valuelt;1.00e-05,则认为该区域是一个peak[9]。MACS的目的是从ChIP-seq的数据集中找出转录因子结合位点和组蛋白修饰富集区域,且不需要正常对照[10]。
Peak相关基因的GO分析,GO 是一种整合性、统一化、动态开放实时更新的分类系统。它包括3大独立的本体(ontology):基因参与的生命过程(biological process),所处的细胞组份和元件(cellular component)及发挥的分子生物学功能(molecular function)。通过 GO 功能富集分析,可以知道peak相关基因涉及到哪些生物学功能的改变[11]。
2 结果
2.1 统计
测序完成后,对原始数据进行去污染,去接头及去除低质量数据处理, 统计clean data产量(表1)。
2.2ChIPSequencing结果与参考基因组序列进行比对
将clean data与目的物种基因组序列进行比对,允许不超过2个碱基的错配,其中比对到基因组上唯一位置的reads(唯一比对reads)将用于后续的信息分析(表2)。
比对软件: SOAP 2.21
参考基因组版本: Hg19
2.3 全基因组Peak扫描
基于一定的分析模型在全基因范围进行peak (ChIP Sequencing富集区域)扫描,得到Peak在基因组上的位置信息(表3)。
2.4 GO分析peaks与注释基因的关联
心脏器官组织中的104个注释基因中,37个基因与细胞组分和元件相关,37个基因发挥着分子生物学功能,30个基因参与生命过程。脾脏器官组织中的137个注释基因中,52个基因与细胞组分和元件相关,45个基因发挥着分子生物学功能,40个基因参与生命过程。从细胞的组分来看,心脏中,亚细胞定位的注释基因中34个作为细胞部分,19个作为细胞器;脾脏中,亚细胞定位的注释基因中46个作为细胞部分,21个作为细胞器。从分子生物学功能来看,心脏中,具有链接和催化活性的基因分别为33个和17个,而脾脏中,具有链接和催化活性的基因分别为36和16个。从参与生命过程来看,心脏中,27个基因参与了细胞过程,20个基因参与了生物的调节过程,而脾脏中,36个基因参与了细胞过程,25个基因参与了生物的调节过程。
2.5 心脏和脾脏中H3K9me3的比较
经过对比分析心脏和脾脏组织样本,可以得出样本各自具有的特有的基因(表4)。经过方差分析统计GO功能聚类,通过热图比较两个样本中所共同包含的峰值区域富集度(图1)。
3 讨论
通过以上对测序结果的分析,可以得出心脏和脾脏间169个基因表现了显著地H3K9me3差异,其中64个基因是心脏中的表现H3K9me3的特有基因,87个基因是脾脏中的表现H3K9me3的特有基因。从这些基因中,挑选出8个表现H3K9me3明显的基因,然后再从这8个基因中挑选出两个心脏和脾脏间表现H3K9me3差异最明显的基因,分别是PTPN3和RBMS3。

表1 Clean data产量统计Table 1 Clean data production statistics
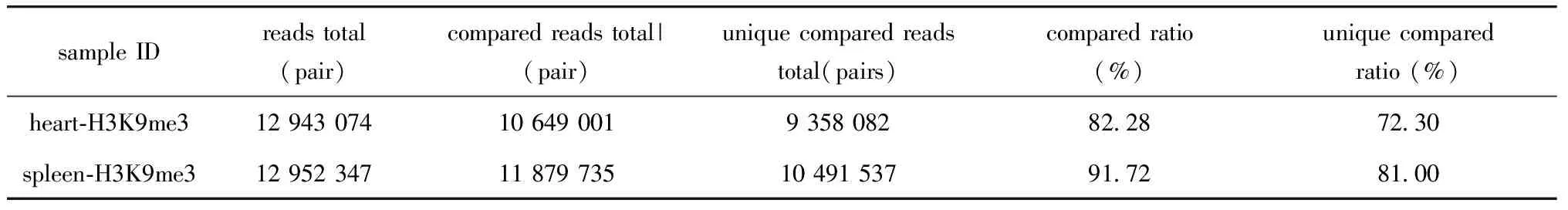
表2 序列比对结果统计Table 2 Sequence alignment result statistics

表3 Peak信息统计Table 3 Peak statistics
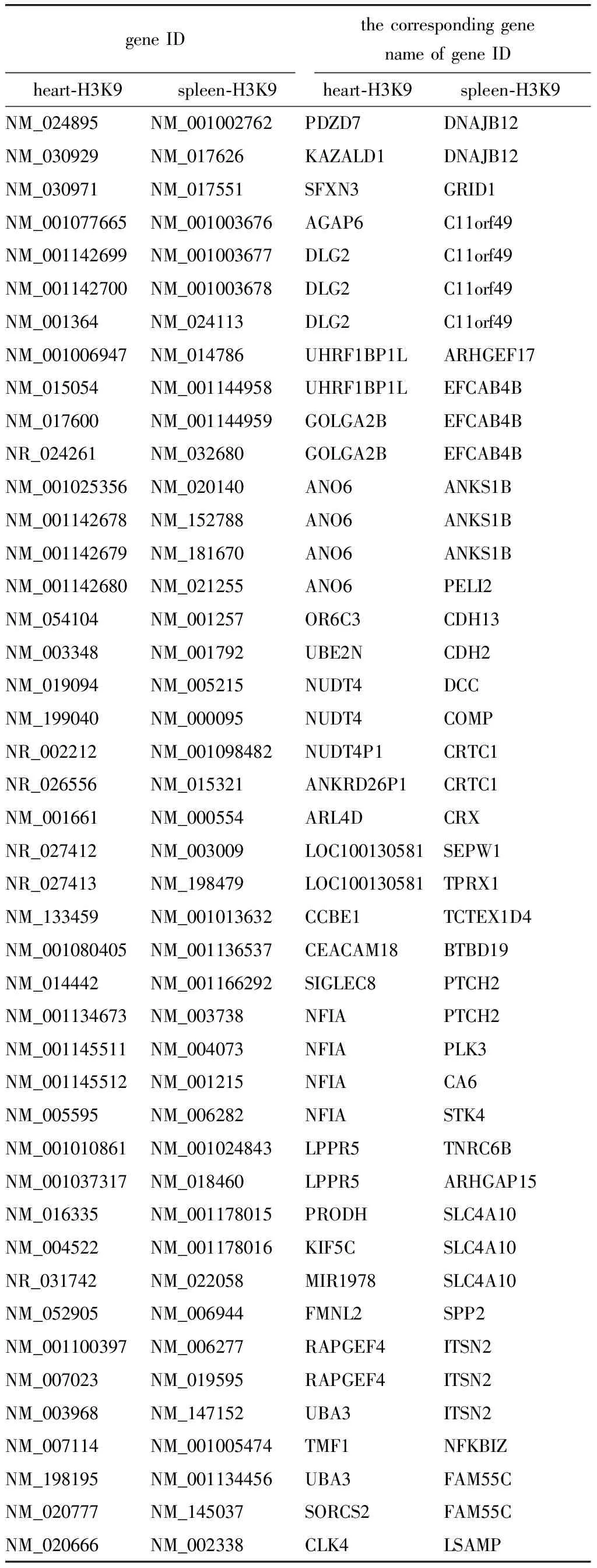
表4 样本各自具有的特有的基因Table 4 The unique genes of each sample

续表4

图1 两个样本中所共同包含的峰值区域富集度Fig 1 The common area of the peak enrichment between the two samples
新一代测序技术分析非小细胞肺癌(non-small-cell lung carcinoma, NSCLC)细胞系H2228的转录水平,发现了一个由多个外显子组成的融合转录子间变性淋巴瘤受体酪氨酸激酶(anaplasticlymphoma kinase, ALK)和PTPN3。基因组结构的详细分析表明,ALK的部分基因区域的外显子10和11 易位到了PTPN3的外显子2和3之间[12]。
RNA结合模体,单链相互作用蛋白3(RNA binding motif, single stranded interacting protein, RBMS 3),最初被发现在RNA结合蛋白中,作用于DNA结合过程中,并且与DNA复制,基因转录,细胞周期进程和细胞凋亡等多样化的功能相关联。RBMS3还能抑制微血管的形成,这可能是通过下调MMP2和β-catenin和灭活其下游的目标,包括细胞周期蛋白D1,c-Myc基因,MMP7和MMP9来实现的[13]。
综上所述,本文系统地评估了心脏和脾脏中H3K9me3差异的状态,并且通过关键基因和组蛋白甲基化对于人体器官之间的关联获得了新的见解。研究也表明下一代测序用于研究DNA甲基化具有很好的实用性,并且具有疾病的鉴别诊断,预后和治疗的重要性。这些新的研究结果表明,H3K9me3具有作为一个潜在的生物标志物或表观遗传疾病治疗靶点的意义。
感谢:所有捐献血液的志愿者。
[1] Lessard JA, Crabtree GR. Chromatin regulatory mechanisms in pluripotency [J]. Annu Rev Cell Dev Biol, 2010, 26: 503- 532.
[2] Jin-Ah P, Ae-Jin K, Yoonsung K,etal. Deacetylation and methylation at Histone H3 Lysine 9 (H3K9) coordinate chromosome condensation during cell cycle progression [J]. Molecules and Cells, 2011, 31:343- 349.
[3] Yoichi S, Makoto T. H3K9 methyltransferase G9a and the related molecule GLP [J]. Genes Dev, 2011, 25:781- 788.
[4] Debasish R, Miyoung H, Michael S. ChIP-Seq: A method for global identification of regulatory elements in the genome [J]. Curr Protocols Mol Biol, 2010, 21:1- 14.
[5] Wacker DA, Kim TH. From sextant to GPS: Twenty-five years of mapping the genome with ChIP [J]. J. Cell. Biochem, 2009, 107:6- 10.
[6] Euskirchen GM, Rozowsky JS, Wie CL,etal. Mapping of transcription factor binding regions in mammalian cells by ChIP: Comparison of array-and sequencing-based technologies [J]. Genome Res, 2007, 17:898- 909.
[7] Lin B, Wang J, Hong X,etal. Integrated expression profiling and ChIP-seq analyses of the growth inhibition response program of the androgen receptor [J]. PLoS One, 2009, 4:e6589. doi: 10.1371/journal.pone.0006589.
[8] Langmead B, Schatz MC. Searching for SNPs with cloud computing [J]. Genome Biol, 2009, 10:R134. doi: 10.1186/gb-2009-10-11-r134.
[9] Feng J, Liu T. Using MACS to identify peaks from ChIP-Seq data [J]. Curr Protoc Bioinformatics, 2011, 2:14.
[10] Joo HS, Robert W Li, Yuan G,etal. Genome-wide ChIP-seq mapping and analysis reveal butyrate-induced acetylation of H3K9 and H3K27 correlated with transcription activity in bovine cells [J]. Funct Integr Genomics, 2012, 12:119- 130.
[11] Seth C, Amelia I, Christopher J,etal. AmiGO: online access to ontology and annotation data [J]. Bioinformatics, 2009, 25:288- 289.
[12] Jung Y, Kim P, Jung Y,etal. Discovery of ALK-PTPN3 gene fusion from human non-small cell lung carcinoma cell line using next generation RNA sequencing [J]. Gen Chromosome Cancer, 2012, 51:590- 597.
[13] Chen J, Kwong DL, Zhu CL,etal. RBMS3 at 3p24 inhibits nasopharyngeal carcinoma development via inhibiting cell proliferation, angiogenesis, and inducingapoptosis [J]. PloS One, 2012, 7:e44636. doi: 10.1371/journal.pone. 0044636.
Analysis of difference in histone H3K9 trimethylations in normal human heart and spleen by ChIP-seq
XUE Wen1, CAO Cui-hui1,2, SUI Wei-guo1, CHE Wen-ti1, CHEN Jie-jing1, GUO Li1, DAI Yong3*
(1.Guangxi Key Laboratory of Metabolic Diseases Research, Dept. of Nephrology, Guilin 181st Hospital, Guilin 541002;2.College of Life Science, Guangxi Normal University, Guilin 541004; 3.Clinical Medical Research Center, the Second Clinical Medical College of Jinan University, Shenzhen People’s Hospital, Shenzhen 518020, China)
ObjectiveThe modifications are closely-associated with tissue-specific expression, function and development by generating the genome-wide maps of H3K9me3 of human heart and spleen.MethodsThe DNA of samples was extracted by chromatin immunoprecipitation. The ChIP results were validated by qPCR.Then building the ChIP sequencing library, contrasting with the target genome sequence to obtain compared reads, and peak analysis of whole genome, GO enrichment analysis of the biological functions of the related genes of peak.Results169 genes displayed significant H3K9me3 differences between heart and spleen. Among these genes, 64 genes were the special genes in heart-H3K9me3; 87 genes were special genes in spleen-H3K9me3. From these genes, we selected eight genes of H3K9 which were of significantly expression. Then selecting two genes of H3K9 which were the most significant difference expression from the eight genes.They are PTPN3 and RBMS.ConclusionsH3K9me3 may be a potential biomarker or promising target for epigenetic-based disease treatment.
H3K9me3; ChIP-seq; epigenetic
2013- 09- 26
2014- 01- 13
广西自然科学基金(2012GXNSFDA053017);广西科技基础设施建设项目(11-031-33);广西重点实验室建设项目(12-071-32)
*通信作者(correspondingauthor): daiyong2222@gmail.com
1001-6325(2014)07-0939-06
研究论文
Q593+.9
A

