2个不同来源真姬菇子实体同源性的分子鉴定*
黄龙花,夏凤娜,邵满超,张一帆,陈多扬,胡惠萍**
(1.广东省微生物研究所,广东省菌种保藏与应用重点实验室,广东省微生物应用新技术公共实验室,
广东省华南应用微生物重点实验室,广东 广州 510070;2.广东粤微食用菌技术有限公司,广东 广州 510070)
真姬菇Hypsizygus marmoreus(PK)Sing.属于担子菌亚门、伞菌目、白蘑科、玉蕈属[1],又名斑玉蕈、蟹味菇、玉蕈、假松茸、灵芝菇等,是北温带一种品质优良的食用菌[2]。真姬菇营养价值较高,是1种具有良好保健功能的食用菌,其子实体热水和有机溶剂 (甲醇和乙醇等)提取物,具有清除体内自由基、降血压、提高免疫力和延缓衰老等功效[3]。在日本有“香在松口蘑,味在玉蕈”之称。由于我国真姬菇品种名称使用不规范,造成同种异名、同名异种现象较多,给真姬菇品种交流和产品出口带来不便[4]。因此建立一套准确可靠、稳定性好的真姬菇菌株分类鉴定方法迫在眉睫。
本研究将ERIC-PCR指纹图谱分析和ITS序列分析相结合,对2个不同企业提供的真姬菇子实体进行同源性研究,为快速准确鉴定真姬菇品种提供参考。其中利用ERIC-PCR技术对真姬菇进行同源性分析在国内外尚属首次。
1 材料与方法
1.1 材料
2个供试真姬菇子实体样品,由广东省食用菌行业协会会员单位提供,具体见表1。

表1 供试菇品
1.2 子实体前处理
对获得的子实体进行实地拍照记录,并将新鲜子实体在恒温鼓风干燥箱中50℃烘干,保存备用。
1.3 子实体基因组DNA的提取
子实体总DNA的提取纯化按黄龙花等[5]的方法进行。
1.4 PCR扩增
1.4.1 ERIC-PCR
采用ERIC-PCR通用引物ERIC2和ERICR(ERIC2:5'-ATG TAA GCT CCT GGG GAT TCA C-3';ERICR:5'-AAG TAA GTG ACT GGG GTG AGC G-3')进行PCR扩增,引物由上海生工合成,扩增在Biometra PCR仪上进行。反应体系为2 ×PCR Taqmix 12.5 μL,DNA 模板 (浓度适中)2.5 μL,ddH2O 6 μL,引物 (10 μmol·L-1)各 2 μL,总体积 25 μL。反应条件为95℃反应5 min;94℃反应1 min,40℃~60℃(温度梯度)反应1 min,72℃反应2 min,30个循环;72℃反应8 min。
1.4.2 ITS-PCR
采用真菌核糖体基因间隔区通用引物 ITS1和 ITS4[6](ITS1:TCC GTA GGT GAA CCT GCG G;ITS4:TCC TCC GCT TAT TGA TA TGC)进行PCR扩增,引物由Sangon公司合成,扩增在Biometra PCR仪上进行。反应体系为2×PCR Taqmix 25 μL,DNA 模板 (浓度适中)5 μL,ddH2O 12 μL,引物(10 μmol·L-1)各 4 μL,总体积 50 μL。反应条件为 94℃反应5 min;94℃反应1 min,55℃反应1 min,72℃反应1 min,30个循环;72℃反应10 min。
1.5 电泳成像及测序
对ERIC-PCR及ITS-PCR产物进行1.0%的琼脂糖凝胶电泳,在UVP凝胶成像系统 (GE公司)下成像保存。ITS-PCR产物回收纯化后,以扩增引物为测序引物进行双向测序拼接,由华大基因完成。
1.6 图谱分析及序列比对分析
对电泳指纹图谱进行分析 (样品多的情况下,需要用UVIband等软件构建系统进化树,进行进化关系分析);对测得碱基序列,应用Clustal X进行序列完全比对分析 (样品多的情况下,需要进一步用MEGA等软件构建系统进化树,进行进化关系分析)。
2 结果与分析
A公司灵芝菇子实体、B公司真姬菇子实体特征见图1、图2。


由图1、图2可以看出,2种鲜菇子实体外观差异极小,难以区分 (色差由拍照引起)。观察发现,A公司灵芝菇整体较为矮胖,纤维感较强,因而提取DNA时,干燥子实体较难磨碎。对照1和对照2是由同一菌种分离出的不同菌株,结果见图3。
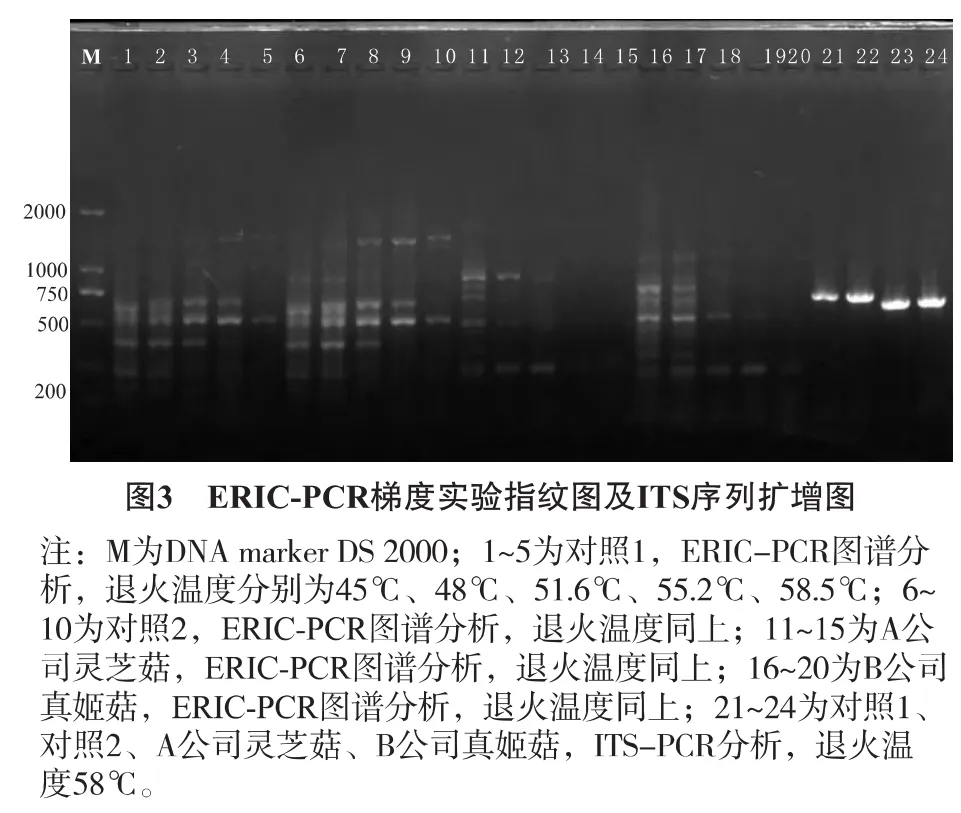
从图3可以看出,退火温度为45℃和48℃时,条带多态性更为丰富,说明这些菌种的ERIC-PCR最佳退火温度为(45±5)℃,其中对照1和对照2的指纹图谱极相近,为同一品系的菌种 (图4进一步验证);而A公司灵芝菇和B公司真姬菇的指纹图谱则差异比较大,应该不是同一品系 (图4进一步验证)。为探索最适退火温度,进一步进行40℃~55℃退火温度的梯度PCR,结果见图4。

由图4可以看出,对照1和对照2在同一退火温度下的ERIC-PCR指纹图谱一致,应为同一品种,其子实体应栽培自同一品种的同一品系;而A公司灵芝菇和B公司真姬菇在同一退火温度下的ERIC-PCR指纹图谱差异明显,可能为不同品种或者同一品种的不同品系。真姬菇ITS序列完全比对图见图5。
由图5可以看出,测序所得的A公司灵芝菇和B公司真姬菇的ITS序列长度均为661 bp,对661个碱基进行完全比对,结果发现它们存在3个碱基差异,其中有1个落在ITS1上;2个在ITS2上。将序列在NCBI的GenBank上作Blast,发现2个序列和GenBank上的已知真姬菇 (Hypsizigus marmoreus)序列相似性均达99%,所以2个均为真姬菇,但应来自不同品系。

3 结论
ERIC-PCR指纹图谱分析和ITS序列分析相结合,对A公司灵芝菇和B公司真姬菇2组外观相近的菇品子实体进行同源性研究。结果发现,A公司灵芝菇和B公司真姬菇的ERIC-PCR指纹图谱存在较大的多态性差异,它们应属于不同菌株 (品系),甚至不同品种;ITS序列测定结果显示它们仅存在3个碱基差异,应属于同一品种。因此2种分子方法结合分析表明,这两个子实体样品均属于真姬菇 (Hypsizigus marmoreus)品种,来自同一个品种的不同亚种/菌株 (品系),它们拥有各自独立知识产权。
[1]黄年来.中国大型真菌原色图鉴[M].北京:中国农业出版社,1998.
[2]杨新美.食用菌栽培学[M].北京:农业出版社,1986.
[3]孙培龙,魏红福,杨开,等.真姬菇研究进展 [J].食品科技,2005,26(9):54-57.
[4]王波.几种食用菌品种名称订正 [J].中国食用菌,2006,25(5):27-28.
[5]黄龙花,吴清平,杨小兵,等.基于ITS序列分析探讨我国栽培风尾菇的分类地位 [J].食用菌学报,2009,16(2):30-35.
[6]White TJ,Bruns T,Lee S.Analysis of phylogenetic relationships by amplification and direct sequencing of ribosomal RNA genes[C].//Innis MA.Ed.PCR Protocols:A guide to methods and applications.New York:Academic,1990:15-22.

