亚胺及其相关离子在区分赖氨酸和谷氨酰胺残基中的应用
李水明,何曼文,王 勇
(1. 深圳大学生命科学学院,深圳市海洋生物资源与生态环境重点实验室,广东 深圳 518060;2. 深圳大学生命科学学院,深圳市微生物基因工程重点实验室,广东 深圳 518060)
亚胺及其相关离子在区分赖氨酸和谷氨酰胺残基中的应用
李水明1,何曼文2,王 勇1
(1. 深圳大学生命科学学院,深圳市海洋生物资源与生态环境重点实验室,广东 深圳 518060;2. 深圳大学生命科学学院,深圳市微生物基因工程重点实验室,广东 深圳 518060)
完全从头测序要求区分肽段中的同量异序氨基酸谷氨酰胺和赖氨酸,而这通常需要借助高分辨质谱。谷氨酰胺和赖氨酸都可以产生m/z101亚胺离子和m/z84亚胺相关离子,尽管已知赖氨酸亚胺离子m/z101有时强度较弱,以及m/z84离子可预示赖氨酸的存在,但该特征能否用于区分赖氨酸和谷氨酰胺仍存在争议。本工作以合成肽段和蛋白质胰酶水解肽段为研究体系,利用基质辅助激光解吸电离的高能碰撞诱导解离模式产生的亚胺及其相关离子的相对强度区分这两种氨基酸。结果表明:谷氨酰胺更易产生m/z101离子,而赖氨酸更易产生m/z84 和m/z129离子。对于不同时含有赖氨酸和谷氨酰胺的肽段,使用m/z84离子与亚胺离子m/z101的相对强度比值,而非它们的绝对强度,以及m/z101离子的测量值,可以直接判断序列中存在的是赖氨酸还是谷氨酰胺。对于含有谷氨酰胺,并以赖氨酸结尾或含有漏切赖氨酸的肽段,该强度比可为序列中谷氨酰胺的个数和位置提供参考。因此,低质量区提供的亚胺及其相关离子信息有助于区分赖氨酸和谷氨酰胺。
亚胺离子;基质辅助激光解吸电离-串联飞行时间质谱;赖氨酸;谷氨酰胺;同量异序氨基酸
数据库搜索和从头测序是利用质谱技术鉴定蛋白质的两种基本方法[1-2],尽管数据库搜索在目前蛋白质组学研究中最为常用,但后者对于鉴定基因组未测序生物和高置信度鉴定已知蛋白更为必要[3-5]。完全的从头测序不仅要求获得连续的子离子序列,还要能够区分同量异序的氨基酸或氨基酸组合。准确测定生物分子的单同位素质量可以获得肽段的组成信息,例如,区分质量为1 100 u肽段的质量准确度为0.1×10-6(百万分之一)[6]。赖氨酸(lysine,Lys,K)和谷氨酰胺(glutamine,Gln,Q)是分子质量相差36.4 mu的同量异序氨基酸。由于胰蛋白酶特异性水解赖氨酸和精氨酸,在理论上,肽段中间不应有赖氨酸残基,但由于漏切现象,利用质谱技术区分这两种氨基酸仍有必要。它们可以被四极杆-飞行时间质谱区分,但是傅里叶变换-离子回旋共振和静电轨道场离子阱质谱是更好的选择[7-10]。
典型情况下,串联飞行时间质谱(TOF/TOF)的外标法准确度是5.0×10-5,因此,该类仪器有时不能区分赖氨酸和谷氨酰胺[11-12]。然而,在TOF/TOF类仪器上实现的高能碰撞诱导解离可以为从头测序分析提供充分断裂和较完全的子离子序列。实际上,在基质辅助激光解吸电离-串联飞行时间(MALDI-TOF/TOF)质谱中,高能断裂和低能断裂都可以发生,加之其高通量特性和操作方便等优点,该类质谱已被广泛用于蛋白质鉴定和蛋白质相互作用等研究中[13-18]。高能碰撞诱导解离(CID)的另一个显著特征是可以产生亚胺离子(RCH=NH2+)。早期研究表明,亚胺离子可作为肽段序列中存在或缺少某些特定氨基酸的标志[19-20]。然而,利用亚胺离子辅助从头测序的研究尚且不多[10],可能是近年来高分辨和高准确度质谱仪发展迅速,而TOF/TOF类仪器的准确度较低[21-22],也可能是由于离子阱质谱在蛋白质组学研究中的快速发展,使得亚胺离子的作用被忽略[23]。
基于四极磁质谱,Falick等[24]指出,不可能利用m/z84和m/z101离子区分赖氨酸和谷氨酰胺。然而,Papayannopoulos等[19]认为,m/z84离子是序列中存在赖氨酸的可信标志。因此,能否利用亚胺离子区分赖氨酸和谷氨酰胺仍存在争议,目前大多数从头测序软件也没有利用亚胺离子提高分析结果的确定性。本研究利用合成多肽和蛋白质胰酶切的实际样品,构建了由184个来源于双向电泳或纯化蛋白的实际肽段及22个合成肽段组成的数据集,并将数据集分成含有谷氨酰胺、赖氨酸和同时含有这两种氨基酸三类,探讨利用亚胺和亚胺相关离子相对强度比和所测质荷比区分这两个同量异序氨基酸的可能性。
1 实验部分
1.1 仪器与试剂
4800 MALDI TOF/TOFTMAnalyzer基质辅助激光解吸电离-串联飞行时间质谱仪:美国Applied Biosystems/MDX Sciex公司产品,配有4700 Series Explorer Software软件;Milli-Q超纯水处理系统:美国Millipore公司产品。
胰蛋白酶(质谱级):美国Promega公司产品;α-腈基-4-羟基肉桂酸:美国Sigma公司产品;乙腈和三氟乙酸等试剂均为分析纯或优级纯;多肽:由上海强耀公司合成。
1.2 胶内胰酶水解条件
切胶:将双向电泳分离后的目标条带用移液枪枪头切下,小心地放入PCR管中。脱色:用100 μL高纯水振荡洗涤10 min,重复3次;然后用100 μL 25 mmol/L NH4HCO3室温振荡平衡20 min;再用100 μL 25 mmol/L NH4HCO3和50%乙腈室温振荡30 min。干燥胶粒:100 μL 25 mmol/L NH4HCO3室温振荡平衡20 min;加入5 μL 50%乙腈溶液,让胶粒脱水;再加100%乙腈,让胶粒变白完全脱水。酶解:加入5 μL 20 mg/L胰蛋白酶(质谱用测序级),4 ℃放置30 min,使酶液完全浸透胶粒,吸掉多余的酶液;冰上充分吸涨变透明后,再加入20 μL含10% 乙腈的40 mmol/L NH4HCO3溶液覆盖,37 ℃水浴恒温8 h,直接取上清酶解液,进行质谱分析。
1.3 质谱分析条件
基质制备:将6 g/Lα-腈基-4-羟基肉桂酸和2 g/L柠檬酸氢二胺溶于10 mL 50%乙腈(含0.1%三氟乙酸)中,V(基质溶液)∶V(蛋白酶解液)=1∶1混合后点于不锈钢靶板,自然干燥结晶后,待测。
采用正离子反射模式,一级质谱每张谱图累加800次,二级质谱累加1 200次,碰撞诱导解离条件为1 kV,碰撞能加空气碰撞(1 kV, gas on),源内电压8 kV,碰撞池电压7 kV,二级源加速电压15 kV。
1.4 数据分析条件
数据库搜索条件:串联质谱数据信噪比设为5,一级和二级质谱质量误差均设为0.3 u,利用Mascot软件分析,所用数据库NCBI 于2013年1月发布,甲硫氨酸氧化和谷氨酸甲基化作为可变修饰。当Mascot分数接近或小于可信阈值时,利用Data Explorer提供的离子碎片计算器和Protein-Prospector MS-Product program辅助,进行人工解析。
2 结果与讨论
2.1 HDLYGK系列肽段的MALDI-TOF/TOF质谱分析
由于亚胺离子相对强度与肽段序列和仪器条件(如碰撞气压力和激光强度)等因素有关,用亚胺离子m/z101和亚胺相关离子m/z84的强度比值更能区分谷氨酰胺和赖氨酸。例如,在肽段HDLYGK的二级串联质谱图中,组氨酸亚胺离子m/z110.08是基峰,而m/z84.08 和m/z101.11离子强度都很弱,分别为4.29%和0.39%,但它们的比值为11,说明赖氨酸确实更易产生亚胺相关离子m/z84,而非亚胺离子m/z101,示于图1a。以亮氨酸亚胺离子m/z86的强度作为参照,发现在肽段HQDLYGK的二级串联质谱中,m/z84离子的强度没有增加,但m/z101离子的强度显著增加,同时,检测到的质荷比从m/z101.11变为m/z101.08,这从强度和质荷比两个方面说明,m/z101离子主要由谷氨酰胺贡献,示于图1b。相反,在肽段HKDLYGK的TOF/TOF谱中,m/z84离子和m/z129离子的强度均明显高于m/z101.12离子,示于图1c。类似的,在肽段HQKDLYGK的TOF/TOF谱图中,m/z101离子强度有所增加,示于图1d。另一个例子,在实际样品肽段EYWQNLR的串联质谱中,观察到了较明显的m/z101离子,而在肽段KEYWQNLR的串联质谱中,m/z84和m/z129离子的相对强度明显增加,示于图2。以上分析提示,赖氨酸和谷氨酰胺之间的替换会使m/z84离子与m/z101离子的相对强度比例发生明显变化,下面将在较大数据集上验证该假设。
2.2 三种类型肽段中m/z84和m/z129离子与m/z101离子的强度比值分析
本研究共统计了84个以精氨酸结尾的含有1个或多个谷氨酰胺的肽段,包括6个合成肽段和78个酶解肽段。在84个串联质谱图中都检测到了m/z101离子,而m/z84离子仅在67个肽段的质谱图中被发现,m/z84与m/z101离子的强度比范围为1.5~0.056,只有3个肽段的比值大于1,分别为AQIHDLVLVGGSTR(1.5),QYDISDTR(1.33)和NQGDHLLHSTR(1.08)。类似的,在82个肽段的串联质谱中检测到了m/z129离子,大多数情况下,m/z129离子的相对强度均小于m/z101离子,用平均值和标准差表示为0.467±0.384。以上结果说明,对于含有谷氨酰胺但不含赖氨酸残基的肽段,m/z101离子的强度在大多数情况下高于m/z84和m/z129离子。3个例外情况下,谷氨酰胺位于氨基端的第1位或第2位,在实验条件不变的情况下,位置效应和肽段结构的特殊性可能是造成这种例外的原因,即,如果谷氨酰胺或赖氨酸位于N端附近,可能容易生成亚胺相关离子m/z84。为此,合成了肽段QMNGTFR和KMNGTFR进行串联质谱比较,结果示于图3。

注:a. HDLYGK;b. HQDLYGK低质量区;c. HKDLYGK低质量区;d. HQKDLYGK低质量区图1 合成肽段HDLYGK系列的MALDI-TOF/TOF质谱图Fig.1 The MALDI-TOF/TOF spectra of synthetic peptides of HDLYGK series
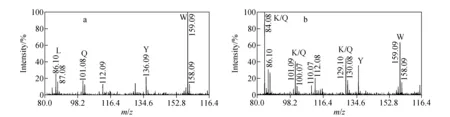
图2 酶切肽段EYWQNLR(a)和KEYWQNLR(b)的低质量区MALDI-TOF/TOF质谱图Fig.2 The low mass region of MALDI-TOF/TOF spectrum of digest peptide EYWQNLR (a) and KEYWQNLR(b)

图3 合成肽段QMNGTFR(a)和KMNGTFR(b)的低质量区MALDI-TOF/TOF质谱图Fig.3 The low mass region of MALDI-TOF/TOF spectrum of synthetic peptide QMNGTFR(a) and KMNGTFR(b)
在肽段QMNGTFR的TOF/TOF谱图中,谷氨酰胺亚胺离子m/z101.08的丰度仅略高于亚胺相关离子m/z84.06,未明显观察到m/z129离子。这是由于肽段较短,断裂充分,并且谷氨酰胺位于肽段氨基端。而对于肽段KMNGTFR,这种趋势更为明显,m/z84.09离子在低质量区是最高峰,而赖氨酸亚胺离子m/z101.11强度较低,还观察到了明显的m/z129离子。
在63个含有赖氨酸但不含有谷氨酰胺的肽段中,48个肽段仅含有1个赖氨酸残基,其中33个以赖氨酸结尾,13个以精氨酸结尾,另外2个以丝氨酸或甘氨酸结尾;剩余的15个肽段以赖氨酸结尾,但在序列中还含有另外1个赖氨酸残基,造成这种现象的原因多为2个赖氨酸残基相邻或者赖氨酸与天冬氨酸(E)相邻。在这63个肽段的TOF/TOF谱图中,均检测到了m/z84离子,而m/z101和m/z129离子的检测频次分别是38次和57次。m/z84与m/z101离子的强度比为1.37~25.6,平均值为4.24;而m/z84与m/z129的离子强度比值的平均值为1.06。因此,用m/z84和m/z129离子同时具有较高强度判定肽段中存在赖氨酸残基更为可靠。
当肽段序列中同时存在赖氨酸和谷氨酰胺时,离子强度的比值可看作是上述2种情况的叠加,用10个合成肽段和49个酶切肽段构成了这个数据集。在总共59个多肽质谱图中,均检测到m/z101离子,而m/z84 和m/z129离子分别在53个和57个肽段中被观察到。m/z84与m/z101离子的相对强度比值的最小值是0.208,对应肽段QVQAVQQFEVK;最大值是7.46,对应肽段VYVEELKPTPEGDLEILLQK,平均值是0.72。当肽段中同时存在赖氨酸和谷氨酰胺时,较难像上述2种情况下利用离子强度的比值进行直接判断,这时需结合谷氨酰胺的个数和位置以及赖氨酸的漏切进行综合分析。总体上,谷氨酰胺个数越多或越接近于N端,其对m/z101离子的贡献越大,亚胺离子在TOF/TOF质谱中的位置效应将在后继工作中分析。
2.3 三种类型肽段中所测m/z101离子质荷比的差别
赖氨酸和谷氨酰胺亚胺离子的理论质荷比分别是m/z101.107 3和m/z101.070 9,区分二者要求3.6×10-5的准确度,串联飞行时间质谱有可能达到该指标。在外标法条件下,对于84个含有谷氨酰胺肽段、63个含有赖氨酸肽段和59个同时含有这2个氨基酸残基的肽段,检测到的m/z101离子的质荷比平均值分别是101.078 1±0.008 1,101.103±0.033 4和101.082±0.010 2。该结果说明,串联飞行时间质谱检测到的m/z101离子的质荷比,对于判断序列中这2种同量异序氨基酸残基具有一定参考作用,由于m/z101离子主要由谷氨酰胺贡献,在同时含有谷氨酰胺和赖氨酸的肽段中,其质荷比更接近与谷氨酰胺的理论值。
3 结论
本研究利用串联飞行时间质谱的高能碰撞诱导解离技术产生的亚胺离子和亚胺相关离子的强度比区分谷氨酰胺和赖氨酸。该类仪器有2种操作模式:即加碰撞气和不加碰撞气,二者在数据库检索模式下的结果差别不大,但前者对于从头测序更为合适,因为其提供了更为丰富的断裂和亚胺离子信息,还可确认翻译后修饰的鉴定结果[25]。研究发现,相比于赖氨酸,谷氨酰胺更容易产生m/z101离子,而赖氨酸更易形成m/z84 和m/z129离子,当存在非C端赖氨酸时,这种趋势尤为明显,这是因为亚胺离子强度随着其与N端距离的增加而减弱[23]。 因此, 相对较强的m/z101离子提示序列中存在谷氨酰胺,而相对较强的m/z84和m/z129离子则预示存在赖氨酸漏切。
与前人工作相比,本研究使用离子相对强度比值而非相对强度值作为判断数据,这是因为多种因素影响目标离子的相对强度。例如,肽段很长时,所有低质量区的离子强度都下降,此外,以脯氨酸结尾的y离子可能为基峰,也抑制了亚胺离子和亚胺相关离子的强度,因此,比较相对强度更为合理,它反映了排除仪器条件和序列特异性后,2种同量异序氨基酸的自身断裂特性。在实验中还发现,样品对亚胺离子强度的影响较小,只要检测器信号不出现饱和,该规律都适用。此外,m/z101离子的测量值也可能为区分赖氨酸和谷氨酰胺提供线索。尽管MALDI-TOF质谱只具有中等质量分辨率和准确度,但如果充分利用亚胺离子信息,则有可能发挥其操作简便和高通量的特性,为完全从头测序提供更多信息。
[1] PEVTSOV,FEDULOVA I,MURZAEI H,et al. Performance evaluation of existing de novo sequencing algorithms [J]. Proteome Research,2006,5(11):3 018-3 028.
[2] MENSCHAERG,VANDEKERCKHOV T T M,BAGGERMANG,et al. A hybrid, de novo based, genome-wide database search approach applied to the sea urchin neuropeptidome [J]. J Proteome Res,2010,9(2):990-996.
[3] CARPENTIER S C,PANISB,VERTOMMEN A,et al. Proteome analysis of non-model plants: A challenging but powerful approach [J]. Mass Spectrom Rev, 2008,27(4):354-377.
[4] STANDING K G. Peptide and protein de novo sequencing by mass spectrometry[J]. Curr Opin Stru Biol, 2003,13(5):595-601.
[5] SAVITSK M M, NIELSEN M L, KJELDSEN F, et al. Proteomics-grade de novo sequencing approach[J]. J Proteome Res, 2005,4(6):2 348-2 354.
[6] SPENGLER B. De novo sequencing, peptide composition analysis, and composition-based sequencing:a new strategy employing accurate mass determination by Fourier transform ion cyclotron resonance mass spectrometry[J]. J Am Soc Mass Spectrum, 2004, 15(5):703-714.
[7] SEIDLER J,ZINN N,BOEHM M E,et al. De novo sequencing of peptides by MS/MS[J]. Proteomics, 2010,10(4):634-649.
[8] HORN D M,ZUBAREV R A,MCLAFFERTY F W. Automated de novo sequencing of proteins by tandem high-resolution mass spectrometry [J]. Proc Natl Acad Sci, 2000,97(19):10 313-10 317.
[9] HAM B M. Even electron mass spectrometry with biomolecule applications[M]. USA: John Wiley & Sons Inc, 2008.
[10] CHI H,SUN R X,YANG B,et al. De novo peptide sequencing and identification using HCD spectra[J]. J Proteome Res, 2010,9(5):2 713-2 724.
[11] OLSEN M T, EPSTEIN J A, YERGEY A L. De novo peptide sequencing using exhaustive enumeration of peptide composition[J]. J Am Soc Mass Spectrum, 2006,17(8):1 041-1 049.
[12] RASH L D, MORALES R A V, VINK S, et al. De novo sequencing of peptides from the parotid secretion of the cane toad, Bufo marinus (Rhinella marina) [J]. Toxicon, 2011,57(2):208-216.
[13] STIMSON E, TRUONG O, RICHERW J, et al. Enhancement of charge remote fragmentation in protonated peptides by high-energy MALDI-TOF-MS “cold” matrices [J]. Int J Mass Spectrom Ion Process, 1997,(169/170):231-240.
[14] YATESJ R, ENG J K, CLAUSER K R, et al. Search of sequence databases with uninterpreted high-energy collision-induced dissociation soectra of peptides[J]. J Am Soc Mass Spectrum, 1996, 7(11):1 089-1 098.
[15] MEDZIHRADSEKY K F, CAMPBELL J M, BA- LDWIN M A, et al. The characteristics of peptide collision-induced dissociation using a high-performance MALDI-TOF/TOF tandem mass spectrometer[J]. Anal Chem, 2000,72(3):552-558.
[16] VESTAL M L, CAMPBELL J M. Tandem time-of-flight mass spectrometry[J]. Methods in enzymology,2005,402:79-108.
[17] KHATUN J, RAMKISSOON K, GIDDINGSM C. Fragmentation characteristics of collision-induced dissociation in MALDI TOF/TOF mass spectrometry[J]. Anal Chem, 2007,79(8):3 032-3 040.
[18] HUANG L, BALDWIN M A, MALTBY D A, et al. The identification of protein-protein interactions of the nuclear pore complex of saccharomyces cerevisiae using high throughput matrix-assisted laser desorption ionization time-of-flight tandem mass spectrometry[J]. Mol Cell Proteomics, 2002,1(6):434-450.
[19] PAPAYANNOPOULOS I A. The interpretation of collision-induced dissociation tandem mass spectra of peptides[J]. Mass Spectrom Rev, 1995,14(1):49-73.
[20] MEDZIHRADSZKYE K F. Peptide sequence analysis[J]. Methods in Enzymology,2005,402:209-244.
[21] BERKOUT V D. Fragmentation of singly protonated peptides via interaction with metastable rare gas atoms[J]. Anal Chem, 2009,81(2):725-731.
[22] YERGEY A L, COORSSEN J R, BACKLUND P S, et al. De novo sequencing of peptides using MALDI/TOF-TOF[J]. J Am Soc Mass Spectrum,2002,13(7):784-791.
[23] HOHMANN L J,ENG J K,GEMMILL A,et al. Quantification of the compositional information provided by immonium ions on a quadrupole-time-of flight mass specrometer[J]. Anal Chem, 2008, 80(14):5 596-5 606.
[24] FALICK A M,HINES W M,MEDZIHRADSZKY K F,et al. Low-mass ions produced from peptides by high-energy collision-induced dissociation in tandem mass spectgrometry[J]. J Am Soc Mass Spectrum, 1993, 4(11):882-893.
[25] 王 勇,李水明,何曼文. 溶菌酶基质辅助激光解吸电离-串联飞行时间质谱分析中的不常见修饰及脱水反应[J]. 分析化学,2013,41(4):494-499. WANG Yong,LI Shuiming,HE Manwen. The uncommon modifications and dehydration reaction in the analysis of lysozyme by matrix-assisted laser desorption ionization tandem time of flight mass spectrometry [J]. Chinese Journal of Analytical Chemistry, 2013,41(4):494-499(in Chinese).
Application of Immonium and Their Related Ions in Differentiation of Lysine and Glutamine Residues
LI Shui-ming1, HE Man-wen2, WANG Yong1
(1.CollegeofLifeScience,ShenzhenKeyLaboratoryofMarineBioresourcesandEcology,ShenzhenUniversity,Shenzhen518060,China;2.CollegeofLifeScience,ShenzhenKeyLaboratoryofMicrobialGeneticEngineering,ShenzhenUniversity,Shenzhen518060,China)
Differentiation of isobaric glutamine (Gln) and lysine (Lys) is needed for complete de novo sequencing and require high resolution mass spectrometry generally. Both Gln and Lys can produce immonium ions atm/z101 and their related ions atm/z84. Although it is known that the peak atm/z101 due to Lys is sometimes weak and abundant ions atm/z84 is a good indicator of the presence of Lys, whether this feature can be used to discriminate Lys and Gln is still a controversial issue. In this study, investigations were used to distinguish Gln and Lys based on immonium and immonium-related ions produced by matrix-assisted laser desorption ionization tandem time-of-flight (MALDI-TOF/TOF) with high energy collision induced dissociation (CID). The work focused on a series of synthetic peptides and 184 tryptic peptides. The result shows that Gln is more prone to generate immonium ions atm/z101comparede to Lys, which generatesm/z84 andm/z129 ions mainly. It is the ratios of intensity ofm/z101 ion versusm/z84, rather than the intensity value of them, can be used to distinguish Gln versus Lys for practical samples for those peptides that containing Lys or Gln, or provide clue for the number and position of Gln for Lys-terminated peptides. Additionally, the mass measurement atm/z101 is also relevant for distinguishing Lys and Gln. This observation has demonstrated that the usefulness of low mass region information of TOF/TOF spectra in distinguishing isobaric Lys and Gln.
immonium ions; matrix-assisted laser desorption ionization tandem time-of flight mass spectrometry; lysine; glutamine; isobaric amino acids
2013-08-12;
2013-09-24
国家自然科学基金(21271131; 31070731)资助
李水明(1979~),男(汉族),湖南人,工程师,从事生物质谱分析研究。E-mail: 414004365@qq.com
王 勇(1970~),男(汉族),吉林人,副教授,从事生物质谱分析研究。E-mail: wyong@szu.edu.cn
时间:2014-03-24; 网络出版地址:http://www.cnki.net/kcms/doi/10.7538/zpxb.youxian.2014.0018.html
O 657.63
A
1004-2997(2014)03-0256-06
10.7538/zpxb.youxian.2014.0018

