肠炎沙门氏菌血清型特异性基因挖掘及生物信息学分析
余水静, 彭艳平, 邓扬悟, 郭燕华, 梁长利, 欧阳城添
(江西理工大学资源与环境工程学院,江西 赣州341000)
肠炎沙门氏菌 (Salmonella enterica serovar Enteritidis,SE)是肠道沙门氏菌(S.enteriea)重要血清型之一,能引起畜禽的胃肠炎以及人类肠炎[1].SE已成为近年来引起胃肠炎和食物中毒最为常见的病原菌之一.当前SE的检测鉴定主要基于传统方法,即增菌培养、选择分离、生化鉴定、血清分型,通常耗时5~7 d,不利于及时诊断病情控制疫情蔓延[2].鉴于SE严重威胁人类生命健康,国内外学者一直致力于SE快速检测技术的研究.采用PCR分子方法对沙门氏菌进行血清分型,只用一个简单的PCR反应在数小时内即可完成,省时省力、操作简便、成本低廉,而且结果判断准确[3-4],可满足SE流行病学研究和食品安全预警的实际需求.
PCR检测方法的准确度主要取决于检测基因的特异性,因此,挖掘特异性基因是构建PCR检测方法的关键.测序技术的快速发展使得越来越多的微生物基因组信息可利用,目前,GenBank数据库(http://www.ncbi.nlm.nih.gov/genomes/lproks.cgi)中已公布有45个完全测序的沙门氏菌菌株基因组信息.如此丰富的基因组信息有助于通过比较基因组学方法有效地挖掘SE血清型特异性基因.为此,拟利用比较基因组学工具挖掘出SE血清型特异性基因,并从生物信息学角度分析这些基因特征,以期为SE血清型分子鉴定提供检测靶点和理论依据.
1 材料与方法
1.1 基因组数据收集
从美国国立生物技术信息中心网站(National Center for Biotechnology Information, NCBI)(http://www.ncbi.nlm.nih.gov/genomes/lproks.cgi)收集已完全测序的沙门氏菌共45菌株基因组信息,包括1株SE、2株邦戈尔沙门氏菌(S.bongori)和42株肠道沙门氏菌(S.enterica),其中肠道沙门氏菌涉及21种血清型.
1.2 SE血清型特异性基因挖掘
利用文献[5]中特异性靶点挖掘平台SMM-system挖掘SE血清型特异性基因,具体过程为:去除SE全基因组序列,将其余44个沙门氏菌基因组并成一个基因组数据库;以SE的4318个蛋白编码序列(Coding Sequence,CDS)作为查询序列,进行BLASTN核酸序列比对,如果比对返回来的CDS最低e-value≥0.001,那么这个CDS对于数据库任何核酸序列很少或没有序列相似性,鉴定该CDS为特异性基因,对这些基因进行NCBI网站在线BLASTN核酸比对,最终确定SE血清型特异性基因,挖掘流程见图1.
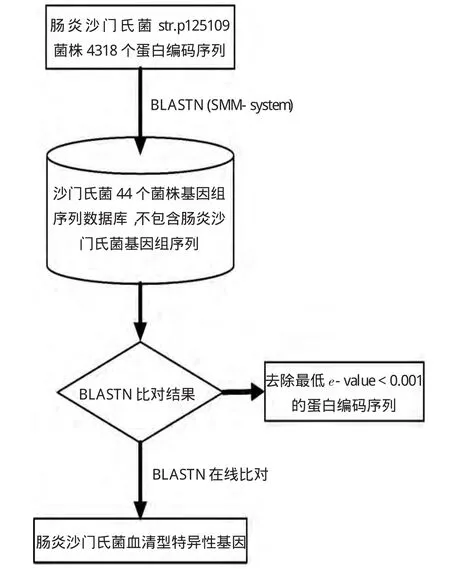
图1 SE血清型特异性基因挖掘流程
1.3 SE血清型特异性基因生物信息学分析
对鉴定的SE血清型特异性基因进行生物信息学分析,包括利用ProtParam在线工具计算SE血清型特异性基因的理化性质[6];蛋白二级结构使用Hnn Secondary Prediction Method程序预测[7];TMpredict进行蛋白质序列跨膜区分析[8];利用SignalP v4.0进行信号肽预测[9];利用PSORT进行亚细胞定位分析[10].将血清型特异性基因氨基酸序列提交至NCBI网站PDB数据库进行PSI-BLAST比对,以获得同源性PDB-ID,利用SWISS-MODEL在线服务器预测血清型特异性基因编码的蛋白三级结构[11].
2 结果与分析
2.1 SE血清型特异性基因鉴定
通过BLASTN序列比对,最终鉴定了6个SE血清型特异性基因(见表1),通过分析这些基因的功能注释,发现其中4个基因(SEN1382,SEN1936,SEN1945和SEN1959)编码的蛋白与噬菌体(phage)相关,而其余2个基因为假设蛋白(Hypothetical Protein).
2.2 SE血清型特异性基因理化性质分析
对6个SE血清型特异性基因编码的蛋白理化性质进行了分析 (见表2),等电点(Isoelectric Point,PI)分析显示:3个血清型特异性基因蛋白等电点小于7.0,呈酸性,其中SEN1388等电点最低(4.60);另外3个血清型特异性基因蛋白等电点大于7.0,呈碱性,其中SEN1945最高(9.13).脂溶指数(Aliphatic index)分析显示:其中4个血清型特异性基因蛋白脂溶指数大于100,为亲水性蛋白;其余2个血清型特异性基因蛋白脂溶指数均小于100,为疏水性蛋白.不稳定指数(Instability index)分析表明:3个血清型特异性基因蛋白不稳定指数大于40,为不稳定蛋白;其余3个血清型特异性基因蛋白不稳定指数小于40,为稳定蛋白.
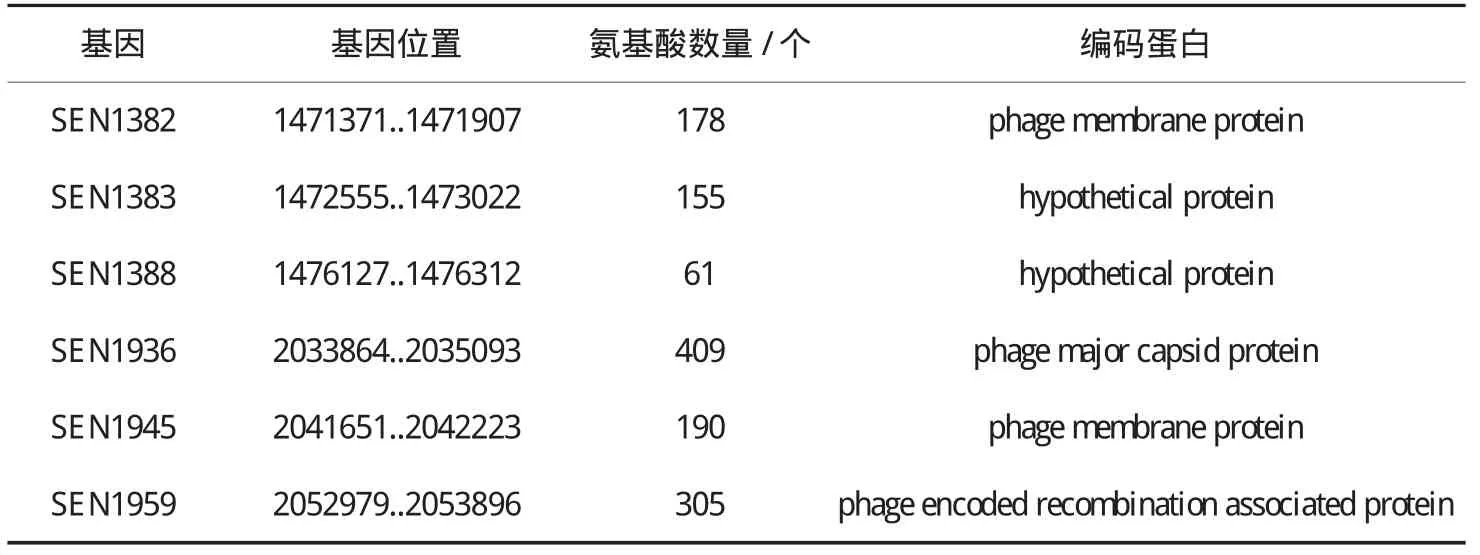
表1 挖掘的SE血清型特异性基因信息

表2 SE血清型特异性基因理化性质
2.3 SE血清型特异性基因二级结构分析
对6个SE血清型特异性基因蛋白质二级结构分析表明(见表3),这些血清型特异性基因蛋白二级结构均由α-螺旋 (Alpha helix)、扩展链(Extended strand)和无规则卷曲(Random coil)共 3种形式组成,其百分比均为:α-螺旋〉无规则卷曲〉扩展链.
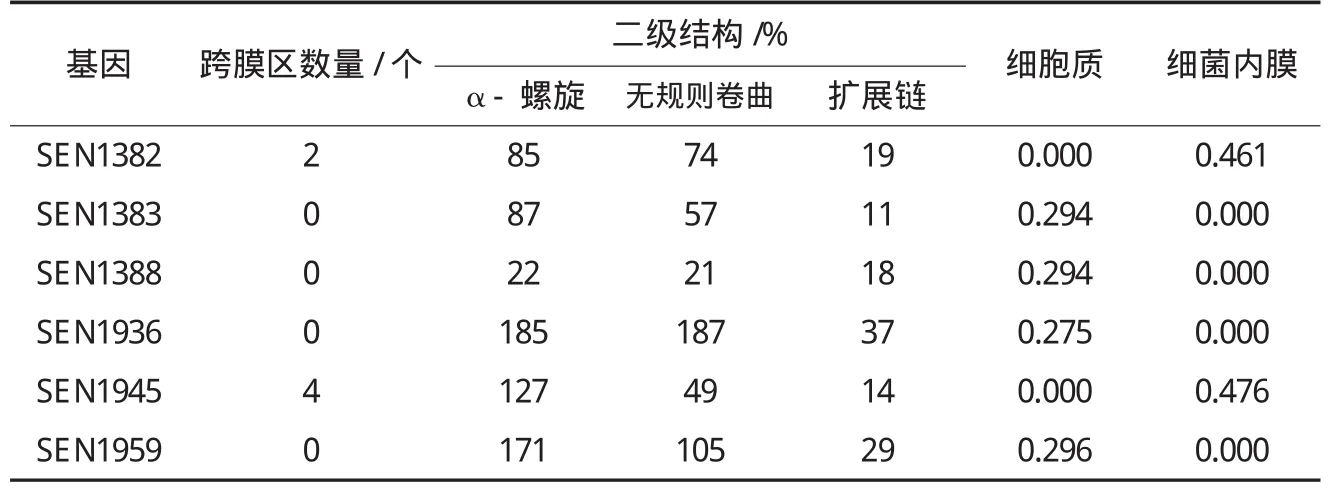
表3 SE血清型特异性基因二级结构、跨膜区及亚细胞定位预测
2.4 SE血清型特异性基因蛋白跨膜区预测和亚细胞定位分析
对6个SE血清型特异性基因编码的蛋白序列进行了跨膜区预测和亚细胞定位分析(见表3),跨膜区预测结果表明:SEN1382和SEN1945基因所编码的噬菌体膜蛋白 (Phage Membrane Protein)均存在跨膜区,其中SEN1382存在2个跨膜区,SEN1945存在4个跨膜区,预测结果与这两个基因功能注释均为膜蛋白相一致;其余4个SE血清型特异性编码的蛋白均不存在跨膜区.
亚细胞定位分析结果表明:SEN1382和SEN1945基因编码的噬菌体膜蛋白(Phage Membrane Protein)位于细菌内膜 (Bacterial Inner Membrane)上,其余4个基因编码的蛋白位于细胞质(Bacterial Cytoplasm)中,亚细胞定位结果进一步证实了跨膜区预测结果,并同时佐证了基因功能注释.此外,信号肽分析结果表明,所有6个SE血清型特异性基因编码的蛋白均无信号肽,可以推测这些基因编码的蛋白不能分泌到细胞外.
2.5 SE血清型特异性基因三级结构分析
将6个SE血清型特异性基因氨基酸序列提交至NCBI网站PDB数据库进行PSI-BLAST比对,获得6个同源性PDB-ID为:1ZTM_A(SEN1382),3KZ3_A(SEN1383),4M3N_A (SEN1388),4C98_A(SEN1936),3R9C_A(SEN1945),2OWL_A(SEN1959).利用SWISS-MODEL在线服务器预测SE血清型特异性基因编码的蛋白三级结构,结果只获得SEN1383和SEN1959两个基因的三级结构模型(图2),其余4个基因的三级结构没有预测结果.对这两个基因的三级结构预测模型进行质量评价,评估模型可信度区间为0~1之间(图3)[11].研究中只预测出两个基因的三级结构,可能与基因序列的特异性有关,在比对获得PDB-ID时发现其相似度不是很理想,因而还需其它分析方法进一步证实.
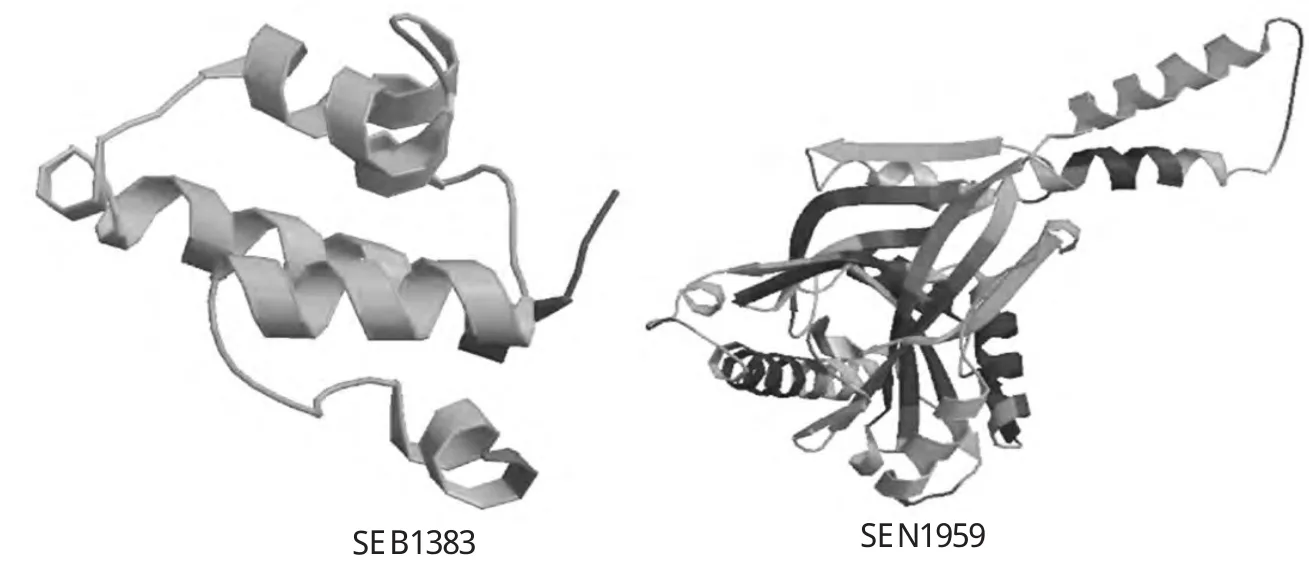
图2 SE血清型特异性基因SEN1383和SEN1959的三级结构模型

图3 SE血清型特异性基因SEN1383和SEN1959的三级结构模型评价
3 讨 论
沙门氏菌血清型已超过2600种,呈现出明显的多样性特征[12].研究表明,噬菌体在沙门氏菌血清型多样性形成过程中扮演着非常重要的角色[13].通过与鼠伤寒沙门氏菌 (S.enterica serovar Typhimurium LT2)基因组比较发现,SE含有独有的许多原噬菌体元件 (Prophage element)[14].挖掘出的SE血清型特异性基因有4个与噬菌体相关,与该报道观点一致.此外,鉴定的6个SE血清型特异性基因不存在其它沙门氏菌血清型菌株中,因而可以作为PCR检测靶标用于构建SE分子鉴定方法.
靶标序列的特异性是特异性检测SE的关键.一个多重PCR方法已构建用于特异性检测SE,其靶向基因为inv A,hil A,spv A,sdf和16S rRNA基因[15].由于靶标基因特异性问题,多重PCR方法避免了由单个基因单重PCR方法无法把所有血清型区分开的难题.然而,由于基因水平迁移或突变等原因,靶标的不稳定性问题也日益突出,因而需要更多的检测靶点来检测SE.研究中挖掘出的SE血清型特异性基因理论上可以用于构建PCR方法特异性检测SE,但仍需进一步设计引物构建PCR方法进行实验验证.
沙门氏菌的血清型由O抗原和H抗原以及Vi抗原联合所决定,抗原的差异导致了血清型的多样性.抗原的多态性是由其编码基因的多样性决定的,主要是rfb、fliC和fljB等基因及其基因簇中一些相关基因的序列存在着差异,甚至有些基因的编码序列完全不同而只有编码的蛋白的高级结构具有相似性[16].SE作为一个独立的血清型必然有独特的抗原相关基因,研究中挖掘的6个SE血清型特异性基因4个涉及到噬菌体相关蛋白,2个为假设蛋白.当前,并没有理由推测这些血清型特异性基因与O抗原、H抗原或Vi抗原的生物合成相关.因此,沙门氏菌血清型多样性的形成仍需从多角度研究.
4 结 论
(1)通过比较基因组学方法,挖掘获得了6个SE血清型特异性基因,可为鉴定SE分子鉴定提供靶标.
(2)鉴定的4个SE血清型特异性基因编码的蛋白与噬菌体相关,因而可以推测,噬菌体与沙门氏菌血清型多样性的形成密切相关.
(3)对SE血清型特异性基因的理化性质、二级结构、亚细胞定位、信号肽和跨膜区分析,显示出SE血清型特异性基因具有多样性特征,暗示了沙门氏菌血清型形成过程的复杂性.
[1]Garcia-Huidobro D,Carreno M,Alcayaga S,et al.Clinical and epidemiological description of severe outbreak of foodborne infection by Salmonella Enteritidis[J].Revista Chilena de Infectologia: Organo Oficial de la Sociedad Chilena de Infectologia,2012,29(2):132-137.
[2]曹际娟.食品微生物学与现代检测技术[M].大连:辽宁师范大学出版社,2006.
[3]Cardona-Castro N,Sanchez-Jimenez M,Lavalett L,et al.Development and evaluation of a multiplex polymerase chain reaction assay to identify Salmonella serogroups and serotypes[J].Diagnostic Microbiology and Infectious Disease,2009,65(3):327-330.
[4]Levy H,Diallo S,Tennant S M,et al.PCR method to identify Salmonella enterica serovars Typhi,Paratyphi A,and Paratyphi B among Salmonella Isolates from the blood of patientswith clinical enteric fever[J].JournalofClinicalMicrobiology,2008,46(5):1861-1866.
[5]Yu S,Liu W,Shi C,et al.SMM-system:A mining tool to identify specific markers in Salmonella enterica[J].Journal of Microbiological Methods,2011,84(3):423-429.
[6]Wilkins M R,Gasteiger E,Bairoch A,et al.Protein identification and analysis tools in the ExPASy server[J].Methods in Molecular Biology,1999,112:531-552.
[7]Combet C,Blanchet C,Geourjon C,et al.NPS@:network protein sequence analysis[J].NPS@:Network Protein Sequence Analysis,2000,25(3):147-150.
[8]Krogh A,Larsson B,Von Heijne G,et al.Predicting transmembrane protein topology with a hidden Markov model:application to complete genomes[J].Journal of Molecular Biology,2001,305(3):567-580.
[9]Petersen T N,Brunak S,Von Heijne G,et al.SignalP 4.0:discriminating signal peptides from transmembrane regions[J].Nature Methods,2011,8(10):785-786.
[10]Nakai K,Horton P.PSORT:a program for detecting sorting signals in proteins and predicting their subcellular localization[J].Trends in Biochemical Sciences,1999,24(1):34-36.
[11]Benkert P,Biasini M,Schwede T.Toward the estimation of the absolute quality of individual protein structure models[J].Bioinformatics,2011,27(3):343-350.
[12]Guibourdenche M,Roggentin P,Mikoleit M,et al.Supplement 2003-2007(No.47)to theWhite-Kauffmann-LeMinor scheme[J].Research in Microbiology,2010,161(1):26-29.
[13]Thomson N,Baker S,Pickard D,et al.The role of prophage-like elements in the diversity of Salmonella enterica serovars[J].Journal ofMolecular Biology,2004,339(2):279-300.
[14]Thomson N R,Clayton D J,Windhorst D,et al.Comparative genome analysis of Salmonella Enteritidis PT4 and Salmonella Gallinarum 287/91 provides insights into evolutionary and host adaptation pathways[J].Genome Research,2008,18(10):1624-1637.
[15]Trafny E A,Kozlowska K,Szpakowska M.A novelmultiplex PCR assay for the detection of Salmonella enterica serovar Enteritidis in human faeces[J].Letters in Applied Microbiology,2006,43(6):673-679.
[16]Liu D,Verma N K,Romana L K,et al.Relationships among the rfb regions of Salmonella serovars A,B,and D[J].Journal of Bacteriology,1991,173(15):4814-4819.

