人工湿地污水处理系统春季空气微生物群落结构分析
吴等等,宋志文,王 琳,徐爱玲,夏 岩 (青岛理工大学环境与市政工程学院,山东 青岛 266033)
人工湿地污水处理系统春季空气微生物群落结构分析
吴等等,宋志文*,王 琳,徐爱玲,夏 岩 (青岛理工大学环境与市政工程学院,山东 青岛 266033)
通过构建16S/18S rDNA基因文库,分析自由表面流人工湿地污水处理系统春季空气细菌和空气真菌群落结构特征.结果表明,空气细菌分布在变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)、蓝藻门(Cyanophyta)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes),主要为β-变形菌纲(71.04%)、γ-变形菌纲(12.03%)、α-变形菌纲(3.83%)、蓝藻纲(4.38%)、芽孢杆菌纲(3.28%)和鞘脂杆菌纲(2.19%),优势菌属是马赛菌属(Massilia 66.66%)、假单胞菌属(Pseudomonas 4.37%)、蓝丝细菌属(Cyanothece 3.83%)和沙雷氏菌属(Serratia 3.28%).空气真菌主要类群为座囊菌纲(Dothideomycetes 61.18%),其次是接合菌纲(Zygomycetes 16.47%)、盘菌纲(Discomycetes 14.12%),优势菌属是核腔菌属(Pyrenophora 48.31%)、被孢霉属(Mortierella 15.7%)、缘刺盘菌属(Cheilymenia 12.4%)、Boothiomyces (4.5%).人工湿地空气微生物中未检测出大肠杆菌(Escherichia coli)、沙门氏菌(Salmonella spp.)和产气荚膜梭菌(Clostridium perfringens),但存在粘质沙雷氏菌(S. marcescens)、恶臭假单胞菌(P. putida)、表皮葡萄球菌(Staphylococcus epidermidis)等致病菌或条件致病菌.
人工湿地;空气细菌;空气真菌;群落结构;基因文库
生活污水中含有沙门氏菌(Salmonella spp.)、志贺氏菌(Shigella spp.)、霍乱弧菌(Vibrio cholerae)和肠道病毒等多种病原微生物,其处理过程中由于污水流动、溢流跌落、搅拌或曝气产生的微生物气溶胶增加了污水处理厂工作人员及周边人群的健康风险[1].对污水处理厂工作人员和周边人群健康状态研究显示,呼吸道疾病、肠道疾病、污水源病毒感染等疾病与微生物气溶胶密切相关.
人工湿地具有净化污染物效果好、运行费用低、易维护等优点,在污水处理、污染物控制和改善环境等方面应用广泛[2].与传统的二级污水处理工艺相比,人工湿地微生物气溶胶对公众健康和生态安全的影响更应该引起足够重视[3],主要表现在:(1)随着城市化进程加快,原来远离城区的人工湿地距离商业居住区越来越近,随之也带来环境卫生安全问题.(2)与传统污水处理工艺的严格封闭式管理不同,人工湿地是相对开放的生态系统,部分人工湿地还作为旅游景点,接待游客.(3)在污水处理中,人工湿地通常作为二级或三级处理工艺,研究发现,即使作为三级污水处理工艺,人工湿地中仍可检测出一定数量的沙门氏菌和产气荚膜梭菌(Clostridium perfringens)[4].(4)人工湿地基质是污水中病原微生物的汇,在特殊情况下,人工湿地有可能成为周围环境病原微生物的源,并以微生物气溶胶形式传播[5].(5)研究表明,自由表面流人工湿地细菌气溶胶和真菌气溶胶易进入肺部的细菌和真菌气溶胶(0.65~4.7µm)颗粒数分别占总数的22.2%~62.3%和54.2%~87.6%[6]. (6)除生活污水外,人工湿地内恒温动物和鸟类粪便也可能成为病原微生物的源,并且存在季节性变化,有较大的不确定性.(7)人工湿地植物叶片可以成为真菌孢子栖息和繁殖场所,使得人工湿地既是真菌孢子的汇也是周边环境的源[7].
目前,国内外一些学者已在污水处理系统空气微生物检测、曝气系统对微生物气溶胶影响、不同处理单元微生物气溶胶浓度变化、工艺改造对微生物气溶胶的影响、微生物气溶胶季节变化及控制措施等领域开展了一些工作[8-17].这些研究大多采用传统微生物培养方法,由于“可培养类”仅占空气微生物总数的不到1%[18-19],并且还有部分微生物处于活的不可培养状态,同时培养结果受培养基组分影响较大,导致测得的数据与实际情况有较大偏差.相对于纯培养法,分子生态学手段能够克服上述缺陷[20],但目前尚未见到利用分子生态学手段研究人工湿地空气微生物群落结构的报道.
本研究以自由表面流人工湿地污水处理系统为研究对象,通过构建 16S/18S rDNA基因文库,对春季空气细菌和空气真菌群落结构组成和系统发育进行分析,确定优势菌群,明确其中致病菌及条件致病菌种类,以期为其环境卫生评价提供参考.
1 材料与方法
1.1 研究地点概况
人工湿地污水处理系统位于山东省青岛市,东面和南面临黄海,西面为人工湿地进水前的倒置A2O预处理区,北面为住宅区.人工湿地为自由表面流芦苇湿地,由99个并联运行湿地单元组成,每个单元大小为140m×32m,总占地面积76.7ha,处理规模 3×104m3/d,污水来源为生活污水和工业废水(约1:1).
1.2 空气样品采集
采样时间为2013年5月,采用KC-6120空气综合采样器采集空气微生物,空气流量100L/min,采样时间持续 3d.采样结束后取下滤膜,用灭菌生理盐水冲洗滤膜,使沉积微生物粒子溶于其中,然后12000r/min离心20min,浓缩微生物粒子于2mL离心管,作为实验样品备用.
1.3 DNA提取
用DNA提取试剂盒(OMEGA)提取上述样品中环境总DNA,操作过程参照DNA提取试剂盒说明书.DNA浓度和纯度通过Nanodro检测.
1.4 16S/18S rDNA序列PCR扩增
采用细菌通用引物 27f/1500R[21](上游: 5'-AGAGTTTGATCCTGGCTCAG-3';下游:5'-AGAAAGGAGGTGATCCAGCC-3');真菌通用引 物 EF4f/EF3r[22](上 游 :5’-GGAAGGGGT GTATTTATTAG-3’;下游:5’-TCCTCTAAATGACCAGTTTG-3’)扩增空气微生物16S/18S rDNA片段.PCR 反应体系:ddH2O19µL,上游引物(5µmol/L)2µL,下游引物(5µmol/L)2µL,DNA样品2µL,Master mix 25µL,总体积50µL;PCR反应程序:94℃预变性 5min,94℃变性 45s,58.5退火45s,72℃延伸 90s,共 36个循环;72℃延伸10min;4℃保存.PCR产物用经EB染色的1%琼脂糖凝胶电泳检测.
1.5 16S/18S rDNA克隆文库构建
用琼脂糖凝胶回收纯化试剂盒(OMEGA)对PCR产物进行回收纯化,将纯化后的DNA片段通过T4连接酶克隆试剂盒(MBI Fermentas)连接在载体上,转化到感受态大肠杆菌(Top10)细胞中,涂布到含有氨苄青霉素、IPTG和 X-Gal的LB(Luria-Bertani)培养基上,37℃培养 16h.随机选取一定数量白色克隆子,采用菌体直接扩增方式,用pTZ57R/T Vector通用引物M13/PUCR和M13/PUCF扩增外源插入片段,重新获得16S/18S rDNA片段.菌落PCR扩增程序:94℃预变性5min;94℃变性45s,55℃退火45s,72℃延伸1.5min,共36个循环;72℃延伸10min;4℃保存.通过含有EB的1%琼脂糖凝胶电泳筛选含有插入片段的克隆子,完成克隆文库构建.
1.6 RFLP分析
用限制性核酸内切酶HhaI(Thermo, FD1854)消化从各克隆子扩增的16S rDNA片段,用限制性核酸内切酶 RsaI(Thermo, FD1123)消化 18S rDNA片段.酶切反应体系:ddH2O5.25µL、10×酶切缓冲液 1µL、PCR产物 3.5µL、HhaI(RsaI) 0.25µL.酶切反应条件:37℃ 20min,80℃ 5min,4℃保存.用含有EB染色的3%琼脂糖凝胶分离酶切片段,200V下电泳 2.5h,用凝胶成像仪拍照,并对琼脂糖凝胶电泳图谱进行分析,将每个酶切分型结果作为一个OUT (operational taxonomic unit).
1.7 多样性指数分析

表1 克隆文库多样性指数与覆盖度计算方法Table 1 The Formulae for the analysis of genotypic diversity and community coverage
采用覆盖度(Coverage Value, C)、香农多样性指数(Shannon Diversity Index, H’)、辛普森指数 (Simpson Index, D)、 丰 富 度 (Species Richness)、均匀度(Species evenness, E)对16S/18S rDNA克隆文库进行微生物多样性分析.计算方法[23]见表1.
1.8 测序及文库分析
根据酶切分型结果,从每个OTU中选取一个克隆子对其插入片段测序.测序委托生工生物工程(上海)股份有限公司完成.测序结果在NCBI中利用 BLAST程序(http://www.ncbi. nlm.nih. gov/BLAST/)查找相似序列,分析和鉴定克隆文库细菌、真菌群落结构组成,并采用MEGA5.0软件进行细菌、真菌系统发育分析(建树模型为kimura 2-parameter, bootstrap值为1000).
2 结果与分析
2.1 克隆文库构建和RFLP分析
人工湿地空气微生物16S/18S rDNA PCR产物琼脂糖凝胶电泳图谱如图 1所示.扩增片段长度均在1500bp左右,电泳条带均一、信号较强,能够满足PCR产物胶回收纯化要求.
人工湿地空气微生物16S/18S rDNA克隆文库部分菌落PCR产物琼脂糖凝胶电泳图谱如图2所示, PCR片段长度分别为1600bp、1700bp,大部分电泳条带均一且信号较强,但有少部分菌落 PCR产物电泳条带信号较弱且不均一,同时也存在一些假阳性克隆.因此,根据菌落 PCR产物琼脂糖凝胶电泳图谱,挑选信号较强且均一性好的电泳条带对应的克隆子,完成克隆文库构建.
人工湿地空气微生物16S/18S rDNA基因文库部分菌落PCR产物酶切产物电泳图谱如图3所示.电泳条带较丰富,且位置差异性较大,能够达到区分空气不同类型细菌/真菌的目的.酶切分型结果表明,人工湿地空气细菌16S rDNA克隆文库包括190个克隆子,被分为54个OTUs;空气真菌18S rDNA克隆文库包括89个克隆子,被分为14个OTUs.

图1 16S/18S rDNA PCR产物电泳图谱Fig.1 Agarose gel electrophoresis of 16S/18S rDNA gene of bacteria and fungi
图2 16S/18S rDNA克隆文库部分菌落PCR产物电泳图谱Fig.2 Agarose gel electrophoresis of PCR products of 16S/18S rDNA clone libraries
图3 16S/18S rDNA克隆文库部分菌落PCR产物酶切电泳图谱Fig.3 Partial restriction fragment length profiles of 16S/18S rDNA fragments: The 16S/18S rDNA amplifer was digested with restriction endonucleases and analyzed by agarose gel electrophoresis
2.2 微生物群落结构和系统发育分析
克隆文库能够反映研究区域微生物群落结构和种群多样性,但由于方法本身局限性,其并不能包含实际环境中所有微生物类群,为了明确构建的克隆文库是否可以准确反映实际环境微生物群落结构组成和种群多样性,需要计算克隆文库各种多样性参数.其中,覆盖度(C)表示克隆文库所包含微生物种类占样品全部微生物种类的比例,其数值越大,表明所构建的克隆文库越能真实的反映该样品中微生物多样性特征;香农多样性指数(H’)、辛普森指数(D)、丰富度和均匀度(E)等均能从不同侧面反映克隆文库微生物多样性.表2是人工湿地克隆文库多样性参数,可以看出,细菌16S rDNA克隆文库包括190个克隆子,覆盖率85.3%,真菌18S rDNA克隆文库包括89个克隆子,覆盖率 91.0%,克隆文库包含了大部分空气细菌和真菌类群,能够代表环境样品微生物多样性16S rDNA克隆文库Shannon指数、丰富度指数和均匀度指数均大于 18S rDNA,说明人工湿地空气细菌多样性高于真菌,且个体分配均匀性也较高.
根据人工湿地空气微生物 16S/18S rDNA克隆文库分析结果(表 3和表 4),构建空气细菌、空气真菌系统发育树(图4和图5).人工湿地春季空气细菌16S rDNA克隆文库中与已公布的模式菌株有着 81%~99%的相似性,其中, 72.1%克隆子序列能在 NCBI中找到相似度大于 97%的菌株. 18S rDNA克隆文库中与已公布的模式菌株有着 91%~99%的相似性,74.2%克隆子序列能在NCBI中找到相似度大于97%的菌株.
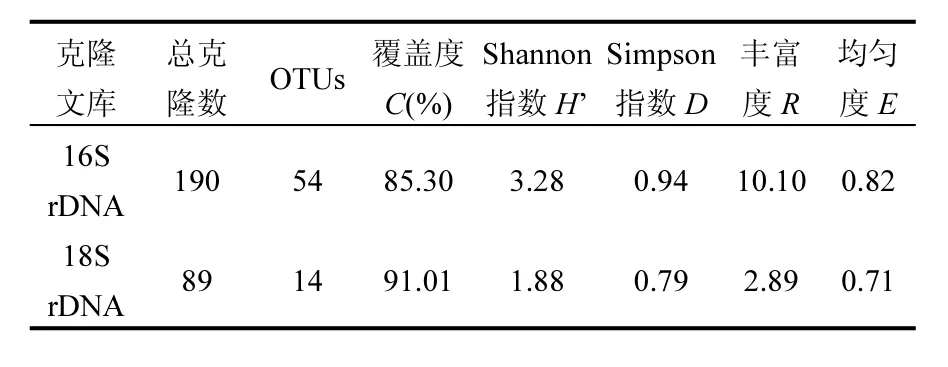
表2 克隆文库微生物多样性及覆盖度Table 2 Diversity index and coverage in 16S/18S rDNA clone library
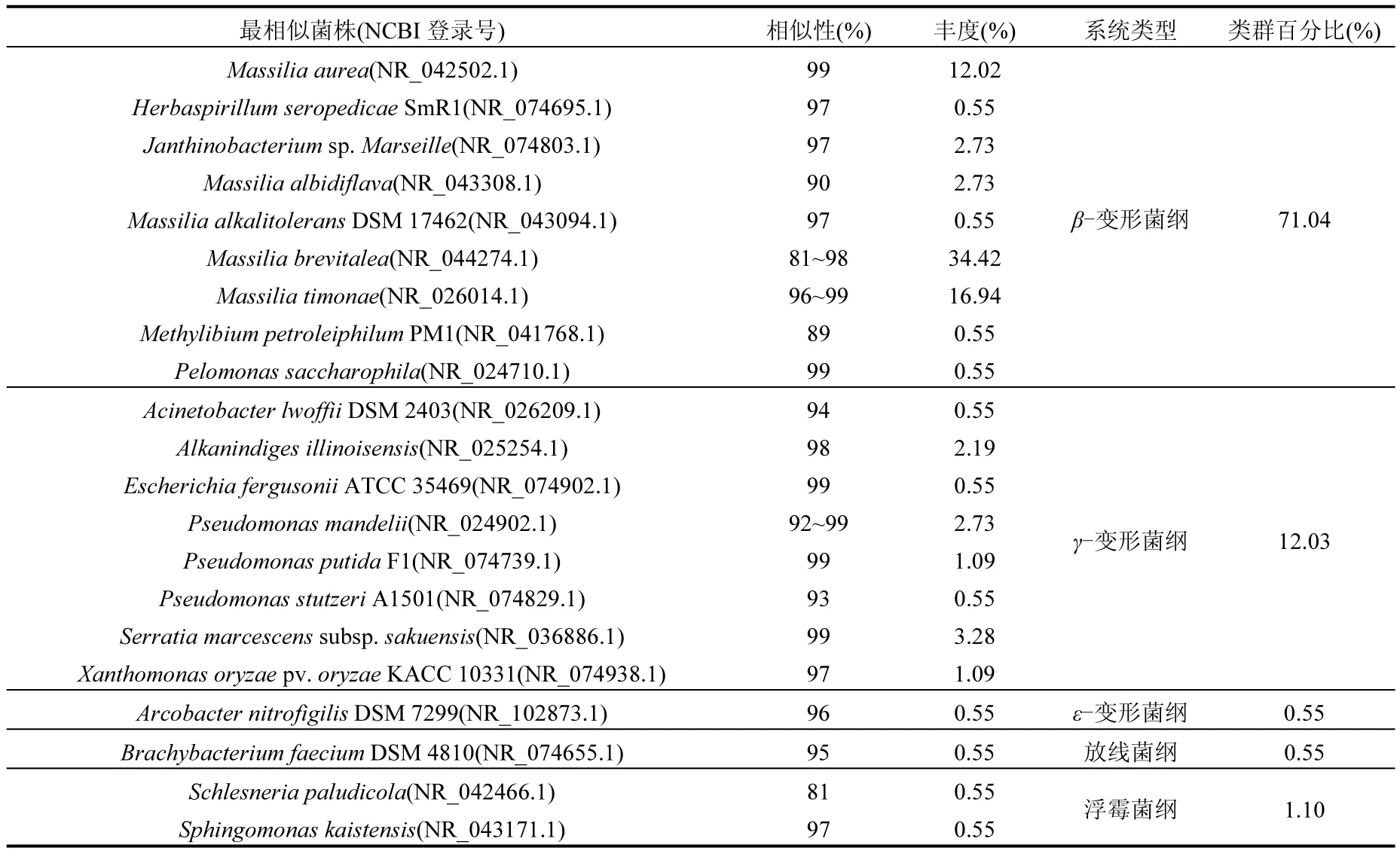
表3 人工湿地16S rDNA克隆文库分析结果Table 3 Inventory of bacterial 16S rDNA cloned fragments in constructed wetlands, arranged in groups according to their sequence similarities
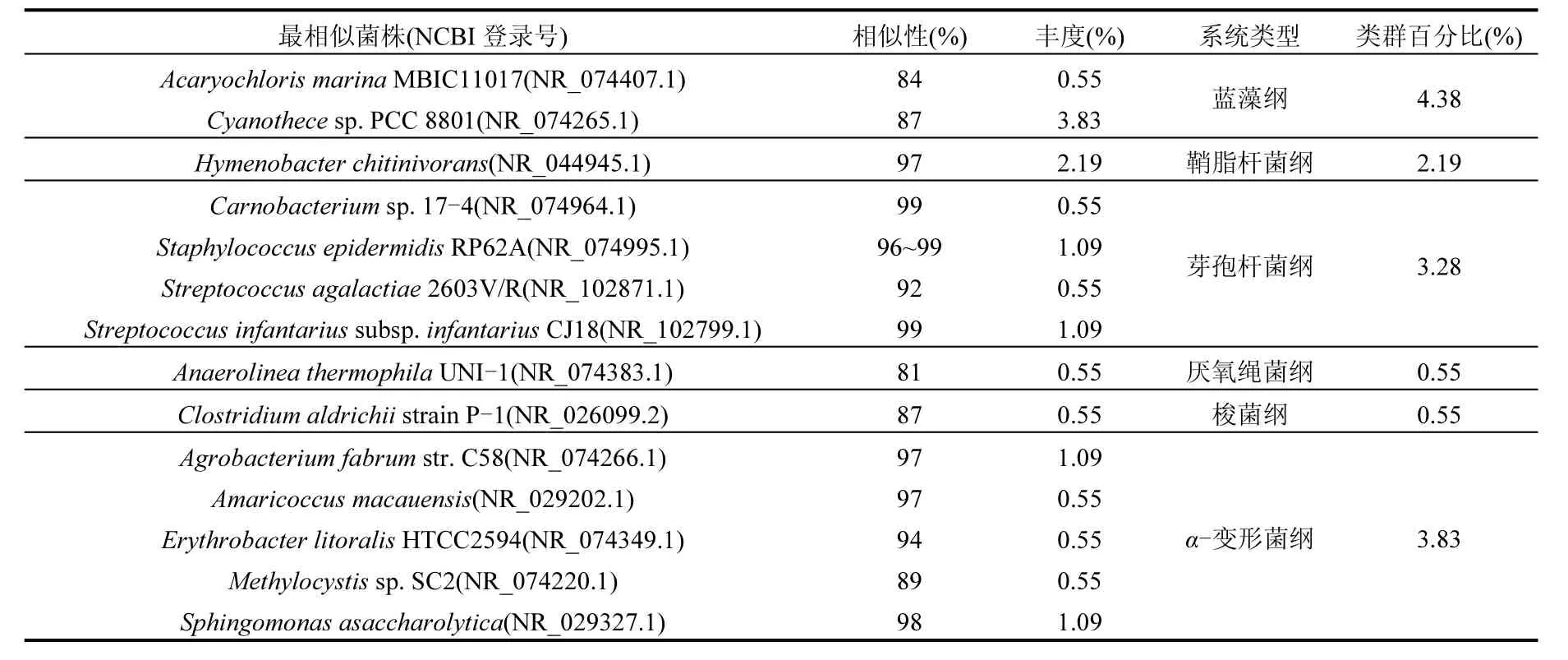
续表3
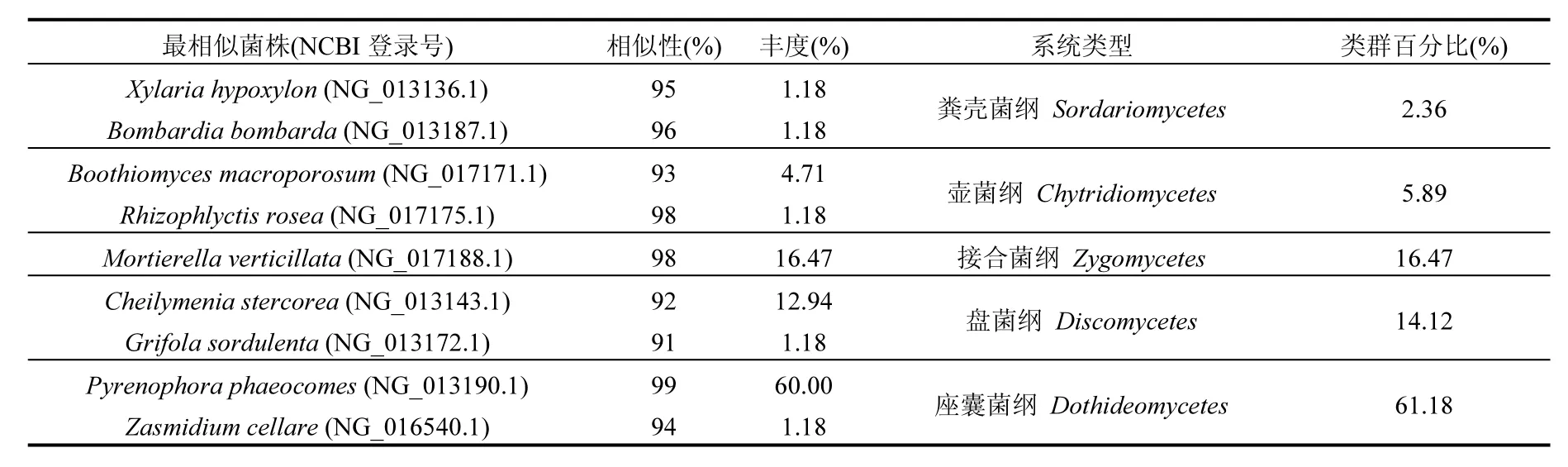
表4 人工湿地18S rDNA克隆文库分析结果Table 4 Inventory of fungal 18S rDNA cloned fragments in constructed wetlands, arranged in groups according to their sequence similarities
人工湿地空气细菌群落具有较高的生物多样性,分布在 7 个门,包括变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)、蓝藻门(Cyanophyta)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes).其中大部分属变形菌门中的β-变形菌纲(71.04%)、γ-变形菌纲(12.03%)和α-变形菌纲(3.83%),蓝藻门中的蓝藻纲(4.38%),厚壁菌门中的芽孢杆菌纲(3.28%),拟杆菌门中的鞘脂杆菌纲(2.19%).此外,还有少量属浮霉菌门中的浮霉菌纲(1.10%)、变形菌门中的 ε-变形菌纲(0.55%)、放线菌门中的放线菌纲(0.55%)、厚壁菌门中的梭菌纲(0.55%)和绿弯菌门中的厌氧绳菌纲(0.55%)的细菌.
空气真菌相似度最高的菌株分布在 5个类群中,座囊菌纲(Dothideomycetes61.18%)占绝对优势,其次是接合菌纲(Zygomycetes16.47%),盘菌纲(Discomycetes14.12%),壶菌纲(Chytridiomycetes5.89%)和粪壳菌纲(Sordariomycetes2.36%)相对较少.
3 讨论
16S/18S rDNA克隆文库法是微生物分子生态学中用来调查环境微生物组成的常用方法之一[24],突破了传统微生物分离纯化方法的局限性,能较全面揭示各种生态环境中微生物多样性[25].为了客观反映环境样品微生物群落结构,构建的克隆文库应包含环境样品中所有微生物,即库容值是100%.但Kemp等[26]分析比较文献中225个来自多种环境的 16S rDNA组成,发现随 16S rDNA克隆文库库容增大,检测到的OTU数目总在增加,说明在实际克隆文库构建过程中,由于PCR反应偏向性等因素影响,克隆文库不能穷尽样品中微生物种类和组成,覆盖度不可能达到100%.本研究中空气细菌和空气真菌 16S/18S rDNA克隆文库库容值分别为85.3%和91.0%,说明构建的克隆文库包括样品中大部分细菌和真菌种类,能够较好的反映人工湿地空气细菌和真菌群落结构.克隆子测序结果与 NCBI比对结果显示,空气细菌和空气真菌中分别有 72.1%和74.2%克隆子序列能在NCBI中找到相似度大于97%的菌株.一般认为,原核生物16S rDNA序列3%之内的变异属于种间变异[27],说明人工湿地空气微生物中存在部分未被认知的微生物种类.相对于传统培养方法,构建 16S/18S rDNA克隆文库方法能够获得对空气微生物群落结构多样性相对全面的认识,为空气中未知的微生物纯化培养及功能研究奠定基础.
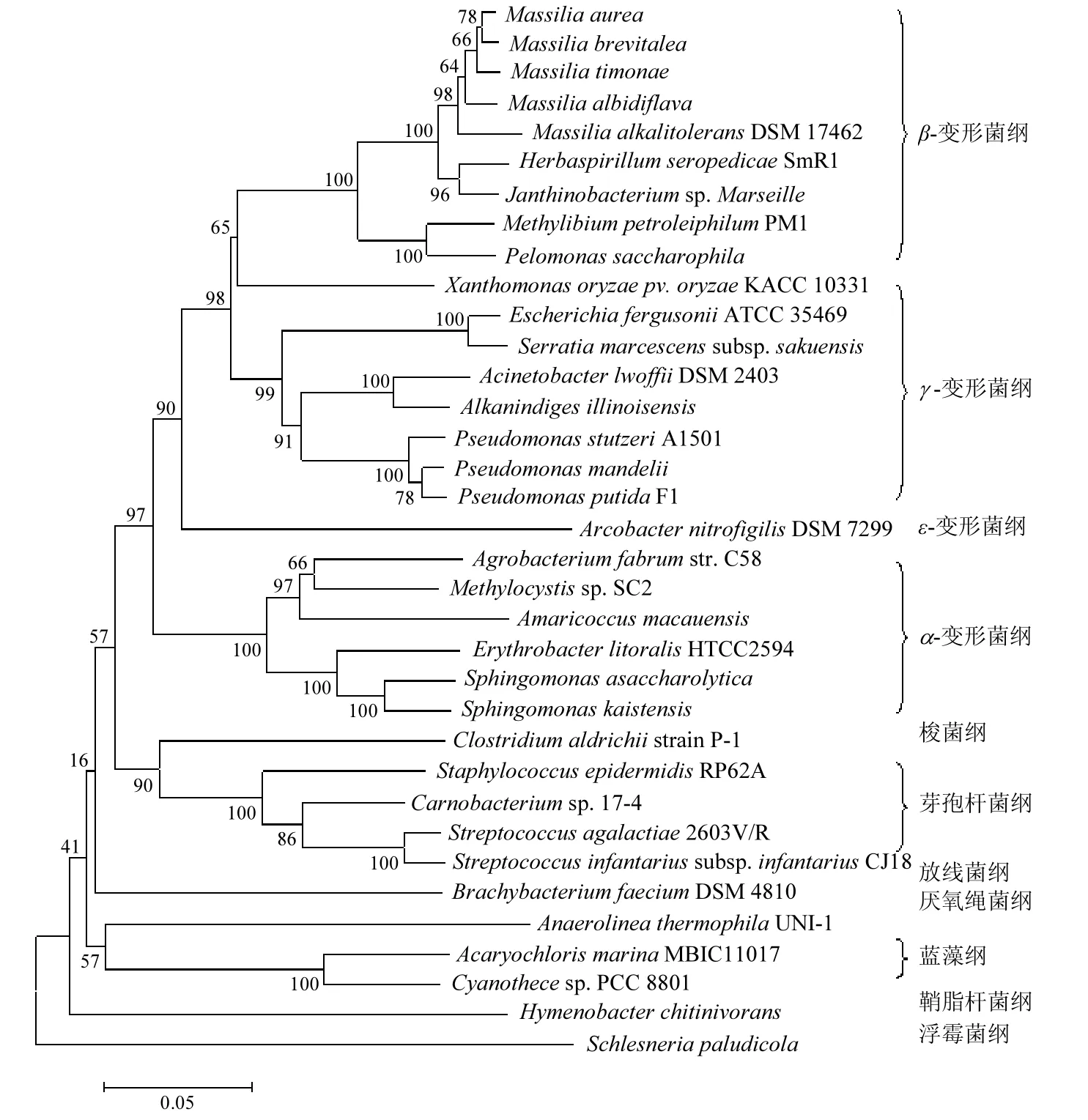
图4 人工湿地空气细菌系统发育树Fig.4 Phylogenetic tree based on 16S rDNA clones of airborne bacteria in constructed wetlands
人工湿地利用基质、植物、微生物的物理、化学和生物三重协同作用净化污水,其空气微生物群落结构与地理环境、社会环境及人类活动密切相关. 16S rDNA克隆文库分析结果表明,β-变形菌纲和 γ-变形菌纲属优势菌群,说明变形菌门细菌适合大气环境生长,这与国内外的研究一致[28-30].革兰氏阴性菌明显多于革兰氏阳性菌,符合革兰氏阴性菌可生活在低温,甚至嗜冷环境以及对辐射有抗性的结论[31-32].从人工湿地空气细菌菌属组成上看,马赛菌属(Massilia 66.66%)占绝对优势,其次是假单胞菌属(Pseudomonas 4.37%)、蓝丝细菌属(Cyanothece 3.83%)和沙雷氏菌属(Serratia 3.28%). Massilia以易降解有机物质为底物,属于生长速度较快的富营养菌[33],当环境中存在足够碳源和能源时,能够以高增殖率繁殖,是许多植物土壤根际的优势菌群[34-45],从戈壁和沙漠表层沙[46]、土壤[47-49]、湖泊(水库)沉积物[50-51]、垃圾填埋场气溶胶[52-53]、饮用水[54]中也检测到其存在,说明人工湿地基质(土壤)是空气微生物的重要源.假单胞菌在大气中普遍存在,是寄生于植物的潜在病原菌,表明植被也是人工湿地空气微生物的源[55].蓝丝细菌属对盐度具有一定耐受性[56],可能来源于海洋环境.粘质沙雷氏菌则广泛分布于自然界,是水和土壤中的常居菌群.

图5 人工湿地空气真菌系统发育树Fig.5 Phylogenetic tree based on 18S rDNA clones of airborne fungi in constructed wetlands
人工湿地空气微生物群落组成与传统污水处理系统存在一定差异.刘建伟等[16]对北京某污水处理厂研究发现,格栅间空气中异养细菌种类相对较多,包括芽孢杆菌、大肠杆菌、假单胞菌、葡萄球菌和短杆菌,污泥浓缩池和污泥脱水池优势异养细菌主要是假单胞菌、大肠杆菌和芽孢杆菌,且在不同时间内在各采样点所取的样品中,假单胞菌一直保持优势地位;余贵英等[17]对污水处理厂反应池、污泥浓缩池、厂前区等功能区微生物气溶胶研究表明,空气中主要为大肠杆菌、绿色链球菌、枯草芽孢杆菌、不动杆菌、变形杆菌和表皮葡萄球菌,污泥浓缩池主要为大肠杆菌.从本研究的分析结果看,在上述细菌类型中,除假单胞菌为人工湿地空气细菌优势菌属外,其他大部分种类虽然检测到,但均不占优势,并且未检测到大肠杆菌,只检测到同属的E.fergusonii.说明人工湿地与传统二级污水处理系统中空气微生物来源有较大差异,本研究地点位于海滨区域,空气微生物构成受到基质、污水、植被、鸟类与恒温动物、海洋等诸多因素影响,其空气微生物来源更为复杂.另外,不同研究方法也可能造成空气微生物群落结构差异.于淼等[6]采用传统培养方法对自由表面流人工湿地微生物气溶胶进行研究,发现空气真菌主要有酵母菌、镰刀菌属、枝孢属、毛霉属、交链孢属、肉座菌属、枝霉属、青霉属和曲霉属,而本研究结果表明人工湿地空气真菌优势菌属为核腔菌属、被孢霉属、Boothiomyces和缘刺盘菌属.
人工湿地空气中存在部分致病菌或条件致病菌.其中,粘质沙雷氏菌(S.marcescens)可引起泌尿系统、下呼吸道、伤口感染及菌血症,机体抵抗力低下、免疫功能不全、慢性虚弱人群容易感染;表皮葡萄球菌(S.epidermidis)常引起人类呼吸道、伤口、泌尿生殖道、眼部等感染;恶臭假单胞菌(P. putida)是人类重要的临床病源,已从败血症、化脓性中耳炎、角膜巩膜炎和痢疾样腹泻患者标本中分离出该菌;织片草螺菌(H. seropedicae)可导致菌血症.水稻黄单胞菌水稻致病变种(Xanthomonas oryzae pv. oryzae)可以引起水稻白叶枯病,是世界水稻生产上重要的细菌病害之一[57].值得注意的是,尽管前期研究发现人工湿地污水中存在一定数量的沙门氏菌和产气荚膜梭菌[4,58],从空气样品中并未检测到其存在,为此今后拟采用高通量测序和实时荧光定量PCR等高灵敏度方法进一步研究.
4 结论
4.1 人工湿地春季空气细菌群落具有较高的多样性,分布在7个门,其中β-变形菌纲和γ-变形菌纲属优势菌群.优势菌属为马赛菌属、假单胞菌属、蓝丝细菌属和沙雷氏菌属.空气真菌中座囊菌纲占绝对优势,其次是接合菌纲和盘菌纲, 优势菌属为核腔菌属、被孢霉属、Boothiomyces和缘刺盘菌属.
4.2 人工湿地春季空气微生物中未检测出大肠杆菌、沙门氏菌和产气荚膜梭菌,但存在粘质沙雷氏菌、施氏假单胞菌、表皮葡萄球菌等致病菌或条件致病菌.
4.3 人工湿地空气微生物中存在部分未被认知的种类.采用构建 16S/18S rDNA克隆文库方法分析污水处理系统空气微生物群落结构特征,其结果与传统微生物培养方法存在较大差异.
[1] Pillai S D, Ricke S C. Bioaerosols from municipal and animal wastes: background and contemporary issues [J]. Canadian Journal of Microbiology, 2002,48:681-696.
[2] Song Z W, Zheng Z P, Li J, et al. Seasonal and annual performance of a full-scale constructedwetland system for sewage treatment in China [J]. Ecological Engineering, 2006,26: 272—282
[3] 徐 敏,宋志文,杨 光,等.人工湿地与环境卫生安全 [J]. 生态学杂志, 2007,26(11):1873-1877.
[4] 孙 群,吴 蕾,夏文香,等.人工湿地中指示和病原微生物分布与衰减研究 [J]. 安全与环境学报, 2009,9(5):63-66.
[5] Karim M R, Manshadi F D, Karpiscak M M. The persistence and removal of enteric pathogens in constructed wetlands [J]. Water Research, 2004,38:1831-1837.
[6] 于 淼,孙 群,宋志文,等.自由表面流人工湿地微生物气溶胶研究 [J]. 环境污染与防治, 2010,32(2):8-12.
[7] Levetin E, Dorsey K. Contribution of leaf surface fungi to the air spora [J]. Aerobiologia, 2006,22(1):3-12.
[8] Patentalakis N, Pantidou A, Kalogerakis N. Determination of enterobacteria in air and wastewater samples from a wastewater treatment plant by epi-fluorescence microscopy [J]. Water, Air and Soil Pollution, 2008,8:107-115.
[9] Korzeniewska E, Filipkowska Z, Gotkowska-plachta A, et al. Determination of emitted airborne microorganisms from a BIOPAK wastewater treatment plant [J]. Water Research, 2009,43: 2841-2851.
[10] Heinonen-Tanski H, Reponen T, Koivunen J. Airborne enteric coliphages and bacteria in sewage treatment plants [J]. Water Research, 2009,43:2558-2566.
[11] Sanchez-Monedero M A, Aguilar M I, Fenoll R, et al. Effect of the aeration system on the levels of airborne microorganisms generated at wastewater treatment plants [J]. Water Research, 2008,42:3739-3744.
[12] Karra S, Katsivela E. microorganisms in bioaerosol emissions from wastewater treatment plants during summer at a Mediterranean site [J]. Water Research, 2007,41:1355-1365.
[13] Hung H F, Kuo Y M, Chien C C, et al. Use of floating balls for reducing bacterial aerosol emissions from aeration in wastewater treatment processes [J]. Journal of Hazardous Materials, 2010, 175(1-3):866-871.
[14] Fernan N L, Fedorak P M. Changes at an activated sludge sewage treatment plant alter the umbers of airborne aerobic microorganisms [J]. Water Research, 2005,39:4597-4608.
[15] 吴万宁,查 勇,王 强,等.南京地区冬夏季大气重污染个例对比分析 [J]. 中国环境科学, 2014,34(3):581-587.
[16] 刘建伟,周竞男,马文林,等.北京某城市污水处理厂微生物气溶胶污染特性研究 [J]. 环境污染与防治, 2013,35(6):1-4.
[17] 余贵英,赵振新,宋 宏,等.某污水处理厂微生物气溶胶污染调查 [J]. 疾病控制杂志, 1999,3(1):1-4.
[18] Brock T. The study of microorganisms in situ: Progress and problems [J]. Symposium of the Society for General Microbiology, 1987,41:1-17.
[19] Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation [J]. Microbiology Reviews, 1995,59(1):143-169.
[20] Grif fi n D W, Kubilay N, Kodak M, et al. Airborne desert dust and aeromicrobiology over the Turkish Mediterranean coastline [J]. Atmospheric Environment, 2007,41(19):4050-4062.
[21] Spröer C, Reichenbach H, Stackebrandt E. The correlation between morphogenetic classification of myxobacteria [J]. International Journal of Systematic Bacteriology, 1999,49:1255-1262.
[22] Smit E, Leeflang P, Glandorf B, et al. Analysis of fungal diversityin the wheat rhizosphere by sequencing of cloned PCR-amplified genes encoding 18S rRNA and temperature gradient gel electrophoresis [J]. Applied and Environmental Microbiology, 1999,65(6):2614-2621.
[23] 王 英,滕齐辉,崔中利,等.免耕水稻土壤中细菌多样性及其空间分布的研究 [J]. 土壤学报, 2007,44(1):137-143.
[24] Chen A, Imachi H, Sekiguchi Y, et al. Archaeal community compositions at different depths of a municipal solid waste landfill in Taiwan as revealed by 16S rDNA cloning analyses [J]. Biotechnology Letter, 2003,(25):719-724.
[25] Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation [J]. Microbial. Rev., 1995,59:143-169.
[26] Kemp P F, Aller J Y. Bacterial diversity in aquatic and other environments: what 16S rDNA libraries can tell us [J]. FEMS Microbiology Ecology, 2004,47:161-177.
[27] Stackebrandt E, Goebel B M. Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology [J]. International Journal of Systematic and evolutionary Microbiology, 1994,44(4): 846-849.
[28] 梁宗敏,杜 睿,杜鹏瑞,等.北京大气降水中细菌气溶胶的多样性研究 [J]. 中国环境科学, 2014,34(2):317-323.
[29] Despres V R, Nowoisky J F, Klose M, et al. Characterization of primary biogenic aerosol particles in urban, rural, and high-alpine air by DNA sequence and restriction fragment analysis of ribosomal RNA genes [J]. Biogeosciences, 2007,4:1127-1141.
[30] Maron P A, Lejon D P H, Carvalho E, et al. Assessing genetic structure and diversity of airborne bacterial communities by DNA fingerprinting and 16S rDNA clone library [J]. Atmos. Environ., 2005,39:3687—3695.
[31] Amato P, Parazols M, Sancelme M, et al. Microorganisms isolated from the water phase of tropospheric clouds at the Puy de Dôme: major groups and growth abilities at low temperatures [J]. FEMS Microbiol. Ecol., 2007,59:242—254.
[32] Christner B C, Mosley-Thompson E, Thompson L G, et al. Bacterial recovery from ancient glacial ice [J]. Environmental Microbiology, 2003,5:433-436.
[33] Li X, Rui J, Mao Y, et al. Dynamics of the bacterial community structure in the rhizosphere of a maize cultivar [J]. Soil Biology and Biochemistry, 2014,68:392-401.
[34] Ofek M, Hadar Y, Minz D. Ecology of root colonizing Massilia (Oxalo-bacteraceae) [J]. PLoS One, 2012,7:e40117.
[35] Hrynkiewicz K, Baum C, Leinweber P. Density, metabolic activity, and identity of cultivable rhizosphere bacteria on Saliχ viminalis in disturbed arable and landfill soils [J]. J. Plant Nutr. Soil Sci., 2010,173:747—756.
[36] Weinert N, Meincke R, Gottwald C, et al. Effects of genetically modified potatoes with increased zeaxanthin content on the abundance and diversity of rhizobacteria with in vitro antagonistic activity do not exceed natural variability among cultivars [J]. Plant Soil, 2010,326: 437—452.
[37] Brooks D D, Chan R, Starks E R, et al. Ectomycorrhizal hyphae structure components of the soil bacterial community for decreased phosphate production [J]. FEMS Microbiol. Ecol., 2011, 76:245-255.
[38] Gro¨nemeyer J L, Burbano C S, Hurek T, et al. Isolation and characterization of root-associated bacteria from agricultural crops in Kavango region of Namibia [J]. Plant Soil, 2012,356: 67-82.
[39] Pisa G, Magnani G S, Weber H, et al. Diversity of16S rRNA genes from bacteria of sugarcane rhizosphere soil [J]. Braz. J Med. BiolRes., 2011,44:1215—1221.
[40] Ulrich K, Ulrich A, Ewald D. Diversity of endophytic bacterial communities in poplar grown under field conditions [J]. FEMS Microbiol. Ecol., 2008,63:169—180.
[41] Kuffner M, De Maria S, Puschenreiter M, et al. Culturable bacteria from Zn- and Cd- accumulating Saliχ Caprea with differential effects on plant growth and heave metal availability [J]. J. Appl. Mibrobiol., 2010,108:1471—1484.
[42] Dohrmann A B, Tebbe C C. Effect of elevated tropospheric ozone on the structure of bacterial communities inhabiting the rhizosphere of herbaceous plants native to Germany [J]. Appl. Environ. Microbiol., 2005,71:7750—7758.
[43] Green S J, Michel F C, Hadar Y, et al. Contrasting patterns of seed and root colonization by bacteria from the genus Chrysobacterium and from the family Oxalobacteraceae [J]. ISME J., 2007,1:291—299.
[44] Abou-Shanab RAI, van Berkum P, Angel J S. Heavy metal resistance and genotypic analysis of metal resistance genes in Gram-positive and Gram-negative bacteria present in Ni-rich serpentine soil and in the rhizosphere of Alyssum murale [J]. Chemospehre, 2007,68:360—367.
[45] Abou-Shanab R A I, van Berkum P, Angle J S, et al. Characterization of Ni-resistant bacteria in the rhizosphere of the hyperaccumulator Alyssum murale by 16S rRNA gene sequence analysis [J]. World J. Microbiol. Biotechnol., 2010,26:101—108.
[46] An S, Coutea C, Luo F, et al. Bacterial Diversity of Surface Sand Samples from the Gobi and Taklamaken Deserts [J]. Microb. Ecol., 2013,66:850-860.
[47] Zhang W, Wang H, Zhang R, et al. Bacterial communities in PAH contaminated soils at an electronic-waste processing center in China [J]. Ecotoxicology, 2010,19:96-104.
[48] Ferrari B C, Binnerup S J, Gillings M. Microcolony cultivation ona soil substrate membrane system selects for previously uncultured soil bacteria [J]. Appl. Environ. Microbiol., 2005,71: 8714-8720.
[49] Nagy M L, Pe’rez A, Garcia-Pichel F. The prokaryotic diversity ofbiological soil crusts in the Sonoran Desert (Organ Pipe Cactus NationalMonument, AZ) [J]. FEMS Microbiol. Ecol., 2005,54: 233-245.
[50] Foti M J, Sorokin D Y, Zacharova E E, et al. Bacterial diversity and activity along a salinity gradient in soda lakes of the Kulunda Steppe (Altai, Russia) [J]. Extremophiles, 2008:12:133-145.
[51] Cheng W, Zhang J, Wang Z, et al. Bacterial communities in sediments of a drinking water reservoir [J]. Ann. Microbiol., 2014,64:875-878.
[52] Bru-Adan V, We´ry N, Moletta-Denat M, et al. Diversity of Bacteria and Fungi in Aerosols During Screening in a Green Waste Composting Plant [J]. Curr. Microbiol., 2009,59:326-335.
[53] Blatny J M, Ho J, Skogan G., et al. Airborne Legionella bacteria from pulp waste treatment plant: aerosol particles characterized as aggregates and their potential hazard [J]. Aerobiologia, 2011, 27:147-162.
[54] Gallego V, Sa′ nchez-Porro C, Garc′ a MT, et al. Massilia aurea sp.nov., isolated from drinking water [J]. Int. J. Syst. Evol. Microbiol., 2006,56:2449-2453.
[55] Fahlgren C, Bratbak G, Sandaa R A, et al. Diversity of airborne bacteria in samples collected using different devices for aerosol collection [J]. Aerobiologia, 2010,27:107-120.
[56] 陶天申,杨瑞馥,东秀珠.原核生物系统学 [M]. 北京:化学工业出版社, 2007.
[57] 张 帆,周永力.白叶枯病菌(Xanthomonas oryzae pv. oryzae)与水稻抗病基因识别的分子机理[J].中国水稻科学, 2013,27(3): 305-311.
[58] 宋志文,孙 群,徐爱玲,等.人工湿地中指示与病原微生物动态分布及相关性分析 [J]. 生态学杂志, 2013,32(1):91-97.
致谢:本实验的现场采样工作由藏云生工程师等协助完成,在此表示感谢.
Community structure of airborne microbes in spring in a constructed wetland system for sewage treatment.
WU Deng-deng, SONG Zhi-wen*, WANG Lin, XU Ai-ling, XIA Yan (College of Environmental and Municipal Engineering,Qingdao Technological University, Qingdao 266033, China). China Environmental Science, 2014,34(12):3164~3174
constructed wetland;airborne bacteria;airborne fungi;community structure;genomic library
X172
A
1000-6923(2014)12-3164-11
吴等等(1988-),女,甘肃省庆阳人,青岛理工大学硕士研究生,主要从事环境微生物方面研究.发表论文5篇.
2014-03-20
国家自然科学基金资助项目(31170509)
* 责任作者, 教授, songzhiwen@qtech.edu.cn

