中国汉族人群DNMT基因单核苷酸多态性与弱精子症的相关性研究
李 琦欧光鑫杨学习余庆锋叶禄伟毛向明*
1. 南方医科大学附属南方医院泌尿外科(广州 510515); 2. 肇庆市第一人民医院泌尿外科; 3. 南方医科大学生物技术学院抗体工程研究所; 4. 汕头市中心医院泌尿外科
中国汉族人群DNMT基因单核苷酸多态性与弱精子症的相关性研究
李 琦1△欧光鑫2△杨学习3余庆锋4叶禄伟1毛向明1*
1. 南方医科大学附属南方医院泌尿外科(广州 510515); 2. 肇庆市第一人民医院泌尿外科; 3. 南方医科大学生物技术学院抗体工程研究所; 4. 汕头市中心医院泌尿外科
目的探讨DNMT基因(DNA methyltransferase)单核苷酸多态性(SNP)与弱精子症男性不育的关系。方法本研究检测了中国南方人群中DNMT1 (rs2114724, rs2228611, rs2228612, rs8101866, rs16999593), DNMT2 (rs11254413),DNMT3A (rs1550117, rs11887120, rs13420827, rs13428812, rs6733301), DNMT3B(rs2424908,rs2424913)和DNMT3L (rs113593938)14个SNPs与弱精子症的相关性。运用Sequenom MALDI-TOFMS 平台技术对特发性弱精子症不育患者(病例组,n=195)和正常精子活力男性(对照组,n=184)精液样本DNA进行基因分型。通过遗传平衡检验(Hardy-Weinberg Equilibrium test, HWE)样本结果,卡方检验和logistic回归分析所有基因型分布和等位基因频率。结果这些检测的SNPs中,DNMT3A基因rs13428812位点与弱精子症相关(OR=1.54;95%CI:1.02-2.13;P= 0.039)。其余位点未检查出与弱精子症的相关性。结论 DNMT3A基因rs13428812位点可能与弱精子症密切相关,但是机制尚不明确。
弱精子症; DNA甲基转移酶; 多态性, 单核苷酸; 精子能动性; 汉族
DNA甲基化(DNA methylation)是一种复制后修饰,发生在绝大多数原核生物和真核生物基因组中,对维持基因表达、基因印记、染色体的完整和X染色体的失活都起重要的作用。在哺乳动物中,DNA甲基化是由DNA甲基转移酶(DNA methyltransferase,DNMT)建立和维持的。最新研究发现,异常的基因甲基化与男性不育和精子活力不足具有紧密的联系[1-4]。然而,弱精子症与DNA甲基转移酶基因单核苷酸多态性(SNP)的相关性研究很少。本文旨在研究DNMT基因14个SNPs位点是否与弱精子症男性不育相关。
材料与方法
一、研究对象
研究对象为在我院以不育为主诉就诊的精子活力不足的男性患者,所有患者均为婚后未行避孕措施1年以上而未能生育,均排除生殖道感染、精索静脉曲张、不良生活习惯、激素水平异常及系统性疾病等可能导致精子活力下降的因素,配偶检查未见明显异常。弱精子症的诊断按照1999年WHO的诊断标准[5],病例组的纳入标准:a级精子<25%并且(a+b)级精子<50%,其他精液参数无明显异常。对照组纳入标准为:任意一次精液检查提示精子活力正常(a级精子≥25或a级+b级精子≥50%),其他精液参数无明显异常者。所有纳入研究者均精子浓度>20×106/mL并且精液白细胞浓度<2×106/mL。共有195位弱精子症男性(年龄范围21~56岁)及184位正常男性志愿者(年龄范围21~56岁)纳入研究。该研究经本院伦理委员会批准,所有研究对象均予告知并签署知情同意书。
二、方法
(一)精液获取及精子活力分析
所有纳入研究对象均禁欲2~7d,手淫取出精液,置于37℃水浴箱内液化1h,采用MicroPtic公司生产的SCA精子质量分析仪对样本进行精液常规分析。
(二)精子提纯和DNA的提取
经液化及活力分析后的精液,采用50% percoll提纯精子,离心机2 000×g,离心15min,然后用1倍的PBS清洗2次备用。采用基因组DNA提取试剂盒(北京百泰克生物技术有限公司)提取精子总DNA,储存于-80℃。Nanodrop 分光光度计测定所提取基因组DNA的浓度与纯度。
(三)SNP位点选择和基因型分析
从公共单核苷酸多态性数据库(single nucleotide polymorphism database,dbSNP)[6]中选择DNMT基因的14个SNPs位点。14个SNPs位点的具体信息见表1。用Assay Design 4.0软件设计PCR引物(Sequenom, San Diego, CA),利用MassARRAY平台进行基因型分型(Sequenom, San Diego, CA)。DNA样品质控设置在90%水平。

表1 从DNMT基因中选择出的SNP位点
三、统计学分析
采用SPSS13.0统计软件处理数据,各SNP位点基因型频率的Hardy- Weinberg平衡适合度检验采用x2适合性检验,基因型和等位基因频率分布差异采用x2独立性检验,DNMTs基因型与弱精子症之间的相关性采用比值比(OR)和95%可信度区间(95%CI)分析。
结 果
在病例组和对照组中,我们总共分析了14个SNPs。基因型分析结果如表2。其中,有两个位点(rs2228612和rs11254413)的基因型分布频率不符合Hardy-Weinberg遗传平衡定律(P<0.05)。此外,DNMT3A基因rs13428812位点与弱精子症易感性密切相关。在病例组中,rs13428812位点基因型AA、GA-GG分布频率分别为49.2%、50.8%,对照组中分别为59.8%、40.2%,两组比较差异有统计学意义(P=0.039)。对比AA与GG,杂合子基因型AG可能会增加患弱精子症的风险(OR=1.54; 95%CI: 1.02-2.13;P= 0.039)。DNMT3L基因rs113593938位点可能与弱精子症密切相关(P=0.009),但是,在对照组中没有发现基因型TC的分布,所以OR值无法计算。其余位点未检出与弱精子症有相关性。
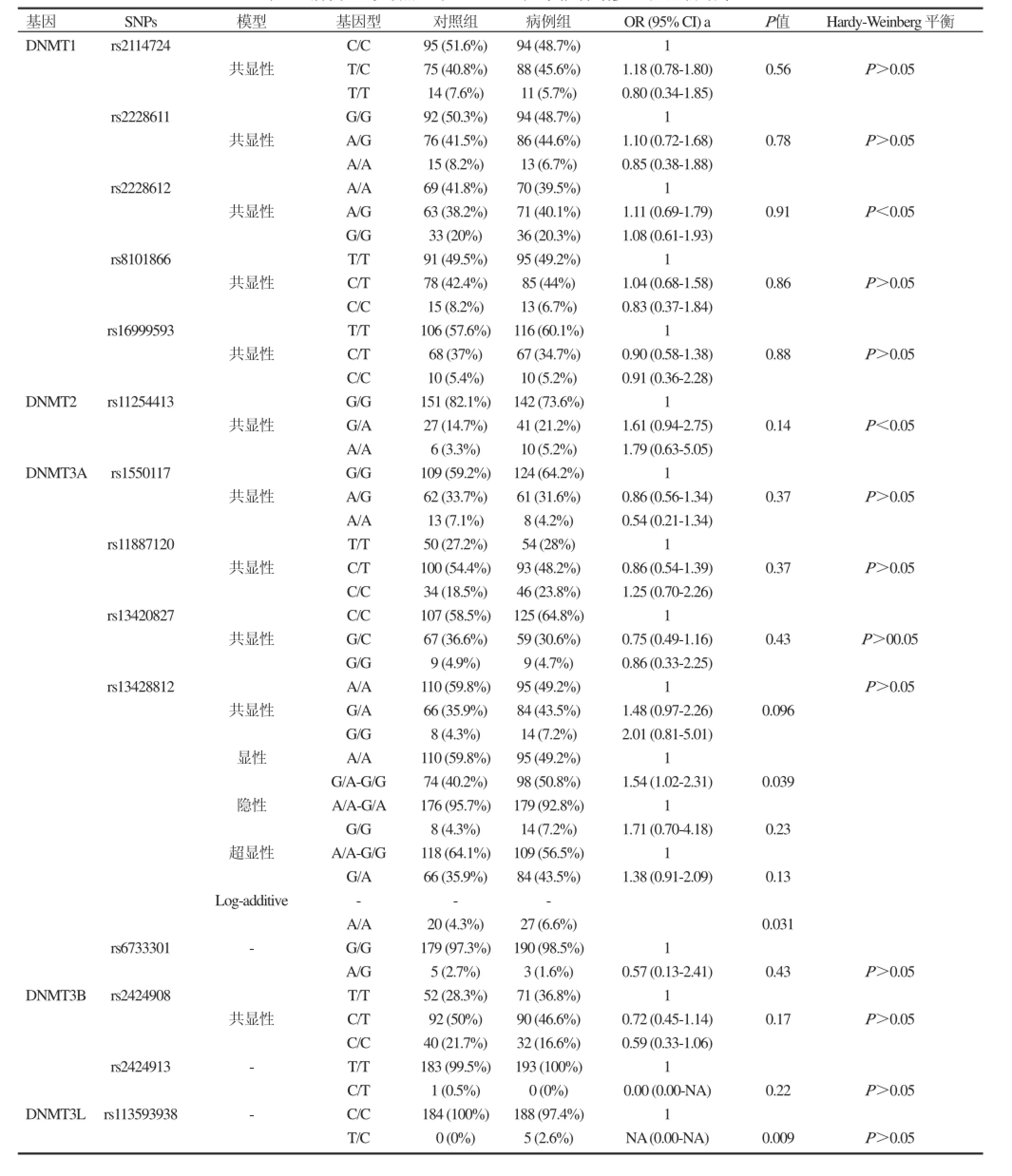
表2 病例组与对照组中DNMT基因单核苷酸多态性分析结果
讨 论
文献报道称目前15%的育龄夫妇存在不孕不育,其中男性因素占45%~50%[7]。Curi等[8]的研究发现弱精子症或精子活力不足是引起男性不育的常见因素。
DNA甲基化是表观遗传学的重要组成部分,在维持正常细胞功能、基因印记、胚胎发育以及人类肿瘤发生中起着重要的作用。目前有研究提示,男性不育症患者,包括少弱精子症、无精子症等,其精子中DNA甲基化有着明显的异常[9],人们推测,精子发生相关基因的异常DNA甲基化与不育之间有着密切的联系,并且有遗传倾向,造成子代不育。
异常的DNA甲基化主要发生于精子形成时。根据Pacheco等[10]的研究发现,CpG岛甲基化和mRNA的改变与精子活力不足相关。Nanassy等[1]研究发现CREM基因启动子异常甲基化会导致精液中鱼精蛋白异常和严重少精子症不育,同时,此甲基化作用越强,精液质量越差。Hammoud等[3]研究证实,与正常人相比,精液中鱼精蛋白异常和严重少精子症不育患者在印迹基因位点(LIT1, SNRPN, MEST, ZAC, PEG3)存在整体甲基化模式的显著改变。除此之外,研究还发现,H19, DAZL, MTHR基因的异常甲基化与精子形成密切相关[4,11,12]。
到目前为止,在哺乳动物中发现了三种DNA甲基转移酶,分别为DNMT1, DNMT2 和 DNMT3(DNMT3A and DNMT3B DNMT3L)[13]。DNMT1的主要作用是维持DNA甲基化,这种维持作用可以将DNA甲基化信息传递给子代细胞。DNMT2,主要为tRNA的甲基转移酶,其生物学功能尚不十分明确。目前认为,DNMT2的DNA甲基化酶活性非常弱,但是Goll等[14]的研究发现,它能够特异性甲基化tRNAAsp反密码子环38C。DNMT3A、DNMT3B具有类似的结构,被称之为从头甲基转移酶。DNMT3L是一种DNMT3类似蛋白,与DNMT3A和DNMT3B相结合,但是缺乏单独的催化活性。它能增强从头甲基转移酶的催化活性,并且只在处于从头甲基化发生的生殖细胞中表达[15]。
越来越多的研究表明,DNA甲基转移酶与精子发生密切相关[4,16-18]。DNMT3A在原始生殖细胞发育过程中起着至关重要的作用,其异常可能会导致DNA甲基化改变从而导致癌症的发生。La Salle等[16]在2004年的研究显示,DNMT3家族(DNMT3A, DNMT3B and DNMT3L)具有显著的基因独特性和生殖细胞系特有的表达模式,并且DNMT3A和DNMT3L在男性生殖细胞系的从头甲基化过程中存在相互作用。在随后2006年的研究中发现,DNMT3A在增殖和分化的男性生殖细胞甚至精子细胞中表达。La Salle的研究证实,在粗线期,无论是RNA水平还是蛋白质水平,DNMT3A的表达都是下调的[19]。Marques等[4]研究表明,在精子形成的整个过程中,DNMT3均有表达,作用可能是维持DNA甲基化模式以避免基因组印记错误的雄性配子的传递。然而,Lees Murdock等[17]却发现DNMT3家族仅仅在生殖细胞处于从头甲基化的时期出现。
虽然之前的很多研究均表明DNMT与精子发生密切相关,但是DNMT基因单核苷酸多态性与精子活力不足的相关性还未见报道。因此,我们的研究第一次证实了DNMT3A基因rs13428812位点与弱精子症男性不育可能存在密切关系。另外,如果样本量足够大的话,DNMT3L基因rs113593938位点也可能检出与弱精子症相关。但是,本次研究纳入的样本数较少,病例组与对照组均少于300例,且局限于中国南方人群,并且精液质量特性中一些变异基因型之间的差异性尚不清楚。因此,针对更大样本、不同地区人群以及体内外功能研究是非常必要的,以此来证实本文的结果及其探讨造成弱精子症的机制。
1 Nanassy L, Carrell DT. Abnormal methylation of the promoter of CREM is broadly associated with male factor infertility and poor sperm quality but is improved in sperm selected by density gradient centrifugation.Fertil Steril2011; 95(7): 2310-2314
2 Sato A, Hiura H, Okae H,et al.Assessing loss of imprint methylation in sperm from subfertile men using novel methylation polymerase chain reaction Luminex analysis.Fertil Steril2011; 95(1): 129-134, 134.e1-e4
3 Hammoud SS, Purwar J, Pf ueger C,et al. Alterationsin sperm DNA methylation patterns at imprinted loci in two classes of infertility.Fertil Steril2010; 94(5): 1728-1733
4 Marques CJ, Francisco T, Sousa S,et al.Methylation defects of imprinted genes in human testicular spermatozoa.Fertil Steril2010; 94(2): 585-594
5 WHO.WHO Laboratory manual for the examination of human semen and semen-cervica mucus interaction(Aed). Cambridge. London: Cambridge University Press,1999
6 NCBI SNP Database. Available online: http://www.ncbi. nlm.nih.gov/projects/SNP/ (accessed on 13 October 2011)
7 Jungwirth A, Giwercman A, Tournaye H, et al. European Association of Urology guidelines on Male Infertility: the 2012 update.Eur Urol2012; 62(2): 324-332
8 Curi SM, Ariagno JI, Chenlo PH,et al.Asthenozoospermia: analysis of a large population.Arch Androl2003; 49(5): 343-349
9 Carrell DT. Epigenetics of the male gamete.Fertil Steril2012; 97(2): 267-274
10 Pacheco SE, Houseman EA, Christensen BC,et al.Integrative DNA methylation and gene expression analyses identify DNA packaging and epigenetic regulatory genes associated.PLoS One2011; 6(6): e20280
11 Navarro-Costa P, Nogueira P, Carvalho M,et al. Incorrect DNA methylation of the DAZL promoter CpG island associates with defective human sperm.Hum Reprod2010; 25(10): 2647-2654
12 Wu W, Shen O, Qin Y,et al. Idiopathic male infertility is strongly associated with aberrant promoter methylation of methylenetetrahydrofolate reductase (MTHFR).PLoS One2010; 5(11): e13884
13 Goll MG, Bestor TH. Eukaryotic cytosine methyltransferases.Annu Rev Biochem2005; 74: 481-514
14 Goll MG, Kirpekar F, Maggert KA, et al. Methylation of tRNAAsp by the DNA methyltransferase homolog Dnmt2.Science2006; 311(5759): 395-398
15 Bourc'his D, Xu GL, Lin CS,et al.Dnmt3L and the establishment of maternal genomic imprints.Science2001; 294(5551): 2536-2539
16 La Salle S, Mertineit C, Taketo T,et al. Windows for sex-specif c methylation marked by DNA methyltransferase expression prof les in mouse germ cells.Dev Biol2004; 268(2): 403-415
17 Lees-Murdock DJ, Shovlin TC, Gardiner T,et al. DNA methyltransferase expression in the mouse germ line during periods of de novo methylation.Dev Dyn2005; 232(4): 992-1002
18 Takashima S, Takehashi M, Lee J,et al.Abnormal DNA methyltransferase expression in mouse germline stem cells results in spermatogenic defects.Biol Reprod2009; 81(1): 155-164
19 La Salle S, Trasler JM. Dynamic expression of DNMT3a and DNMT3b isoforms during male germ cell development in the mouse.Dev Biol2006; 296(1): 71-82
(2014-01-10收稿)
Association of DNMT Polymorphisms with Asthenozoospermia in a Chinese Han population
Li Qi1△, Ou Guangxin2△, Yang Xuexi3, Yu Qingfeng4, Ye Luwei1, Mao Xiangming1*
1. Department of Urology, Nanfang Hospital of Southern Medical University, Guangzhou 510515,Guangdong, China; 2. Department of Urology, the First People's Hospital of Zhaoqing;3. School of Biotechnology, Southern Medical University;
4. Department of Urology, Shantou Central Hospital
Mao Xiangming,E-mail:mxm@f mmu.com
ObjectiveTo investigate the association betweenDNMTgene polymorphism and asthenozoospermia.MethodsThe associations of 14 single nucleotide polymorphisms (SNPs) from DNMT1 (rs2114724, rs2228611, rs2228612, rs8101866, rs16999593), DNMT2 (rs11254413), DNMT3A (rs1550117, rs11887120, rs13420827, rs13428812, rs6733301), DNMT3B (rs2424908, rs2424913) and DNMT3L (rs113593938) with asthenozoospermia in the population of South China were assessed in the study. The SNPs genotyping was carried out in 195 idiopathic asthenospermia patients and 184 age-matched healthy volunteers using the Sequenom MALDI-TOF-MS platform. After HWE, the genotype and allele frequencies were calculated and analyzed with Chi-square test and logistic regression.ResultsAmong these SNPs, rs13428812 in DNMT3A was signif cantly associated with increased asthenozoospermia risk (odds ratio [OR], 1.54; 95% conf dence interval [95%CI], 1.02-2.13; P = 0.039). However, no other associations were found. Conclution These results indicate that DNMT3A (rs13428812) may contribute to asthenozoospermia, but the detail mechanisms were still unknown.
asthenozoospermia; DNA methyltransferase; polymorphism, single nucleotide; sperm motility; Han nationality
10.3969/j.issn.1008-0848.2014.05.004
R 698.2
△共同第一作者
*通讯作者, E-mail: mxm@f mmu.com

