两种鲨鱼鳃部真菌菌群的PCR-DGGE分析*
张 翼 穆 军 冯 妍 阎 松
两种鲨鱼鳃部真菌菌群的PCR-DGGE分析*
张 翼1, 2穆 军1, 3①冯 妍1阎 松1
(1. 大连交通大学环境与化学工程学院 大连 116028; 2. 大连理工大学生命科学与技术学院 大连 116024; 3. 浙江海洋学院海洋科学与技术学院 舟山 316022)
以提取自东海海域两种鲨鱼(尖吻鲭鲨和噬人鲨)鳃耙组织的总DNA为模板, 对其真菌菌群rDNA的基因内转录间隔区ITS1序列进行了PCR扩增, 扩增产物经变性梯度凝胶电泳(DGGE)分离, 电泳图谱中的7条主要条带经过二次PCR扩增后进行了克隆测序。NCBI-BLAST比对及分子系统发育分析结果表明, 尖吻鲭鲨鳃部真菌菌群的优势种来自四个分类单元, 其中一个来自青霉属, 三个来自曲霉属; 噬人鲨鳃部真菌的优势种来自四个分类单元, 其中一种来自青霉属、两个来自梗孢酵母属、一个来自枝顶孢属; 青霉属为两种鲨鱼鳃部共同的优势菌; 另外DGGE指纹图谱显示还有种类较丰富的劣势菌存在。数据库检索显示多数真菌分类单元具有较好的产生生物活性天然产物的潜力。该PCR-DGGE分析揭示鲨鱼鳃部栖生有较丰富的真菌类群, 它们的代谢产物及其与宿主之间的相互作用还有待深入研究。
鲨鱼; 鳃; 真菌菌群; ITS rDNA; 变性梯度凝胶电泳
在海洋动植物的体内或体表普遍栖生有真菌等各类微生物, 它们与宿主之间存在着共栖、共生或寄生关系。某些共附生微生物能产生一些具有细胞毒活性或抗菌活性的化学物质, 参与宿主对外界虫害、捕食者、污损生物或病原微生物的化学防御, 提高宿主的环境适应性和生存能力(Haygood, 1999; Armstrong, 2001; Taylor, 2007), 因此成为海洋天然产物化学研究的焦点资源, 其中共附生真菌因其代谢潜力强大而备受关注(Bugni, 2004; Saleem, 2007)。但目前国内外海洋共附生真菌等药源微生物的宿主来源主要集中于海绵、红树林、海藻等营固着生活的海洋动植物, 对于鱼类等营游泳生活的宿主的相关研究报道非常少(林永成等, 2003; Bugni, 2004; 周荣丽等, 2008)。
鲨鱼是体型较大的鱼类, 广泛分布于各大洋, 也是我国海域主要的软骨鱼类(朱元鼎等, 2001; 张清榕等, 2005)。它们的一些生理特点不同于硬骨鱼类, 如没有鳔, 而仅靠肝脏油脂浮力不足, 因此多数鲨鱼一直在积极游泳靠鳍提供升力以避免下沉, 在游泳时大流量的海水流经鳃裂给鲨鱼提供氧气, 以姥鲨为例, 每小时须过滤2000m3的海水(孟庆闻等, 1987; Maddalena, 2007; Knickle, 2011), 这非常有利于截获富集广泛海域内的丰富多样的海洋微生物。而且鳃组织充足的氧气、毛细血管网提供的丰富营养都非常有利于真菌等好氧海洋微生物的栖居生长, 可视为一种“自航式微生物采样器”, 但目前关于其药源微生物的研究报道非常少(王灿等, 2009)。
众所周知, 可培养微生物通常只占环境微生物总数的冰山一角, 而变性梯度凝胶电泳技术(DGGE)则可不依赖于传统的菌株分离培养策略, 可直接揭示样品中的微生物种类及其组成, 因此也被越来越多地用于研究珊瑚、海绵、海藻等海洋生物宿主中共附生微生物菌群组成(Bourne, 2005; Gao, 2008; Zuccaro, 2008)。由于鲨鱼鳃具有较明显的海洋微生物采集优势, 而其共附生微生物菌群组成的研究尚为空白, 本实验中运用PCR-DGGE技术分析了两种鲨鱼鳃部真菌菌群的组成, 以期对其生物多样性提供一个全景视角, 为进一步的药用真菌资源的开发及化学生态学的研究提供参考依据。
1 材料与方法
1.1 实验材料
两种鲨鱼鳃样品于2010年4月取自浙江省乐清市一水产品加工厂刚运输抵岸的东海海域渔获的新鲜鲨鱼, 使用无菌工具切割鳃耙样品, 无菌海水冲洗后密封于无菌容器中, 于4°C保存并迅即空运至实验室, 于超净工作台中用无菌海水冲洗后, 切割为小块后置于25%无菌甘油冻存液中-70°C密封冻存。取样时对鲨鱼拍照并测量其身体尺寸, 经大连自然史博物馆鱼类学专家赵永波研究员鉴定分别为尖吻鲭鲨()(本实验中编号为A)和噬人鲨() (本实验中编号为B)。
1.2 样品总DNA提取与纯化
各取5 g每种鲨鱼鳃样品放入灭菌研钵中, 加入液氮研磨至粉末状, 转入50 mL离心管中。加入5 mL CTAB提取液(100 mmol/L Tris·Cl, 100 mmol/L EDTA-Na2, 200 mmol/L NaCl, 2% CTAB, pH 8.0), 37°C振荡45 min。加入20% SDS至终浓度为2%, 65°C水浴2 h。然后用酚-氯仿法完成DNA的提取(Sambrook, 2002)。测定提取的总DNA浓度, 并用TE稀释至100 ng/mL。而后使用上海生工UNIQ-10离心柱式胶回收试剂盒对模板DNA进行纯化, 最终回收的DNA片段溶于40 μL TE缓冲液, 保存于-20°C备用。
1.3 PCR扩增
所用引物真菌ITS1-GC、ITS2-GC序列见表1, 反应条件参照Doare-Lebrun(2006)及Gao等(2008)的方法。反应体系为50mL总体积, ddH2O 40 μL, 10× Buffer (含2.0 mmol/L MgCl2) 5 μL, dNTP (10 mmol/L) 1mL, 正反引物各1mL (10mmol/L),酶(5 U/mL) 0.25mL, 模板DNA 2mL, 不足部分以ddH2O补齐。反应程序: 94°C 5min预变性; 94°C变性30 s, 54°C退火45 s, 72°C延伸1 min, 一共35个循环。最后延伸7 min。取PCR产物各2mL, 1.5%琼脂糖凝胶加5% Goldview染液, 120 V稳压电泳15 min, 检查扩增效果。
1.4 变性梯度凝胶电泳(DGGE)分析及条带测序
采用美国Bio-Rad公司D-CodeTM基因突变检测系统对A、B样品进行DGGE分析(龙良鲲等, 2005; Gao, 2008)。所用的聚丙烯酰胺凝胶浓度为8%, 变性剂浓度从15%—40% (100%的变性剂为7 mol/L尿素, 40%甲酰胺)。在70 V电压下, 60°C电泳13 h。而后用超纯水冲洗胶, 5% Goldview的染液染色30min, 凝胶成像拍摄图谱。用洁净的手术刀片切取代表性DGGE条带, 按上海生工UNIQ-10柱式PAGE胶DNA回收试剂盒(SK1135)方法回收, 以回收的DNA片段为模板, 用引物对ITS1和ITS2(表1)对其进行第二次PCR扩增, 反应程序同1.3。扩增产物使用1.5%琼脂糖凝胶电泳纯化, 切胶后使用SanPrep柱式DNA胶回收试剂盒回收DNA。使用T4 DNA连接酶将所得DNA片段连接到载体pUCm-T, 用生工SSCS快速一步法转化感受态大肠杆菌(SK2301), 以蓝白斑法挑选阳性克隆, 每个条带取3个克隆, 培养过夜后使用SK1191 UNIQ-10柱式质粒小量抽提试剂盒提取质粒DNA, 并交上海生工生物工程公司使用通用引物M13+/-正反向测序。所得序列均提交Genbank, 其登录号见表2。
1.5 序列比对及系统树构建
用BLAST 软件将测序结果与GenBank 中的相关序列进行同源性比较。并将序列结果及从Genbank中下载的相关种属真菌的ITS1序列利用Clustal X 1.81软件进行DNA 序列排列剪切, 使用MEGA 4.0软件构建NJ树、UPGMA树(Thompson, 1997; Tamura, 2007)。结合BLAST比对及聚类分析系统树结果, 确定优势条带所对应的分类单元。
表1 实验所用真菌引物

Tab.1 Fungal primers used in this experiment
GC: CGCCCGCCGCGCCCCGCGCCCGGCCCGCCGCCCCCGCCCC
2 结果
2.1 PCR扩增
如图1所示, 使用真菌rDNA通用引物对ITS1-GC、ITS2-GC从两种鲨鱼的鳃组织中都扩增出了较明亮的条带, 其长度约为320 bp, 符合真菌ITS1 rDNA区的长度特征, 这表明这两种鲨鱼鳃部都栖息有较丰富的真菌。实验中也曾使用通用引物对ITS1、ITS4试图扩增出ITS1-5.8S-ITS2全长序列, 但发现扩增条带亮度较为微弱, 不宜用于下一步的变性梯度凝胶电泳, 故未采用。
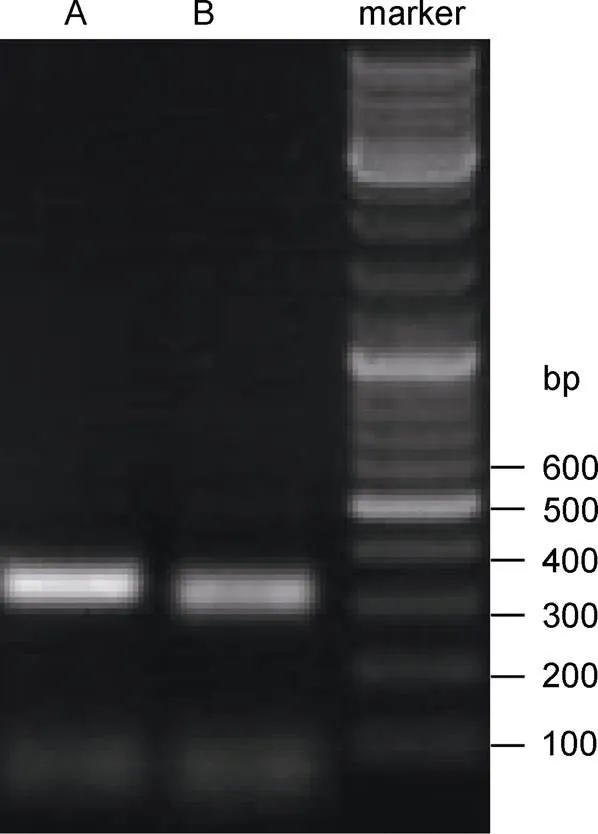
图1 两种鲨鱼鳃部真菌ITS1 rDNA及邻接区PCR扩增结果(A.尖吻鲭鲨; B.噬人鲨)
2.2 变性梯度凝胶电泳DGGE
将PCR扩增得到的产物(ITS1全长序列及相邻的上游18S和下游5.8S的部分序列)经变性梯度凝胶电泳后, 可用于分析两种鲨鱼鳃中的真菌菌群。如图2结果显示, 两种鲨鱼鳃都显示了较丰富的DGGE指纹, 样品A、B各含有4条较亮带, 这表明它们各自含有至少四种优势菌, 其中带1为两样品的共有条带, 除此之外, 两样品中还都显示了较丰富的微量条带, 该结果表明这两种鲨鱼鳃部均栖息有较丰富的真菌类群。而这两种鲨鱼鳃中的优势均又存在明显差异, 这可能与其生活环境有关, 也可能是由种间差异或个体差异造成的。
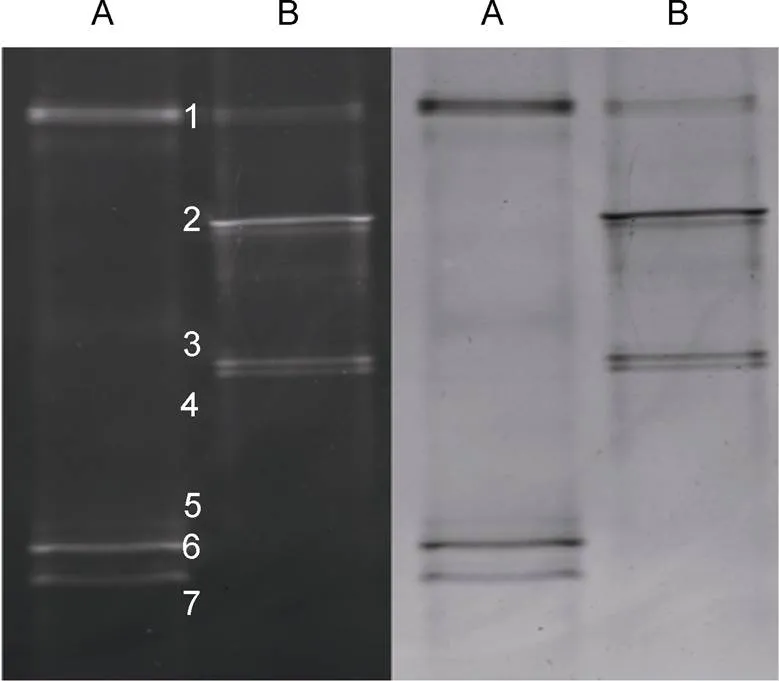
图2 两种鲨鱼鳃部真菌菌群的DGGE指纹图谱(左: 紫外成像; 右: 自然光成像。A.尖吻鲭鲨; B.噬人鲨)
为提高扩增反应的可靠性、提高测序成功率, 利用引物对ITS1、ITS2对上述优势条带进行了第二次PCR扩增, 如图3所示, 7条带均获得了较浓的扩增产物, 其序列长度符合真菌ITS1区特征。经电泳纯化、连接载体、转化大肠杆菌感受态细胞后, 从每个条带获得的3个阳性克隆提取质粒测序, 结果一致, 并将序列提交GenBank (表2), 通过BLAST与GenBank收录的真菌相应序列数据比对后, 发现带1为青霉属(), 带2、3为梗孢酵母属(), 带4为枝顶孢属(), 带5、6、7为曲霉属(), 它们与数据库中相应种属菌株序列的相似性达到了99%—100%。

图3 经第二次PCR扩增后的优势条带电泳图
由于BLAST结果显示每个条带与GenBank收录的相应属中多个种的序列相似性数值均相同, 为进一步了解优势条带所代表真菌的分类地位, 从GenBank数据库下载了相应属中多个种的该段DNA序列进行比较, 这些序列均来自一些主要菌种库, 如美国典型培养物保藏中心(ATCC)、美国农业部农业研究局菌种保藏中心(ARS/NRRL)、荷兰微生物菌种保藏中心(CBS)、英国真菌保藏中心(CABI)及丹麦科技大学真菌保藏中心(IBT)的保藏菌株。利用MEGA4.0软件将其与本实验中条带序列一起构建了系统树(NJ法、UPGMA法)。结果显示(NJ树见图4), 条带1中的DNA序列与产黄青霉()、灰黄青霉()及袋鼠鼠青霉()的DNA序列具有较近亲缘关系, 条带5、6、7的DNA序列与聚多曲霉()和肉色曲霉()的DNA序列具有较近亲缘关系, 条带4的DNA序列与基利枝顶孢霉()的DNA序列具有较近亲缘关系, 条带2、3的DNA序列与嗜盐梗孢酵母()的DNA序列关系较近。UPGMA树也显示相同亲缘关系(未附图)。
表2 DGGE优势条带序列BLAST比对结果

Tab.2 Sequences’ BLAST result of dominant DGGE bands
3 讨论
众所周知, 鲨鱼被发现患恶性肿瘤的几率非常低, 约为百万分之一, 其同类之间因争夺食物经常撕咬造成的外伤能很快愈合, 很少因此导致严重微生物感染, 这种海洋生物可能具有强大的化学防御机制(Lee, 1983; 贺丽虹等, 2005; Luer, 2012)。目前国内外对于鲨鱼化学防御及药用研究主要集中在鲨鱼自身成分如角鲨烯、鲨鱼肝及胃中的具有抗肿瘤和广谱抗菌活性的角鲨胺Squalamine以及鲨鱼软骨抗肿瘤成分的研究与开发(Moore, 1993; Cho, 2002; 贺丽虹等, 2005)。
鲨鱼是否存在其他化学防御机制, 如共附生微生物的协助防御呢?这是一个值得研究的问题。尽管迄今对于鲨鱼共附生微生物研究报道还比较少, 但已有研究表明共附生微生物是有可能参与鲨鱼的化学防御的。如郑忠辉等(1998)曾报道从鲨鱼的肠道中分离的一株弧菌和一株未鉴定菌株具有抑制金黄色葡萄球菌、大肠杆菌和鳗弧菌的活性; 另外王灿等(2009)报道23株分离自姥鲨消化道的细菌中有65%具有抗菌活性; Luer(2012)报道从鲨鱼的近亲——两种魟鱼的体表粘液中分离到的共生细菌中分别有12.0%或21.6%的菌株可拮抗与外伤感染相关的病原菌。上述王灿等的报道中曾提及从姥鲨鳃中分离到5株分类地位及生物活性不详的微生物, 但总而言之, 鲨鱼鳃作为一个新颖的海洋共附生药源微生物来源, 迄今国内外有关研究报道仍非常少。
本文研究表明, 在作者采样的两个鲨鱼样本鳃部的确生活有组成较丰富的真菌菌群, 且两者的优势菌群存在明显差异, 其中青霉属是它们共同的优势菌, 曲霉属为尖吻鲭鲨鳃部独有优势菌, 枝顶孢属和梗孢酵母属则为噬人鲨鳃部独有优势菌。其中青霉属、曲霉属、枝顶孢属隶属于子囊菌亚门, 而梗孢酵母属隶属于担子菌亚门。值得注意的是青霉属、曲霉属也是被报道最多的海洋真菌类群, 这被认为跟它们对于盐度的较强适应能力有关(Bugni, 2004)。另外, 曲霉、青霉也是最重要的生物活性次生代谢产物产生菌, 检索天然产物数据库Dictionary of Natural products on DVD (Buckingham, 2011)显示库中源自海洋青霉的天然产物多达235个(所有已收录青霉天然产物达1435个), 而源自海洋曲霉的多达244个(所有已收录曲霉天然产物达1524个)。枝顶孢属也是重要的天然产物来源, 来自整个自然界的枝顶孢属天然产物已达200个, 而源自海洋枝顶孢属的已达47个。梗孢酵母属最早是从海洋中发现的一个属(Fell, 1966), 但尚未见到有来自梗孢酵母属的天然产物收录。除上文所述优势菌外, DGGE指纹图谱显示这两种鲨鱼鳃中还含有丰富的微量真菌类群, 对这些微量条带区域的进一步PCR-DGGE分析有助于更多地了解鲨鱼鳃中真菌菌群组成, 而这些分类学背景将有可能用于指导选择性培养策略来富集培养尽可能多的可培养真菌, 或运用非培养依赖性的宏基因组技术来有针对性的克隆一些具有药用潜力物种的药源基因。
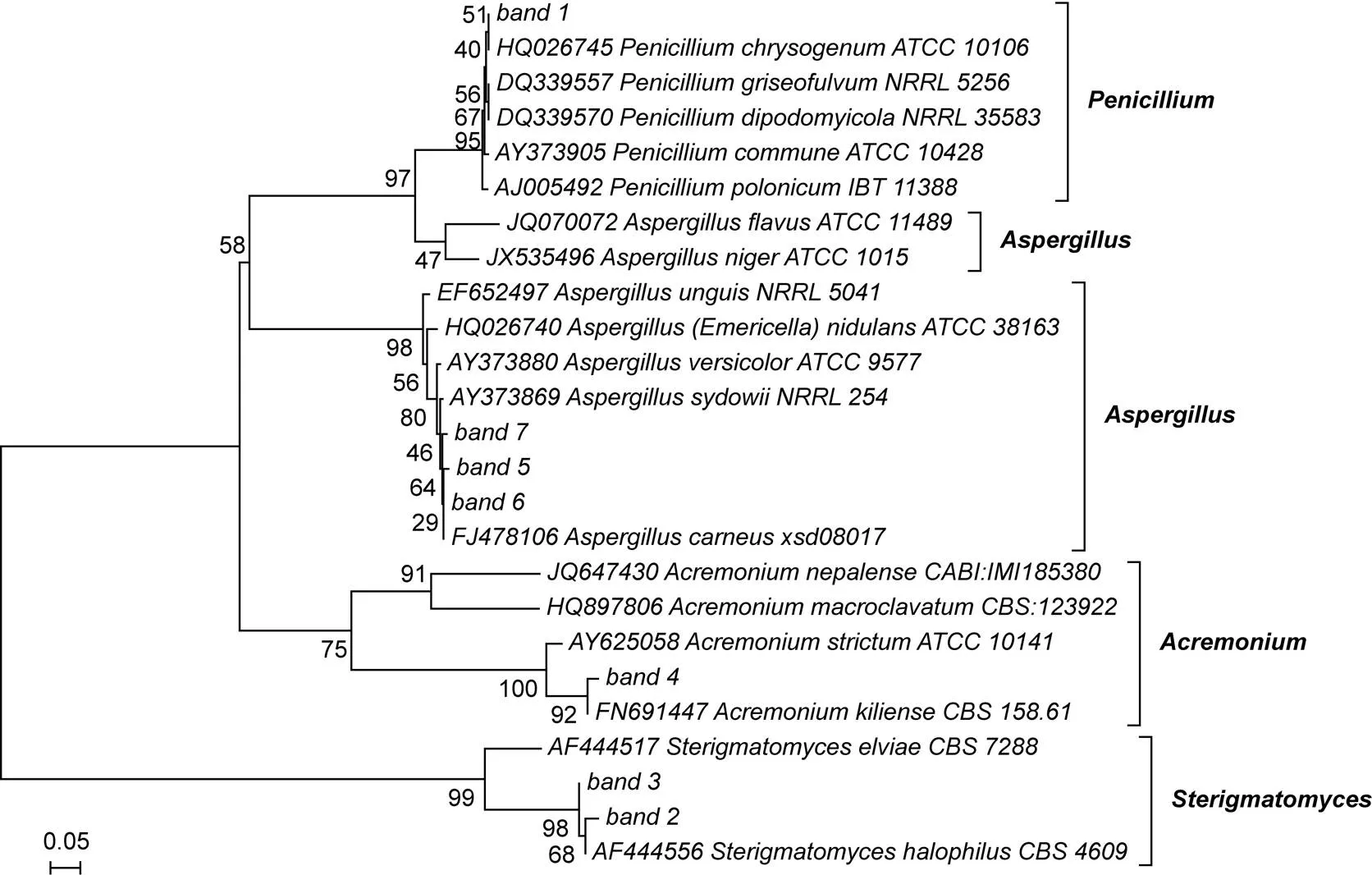
图4 基于优势真菌菌群ITS1及相邻序列构建的NJ聚类分析树(自展1000次验证节点置信度)
鲨鱼属于较特殊实验材料, 从活体鲨鱼鳃部现场即时取样具有一定危险性, 而死亡鲨鱼不排除受到空气中真菌的污染。不过, 由于鲨鱼鳃裂外皮对鳃耙具有一定封闭作用, 且本文鳃样品采自死亡时间不足一天且刚运抵港口的新鲜鲨鱼, 样品经过无菌水重复冲洗, 可能存在的附着不牢的少量陆源污染真菌被较大程度上排除, 经鉴定的DNA条带确属报道较多的海洋真菌类群, 且与GenBank中一些海洋来源菌株序列的相似性达到了99%—100%。此外, 大量获取新鲜鲨鱼样本也具有一定困难, 限于实验条件, 本文所采鲨鱼样本数量有限, 尚不足以说明这两条不同种鲨鱼鳃部真菌菌群的差异是来自于种间差别抑或个体差别。对于以上不足之处, 如条件具备, 仍以从大量活体鲨鱼样本现场即时取样更为科学严谨。另外, 噬人鲨和尖吻鲭鲨均广布于热带、亚热带和温带海洋, 活动范围广、行踪难溯, 关于其鳃部真菌菌群的差异与其生活环境的关系尚难做出推论。
PCR-DGGE作为一种可不依赖于菌株分离培养的技术, 对于海洋生物宿主中共附生微生物菌群组成的研究具有独特的优势。当然, 该技术也存在一定局限性, 如Vallaeys等(1997)报道过DGGE实验中具有不同序列DNA片段的共迁移现象, 因此为了得到可靠结果, 本文中对于DGGE条带采用克隆测序是确有必要的。另外动物宿主组织DNA样品成分较为复杂, 干扰因素多, 这可能也导致了本文中共附生真菌ITS1-5.8S-ITS2 rDNA长序列扩增产物浓度过稀, 不足以进行DGGE及切胶分离, 从而未能获得更长序列信息, 以进一步鉴定同一属中的不同种。该技术有必要与其他分子生物学、微生物学方法配合使用, 以提供共附生微生物菌群组成更丰富的信息。
综上所述, 对具有药用潜力的鲨鱼鳃部共附生真菌进行深入研究, 将有望发现新颖的活性化合物, 用于治疗人类恶性肿瘤、病原微生物感染的新药的开发, 另外也有助于进一步揭示鲨鱼的化学防御机制, 对环境友好的海水养殖抗菌、抗寄生虫病害药物的开发也有借鉴启示作用。
王 灿, 曹张军, 邵志宇等, 2009. 姥鲨共附生微生物抗菌活性筛选及系统发育分析. 中华航海医学与高气压医学杂志, 16(5): 265—268
龙良鲲, 羊宋贞, 姚 青等, 2005. AM真菌DNA的提取与PCR-DGGE分析. 菌物学报, 24(4): 564—569
朱元鼎, 孟庆闻, 2001. 中国动物志(圆口纲、软骨鱼纲). 北京: 科学出版社, 1—552
张清榕, 杨圣云, 2005. 中国软骨鱼类种类、地理分布及资源. 厦门大学学报(自然科学版), 44(Sup.): 207—211
林永成, 周世宁, 2003. 海洋微生物及其次生代谢产物. 北京: 化学工业出版社, 23—27
周荣丽, 穆 军, 张 翼等, 2008. 海洋真菌活性代谢产物最新研究进展. 天然产物研究与开发, 20(4): 741—747
郑忠辉, 陈连兴, 1998. 厦门海区潮间带海洋动植物共附生微生物的抗菌活性. 台湾海峡, 17(4): 439—444
孟庆闻, 苏锦祥, 李婉端, 1987. 鱼类比较解剖. 北京: 科学出版社, 205, 211—218
贺丽虹, 黄建华, 沈颂东等, 2005. 鲨鱼中的抗肿瘤活性物质及其作用机制. 海洋科学, 29(11): 63—66, 91
Sambrook J, Fritsch E F, Maniatis T著, 2002. 黄培堂译, 2002. 分子克隆实验指南(第3版). 北京: 科学出版社, 479—483
Armstrong E, Yan L M, Boyd K G, 2001. The symbiotic role of marine microbes on living surfaces. Hydrobiologia, 461: 37—40
Bourne D G, Munn C B, 2005. Diversity of bacteria associated with the coralfrom the Great Barrier Reef. Environ Microbiol, 7(8): 1162—1174
Buckingham J, 2011. Dictionary of Natural Products on DVD. Chapman and Hall/CRC, London, UK
Bugni T S, Ireland C M, 2004. Marine-derived fungi: a chemically and biologically diverse group of microorganisms. Nat Prod Rep, 21(1): 143—63
Cho J, Kim Y, 2002. Sharks: a potential source of antiangiogenic factors and tumor treatments. Mar Biotechnol (NY), 4(6): 521—525
Doare-Lebrun E, El Arbi A, Charlet M, 2006. Analysis of fungal diversity of grapes by application of temporal temperature gradient gel electrophoresis-potentialities and limits of the method. J Appl Microbiol, 101(6): 1340—1350
Fell J W, 1966., a new fungal genus from marine areas. Antonie van Leeuwenhoek, 32: 99—104
Gao Z, Li B L, Zheng C C, 2008. Molecular Detection of Fungal Communities in the Hawaiian Marine Spongesand. Appl Environ Microbiol, 74: 6091—6101
Haygood M G, Schmidt E W, Davidson S K, 1999. Microbial symbionts of marine invertebrates: opportunities for microbial biotechnology. J Mol Microbiol Biotechnol, 1(1): 33—43
Knickle C, Billingsley L, DiVittorio K, 2011. Biological Profiles basking shark. Florida Museum of Natural History. http://www.flmnh.ufl.edu/fish/gallery/descript/baskingshark/baskingshark.html
Lee A, Langer R, 1983. Shark cartilage contains inhibitors of tumor angiogenesis. Science, 221: 1185—1187
Luer C, 2012. Novel Compounds From Shark and Stingray Epidermal Mucus With Antimicrobial Activity Against Wound Infection Pathogens. Annual Report of Mote Marine Laboratory, Inc. Sarasota, Florida. 4—30
Maddalena A D, 2007. Great White Sharks Preserved in European Museums. Newcastle: Cambridge Scholars Publishing, 10
Moore K S, Wehrli S, Roder H, 1993. Squalamine: an aminosterol antibiotic from the shark. Proc Nat Acad Sci USA, 90(4): 1354—1358
Saleem M, Ali M S, Hussain S, 2007. Marine natural products of fungal origin. Nat Prod Rep, 24: 1142—1152
Tamura K, Dudley J, Nei M, 2007. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0. Mol Biol Evol, 24: 1596—1599
Taylor M W, Radax R, Steger D, 2007. Sponge-associated microorganisms: evolution, ecology, and biotechnological potential. Microbiol Mol Biol Rev, 71(2): 295—347
Thompson J D, Gibson T J, Plewniak F, 1997. The CLUSTALX windows inter-face: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res, 25: 4876—4882
Vallaeys T, Topp E, Muyzer G, 1997. Evaluation of denaturing gradient gel electrophoresis in the detection of 16S rDNA sequence variation in rhizobia and methanotrophs. FEMS Microbiol Ecol, 24(3): 279—285
Zuccaro A, Schoch C L, Spatafora J W, 2008. Detection and identification of fungi intimately associated with the brown seaweed. Appl Environ Microbiol, 74(4): 931—941
THE PCR-DGGE ANALYSIS OF FUNGAL COMMUNITY IN THE GILLS OF TWO SHARK SPECIES
ZHANG Yi1, 2, MU Jun1, 3, FENG Yan1, YAN Song1
(1. School of Environmental and Chemical Engineering, Dalian Jiaotong University, Dalian 116028, China; 2. School of Life Science and Biotechnology, Daian University of Technology, Dalian 116024, China; 3. Marine Science and Technology College, Zhejiang Ocean University, Zhoushan 316022, China)
Gill tissue DNA samples were extracted from two shark specimens from East China Sea,and, and used as templates for PCR amplification for the sequences of internal transcribed spacer 1 in ribosomal DNA gene of the fungal communities in the gill tissue. The PCR products were subsequently separated by DGGE (denatured gradient gel electrophoresis). Then, seven main electrophoresis stripes in the spectrum were amplified by secondary PCR, cloned, and sequenced. NCBI-BLAST comparison and molecular phylogenetic analysis indicated that the dominant fungal species in the gills ofcome from four taxonomical units including one from genusand three from. For, one dominant taxonomical unit comes from, two fromand one from A.is the common dominant fungal group in gills of the two shark species. In addition, the DGGE fingerprint also shows the presence of diverse minor fungal groups. Database searching suggested that most of these fungal taxonomical units have good potential in the production of natural bioactive substances. The PCR-DGGE analysis revealed that rich fungal community inhabits the two shark specimens’ gills. More investigations are demanded for understanding the metabolites and interaction with their hosts.
sharks; gills; fungal community; internal transcribed spacer ribosomal DNA; denatured gradient gel electrophoresis
10.11693/hyhz20121205002
* 国家自然科学基金项目, 20902009号; 中国博士后科学基金项目, 2011M500051号, 2012T50258号; 辽宁省教育厅优秀人才支持计划, 2009R08号。张翼, 博士, 副教授, E-mail: hubeizhangyi@163.com
穆军, 博士, 教授, E-mail: mujun1971@126.com
2012-12-05,
2013-04-22
Q938

